青藏高原素有“世界屋脊”“地球第三极”之称[1],具有紫外线辐射强、光照充足、大气氧分压低、全年气温偏低的气候特点[2]。大气氧分压随海拔的升高逐渐降低,当海拔达到4 000 m时,其大气氧分压仅为海平面的60%左右,高寒、低氧这种独特的气候条件成为了高原动物生长发育主要的限制因素,给当地动物的生存带来了极大挑战[3-4]。为促进藏区奶业发展,西藏自治区曾引进荷斯坦奶牛进行饲养,赵丽等[5]研究发现,西藏自治区地处高海拔地区,荷斯坦奶牛较本地牦牛体型大、代谢旺盛、耗氧量大,高原适应性差,高原病致死亡率高。犊牛阶段的饲养管理关系奶牛一生的生产潜能,同样也可能影响荷斯坦奶牛的高原适应性。其中,断奶阶段对于幼年反刍动物来说尤其重要。在这个阶段,犊牛的主要营养物质来源从牛奶转移到固体饲料,例如补饲开食料和粗饲料;同时,犊牛的瘤胃内环境及其功能发生变化。
断奶作为犊牛生长发育最重要的过渡阶段,其严重影响犊牛后续的消化性能、生长性能乃至免疫功能[6]。在反刍动物的幼年时期,采食的牛奶不经过未发育的瘤胃,营养物质在后肠道消化和吸收,不可消化的饲料在后肠道发酵[7]。研究发现,奶牛胃肠道菌群容易受到饲粮变化的影响,肠道微生物区系在胃肠道的发育和功能以及肠道健康中起着基础作用[8],更好地了解断奶前后肠道微生物群落的变化对犊牛培育是至关重要的。本试验拟在海拔3 650 m处研究荷斯坦犊牛早期断奶前后的粪便菌群变化情况,旨在探究高海拔地区荷斯坦犊牛断奶前后肠道菌群的差异,为日后西藏地区饲养荷斯坦牛犊牛提供参考依据。
1 材料与方法 1.1 试验设计及样品采集2021年9月份在西藏自治区拉萨市高标准奶牛养殖中心(海拔3 650 m),从365头荷斯坦犊牛中选取8头体重相近[断奶前采样当天体重(75.67±2.78) kg]、月龄相近的荷斯坦母犊牛为研究对象,采用直肠采粪法采集试验牛只断奶前[(60±2)日龄]和断奶后[(120±2)日龄]的粪便样品,于液氮罐-196 ℃保存。
荷斯坦犊牛每天饲喂3次(08:30、14:30和19:30),犊牛自7日龄时开始补充开食料,3月龄时采用逐渐断奶法进行断奶。断奶后,犊牛自由采食燕麦干草、苜蓿干草和开食料。开食料营养水平如下:粗蛋白质含量为24.08%,乙醚浸出物含量为4.50%,粗灰分含量为11.52%,钙含量为1.42%,磷含量为0.7%。
1.2 粪便菌群的测定 1.2.1 基因组DNA提取、PCR扩增和16S rRNA测序饲养试验采样结束后将粪便样品空运至美吉生物有限公司进行基因组DNA提取、PCR扩增和16S rRNA测序。使用OMEGA试剂盒(Omega Bio-Tek,美国)从1 g粪便样品中提取总微生物基因组DNA。使用Nanodrop 2000分光光度计(Thermo Fisher Scientific,美国)确认提取的DNA的浓度和纯度。使用正向引物338F(5′-ACTCCTACGGGAGGCAGCA-3′)和反向引物806R(3′-GGACTACNNGGGTATCTAAT-5′)扩增瘤胃细菌16S rRNA基因的V3~V4区域。PCR条件如下:95 ℃初始变性5 min,95 ℃ 45 s,55 ℃ 50 s,72 ℃ 45 s,经过28个循环,最后72 ℃延伸10 min。将扩增的片段进行2%琼脂糖凝胶电泳以检测扩增子,使用Agencourt AMPure XP试剂盒(Beckman Coulter Genomics,美国) 进行纯化,然后使用QuantiFluor-ST(Promega,美国)进行定量。纯化的PCR产物在Illumina MiSeq平台(Illumina,美国)上使用2×250 bp测序试剂盒进行测序[9]。
1.2.2 质量控制和统计分析使用QIIME 1.8[10]过滤和去除分数≤20(低质量)、reads < 200 bp以及包含不明碱基或与引物序列不匹配的序列。其余序列通过PEAR 0.9.6[11]组合并使用Flash(版本1.20)解复用[12]。使用UCHIME算法[13]去除组合长度小于230 bp和嵌合序列的读取。为确保样本间物种多样性的可比性,采用标准化操作分类单位(OTU)文件对物种和多样性指数进行分析。然后使用置信度阈值为0.70的核糖体数据库项目分类器[14]基于97%的序列相似性阈值将所得序列聚类为OTU,并与SILVA 128数据库进行比较以进行微生物物种注释[15]。所有数值都是使用UCLUST获得的,以生成具有代表性的OTU表[16]。使用QIIME 1.8和Chao1指数、Shannon指数和Simpson指数等程序确定菌群的OTU级Alpha多样性;使用R(版本4.0.5)[17]的“ggplot2”包(Wickham,2009)可视化结果。基于Bray-Curtis相异矩阵的主坐标分析(PCoA)在R中使用“vegan”包进行Beta多样性分析。将优质序列采用抽平方式生成OTU,进行OTU聚类分析和注释(97%相似水平),并进行Alpha多样性和Beta多样性分析;得到各水平分类信息,进而得到西藏地区荷斯坦犊牛断奶前后粪便菌群组成和结构差异。通过Beta多样性分析法绘制PCoA图,分析高海拔地区犊牛断奶前后粪便菌群物种多样性的差异。
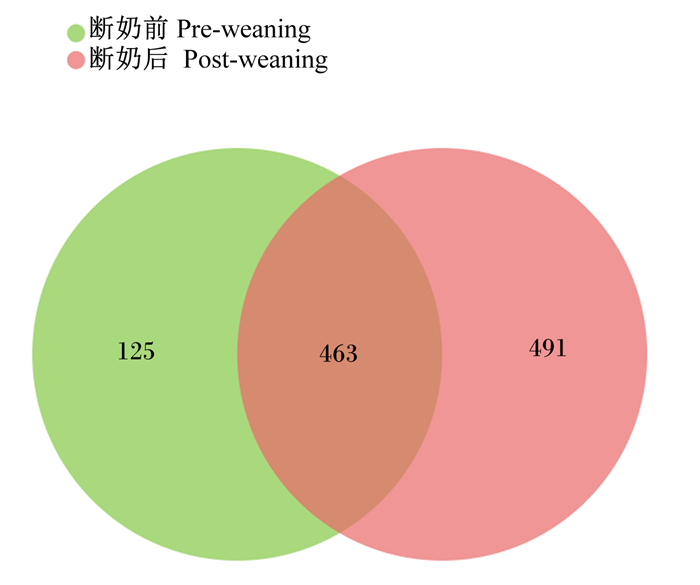
2 结果与分析 2.1 细菌群落基本组成经抽平后对OTU进行生物信息统计分析,从韦恩图(图 1)可以直观地得到样本OTU数目及断奶前后犊牛的菌群重叠情况。如图 1所示,本试验从16个样品中共获得1 070个OTU,其中断奶前独有125个,断奶后独有491个,共享463个。对所有序列进行OTU划分,对97%相似水平下的OTU进行生物信息统计分析,本试验得到的细菌物种包括15个门,21个纲,50个目,91个科,253个属。
|
图 1 断奶前后犊牛粪便菌群韦恩图 Fig. 1 Venn diagram of fecal bacteria community of calves before and after weaning |
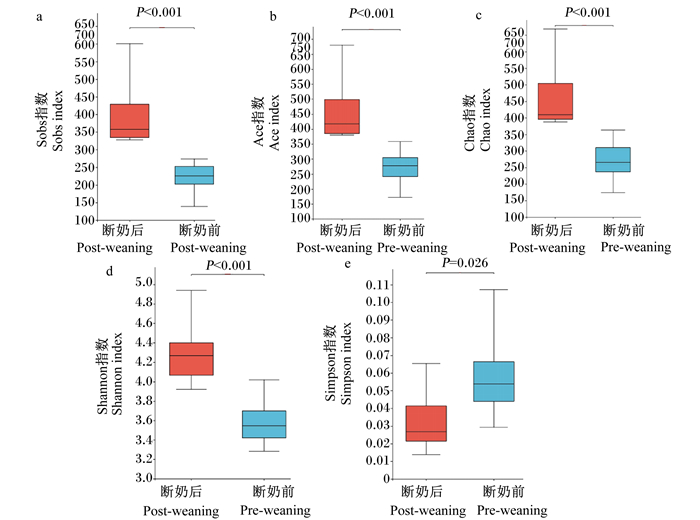
Alpha多样性可反映物种的丰富度和多样性,其包括一系列统计学分析指数:Sobs指数、Ace指数、Chao指数、Shannon指数和Simpson指数。本试验中断奶前后犊牛粪便样本文库覆盖率(coverage)均高于0.99,反映本次测序结果可以代表样本中菌群的真实情况。由图 2可知,在高海拔地区,断奶前犊牛和断奶后犊牛Sobs指数、Ace指数、Chao1指数、Simpson指数、Shannon指数均存在显著差异(P < 0.05)。
|
图 2 断奶前后犊牛粪便菌群的Alpha多样性指数 Fig. 2 Alpha diversity indexes of fecal bacteria community of calves before and after weaning |
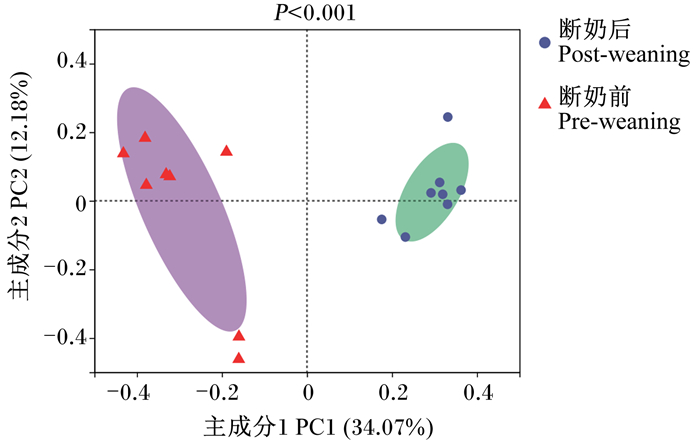
PCoA图中每个样本坐标点表示其菌群多样性,样本和样本之间的距离代表样本之间的物种丰度差异。由图 3可知,断奶前和断奶后犊牛粪便菌群样本距离较远,表明断奶前后犊牛菌群Bate多样性具有一定差异。
|
图 3 断奶前后犊牛粪便菌群的PCoA图 Fig. 3 PCoA diagram of fecal bacteria community of calves before and after weaning |
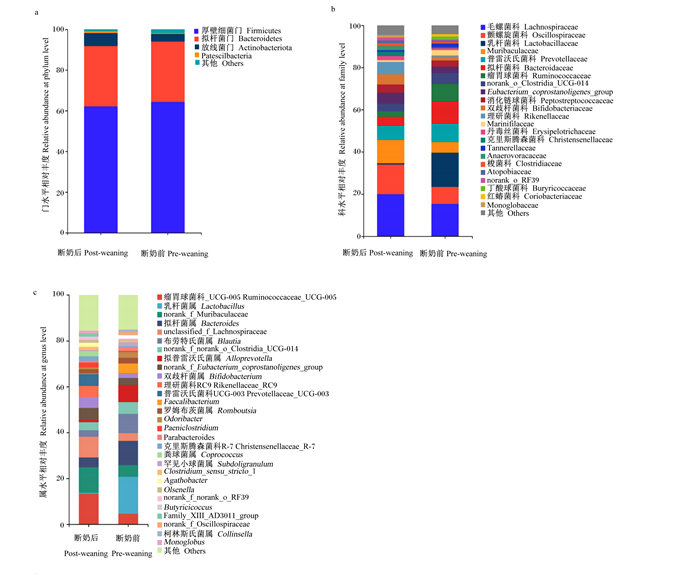
根据本试验样品测序结果,在门水平上,断奶前犊牛和断奶后犊牛均以厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)为优势菌(图 4-a)。如图 5-a所示,与断奶前犊牛相比,断奶后犊牛粪便菌群中Patescibacteria和Campilobacterota的相对丰度显著下降(P < 0.05)。
|
图 4 断奶前后犊牛粪便菌群分布柱状图 Fig. 4 Histograms of fecal bacteria community distribution of calves before and after weaning |
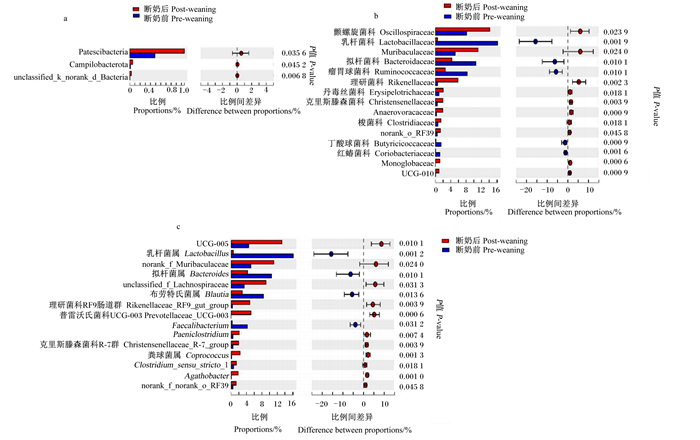
|
图 5 断奶前后犊牛粪便菌群组成差异 Fig. 5 Differences in fecal bacteria community composition of calves before and after weaning |
如图 4-b所示,在科水平上,断奶前犊牛和断奶后犊牛均以毛螺菌科(Lachnospiraceae)、颤螺旋菌科(Oscillospiraceae)、普雷沃氏菌科(Prevotellaceae)为优势菌。图 5-b展示了菌群相对丰度平均值排在前15的菌科,与断奶前犊牛相比,断奶后犊牛粪便菌群中Oscillospiraceae、鼠杆菌科(Muribaculaceae)、理研菌科(Rikenellaceae)、丹毒丝菌科(Erysipelotrichaceae)、克里斯滕森菌科(Christensenellaceae)、Anaerovoracaceae、梭菌科(Clostridiaceae)、norank_o_RF39、Monoglobaceae和UCG-010的相对丰度显著增加(P < 0.05),而乳杆菌科(Lactobacillaceae)、拟杆菌科(Bacteroidaceae)、瘤胃球菌科(Ruminococcaceae)、丁酸弧菌科(Butyricicoccaceae)和红蝽菌科(Coriobacteriaceae)的相对丰度显著降低(P < 0.05)。
2.4.3 断奶前后粪便菌群属水平差异如图 4-c和图 5-c所示,在属水平上,与断奶前犊牛相比,断奶后犊牛粪便菌群中UCG-005、norank_f_Muribaculacea、unclassified_f_Lachnospiraceae、理研菌科RC9肠道群(Rikenellaceae_RC9_gut_group)、普雷沃氏菌科UCG-003(Prevotellaceae_UCG-003)、Paeniclostridium、克里斯滕森菌科R-7群(Christensenellaceae_R-7_group)、粪球菌属(Coprococcus)、狭义梭菌属1(Clostridium_sensu_stricto_1)、不动杆菌属(Agathobacter)、norank_f_norank_o_RF39的相对丰度显著降低(P < 0.05),乳杆菌属(Lactobacillus)、类杆菌属(Bacteroides)、布劳特氏菌属(Blautia)、粪杆菌属(Faecalibacterium)的相对丰度显著增加(P < 0.05)。图 6展示了断奶前后犊牛粪便菌属的互作关系,其中每一节点表示1个属与其他属的相关性(r>0.5或 < -0.5和P < 0.05)。断奶前犊牛和断奶后犊牛的节点数分别为203和219个,表明断奶后犊牛瘤胃菌群复杂程度高于断奶前犊牛。
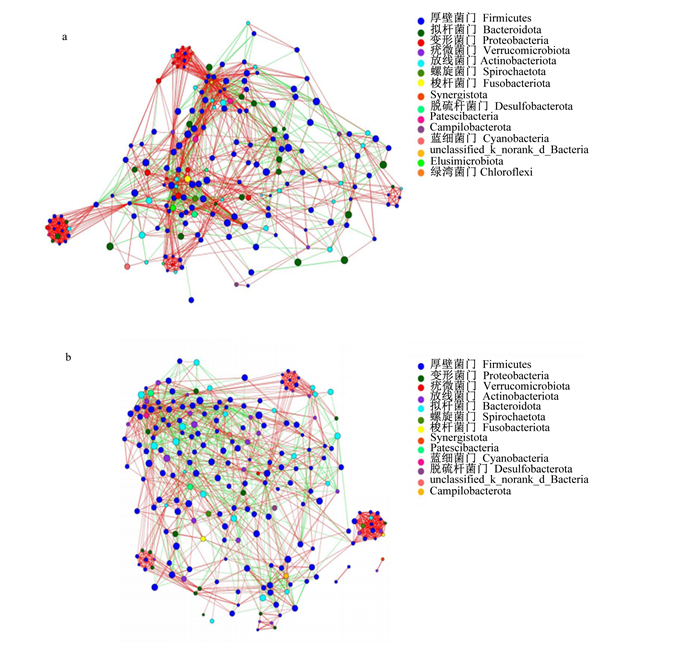
|
a: 断奶前pre-weaning;b: 断奶后post-weaning。 图 6 断奶前后犊牛粪便菌属共生网络图 Fig. 6 Network of co-occurring bacteria genera in feces of calves before and after weaning |
断奶影响犊牛的健康和生长性能,越来越多研究表明早期微生物定植的重要性及其对动物健康和终生生产力的影响[18]。前人研究认为,自然断奶过程在大约10个月大时开始,逐渐发生数周[19-20]。相比之下,早期断奶一般在犊牛8周龄(2月龄)时进行[21],以刺激对固体饲料的采食,进一步促进瘤胃发育[22]。目前我国大规模牧场普遍采用的奶牛犊牛断奶标准是60日龄[23],断奶体重是出生重的2倍,开食料的采食量达到1 kg。在西藏海拔3 650 m的地区,荷斯坦犊牛出生重低于低海拔地区,所以一般当荷斯坦犊牛到达3月龄时体重才能满足通用的断奶标准。早期断奶后,犊牛采食开食料获取更全面的营养提高日增重,降低饲养成本,从而进一步降低第1次产犊的年龄[21]。目前为止,犊牛早期断奶在高海拔地区的研究仍处于空白阶段。本文首次运用16S rRNA测序分析了高海拔地区断奶前后犊牛粪便菌群的差异,为后续高海拔地区反刍动物的研究提供理论依据不同的细菌组成可能形成不同的肠道功能,犊牛肠道微生物菌群多样性随着年龄的增长而增加,直到它成为稳定的成体微生物区系组成为止。
粪便菌群的分析表明,由网络共生图可知高海拔地区断奶后犊牛粪便菌群的复杂程度高于断奶前犊牛。此外,断奶前后犊牛粪便菌群Alpha和Bate多样性均存在差异显著,韦恩图也显示断奶前后犊牛粪便菌群存在差异。具体来说,本研究发现,在高海拔地区,相比于断奶前,在门水平上,断奶后犊牛粪便菌群中Patescibacteria和Campilobacterota的相对丰度显著降低,其中Patescibacteria与肠道生物稳态有关,主要反映肠道上皮功能障碍[24],说明犊牛断奶后肠道菌群逐渐趋于成熟;在科水平上,断奶后犊牛粪便菌群中Oscillospiraceae、Muribaculacea、Rikenellaceae、Erysipelotrichaceae、Christensenellaceae、Anaerovoracaceae、Clostridiaceae、norank_o_RF39、Monoglobaceae和UCG-010的相对丰度显著增加,而Lactobacillaceae、Bacteroidaceae、Ruminococcaceae、Butyricicoccaceae和Coriobacteriaceae的相对丰度显著降低,其中Oscillospiraceae[25]、Muribaculaceae[26]、Rikenellaceae、Erysipelotrichaceae[24]、Christensenellaceae[27]、Clostridiaceae[28]、Lactobacillaceae[29]、Bacteroidaceae[30]、Ruminococcaceae[31]、Butyricicoccaceae[32]和Coriobacteriaceae的细菌与碳水化合物消化并生成短链脂肪酸有关。此外,有报道显示Rikenellaceae[33]、Christensenellaceae[34]与炎症或机体健康有关,Muribaculaceae是存在于犊牛肠道的一种有害菌,容易导致犊牛患病、腹泻等[35]。断奶后犊牛粪便菌群中Muribaculaceae的相对丰度增加可能是犊牛处于断奶应激的状态下,机体免疫低下,机体肠道菌群紊乱造成的。综上所述,犊牛断奶前后与碳水化合物相关的菌群相对丰度发生明显变化,这可能是由于断奶后荷斯坦犊牛不再摄入牛奶,更多地采食开食料和粗饲料造成的;此外,断奶应激也可能造成犊牛肠道有害菌相对丰度升高,但未引起机体表观指标的明显变化。这也提示我们要加强断奶过渡时期的犊牛管理工作,切实把握好高原奶牛养殖过程中的“犊牛关”。
4 结论综上可知,在本试验中,西藏地区(海拔3 650 m)荷斯坦犊牛在3月龄进行断奶,断奶前后犊牛粪便菌群多样性及丰富度出现明显变化,断奶后犊牛肠道发酵产生短链脂肪酸的菌群增多,肠道发酵能力增强,这主要是犊牛肠道菌群随着饲粮改变而发生的变化。
| [1] |
SUN Y X, LIU S L, LIU Y X, et al. Effects of the interaction among climate, terrain and human activities on biodiversity on the Qinghai-Tibet Plateau[J]. Science of the Total Environment, 2021, 794: 148497. DOI:10.1016/j.scitotenv.2021.148497 |
| [2] |
查瑞波, 孙根年, 董治宝, 等. 青藏高原大气氧分压及游客高原反应风险评价[J]. 生态环境学报, 2016, 25(1): 92-98. ZHA R B, SUN G N, DONG Z B, et al. Assessment of atmospheric oxygen practical pressure and plateau reaction of tourists in the Qinghai-Tibet Plateau[J]. Ecology and Environmental Sciences, 2016, 25(1): 92-98 (in Chinese). |
| [3] |
GUO X S, LONG R J, KREUZER M, et al. Importance of functional ingredients in yak milk-derived food on health of Tibetan nomads living under high-altitude stress: a review[J]. Critical Reviews in Food Science and Nutrition, 2014, 54(3): 292-302. DOI:10.1080/10408398.2011.584134 |
| [4] |
CHEVIRON Z A, BRUMFIELD R T. Genomic insights into adaptation to high-altitude environments[J]. Heredity, 2012, 108(4): 354-361. DOI:10.1038/hdy.2011.85 |
| [5] |
赵丽, 冯静, 马金英, 等. 西藏荷斯坦奶牛死亡原因分析[J]. 中国牛业科学, 2015, 41(6): 92-94. ZHAO L, FENG J, MA J Y, et al. Analysis the cause of death in Tibet holstein cows[J]. China Cattle Science, 2015, 41(6): 92-94 (in Chinese). DOI:10.3969/j.issn.1001-9111.2015.06.024 |
| [6] |
HAO Y Y, GUO C Y, GONG Y, et al. Rumen fermentation, digestive enzyme activity, and bacteria composition between pre-weaning and post-weaning dairy calves[J]. Animals, 2021, 11(9): 2527. DOI:10.3390/ani11092527 |
| [7] |
SONG Y, MALMUTHUGE N, STEELE M A, et al. Shift of hindgut microbiota and microbial short chain fatty acids profiles in dairy calves from birth to pre-weaning[J]. FEMS Microbiology Ecology, 2018, 94(3): fix179. |
| [8] |
MALMUTHUGE N, GUAN L L. Understanding the gut microbiome of dairy calves: opportunities to improve early-life gut health[J]. Journal of Dairy Science, 2017, 100(7): 5996-6005. DOI:10.3168/jds.2016-12239 |
| [9] |
CAPORASO J G, LAUBER C L, WALTERS W A, et al. Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms[J]. ISME Journal, 2012, 6(8): 1621-1624. DOI:10.1038/ismej.2012.8 |
| [10] |
CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al. QⅡME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [11] |
ZHANG J J, KOBERT K, FLOURI T, et al. PEAR: a fast and accurate Illumina paired-end reAd mergeR[J]. Bioinformatics, 2014, 30(5): 614-620. DOI:10.1093/bioinformatics/btt593 |
| [12] |
MAGOČ T, SALZBERG S L. FLASH: fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507 |
| [13] |
EDGAR R C, HAAS B J, CLEMENTE J C, et al. UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics, 2011, 27(16): 2194-2200. DOI:10.1093/bioinformatics/btr381 |
| [14] |
COLE J R, WANG Q, CARDENAS E, et al. The ribosomal database project: improved alignments and new tools for rRNA analysis[J]. Nucleic Acids Research, 2009, 37(Database issue): D141-D145. |
| [15] |
QUAST C, PRUESSE E, YILMAZ P, et al. The SILVA ribosomal RNA gene database project: improved data processing and web-based tools[J]. Nucleic Acids Research, 2013, 41(Database issue): D590-D596. |
| [16] |
EDGAR R C. Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics, 2010, 26(19): 2460-2461. DOI:10.1093/bioinformatics/btq461 |
| [17] |
WICKHAM H. Ggplot2:elegant graphics for data analysis[M]. New York: Springer, 2009.
|
| [18] |
JAMI E, ISRAEL A, KOTSER A, et al. Exploring the bovine rumen bacterial community from birth to adulthood[J]. ISME Journal, 2013, 7(6): 1069-1079. DOI:10.1038/ismej.2013.2 |
| [19] |
HORVATH K C, ALLEN A N, MILLER-CUSHON E K. Effects of access to stationary brushes and chopped hay on behavior and performance of individually housed dairy calves[J]. Journal of Dairy Science, 2020, 103(9): 8421-8432. DOI:10.3168/jds.2019-18042 |
| [20] |
ECKERT E, BROWN H E, LESLIE K E, et al. Weaning age affects growth, feed intake, gastrointestinal development, and behavior in Holstein calves fed an elevated plane of nutrition during the preweaning stage[J]. Journal of Dairy Science, 2015, 98(9): 6315-6326. DOI:10.3168/jds.2014-9062 |
| [21] |
LOHAKARE J D, SVDEKUM K H, PATTANAIK A K. Nutrition-induced changes of growth from birth to first calving and its impact on mammary development and first-lactation milk yield in dairy heifers: a review[J]. Asian-Australasian Journal of Animal Sciences, 2012, 25(9): 1338-1350. DOI:10.5713/ajas.2012.12282 |
| [22] |
KHAN M A, BACH A, WEARY D M, et al. Invited review: transitioning from milk to solid feed in dairy heifers[J]. Journal of Dairy Science, 2016, 99(2): 885-902. DOI:10.3168/jds.2015-9975 |
| [23] |
赵书明. 简述犊牛早期断奶的饲养管理要点[J]. 中国畜禽种业, 2022, 18(1): 111-112. ZHAO S M. Briefly describe the key points of feeding and management of early weaning of calves[J]. The Chinese Livestock and Poultry Breeding, 2022, 18(1): 111-112 (in Chinese). DOI:10.3969/j.issn.1673-4556.2022.01.063 |
| [24] |
VAN DEN BRULE S, RAPPE M, AMBROISE J, et al. Diesel exhaust particles alter the profile and function of the gut microbiota upon subchronic oral administration in mice[J]. Particle and Fibre Toxicology, 2021, 18(1): 7. DOI:10.1186/s12989-021-00400-7 |
| [25] |
GUILLOTEAU P, MARTIN L, EECKHAUT V, et al. From the gut to the peripheral tissues: the multiple effects of butyrate[J]. Nutrition Research Reviews, 2010, 23(2): 366-384. DOI:10.1017/S0954422410000247 |
| [26] |
HE Q D, GUO J J, ZHANG Q, et al. Effects of electroacupuncture on the gut microbiome in cisplatin-induced premature ovarian failure mice[J]. Evidence-Based Complementary and Alternative Medicine, 2022, 2022: 9352833. |
| [27] |
WATERS JL, LEY RE. The human gut bacteria Christensenellaceae are widespread, heritable, and associated with health[J]. BMC Biology, 2019, 17(1). |
| [28] |
LUO Y H, LIU Y, LI H, et al. Differential effect of dietary fibers in intestinal health of growing pigs: outcomes in the gut microbiota and immune-related indexes[J]. Frontiers in Microbiology, 2022, 13: 843045. DOI:10.3389/fmicb.2022.843045 |
| [29] |
MEEHAN C J, BEIKO R G. A phylogenomic view of ecological specialization in the Lachnospiraceae, a family of digestive tract-associated bacteria[J]. Genome Biology and Evolution, 2014, 6(3): 703-713. DOI:10.1093/gbe/evu050 |
| [30] |
JANG Y O, LEE S H, CHOI J J, et al. Fecal microbial transplantation and a high fiber diet attenuates emphysema development by suppressing inflammation and apoptosis[J]. Experimental and Molecular Medicine, 2020, 52(7): 1128-1139. DOI:10.1038/s12276-020-0469-y |
| [31] |
GU X, SIM J, WEI L L, et al. Gut Ruminococcaceae levels at baseline correlate with risk of antibiotic-associated diarrhea[J]. .iScience, 2021, 25(1): 103644. |
| [32] |
HAO Y, JI Z, SHEN Z, et al. Effects of total dietary fiber on cecal microbial community and intestinal morphology of growing white Pekin duck[J]. Frontiers in Microbiology, 2021, 12: 727200. DOI:10.3389/fmicb.2021.727200 |
| [33] |
ECKERSALL P D, BELL R. Acute phase proteins: biomarkers of infection and inflammation in veterinary medicine[J]. The Veterinary Journal, 2010, 185(1): 23-27. DOI:10.1016/j.tvjl.2010.04.009 |
| [34] |
WATERS J L, LEY R E. The human gut bacteria Christensenellaceae are widespread, heritable, and associated with health[J]. BMC Biology, 2019, 17(1): 83. |
| [35] |
王丽华, 徐宏建, 张全宇, 等. 不同剂量25羟基维生素D3对断奶前犊牛生长性能、营养物质表观消化率、血浆代谢物及粪便菌群的影响[J]. 动物营养学报, 2022, 34(1): 404-412. WANG L H, XU H J, ZHANG Q Y, et al. Effects of different doses of 25-hydroxyvitamin D3 on growth performance, nutrient apparent digestibility, plasma metabolites and fecal flora of pre-weaning calves[J]. Chinese Journal of Animal Nutrition, 2022, 34(1): 404-412 (in Chinese). |




