2. 动物疫病与人类健康四川省重点实验室, 雅安 625014
2. Key Laboratory of Animal Disease and Human Health of Sichuan Province, Ya'an 625014, China
纤维素广泛分布于自然界,是含量丰富的碳水化合物,占植物界碳含量的1/2以上[1],而位于植物细胞壁纤维素之间起抗压作用的木质素却难以被微生物和酶降解[2]。王占川等[3]研究表明,将秸秆开发为饲料,使其转化为畜产品将具有很高的经济价值。纤维素通过瘤胃菌群发酵,可作为反刍动物的重要能量来源。反刍动物的瘤胃能同时为好氧、兼性厌氧和绝对厌氧微生物提供良好的栖息环境(温度为39~41 ℃,pH为5.5~7.5)[4]。成年牛瘤胃中栖息的微生物包括200多种细菌、5个属的真菌以及原虫和古菌。瘤胃菌群在数量上占有绝对优势,在纤维素分解过程中发挥着重要作用[5]。刘开朗等[6]统计2008年至2009年间在美国奶业科学协会-美国动物科学学会(ADSA-ASAS)年会论文集、中国知网、PubMed等数据库中与瘤胃菌群分子多态性和元基因组学相关的文献有42篇。尽管对瘤胃菌群多样性和功能的研究取得了一定进展,但对其中影响宿主代谢和生产性能的关键功能菌的分离、鉴定、作用模式及其利用的研究仍处于初步阶段,许多占优势的菌群至今还未在实验室里被分离出来。所以,研究瘤胃菌群,尤其是研究分解利用纤维素的菌群具有非常广阔的前景。
长久以来对反刍动物瘤胃菌群结构的研究主要采取细菌分离和培养的方法,不但费时费力,而且结果不够准确,重复性差。通过纯培养技术分离的微生物种类仅为11%左右[7],大部分信息仍处于未知状态。自Muyzer等[8]首次将聚合酶链式反应-变性梯度凝胶电泳(PCR-DGGE)技术应用于微生物群落结构和多样性研究以来,复杂的肠道微生物菌群得到了更加全面准确的反映。由于反刍动物瘤胃菌群数量庞大且种类繁多,直接对其进行非特异的16S rDNA V3区DGGE分析,所得到的图谱通常复杂且难以分辨。因此,本研究拟将瘤胃内容物先进行富集培养,而后采用PCR-DGGE技术分析,并对结果进行聚类分析和主成分分析(PCA),结合共性和特异性条带的克隆和测序,评估反刍动物瘤胃菌群在不同培养条件下的多样性,旨在了解瘤胃细菌种类,为后续分离纤维素降解菌时培养条件的选择提供参考。
瘤胃内容物采自四川省雅安市飞机坝屠宰场。无菌操作采集3份瘤胃内容物,每份20~30 g,置-20 ℃冰箱保存备用。
PCR引物由英潍捷基(上海)生物技术有限公司合成;2×Taq MasterMix(含染料)购自北京康为世纪生物科技有限公司;DGGE成套试剂购自美国Bio-Rad公司;银染药品和试剂购自四川瑞进特科技有限公司;Gel Extraction Kit购自美国OMEGA公司;pMD19-T载体购自宝生物工程(大连)有限公司;PCR仪为MJ Research PTC-200(美国Bio-Rad公司);DGGE电泳仪为Dcode Universal Detection System ;核酸浓度测定仪为ND-1000 UV-Vis Spectrphotometer(美国NanoDrop公司);凝胶成像系统为GS800 Calibrated Densitometer(美国Bio-Rad公司)。
取3份样品(编号为A、B、C),分别称取1 g瘤胃内容物至装有30 mL培养基的100 mL三角瓶中。分别在37和50 ℃静置培养48 h后摇匀分装于1.5 mL EP管中,每管1 mL左右,置-20 ℃冰箱保存备用。
参照文献[13]提取细菌总DNA。用核酸浓度测定仪测定总DNA浓度,置-20 ℃冰箱保存备用。
参照文献[14]根据大肠杆菌16S rRNA基因V3片段(339~539 bp)设计合成引物。上游引物设计为带GC发夹结构(带下划线的碱基序列),序列为:5′- CGC CCG GGG CGC GCC CCG GGC GGG GCG GGG G CA CGG GGG GAC TCC TAC GGG AGG CAG CAG T-3′;下游引物序列为:5′-GTA TTA CCG CGG CTG CTG GCA C-3′。PCR反应体系(25 μL):上、下游引物(10 pmol/μL)各1.0 μL,2×Taq MasterMix 12 μL,总DNA(模板)1.0~2.0 μL,双蒸水补足25 μL,同时设阴性对照管。PCR扩增条件为94 ℃预变性4 min;94 ℃变性30 s,58 ℃退火30 s,72 ℃延伸2 min,30个循环;最后72 ℃延伸10 min。用1.0%琼脂糖凝胶电泳检测PCR产物片段大小和浓度。
参照文献[15]进行PCR-DGGE,凝胶电泳梯度为35%~65%(100%的变性剂包括7 mol/L尿素和40%甲酰胺),变性方向与电泳方向一致。采用1×TAE作电泳缓冲液,100 V、60 ℃电泳14~16 h,结果经硝酸银进行染色后用凝胶成像系统扫描图像。
将DGGE图谱上的共性和特异性条带分别割胶回收并浸泡在30 μL加有0.1%聚乙二醇辛基苯基醚(Triton X-100)的缓冲液中,4 ℃放置2 d。然后取1 μL作为模板,按1.4方法再次扩增16S rDNA V3区,扩增产物进行DGGE电泳,确认回收片段的正确性。重复该过程1~3次,直至在DGGE图谱上得到单一的特定条带。然后,取1 μL回收纯化的DNA为模板,采用1.4方法扩增V3区,不同的是使用不带GC发夹的引物,PCR产物用于下一步克隆。
采用pMD19-T载体试剂盒,参照使用手册对模板PCR产物进行连接,再将其转入大肠杆菌DH5α感受态细胞中,涂布含氨苄青霉素的营养琼脂平板,每个条带选取1~3个阳性克隆送英潍捷基(上海)生物技术有限公司测序。应用Chromas 2软件对测定序列进行编辑,非嵌合体序列在GenBank数据库中进行比对分析,寻找亲缘关系最近的细菌或克隆。
将DGGE图谱数字化、标准化后得到1个记录DGGE胶中条带迁移位置的数字化矩阵,将矩阵导入SAS 9.1软件和NTSYS 2.1软件进行聚类分析及主成分分析。
瘤胃菌群数量庞大且种类繁多(图1),强的电泳条带反映了瘤胃中的优势菌群,条带数量和位置的复杂性代表细菌菌群的多样性。不同纤维素富集培养基和温度培养的瘤胃内容物均产生了丰富的电泳条带,但菌群结构和组成存在较大差别。37 ℃条件下得到的样品与50 ℃相比其细菌种类丰富度较高,用CMC培养基培养的样品在37 ℃条件下平均条带数为23,而在50 ℃条件下仅为15;同样用PCS培养基培养的样品在37 ℃条件下平均条带数为19,而在50 ℃条件下为16。同一温度条件下,条带数多的样品都集中分布在CMC培养基和PCS培养基,这可能与纤维素对细菌的富集有关。
聚类分析结果(图2)显示,不同纤维素富集条件影响样品细菌的多样性,在37 ℃的瘤胃内容物中,J培养基和PCS培养基间的相似性系数为0.77;J培养基和CMC培养基间的相似性系数仅为0.76。在50 ℃的瘤胃内容物中,J培养基和PCS培养基间的相似性系数为0.78;而CMC培养基和PCS培养基间的相似性系数高达0.84。
温度对瘤胃菌群的培养有一定影响,37 ℃的菌群聚为2个大族,而全部50 ℃的菌群聚为1个大族,PCS培养基和J培养基间的相似性系数在37 ℃仅为0.75,而在50 ℃高达0.97,这可能归因于瘤胃菌群生存的环境温度。
对DGGE图谱的主成分分析(图3)同聚类分析的结果相一致。主成分因子1(PC1)的贡献率为18.05%,主成分因子2(PC2)的贡献率为10.02%;PC1明显地将样品分成2个部分,50 ℃的样品主要分布在图的右边,37 ℃的样品主要分布在图的左边;在50 ℃的样品中,PC2将CMC培养基、PCS培养基和J培养基培养的样品同K培养基培养的样品区别开来;37 ℃的样品中,PC2同样将CMC培养基、PCS培养基和J培养基培养的样品同K培养基培养的样品基本分开。
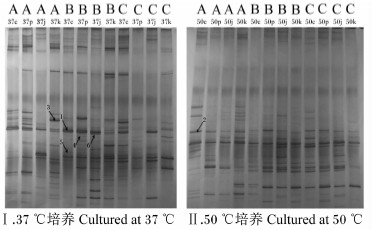 | 图1 不同培养基和温度培养后瘤胃内容物16S rDNA V3 区基因片段的PCR-DGGE图谱 Fig.1 PCR-DGGE profiles of gene fragments of 16S rDNA V3 region of rumen contents after cultured in different mediums at different temperatures |
DGGE图谱上割胶回收的2个共性条带和4个特异性条带(图1箭头所指),2个共性条带为优势菌群,克隆测序后在GenBank数据库中进行序列比对分析,找出了最相近的菌种,结果见表1。不同条件培养后的瘤胃菌群结构丰富,代表优势菌群的条带属于解没食子酸链球菌(Streptococcus gallolyticus)和解脲芽孢杆菌(Ureibacillus themosphaericus)2个菌属。本试验回收的几个主要特异性条带属于解没食子酸链球菌、摩氏假单胞菌(Pseudomonas mosselii)、产碱杆菌(Alcaligenes)和未培养毛螺旋菌(uncultured Lachnospiraceae)4个菌属。
在6个测序结果中,与GenBank数据库中细菌的同源性绝大多数都到达了100%,而条带1和条带4同数据库中与之同源性最高的未培养微生物的相似性也达到了99%,说明试验中所得的序列与已鉴定的微生物亲缘性非常高。
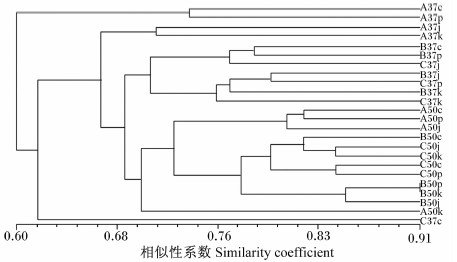 | 图2 PCR-DGGE图谱的聚类分析
Fig.2 Cluster analysis of PCR-DGGE profiles |
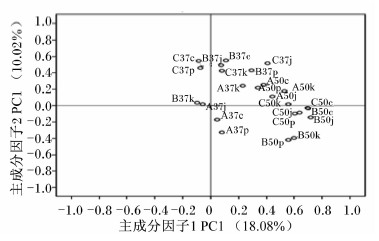 | 图3 PCR-DGGE图谱的主成分分析
Fig.3 PCA analysis of PCR-DGGE profiles |
瘤胃菌群数量大,种类繁多,多样性丰富[16]。DGGE图谱(图1)显示不同培养基和温度条件下,瘤胃细菌种类相当丰富。CMC培养基和PCS培养基富含丰富的CMC-Na、微晶纤维素和纤维素粉等成分,为瘤胃纤维素降解菌提供了丰富的营养原料。瘤胃菌群多样性在37 ℃条件下高于50 ℃时;CMC培养基和PCS培养基培养的瘤胃菌群多样性高于J培养基和K培养基。闫韩韩等[17]研究饲喂添加葡萄糖、尿素和无机盐的饲粮后,羊的瘤胃菌群种类和数量均呈现增加趋势。冯薇等[18]采用PCR-DGGE分析比较了奶牛瘤胃中附着于苜蓿、青贮玉米和羊草3种饲料固相黏附微生物区系,发现其微生物菌群构成存在差异。同样,本研究采用不同培养基和温度评估瘤胃菌群,发现在不同纤维素培养基和温度下瘤胃内容物DGGE图谱存在较大差异,可能是因为瘤胃细菌对不同环境条件表现出不同程度的适应性。
聚类分析及主成分分析结果显示,对于同一份瘤胃内容物样品,37 ℃条件下用CMC培养基和PCS培养基所富集培养的菌群结构较类似,可能是CMC和PCS2种培养基的营养成分类似,为细菌的生长提供了比较类似的营养原料;而在50 ℃富集培养条件下,无论用CMC培养基,还是PCS培养基所富集培养的菌群结构相似度非常高。因为瘤胃菌群的生长环境温度为39~41 ℃,对于大多数瘤胃细菌而言,温度高于50 ℃条件下即引起死亡[19],仅有少数耐高温的细菌能生存下来,如耐高温的解脲芽孢杆菌,所以CMC培养基和PCS培养基在50 ℃条件下的瘤胃菌群多样性差异小。因此,对纤维素分解菌进行富集培养时,可以从CMC培养基和PCS培养基中选取一种,同时选择适当的温度。
| 表1 PCR-DGGE图谱中共性和特异性条带序列的Blast分析
Table 1 Blast analysis of sequences of common and specific bands in PCR-DGGE profiles
|
孔庆亮等[20]应用PCR-16S rDNA鉴定和动态监测奶牛瘤胃菌群,鉴定出具有代表性的细菌为牛链球菌、蜡样芽孢杆菌、地衣芽孢杆菌和嗜热链球菌。同样,本研究在不同纤维素富集培养条件下鉴定出的瘤胃优势菌为解没食子酸链球菌和解脲芽孢杆菌。Kakimoto等[21]通过微生物纯培养方法鉴定了瘤胃内的产脲酶菌主要是链球菌属(Streptococcus)。本试验通过CMC培养基培养后的特异性条带鉴定为产碱杆菌,可能是因为CMC培养基中含有的酵母粉为微生物的生长提供了充足的氮源及维生素等原料。而含丰富纤维素物质的J培养基培养后的特异性条带鉴定为未培养毛螺旋菌。Ghali等[22]从单峰骆驼和黑鹿中均鉴定出牛链球菌,而本试验从富含蛋白胨的PCS培养基培养后的特异性条带鉴定为解没食子酸链球菌,与其为同一属。目前随着分子生物学的发展,越来越多的瘤胃细菌信息被人们掌握,但研究发现瘤胃中仍有89%的细菌未被分离培养[7],大量有价值的瘤胃细菌还有待探究。
① 不同纤维素富集培养基和温度对瘤胃菌群的多样性有一定程度上的影响。
② 37 ℃条件下的瘤胃菌群多样性高于50 ℃,50 ℃条件下可分离培养耐高温细菌,如耐高温解脲芽孢杆菌。
③ 不同培养基得到的瘤胃菌群存在一定差异,采用J培养基培养的瘤胃菌群多样性程度低,但可作为分离某些难培养细菌的培养基,如未培养毛螺旋菌。
| [1] | ENQUIST B J,ECONOMO E P,HUXMAN T E,et al.Scaling metabolismfrom organisms ecosystems[J]. Nature,2003,423(5):639-642. ( 1) 1)
|
| [2] | 赵天章,李慧英,闫素梅.反刍动物饲料纤维物质瘤胃降解规律研究进展[J]. 饲料工业,2010,31(7):28-31. ( 1) 1)
|
| [3] | 王占川,宝鲁德,周子彦,等.秸秆生物饲料技术开发的过去、现在和将来[J]. 畜牧与饲料科学,2011,32(1):53-54. ( 1) 1)
|
| [4] | 张彩英,胡国良,曹华斌.反刍动物瘤胃内环境的特点及调控措施[J]. 中国畜牧兽医,2010,37(4):18-20. ( 1) 1)
|
| [5] | ORPIN C G,JOBLIN K N.The rumen anaerobic fungi[M]//HOBSON P N,STEWART C S.The rumen microbial ecosystem.London:Academic and Professional Publishers,1997:140-195. ( 1) 1)
|
| [6] | 刘开朗,王加启,卜登攀.2008—2009年反刍动物营养研究进展Ⅰ.瘤胃微生物多样性与功能[J]. 中国畜牧兽医,2010,37(2):5-14. ( 1) 1)
|
| [7] | EDWARDS J E,MCEWAN N R,TRAVIS A J,et al.16S rDNA library-based analysis of ruminal bacterial diversity[J]. Antonie Van Leeuwenhoek,2004(3):263-281. ( 2) 2)
|
| [8] | MUYZER G,DE WAAL E C,UIRRERLINDEN A G.Profiling of complex microbial population by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes encoding for 16S rRNA[J]. Applied and Environmental Microbiology,1993,59(3):695-700. ( 1) 1)
|
| [9] | 郝月,杨翔华,张晶,等.秸秆纤维素分解菌的分离筛选[J]. 中国农业通报,2005,21(7):58-60. ( 1) 1)
|
| [10] | 祝令香,徐建,于巍,等.一株纤维素降解菌的鉴定[J]. 农业生物技术学报,2001,9(3):255-257. ( 1) 1)
|
| [11] | 王伟东,崔宗均,杨洪岩,等.高效稳定纤维素分解菌复合系WSC-6的稳定性[J]. 中国环境科学,2005,25(5):567-571. ( 1) 1)
|
| [12] | NAKAJIMA-KAMBE,OKADA T,N TAKEDA M,et al.Screening of novel cellulose-degrading bacterium and its application to denitrification of groundwater[J]. Journal of Bioscience and Bioengineering,2005,99(4):429-433. ( 1) 1)
|
| [13] | LI M,GONG J,COTRILL M,et al.Evaluation of QIAamp® DNA Mini Stoll Kit for microbial ecological studies[J]. Journal of Microbiological Methods,2003,54:13-20. ( 1) 1)
|
| [14] | WALTER J,HERTEL C,TANNOCK G W,et al.Detection of Lactobacillus,Pediococcus,Leuconostoc and Weissellaspecies in human feces by using group-specific PCR primers and denaturing gradient gel electrophoresis[J]. Applied and Environmental Microbiology,2001,67:2578-2585. ( 1) 1)
|
| [15] | 倪学勤,GONG J,YU H,等.采用PCR-DGGE技术分析蛋鸡肠道细菌种群结构及多样性[J]. 畜牧兽医学报,2008,39:955-961. ( 1) 1)
|
| [16] | 王梦芝,徐爱秋,李世霞,等.瘤胃微生物类群的多样性及其综合发酵的研究进展[J]. 饲料工业,2008,29(15):37-40. ( 1) 1)
|
| [17] | 闫韩韩,王红.不同日粮对羊瘤胃微生物数量的影响[J]. 饲料工业,2009,30(13):34-38. ( 1) 1)
|
| [18] | 冯薇,王加启,刘开朗,等.运用PCR-DGGE分析比较瘤胃中不同饲料固相黏附微生物区系[J]. 畜牧兽医学报,2010,41(12):1556-1562. ( 1) 1)
|
| [19] | 施邑屏.温度与微生物[J]. 微生物学通报,1982(6):291-294. ( 1) 1)
|
| [20] | 孔庆亮,王振勇,柴同杰,等.奶牛瘤胃需氧及兼性厌氧菌的PCR-16S rDNA鉴定及日粮的影响[J]. 畜牧兽医学报,2008,39(10):1367-1372. ( 1) 1)
|
| [21] | KAKIMOTO S,OKAZAKI K,SAKANE T,et al.Isolation and taxonomic characterization of acid urease producing bacteria[J]. Agricultural and Biological Chemistry,1989,53(4):1111-1118. ( 1) 1)
|
| [22] | GHALI M B,SCOTT P T,AL JASSIM R A M.Characterization of Streptococcus bovis from the rumen of the dromedary camel and Rusa deer[J]. Letters in Applied Microbiology,2004,39(4):341-346. ( 1) 1)
|




