竹鼠(Rhizomyidae),又名竹根鼠、竹鼬、茅根鼠、芭茅鼠,为哺乳纲啮齿目竹鼠科竹鼠属,主要分布在我国的中部及南部地区,缅甸北部和越南也有分布。竹鼠体大肉多,味道鲜美,营养丰富,毛皮绒厚柔软,有较高的经济价值。竹鼠为植食性动物,一般以苇草根、白茅根、竹叶、竹节、竹根为食,对粗纤维的消化能力特别强。竹鼠的盲肠是消化道的重要组成部分,盲肠中分布的微生物可以促进营养物质的消化和吸收,同时也可以分泌营养素。肠道菌群的多样性和平衡是个体健康和发挥正常生理功能的基础,同时在提高自身生产性能以及对食物的利用率、减少病害等方面有着重要的意义。扩增性rDNA限制性酶切片段多态性分析(amplified ribosomal DNA restriction analysis,ARDRA)技术是美国最新发展起来的一项现代生物鉴定技术[1, 2],它依据原核生物rDNA序列的保守性[3],将扩增的rDNA片段进行酶切,然后通过酶切图谱分析菌群多样性。ARDRA技术可以快速地将大量微生物分群,是一种简便、可靠的方法,在微生物遗传多样性及其分类研究中应用广泛。选择合适的限制性内切酶,可使ARDRA结果与16S rDNA全序列分析结果具有很好的一致性[4]。因此,本试验应用ARDRA技术对竹鼠盲肠细菌菌群的多样性进行初步的探索性研究。
选取6只健康成年竹鼠作为试验动物,采集新鲜的盲肠内容物和盲肠液混合均匀,4层纱布过滤去除食物残渣,1 450 r/min离心30 min,取上清,12 000 r/min离心15 min,弃上清,沉淀用磷酸盐缓冲液(PBS)洗涤3次后悬浮,-80 ℃保存。
在Zhou等[5]和Murray等[6]的方法基础上进行改进,建立竹鼠盲肠细菌总DNA的提取方法,并对所提取的总DNA进行纯化。
用细菌16S rDNA通用引物F27(5′-AGAGTTTGATCCTGGCTCAG-3′)和1492R(5′-TACGGCTACCTTGTTACGACTT-3′)进行扩增。50 μL反应体系包括10×PCR Buffer 5 μL,2.5 mmol/L的dNTP 2 μL,25 mmol/L的MgCl2 3 μL,上、下游引物(10 pm/μL)各1 μL,Taq DNA聚合酶0.5 μL,模板DNA 2 μL,补水至50 μL。PCR反应条件为:95 ℃预变性5 min,95 ℃变性30 s,56 ℃退火30 s,72 ℃延伸90 s,30个循环,最后72 ℃延伸10 min。PCR产物经1.0%的琼脂糖凝胶电泳检测。
用琼脂糖凝胶回收试剂盒对扩增产物进行纯化回收,回收产物连接pGEM-T Easy载体(Promage,美国),连接产物转入大肠杆菌DH 5α高感受态细胞(TaKaRa,大连),构建16S rDNA基因文库。转化后的大肠杆菌在37 ℃振荡培养1.5 h,取100 μL涂布含Amp+/X-gal/IPTG的LB培养基,37 ℃倒置培养过夜,4 ℃放置3 h使蓝白斑充分显色。
用无菌牙签随机挑选白色菌落,采用载体通用引物T7(5′-TAATACGACTCACTATAGGG-3′)和SP6(5′-CATACGATTTAGGTGACACTATAG-3′)进行扩增。PCR产物用限制性内切酶HaeⅢ和HhaⅠ进行酶切,产物经2%的琼脂糖凝胶电泳及溴化乙锭(EB)染色后,在凝胶成像仪上拍照。最后对电泳图谱进行ARDRA,将酶切图谱相同的克隆归为1个操作分类单元(OTU)。
根据16S rDNA酶切带型差异,统计基因文库中每种OTU的种类及与之对应的克隆数,从每种类型的OTU中选择1~2个代表克隆送到生工生物工程(上海)股份有限公司进行测序,所得序列提交GenBank进行Blast序列同源性比对。
从竹鼠盲肠微生物中提取的总DNA条带单一整齐、明亮无拖尾(图1)。以总DNA为模板使用细菌通用引物27F和1492R扩增出约1.5 kb的细菌16S rDNA基因目的片段(图2)。
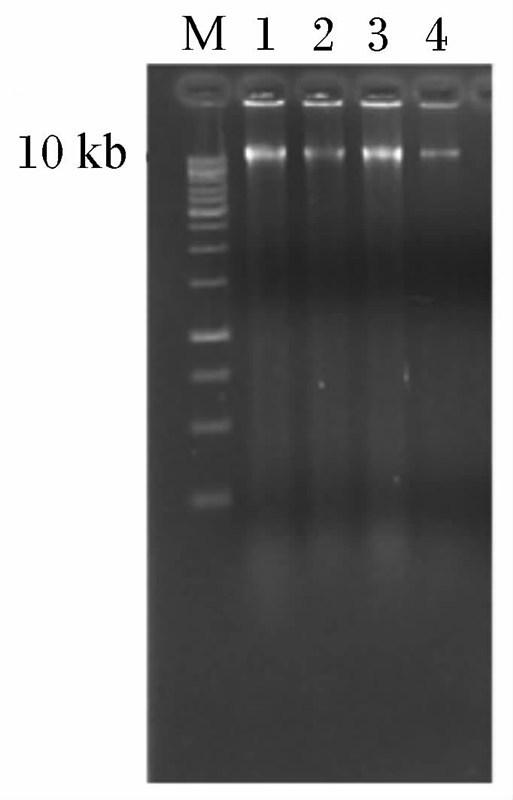 | M:DNA分子质量标准;1~4:总DNA。 M: DNA molecular weight marker; 1 to 4: total DNA. 图1 总DNA电泳结果 Fig.1 Electrophoresis result of total DNA |
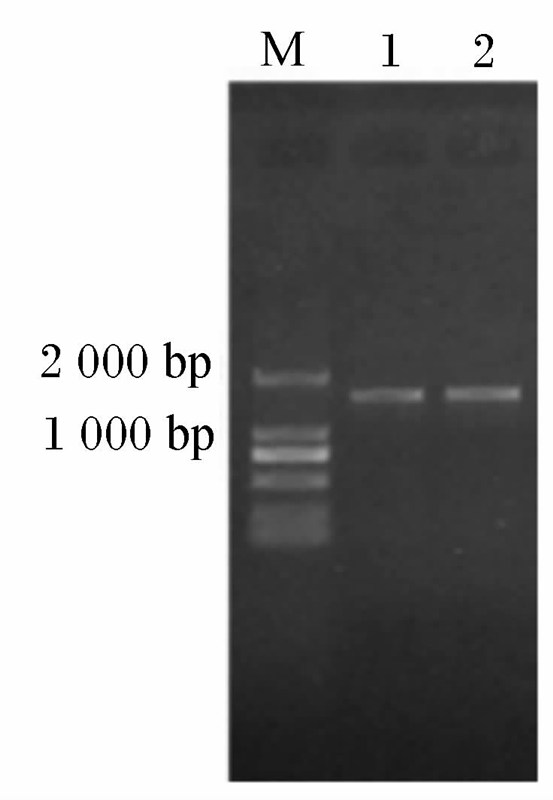 | M:DNA分子质量标准;1和2:16S rDNA目的片段。 M: DNA molecular weight marker; 1 and 2: 16S rDNA target segment.图2 16S rDNA的扩增产物 Fig.2 16S rDNA amplification products |
从基因文库中随机选取克隆250个,通过菌落PCR法进行插入片段检测,阳性率高达87.2%。将所得的218个阳性克隆经菌液PCR后,再用限制性内切酶HaeⅢ和HhaⅠ对大约1.7 kb的序列进行酶切(包括部分载体序列),电泳结果显示,限制性内切酶HaeⅢ和HhaⅠ的酶切谱带存在明显的差异性(图3)。利用Quantity one软件(Bio-Rad,美国)对酶切图谱进行分析,得到34个不同的OTU,每个OTU挑选1个16S rDNA克隆产物进行DNA序列测定,从而得到34个有效序列。经计算,测试盲肠样品中细菌16S rDNA基因文库的库容值C值为88.23%。
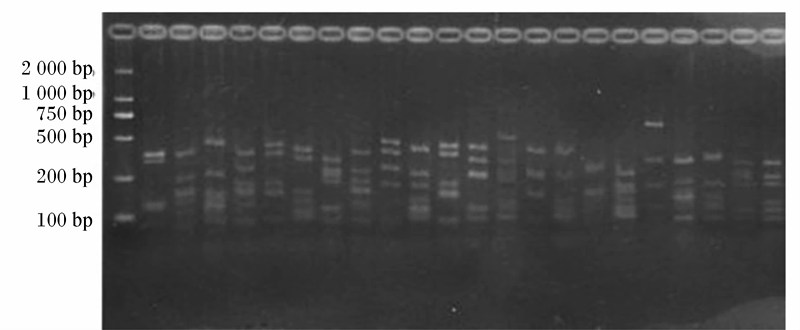 | 图3 克隆的酶切图谱
Fig.3 Restriction fragment patterns of clones |
将测序所得的34个序列与GenBank数据库中已知序列进行比对,其中有4个OTU与基因文库中的序列相似度达100%,占所有序列的11.76%。只有1个OTU与基因文库中比对的相似度低于90%。比对的相似度≥97%的比例占到32.35%。如表1所示,KN110801、KN110831、KN110814和KN110817在克隆库中的克隆数均大于20个,而KN110801和KN110831即为芽孢杆菌属和梭形杆菌属的菌群。KN110811、KN110822、KN110827和KN110833只包含1个克隆子,即它们的HaeⅢ酶切型在这个克隆库中是唯一的。
| 表1 16S rDNA序列分析 Table 1 16S rDNA sequence analysis |
ARDRA通过对16S rDNA酶切产物的琼脂糖凝胶电泳检测,能够较客观地反映样品间微生物种群结构的遗传差异,尤其在环境微生物遗传多样性分析[5, 6, 7]、物种鉴定[8, 9]等研究中已被广泛应用。该方法克服了传统微生物培养方法的弊端,与其他分子生物学研究方法相比,ARDRA对仪器设备的依赖性小、操作简便,更适合开展微生物种群研究。
本次试验首次将ARDRA技术应用于竹鼠盲肠的微生物多样性研究,得到比较理想的结果,通过测序并在GenBank中进行Blast序列同源性比对,结果显示竹鼠盲肠细菌菌群的多样性非常高,且蕴含着丰富的未培养或未知微生物资源,因此可以在优化培养条件和检测手段方面进一步探索。
肠道微生物在宿主能量代谢的调控中有着不可忽视的作用,其主要的代谢功能就是发酵那些不能被小肠吸收的食物残渣和上皮细胞分泌的内生黏液[10]。肠道微生物群落的基因多样性为这个复杂的系统提供了宿主不具备的生化功能和酶,使其可以发酵纤维素、半纤维素、胶质和多糖等产生能量,而这些能量最终以短链脂肪酸供给宿主[11]。Bckhed等[12]研究发现,肠道菌群的作用不仅仅是帮助宿主从食物中获得更多能量,更为重要的是可以直接调节宿主脂肪存储组织的基因表达活性,使宿主增加脂肪的积累,起着保护内脏器官、隔热、保温、防止热量散失、保持正常体温等一系列作用。
因为本次试验只对竹鼠盲肠中细菌菌群组成做了初步的探索性研究,并未对其他的菌群(如真菌和古菌)做全面的研究,因此,有待更加深入全面的研究,为竹鼠盲肠微生物的多样性补充更多的数据,从而提高竹鼠的经济价值和开发未知菌株的生物学功能。
竹鼠盲肠中的细菌菌群大部分是未培养或未知的菌株,在分类学上具有潜在意义的被选菌株;芽孢杆菌属、梭形杆菌属和2个未知菌株的克隆数都大于20个,是优势菌株。这种优势菌群的分布与竹鼠的生活习性和生理特性有着密切的关联。
| [1] | VANEECHOUTTE M,ROSSAU R,DE VOS P,et al.Rapid identification of bacteria of the Comamonadaceae with amplified ribosomal DNA-restriction analysis (ARDRA)[J]. FEMS Microbiology Letters,1992,93(3):227-234. ( 1) 1)
|
| [2] | WEIDNER S,ARNOLD W,PUHLER A.Diversity of uncultured microorganisms associated with the seagrass Halophila stipulacea estimated by restriction fragment length polymorphism analysis of PCR-amplified 16S rRNA genes[J]. Applied and Environmental Microbiology,1996,62(3):766-771. ( 1) 1)
|
| [3] | HEYNDRICKX M,VANDAMME P,VANDAMME P,et al.Applicability of combined amplified ribosomal DNA restriction analysis (ARDRA) patterns in bacterial phylogeny and taxonomy[J]. Journal of Microbiological Methods,1996,26(3):247-259. ( 1) 1)
|
| [4] | LAGUERRE G,ALLARD M R,REVOY F,et al.Rapid identification of rhizobia by restriction fragment length polymorphism analysis of PCR-amplified 16S rRNA genes[J]. Applied and Environmental Microbiology,1994,60(1):56-63. ( 1) 1)
|
| [5] | ZHOU J Z,BRUNS M A,TIEDJE J M.DNA recovery from soils of diverse composition[J]. Applied and Environmental Microbiology,1996,62(2):316-322. ( 2) 2)
|
| [6] | MURRAY A E,PRESTON C M,MASSAWA R,et al.Seasonal and spatial variability of bacterial and archaeal assemblages in the coastal waters near Anvers Island,Antarctica[J]. Applied and Environmental Microbiology,1998,64:2585-2595. ( 2) 2)
|
| [7] | LIU B,ZENG Q,YAN F M,et al.Effects of transgenic plants on soil microorganisms[J]. Plant and Soil,2005,271(1/2):1-13. ( 1) 1)
|
| [8] | YANG Y,SHI W Y,WAN M X,et al.Diversity of bacterial communities in acid mine drainage from the Shen-bu copper mine,Gansu province,China[J]. Electron Journal of Biotechnology,2008,11(1):107-118. ( 1) 1)
|
| [9] | MWIRICHIA R,COUSIN S,MUIGAI A W,et al.Archaeal diversity in the haloalkaline Lake Elmenteita in Kenya[J]. Current Microbiology,2010,60(1):47-52. ( 1) 1)
|
| [10] | ROBERFROID M B,BORNET F,BOULEY C,et al.Colonic microflora:nutrition and health.Summary and conclusions of an International Life Sciences Institute (ILSI)[Europe] workshop held in Barcelona,Spain[J]. Nutrition Reviews,1995,53(5):127-130. ( 1) 1)
|
| [11] | CUMMINGS J H,BEATTY E R,KINGMAN S M,et al.Digestion and physiological properties of resistant starch in the human large bowel[J]. British Journal of Nutrition,1996,75(5):733-747. ( 1) 1)
|
| [12] | BÄCKHED F,DING H,WANG T,et al.The gut microbiota as an environmental factor that regulates fat storage[J]. Proceedings of the National Academy of Sciences of the United States of America,2004,101(44):15718-15723. ( 1) 1)
|




