真核生物的染色质作为调节遗传和变异的物质基础,主要是由DNA和组蛋白组成。染色质组蛋白的共价修饰以及DNA本身的修饰可影响基因的表达,这种可遗传的变异修饰称作表观遗传调控[1, 2]。表观遗传学是在DNA序列不发生改变的情况下,研究基因的表达产生可遗传变化的学科[3]。表观遗传过程的分子基础是复杂的,主要包括染色质结构的改变(DNA甲基化、组蛋白修饰和染色质重组)以及microRNAs(miRNAs)调控[4]。本文先就表观遗传进行概述,然后着重综述各种营养素对机体DNA甲基化状态的影响,并对其机制进行探讨,旨在为从分子的角度调控畜禽营养代谢和生产性能提供新的思路。
DNA甲基化是指在DNA甲基转移酶(DNMTs)催化作用下,以S-腺苷-L-甲硫氨酸(SAH)提供甲基供体,将其甲基转移到脱氧胞嘧啶环第5位碳原子形成甲基化脱氧胞嘧啶的共价修饰[5]。哺乳动物的DNMTs家族主要包括DNMT1、DNMT3a和DNMT3b[6]。哺乳动物的DNA甲基化模式一般是在胚胎生长期由从头合成甲基化转移酶DNMT3a和DNMT3b建立的[7],在随后的复制过程中及复制后由DNMT1维持甲基化状态[2]。而近年来的研究表明必须在DNMT1、DNMT3a和DNMT3b的共同作用下,DNA甲基化状态才能得以稳定维持[8]。DNA甲基化主要发生在胞嘧啶和鸟嘌呤二核苷酸序列岛(CGI)。CGI是指哺乳动物基因启动子及其附近含有大量胞嘧啶和鸟嘌呤二核苷酸(CpG)位点的区域。最近关于胚胎细胞和成年动物组织的研究发现,非CGI也能发生甲基化。非CGI的甲基化主要发生在基因编码区,也有极少数的发生于蛋白结合位点以及增强子区域[9]。另外,研究表明在DNA重复序列的转录沉默子和内源转座子的DNA甲基化在基因组完整性的维护中有非常重要的作用[10]。
作为染色质的基本结构单位,核小体主要由约146 bp的DNA缠绕在2分子的H2A、H2B、H3和H4构成的核心组蛋白上组成。组蛋白的共价修饰是基因表达的重要表观遗传调控方式,主要包括赖氨酸和精氨酸的甲基化、乙酰化、磷酸化、遍在蛋白化、小泛素相关修饰物(SUMO)修饰以及二磷酸腺苷(ADP)核糖基化,通常发生在核心组蛋白的N端[11],其中以组蛋白H3和组蛋白H4氨基酸末端赖氨酸残基的乙酰化和甲基化修饰最为常见。一般来说,组蛋白乙酰化促进转录,而组蛋白去乙酰化抑制转录。乙酰化促进转录的机制主要是在组蛋白乙酰化酶(HAT)的作用下,使染色质“打开”[12],核小体区域乙酰化的N端伸出,降低了组蛋白与DNA之间的亲和力,使染色质有更疏松的结构,从而有利于转录的进行。HAT与去乙酰化酶(HDAC)共同调节体内组蛋白乙酰化的平衡。组蛋白甲基化也能使染色质处于活化或抑制状态,其作用方向主要取决于赖氨酸残基的位置[13]。根据每一位点甲基化程度的不同,赖氨酸残基能分别被单甲基化(me1)、双甲基化(me2,对称或非对称)和三甲基化(me3)[14]。不同组蛋白或不同残基之间可互相调节,如组蛋白H3第10位丝氨酸(H3S10)的磷酸化促进H3第9位赖氨酸(H3K9)与第14位赖氨酸(H3K14)的乙酰化,抑制H3K9的甲基化,从而导致基因呈活化状态。同时,H3第4位赖氨酸(H3K4)的甲基化还可促进H3K9的乙酰化。相反,H3K9的甲基化抑制了H3S10的磷酸化,并且抑制了H3K9、H3K14的乙酰化,从而导致基因沉默[15]。
miRNAs是一种通过抑制翻译过程以及使mRNA失去稳定性来调节基因表达的内源非编码的长度约为23个核苷酸的小RNA(RNAs)[16],它在调节DNA甲基化和组蛋白乙酰化的过程中有着十分重要的作用,而DNA甲基化和组蛋白乙酰化也能反过来调控miRNAs的表达。miRNAs主要通过与mRNA分子的正确配对来降解与之同源的mRNAs,进而调节基因的功能;或通过miRNAs与目标mRNA分子3′端未翻译区域之间的第2~7位核苷酸(也叫“种子序列”)的碱基配对来抑制蛋白转录[17]。研究表明miRNAs主要通过以下几种途径抑制翻译:抑制翻译的开始和延伸、促使共翻译蛋白的降解以及翻译的提前终止[16]。目前在哺乳动物发现的由基因组编码的miRNAs已经多达500余种,而每一种动物miRNAs都可以调控上百种不同的mRNAs,说明miRNAs可调控大部分的转录基因组[18, 19]。
DNA甲基化和组蛋白修饰可以相互触发,并且这2个过程是通过甲基-CpG结合域蛋白(MeCP)相互联系的。一方面,MeCP可以结合CpG二核苷酸,通过招募组蛋白去乙酰化酶(HDTC)和组蛋白甲基转移酶(HMT),促进异染色质形成,启动基因沉默[20]。另一方面,与组蛋白结合的蛋白(如异染色质蛋白)或修饰组蛋白的蛋白(如赖氨酸脱甲基化酶和蛋白精氨酸甲基转移酶)能招募DNMT使DNA甲基化,从而使沉默基因表达[21]。哺乳动物组蛋白甲基化与DNA甲基化状态高度相关,特别是对基因表达具有相反效应的组蛋白H3K4和H3K9,H3K4甲基化上调基因表达,H3K9下调基因表达[22]。
大量研究表明,营养素对畜禽表观遗传具有重要的影响。蜜蜂发展成为蜂王还是工蜂主要取决于其早期食物是蜂王浆还是花粉。蜂王和工蜂头部DNA甲基化的CpG数量基本一致,但有561个基因的甲基化状态呈显著差异,且这些基因多为组蛋白家族基因[23]。用RNA干扰使DNMT3沉默,结果发现幼蜂向蜂王方向发展,即DNA甲基化对幼蜂的发育方向具有重要的决定作用[24]。Choi等[25]对荷斯坦奶牛饲粮所有营养素进行反复的限制与补偿,结果发现整个试验过程中试验组乳腺组织DNA甲基化程度都低于对照组,并且αs1与β酪蛋白的表达量高于对照组,表明营养素的改变可能会导致DNA甲基化状态的改变,并且这种改变很有可能会影响到畜禽的生产性能。
叶酸既可以辅酶形式促进嘌呤和嘧啶的生物合成,还可提供甲基基团合成腺苷蛋氨酸,因此与DNA甲基化之间存在紧密的联系。叶酸缺乏对DNA甲基化的影响具有位点和基因特异性,其甲基化改变的方向也具有细胞、靶器官和转化阶段特异性,并且在基因组和特定基因或特定位点的DNA甲基化程度是不同的[26]。在小鼠上的试验表明,叶酸缺乏使小鼠肝脏和结肠的S-腺苷甲硫氨酸/S-腺苷-L-高半胱氨酸(SAM/SAH)显著降低,DNA呈现低甲基化状态[27],且其基因组DNA甲基化水平与饲粮叶酸水平呈正相关[28, 29],而小肠和脾脏中未见明显的低甲基化水平[30]。补充叶酸还可有效抑制由胃螺旋杆菌导致的总DNA甲基化水平的下降,显著改善胃发育不良[31]以及提高过氧化物酶体增生物激活受体-α(PPAR-α)和糖皮质激素受体(GR)基因启动子甲基化水平,并显著降低胰岛素受体基因启动子甲基化水平[32],表明叶酸除了对消化系统的表观遗传进行调控外,对肝脏糖异生过程中的关键因子也有重要的调控作用。此外,叶酸对神经系统生长发育也存在重要的表观遗传调控。给雄鼠饲喂叶酸缺乏饲粮或含叶酸的饲粮后,发现父源的叶酸缺乏可影响胎儿脑部DNA甲基化程度[33]。塞拉嗪损伤脊索神经元可抑制DNMT3a和DNMT3b蛋白水平,使全基因组DNA甲基化水平下降40%,而用叶酸处理可使其恢复到受伤前的水平[34]。
饲粮中的胆碱可通过调节DNA甲基化状态影响神经系统的生长发育,而母源饲粮胆碱水平对胎儿的脑部发育极其重要。研究表明,小鼠胚胎时期缺乏胆碱可使海马神经上皮层总DNA甲基化水平显著下降,齿状回甲基化程度无显著变化,与细胞周期相关的蛋白激酶抑制因子3(Cdkn3)基因的DNA甲基化程度也显著降低,由于小鼠胚胎时期主要是海马室及室下区域进行细胞的增殖和迁移,此研究结果表明母源饲粮甲基供体的摄入主要影响具有增殖潜力的细胞;此外钙结合蛋白1(Calb1)基因的表达显著增强,而Calb1主要是在成熟的神经元细胞表达,即胆碱缺乏促进了神经元的分化[35]。母源性胆碱缺乏还可使血管内皮细胞的增殖率显著下降,海马血管数量显著下降,血管生成的重要调节因子——血管内皮生长因子C以及血管生成素2基因启动子附近出现低甲基化且表达量显著增加[36]。由此可知,饲粮胆碱缺乏不仅影响神经元细胞的表观遗传状态,对脑部血管内皮细胞及血管的发生也具有一定的表观遗传调控作用。
维生素B12主要通过与钴胺素传递蛋白受体(TCblR)结合,以钴胺素传递蛋白为载体在血液中运输。Silvia等[37]利用TCblR/CD320基因敲除小鼠来控制血液循环中维生素B12的量,从而研究维生素B12缺乏对小鼠大脑表观遗传的影响。结果发现基因敲除小鼠大脑中维生素B12的浓度比对照组显著降低92%,大脑组织全基因组DNA甲基化水平下降44%,即维生素B12缺乏可影响DNA的甲基化状态,并进一步表明维生素B12缺乏可破坏结肠黏膜一碳循环的稳定,进而导致DNA甲基化水平降低,DNA中尿嘧啶的错误掺入增加。严重缺乏维生素B12可导致总DNA及胱硫醚β合酶(CBS)基因启动子均呈低甲基化,DNMT1基因表达显著上调,一碳循环过程中的甲硫氨酸腺苷转移酶1A(MAT1A)、S-腺苷高半胱氨酸水解酶(AHCY)和甜菜碱-高半胱氨酸甲基转移酶(BHMT)以及CBS基因表达显著下调,甲硫氨酸腺苷转移酶2A(MAT2A)和5-甲基四氢叶酸-高半胱氨酸甲基转移酶还原酶(MTRR)基因表达显著上调;添加甲硫氨酸后MAT1A、MAT2A、AHCY以及BHMT基因表达水平恢复到正常水平,CBS及MTRR基因表达水平的改变以及总DNA甲基化状态并未得到改善[38]。MAT、AHCY、BHMT、MTRR都是一碳循环过程中的关键酶(图1),通过维生素B12缺乏与添加甲硫氨酸后各基因甲基化状态的不同改变,我们推测维生素B12可能主要通过影响一碳循环过程中的主要酶而改变DNA甲基化状态,虽然部分酶在甲硫氨酸对甲基化状态的作用路径中也起着一定的作用,但维生素B12与甲硫氨酸对DNA甲基化的影响路径是不同的。
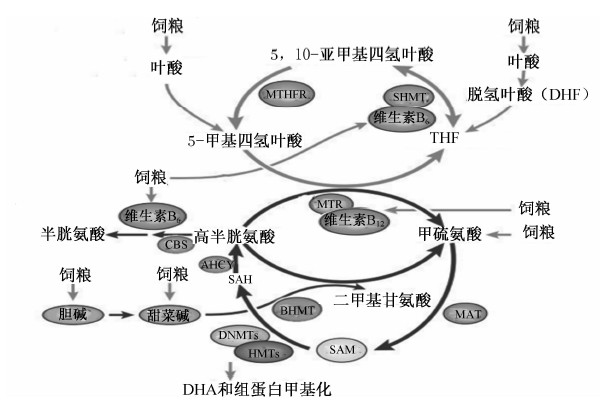 | 图1 饲粮营养素调节DNA和组蛋白甲基化的途径 Fig.1 The regulation pathway of dietary nutrients on DNA and histone methylation |
维生素B12与叶酸在影响DNA甲基化的机制中具有一定协同作用。Kulkarni等[39]发现,维生素B12缺乏使小鼠胎盘二十二碳六烯酸(DHA)水平显著下降;在叶酸水平正常而维生素B12缺乏的条件下,胎盘总DNA甲基化状态无显著变化,维生素B12水平正常和过量叶酸对DNA甲基化水平也无显著影响,但是当添加过量叶酸并缺乏维生素B12时,DNA甲基化水平显著下降。说明叶酸与维生素B12的比例也许是影响DNA甲基化状态的重要因子。然而,Min等[40]对成年小鼠的研究发现,无论是维生素B12缺乏、叶酸过量还是两者并存对DNA甲基化的标志因子SAM、SAH及SAM/SAH均无显著影响,肝脏DNA甲基化状态也未发生改变,表明维生素B12对DNA甲基化的影响也具有生长阶段特异性。Min等[40]还提出维生素B12缺乏可能导致“甲基叶酸陷阱”,从而损害细胞内叶酸的储存系统以提高血液中叶酸水平。此外,限制母羊围妊娠期饲粮中的叶酸、维生素B12和甲硫氨酸,其后代1 400个CGI中4%位点的甲基化状态发生了改变,并伴随着肥胖症和高血压的发生以及胰岛素抗药性的增强和免疫功能的改变[41]。
叶酸、维生素B6、维生素B12、胆碱和甲硫氨酸可通过直接或间接参与一碳循环过程影响SAM的生成,SAM是DNA和组蛋白甲基化的甲基供体[42](图1)。来自饲粮的叶酸首先要被转化成脱氢叶酸,然后转化成四氢叶酸(THF)进入叶酸循环。THF的甲基基团分别在丝氨酸羟甲基转移酶(SHMT)、亚甲基四氢叶酸还原酶(MTHFR)以及5-甲基四氢叶酸-高半胱氨酸甲基转移酶(MTR)的作用下,从THF转移到5,10-亚甲基四氢叶酸再到5-甲基四氢叶酸,最后传递给SAM。随后在DNMTs和组蛋白甲基转移酶(HMTs)作用下利用SAM的甲基基团使DNA和组蛋白发生甲基化。一碳循环也可利用来自甜菜碱的甲基基团。维生素B6和维生素B12是SAM生物合成的重要辅因子,维生素B6还参与高半胱氨酸转化为半胱氨酸以及THF转化为5,10-亚甲基四氢叶酸的过程,维生素B12也是MTR的辅因子。如果半胱氨酸蓄积过量,有利于SAH合成,SAH便反过来抑制甲基转移酶的作用,从而对甲基化反应产生负效应[43]。但是最近有研究表明,肝脏中SAM的浓度非常稳定,预示着特定基因表观遗传机制可能不仅仅与可用于DNA甲基化反应的SAM有关[40, 44]。
瘦素可通过抑制脂肪的合成和促进脂肪分解来调节机体的体重。Milagro等[45]研究发现,采食高能饲粮的大鼠比采食正常能量水平饲粮(对照组)的大鼠更重,此试验中高能饲粮组脂肪组织瘦素启动子区甲基化程度显著高于对照组,其中在-443 pb处CpG甲基化程度显著高于对照组,这一位点的高甲基化与瘦素的低表达相关,并与循环系统中的瘦素水平呈负相关。由此可知,高能饲粮可改变脂肪组织瘦素启动子CGI的甲基化状态,进而引起瘦素的低表达以及循环系统中瘦素水平的降低,从而使脂肪代谢发生紊乱,最终导致体重增加,甚至肥胖症以及相关疾病的发生,这在人类的试验上也得到了验证[46]。黑皮质素4受体是通过降低采食量来调节体重的另一个重要因子,研究表明高能饲粮能导致其外显子甲基化水平降低[47]。饲喂高能饲粮的妊娠小鼠的后代多巴胺再摄取转运体(DAT)、μ-阿片受体(MOR)和前脑啡肽原(PENK)基因的启动子区发生了低甲基化,且其后代相对于对照组更加偏爱蔗糖和脂肪,提示母体摄入高能饲粮除了影响后代的甲基化水平,还可以影响后代的采食行为[48]。DAT、MOR、PENK均与多巴胺和阿片肽有关,而多巴胺和阿片肽通路是影响动物对食物采食偏好的重要神经底物。总而言之,高能饲粮可影响肥胖相关基因的甲基化状态,并通过调控相关基因的表达和水平,最终达到影响机体采食行为以及体重的效果。此外,肥胖相关基因的甲基化状态可能对肥胖症及相关疾病的发生也具有一定的调控作用。
研究表明,一方面,高能饲粮可降低烟酰胺腺嘌呤二核苷酸辅酶与其还原形式的比值(NAD+/NADH),而低能饲粮可提高NAD+/NADH[42],同时NADH与甲硫氨酸合酶的活性有关[49],因此我们推测能量可能通过影响甲硫氨酸的合成来影响DNA甲基化的状态。另一方面,高能饲粮还可通过调节采食量而改变其他各种营养素的摄入量,叶酸和蛋白质以及矿物质摄入量的改变均可能引起DNA甲基化水平的改变。然而这些推测均还需要有更多的试验结果来证明。
作为生命的物质基础,蛋白质是畜禽饲粮必不可缺少的营养素。大量研究表明,母源饲粮中蛋白质水平对其后代DNA甲基化状态也具有一定的调控作用。给雌鼠饲喂低蛋白质饲粮,其F1代PPARα[50]以及糖皮质激素受体110(GR110)[51]基因启动子均出现低甲基化,基因表达量显著增加,DNMT1基因表达水平显著下降,且这种甲基化的改变可传递给F2代[52]。Jia等[53]给妊娠的初产母猪分别饲喂含正常蛋白质(粗蛋白质含量为12%)饲粮和低蛋白质(粗蛋白质含量为6%)饲粮后,低蛋白质组新生小猪初生重及肝、脑、心和肾的重量均显著降低,GR与葡萄糖-6-磷酸激酶(G6PC)启动子的结合也显著降低,并伴随着G6PC启动子区域的低甲基化。小公猪血糖浓度和G6PC活性及基因表达水平均显著下降,而肝脏糖原浓度升高。公母猪组蛋白的修饰也是依赖性别的,低蛋白质组小公猪H3第9位赖氨酸的三甲基化(H3K9me3)以及H3水平显著降低,而H3乙酰化和组H3第4位赖氨酸的三甲基化(H3K4me3)水平显著升高。此外,低蛋白质组小母猪作用于G6PC的3′端未翻译区域的2种miRNA ssc-miR-339-5p和ssc-miR-532-3p水平也显著上调。Jia等[54]在随后的试验中还发现低蛋白质组小公猪肝脏平滑肌磷酸酶(AMP)水平显著升高,NADH脱氢酶6亚基、细胞色素C氧化酶1、2、3亚基和细胞色素B基因表达水平显著升高,并伴随着细胞色素C氧化酶活性的显著增加,同时肝脏线粒体DNA启动子甲基化程度显著降低,而在新生小母猪却完全与之相反,表明母源性的低蛋白质饲粮通过改变糖异生过程中的关键因子如PPARα、GR110和G6PC的甲基化状态和线粒体DNA甲基化水平,调节相关基因以及有氧呼吸过程中相关酶的基因表达水平,进而影响新生小公猪肝脏糖异生的过程,并表现为生长滞后,且这种影响是具有性别特异性的。
目前,关于低蛋白质饲粮影响一碳循环的机制主要有2种可能:一是低蛋白质可降低可用的甘氨酸,从而影响不同代谢终途径之间甲基基团的介入;另一种可能机制是由于营养素限制导致机体产生应激或通过调节GR110基因的表达,从而增加皮质醇活性,降低叶酸利用率。Lillycrop等[55]也指出,叶酸可消除由低蛋白质导致的低甲基化现象并提高DNMT1的表达,这也再次验证了上述的第2种机制的可能。然而,对于蛋白质影响DNA甲基化状态的具体机制还需要更多的研究来进一步说明。
饲粮中矿物元素硒、锌、镁以及铜含量与DNA甲基化也存在一定的联系。在高脂肪饲粮的基础上,添加硒和叶酸的后代其肝脏全基因组DNA甲基化水平显著降低[56]。硒缺乏鼠可导致结肠全基因组DNA甲基化水平显著下降,而对肝脏DNA甲基化水平无显著影响[57]。适量(0.15 mg/kg)和超标(4.00 mg/kg)的硒与硒缺乏饲粮相比显著降低了鼠肝脏总DNA甲基化水平[58]。缺锌使鼠肝脏SAM提供的可利用的甲基供体减少,导致基因组DNA低甲基化[59];还可显著降低卵母细胞的甲基化水平[60]。大鼠怀孕期间缺镁也会使后代的肝脏11β-羟基类固醇脱氢酶2(Hsd11b2)基因启动子超甲基化,并伴随着Hsd11b2基因表达水平的显著降低。Hsd11b2可保护盐皮质激素受体以防止糖皮质激素过量,说明母源镁缺乏可改变后代糖皮质激素的平衡[61]。此外,铜与DNA甲基化可能也存在一定的联系[62]。
矿物元素主要作为一碳循环过程中各种酶的辅因子或者重要组成成分而对甲基化过程产生影响。研究表明,硒影响DNA甲基化的途径至少有2条:硒影响甲硫氨酸的代谢[63]和高半胱氨酸的转硫作用[64],从而扰乱一碳循环;硒还可通过影响DNMTs的活性来限制甲基基团的利用[65]。而锌可影响一碳循环过程中与甲基化反应有关的BHMT和MTR,BHMT是一种含锌金属酶,可催化甲基基团从甜菜碱转移到高半胱氨酸来合成二甲基甘氨酸和SAM的前提物甲硫氨酸;此外,锌还与甲硫氨酸的合成密切相关[60]。
营养素缺乏与过量均会造成机体生长性能与各种表型发生变化,但是这种表型的变化并未涉及到遗传信息的改变,而是表现在DNA甲基化状态的改变。大量研究表明,营养素对机体生长发育过程中各种相关基因的表达具有重要的表观遗传调控作用,并且各营养素之间互相影响、互相协同。近年来,对于DNA甲基化的研究主要集中在其与癌症的相关性上,对畜禽生产性能表观调控的研究虽然较少,但也逐渐成为分子营养研究的热点。从表观遗传的角度来研究饲粮营养素与DNA之间的关系,对于调控畜禽的营养代谢和生产性能将具有十分重要的作用。
| [1] | HENIKOFF S,SHILATIFARD A.Histone modification:cause or cog?[J]. Trends in Genetics,2011,27(10):389-396. ( 1) 1)
|
| [2] | LAW J A,JACOBSEN S E.Establishing,maintaining and modifying DNA methylation patterns in plants and animals[J]. Nature Reviews Genetics,2010,11(3):204-220. ( 2) 2)
|
| [3] | WILFFE A P,GUSCHIN D.Review:chromatin structural features and targets that regulate transcription[J]. Journal of Structural Biology,2000,129(2/3):102-122. ( 1) 1)
|
| [4] | BAYARSAIHAN D.Epigenetic mechanisms in inflammation[J]. Journal of Dental Research,2011,90(1):9-17. ( 1) 1)
|
| [5] | DAVIS C D,UTHUS E O.DNA methylation,cancer susceptibility,and nutrient interactions[J]. Experimental Biology and Medicine,2004,229(10):988-995. ( 1) 1)
|
| [6] | HERMANN A,GOWHER H,JELTSCH A.Biochemistry and biology of mammalian DNA methyltransferases[J]. Cellular and Molecular Life Sciences,2004,61(19/20):2571-2587. ( 1) 1)
|
| [7] | OKANO M,BELL D W,HABER D A,et al.DNA methyltransferases Dnmt3a and Dnmt3b are essential for de novo methylation and mammalian development[J]. Cell,1999,99(3):247-257. ( 1) 1)
|
| [8] | JONES P A,LIANG G N.Rethinking how DNA methylation patterns are maintained[J]. Nature Reviews Genetics,2009,10(11):805-811. ( 1) 1)
|
| [9] | BARRÕS R,OSLER M E,YAN J,et al.Non-CpG methylation of the PGC-1α promoter through DNMT3B controls mitochondrial density[J]. Cell Metabalism,2009,10(3):189-198. ( 1) 1)
|
| [10] | HEDGES D J,DEININGER P L.Inviting instability:transposable elements,double-strand breaks,and the maintenance of genome integrity[J]. Mutation Research,2007,616(1/2):46-59. ( 1) 1)
|
| [11] | FUCHS J,DEMIDOV D,HOUBEN A,et al.Chromosomal histone modification patterns-from conservation to diversity[J]. Trends in Plant Science,2006,11(4):199-208. ( 1) 1)
|
| [12] | CHENG X D,BLUENTHAL R M.Coordinated chromatin control:structural and functional linkage of DNA and histone methylation[J]. Biochemistry,2010,49(14):2999-3008. ( 1) 1)
|
| [13] | BOLLATI V,BACCARELLI A.Environmental epigenetics[J]. Heredity,2010,105(1):105-112. ( 1) 1)
|
| [14] | MARTIN C,ZHANG Y.The diverse functions of histone lysine methylation[J]. Nature Reviews Molecular Cell Biology,2005,6(11):838-849. ( 1) 1)
|
| [15] | JOHNSON L,MOLLAH S,GARCIA B A,et al.Mass spectrometry analysis of Arabidopsis histone H3 reveals distinct combinations of post-translational modifications[J]. Nucleic Acids Research,2004,32(23):6511-6518. ( 1) 1)
|
| [16] | HUNTZINGER E,LZAURRALDE E.Gene silencing by microRNAs:contributions of translational repression and mRNA decay[J]. Nature Reviews Genetics,2011,12(2):99-110. ( 2) 2)
|
| [17] | ZAMORE P D,HALEY B.Ribo-gnome:the big world of small RNAs[J]. Science,2005,309(5740):1519-1524. ( 1) 1)
|
| [18] | BARTEL P D.MicroRNAs:target recognition and regulatory functions[J]. Cell,2009,136(2):215-233. ( 1) 1)
|
| [19] | VOINNET O.Origin,biogenesis,and activity of plant microRNAs[J]. Cell,2009,136(4):669-687. ( 1) 1)
|
| [20] | 刘瑾,汪晖,张本坚,等.表观遗传修饰在胚胎发育过程中的调控作用研究进展[J]. 中国药理学远毒理学杂志,2012,26(4):591-594. ( 1) 1)
|
| [21] | HANDEL A E,EBERS G C,RAMAGOPALAN S V,et al.Epigenetics:molecular mechanisms and implications for disease[J]. Trends in Molecular Medicine,2010,16(1):7-16. ( 1) 1)
|
| [22] | CHOI S W,FRISO S.Epigenetics:a new bridge between nutrition and health[J]. Advances in Nutrition,2010,1(1):8-16. ( 1) 1)
|
| [23] | LYKO F,FORET S,KUCHARSKI R,et al.The honey bee epigenomes:differential methylation of brain DNA in queens and workers[J].、PLoS Biology,2010,8(11):e1000506.、( 1) 1)
|
| [24] | KUCHARSKI R,MALESZKA J,FORET S,et al.Nutritional control of reproductive status in honeybees via DNA methylation[J]. Science,2008,319(5871):1827-1830. ( 1) 1)
|
| [25] | CHOI Y J,JANG K,YIM D S,et al.Effects of compensatory growth on the expression of milk protein gene and biochemical changes of the mammary gland in Holstein cows[J]. The Journal of Nutritional Biochemistry,1998,9(7):380-387. ( 1) 1)
|
| [26] | KIM Y I.Nutritional epigenetics:impact of folate deficiency on DNA methylation and colon cancer susceptibility[J]. The Journal of Nutrition,2009,135(11):2703-2709. ( 1) 1)
|
| [27] | DUTHIE S J,GRANT G,PIRIE L P,et al.Folate deficiency alters hepatic and colon MGMT and OGG-1 DNA repair protein expression in rats but has no effect on genome-wide DNA methylation[J]. Cancer Prevention Research,2010,3(1):92-100. ( 1) 1)
|
| [28] | KEYES M K,JANG H,MASON J B,et al.Older age and dietary folate are determinants of genomic and p16-specific DNA methylation in mouse colon[J]. The Journal of Nutrtion,2007,137(7):1713-1717. ( 1) 1)
|
| [29] | CHOI S W,FRISO S,KEYES M K,et al.Folate supplementation increases genomic DNA methylation in the liver of elder rats[J]. British Journal of Nutrition,2005,93(1):31-35. ( 1) 1)
|
| [30] | LINHART H G,TROEN A,BELL G W,et al.Folate deficiency induces genomic uracil misincorporation and hypomethylation but does not increase DNA point mutations[J]. Gastroenterology,2009,136(1):227-235. ( 1) 1)
|
| [31] | TAMAS A G,KIM Y I,MARTHA C S,et al.Folic acid increases global DNA methylation and reduces inflammation to prevent helicobacter-associated gastric cancer in mice[J]. Gastroenterology,2012,142(4):824-833. ( 1) 1)
|
| [32] | BURDGE G C,LILLYCROP K A,PHILLIPS E S,et al.Folic acid supplementation during the juvenile-pubertal period in rats modifies the phenotype and epigenotype induced by prenatal nutrition[J]. The Journal of Nutrition,2009,139(6):1054-1060. ( 1) 1)
|
| [33] | KIM H W,KIM K N,CHOI Y J,et al.Effects of paternal folate deficiency on the expression of insulin-like growth factor-2 and global DNA methylation in the fetal brain[J]. Molecular Nutrition and Food Reasearch,2013,57(4):671-676. ( 1) 1)
|
| [34] | ISKANDAR B J,RIZK E,MEIER B,et al.Folate regulation of axonal regeneration in the rodent central nervous system through DNA methylation[J]. The Journal of Clinical Investigation,2010,120(5):1603-1616. ( 1) 1)
|
| [35] | NICULESCU M D,CRACIUNESCU C N,ZEISEL S H.Dietary choline deficiency alters global and gene-specific DNA methylation in the developing hippocampus of mouse fetal brains[J]. The FASEB Journal,2006,20(1):43-49. ( 1) 1)
|
| [36] | MAHEDINT M G,CRACIUNESCU C N,ZEISEL S H.Maternal dietary choline deficiency alters angiogenesis in fetal mouse hippocampus[J]. Proceedings of the National Academy of Sciences,2010,107(29):12834-12839. ( 1) 1)
|
| [37] | SILVIA F R,LAI S C,MURPHY M M,et al.Vitamin B12 deficiency in the brain leads to DNA hypomethylation in the TCbIR/CD320 knockout mouse[J].Nutrition and Metanolism,2012,9(1):41. ( 1) 1)
|
| [38] | UEKAWA A,KATSUSHIMA K,OGATA A,et al.Change of epigenetic control of cystathionine beta-synthase gene expression through dietary vitamin B12 is not recovered by methionine supplementation[J]. Journal of Nutrigenetics and Nutrigenomics,2009,2(1):29-36. ( 1) 1)
|
| [39] | KULKARNI A,DANGAT K,KALE A,et al.Effects of altered maternal folic acid,vitamin B12 and docosahexaenoic acid on placental global DNA methylation patterns in wistar rats[J]. PLoS One,2011,6(3):e17706. ( 1) 1)
|
| [40] | MIN H.Effects of dietary supplementation of high-dose folic acid on biomarkers of methylating reaction in vitamin B12-deficient rats[J]. Nutrition Research and Practice,2009,3(2):122-127. ( 3) 3)
|
| [41] | SINCLAIR K D,ALLEGRUCCI C,SINGH R,et al.DNA methylation,insulin resistance,and blood pressure in offspring determined by maternal periconceptional B vitamin and methionine status[J]. Proceedings of the National Academy of Sciences of the United States of America,2007,104(49):19351-19356. ( 1) 1)
|
| [42] | FEIL R,FRAGA M F.Epigenetics and the environment:emerging patterns and implications[J]. Nature Reviews Genetics,2012,13(2):97-109. ( 2) 2)
|
| [43] | JIANG J D,ZHANG J Z,HUANG Y,et al.Homocysteine-mediated expression of SAHH,DNMTs,MBD2,and DNA hypomethylation potential pathogenic mechanism in VSMCs[J]. DNA and Cell Biology,2007,26(8):603-611. ( 1) 1)
|
| [44] | ENGEHAM S F,HAASE A,LANGLEY-EVANS S C.Supplementation of a maternal low-protein diet in rat pregnancy with folic acid ameliorates programming effects upon feeding behaviour in the absence of disturbances to the methionine-homocysteine cycle[J]. British Journal of Nutrition,2010,103(7):996-1007. ( 1) 1)
|
| [45] | MILAGRO F I,CAMPIÓN J,GARCIA-DÍAZ D F,et al.High fat diet-induced obesity modifies the methylation pattern of leptin promoter in rats[J]. Journal of Physiology and Biochemistry,2009,65(1):1-9. ( 1) 1)
|
| [46] | CORDERO P,CAMPION J,MILAGRO F I,et al.Leptin and TNF-alpha promoter methylation levels measured by MSP could predict the response to a low-calorie diet[J]. The Journal of Physiology Biochemistry,2011,67(3):463-470. ( 1) 1)
|
| [47] | WIDIKER S,KARST S,WAGENER A,et al.High-fat diet leads to a decreased methylation of the Mc4r gene in the obese BFMI and the lean B6 mouse lines[J]. Journal of Applied Genetics,2010,51(2):193-197. ( 1) 1)
|
| [48] | VUCETIC Z,KIMMEL J,TOTOKI K,et al.Maternal high fat diet alters methylation and gene expression of dopamine and opioid-related genes[J]. Endocrinology,2010,151(10):4756-4764. ( 1) 1)
|
| [49] | OLTEANU H,BANERJEE R.Human methionine synthase reductase,a soluble P-450 reductase-like dual flavoprotein,is sufficient for NADPH-dependent methionine synthase activation[J]. The Journal of Biological Chemistry,2001,276(38):35558-35563. ( 1) 1)
|
| [50] | LILLYCROP K A,PHILLIPS E S,TORRENS C,et al.Feeding pregnant rats a protein-restricted diet persistently alters the methylation of specific cytosines in the hepatic PPARα promoter of the offspring[J]. British Journal of Nutrition,2008,100(2):278-282. ( 1) 1)
|
| [51] | LILLYCROP K A,SLATER-JEFFERIES J L,HANSON M A,et al.Induction of altered epigenetic regulation of the hepatic glucocorticoid receptor in the offspring of rats fed a protein-restricted diet during pregnancy suggests that reduced DNA methyltransferase-1 expression is involved in impaired DNA methylation and changes in histone modifications[J]. British Journal of Nutrition,2007,97(6):1064-1073. ( 1) 1)
|
| [52] | BURDGE G C,SLATE-JEFFERIES J,TORRENS C,et al.Dietary protein restriction of pregnant rats in the F0 generation induces altered methylation of hepatic gene promoters in the adult male offspring in the F1 and F2 generations[J]. British Journal of Nutrition,2007,97(3):435-439. ( 1) 1)
|
| [53] | JIA Y,CONG R,LI R,et al.Maternal low-protein diet induces gender-dependent changes in epigenetic regulation of the glucose-6-phosphatase gene in newborn piglet liver[J]. The Journal of Nutrition,2012,142(9):1659-1665. ( 1) 1)
|
| [54] | JIA Y, LI R, CONG R,et al.Maternal low-protein diet affects epigenetic regulation of hepatic mitochondrial DNA transcription in a sex-specific manner in newborn piglets associated with GR binding to its promoter[J]. PLoS One,2013,8(5):e63855. ( 1) 1)
|
| [55] | LILLYCROP K A,PHILLIPS E S,JCKSON A A,et al.Dietary protein restriction of pregnant rats induces and folic acid supplementation prevents epigenetic modification of hepatic gene expression in the offspring[J]. The Journal of Nutrition,2005,135(6):1382-1386. ( 1) 1)
|
| [56] | BERMINGHAM E N,BASSETT S A,YOUNG W,et al.Post-weaning selenium and folate supplementation affects gene and protein expression and global DNA methylation in mice fed high-fat diets[J]. BMC Medical Genomics,2013,6(1):7. ( 1) 1)
|
| [57] | DAVIS C D,UTHUS E.Dietary folate and selenium affect dimethylhydrazine-induced aberrant crypt formation,global DNA methylation and one-carbon metabolism in rats[J]. The Journal of Nutrition,2003,133(9):2907-2914. ( 1) 1)
|
| [58] | ZENG H,YAN L,CHENG W H,et al.Dietary selenomethionine increases exon-specific DNA methylation of the p53 gene in rat liver and colon mucosa[J]. The Journal of Nutrition,2011,141(8):1464-1468. ( 1) 1)
|
| [59] | SHARIF R,THOMAS P,ZALEWSKI P,et al.The role of zinc in genomic stability[J]. Mutation Reasearch,2012,733(1/2):111-121. ( 1) 1)
|
| [60] | TIAN X,DIAZ F J.Acute dietary zinc deficiency before conception compromises oocyte epigenetic programming and disrupts embryonic development[J]. Developmental Biology,2013,376(1):51-61. ( 2) 2)
|
| [61] | TAKAYA J,IHARADA A,OKIHANA H,et al.Magnesium deficiency in pregnant rats alters methylation of specific cytosines in the hepatic hydroxysteroid dehydrogenase-2 promoter of the offspring[J]. Epigenetics,2011,6(5):573-578. ( 1) 1)
|
| [62] | LINDER M C.The relationship of copper to DNA damage and damage prevention in humans[J]. Mutation Research,2012,733(1/2):83-91. ( 1) 1)
|
| [63] | UTHUS E O,ROSS S A.Dietary selenium affects homocysteine metabolism differently in Fisher-344 rats and CD-1 mice[J]. The Journal of Nutrition,2007,137(5):1132-1136. ( 1) 1)
|
| [64] | UTHUS E O,YOKOI K,DAVOS C D.Selenium deficiency in Fisher-344 rats decreases plasma and tissue homocysteine concentrations and alters plasma homocysteine and cysteine redox status[J]. The Journal of Nutrition,2002,132(6):1122-1128. ( 1) 1)
|
| [65] | FIALA E S,STARETZ M E,PANDYA G A,et al.Inhibition of DNA cytosine methyltransferase by chemopreventive selenium compounds,determined by an improved assay for DNA cytosine methyltransferase and DNA cytosine methylation[J]. Carcinogenesis,1998,19(4):597-604. ( 1) 1)
|




