2. 吉林大学 动物科技学院, 长春 130062;
3. 农业部奶及奶制品质量监督检验测试中心, 北京 100193;
4. 中国农业科学院 北京畜牧兽医研究所, 动物营养学国家重点实验室, 北京 100193
2. College of Animal Science, Jilin University, Changchun 130062, China;
3. Ministry of Agriculture-Milk and Dairy Product Inspection Center, Beijing 100193, China;
4. Institute of Animal Sciences, Chinese Academy of Agricultural Sciences, State Key Laboratory of Animal Nutrition, Beijing 100193, China
反刍动物瘤胃细菌的数量和种类最多,根据细菌的定植部位可分为3类:瘤胃液相细菌、瘤胃固相细菌和瘤胃壁黏附细菌。20世纪,研究人员主要借助显微镜检和分离培养法对瘤胃壁黏附细菌进行研究,分离出了一些尿素分解菌、蛋白降解菌及纤维素分解菌等,同时还发现瘤胃壁黏附细菌在氧自由基的清除[1]、尿素降解[1]、组织循环[2]及上皮屏障[3]等方面起到重要作用。由于瘤胃中大于90%的细菌无法分离培养,因此,纯培养方法限制了对瘤胃壁细菌的研究,尤其是细菌群落的多样性和功能。近些年来,分子生物学方法,如变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)、限制片段长度多态性(restriction fragment length polymorphism,RFLP)、16S rDNA文库和高通量测序等被广泛地应用在瘤胃微生物研究中,丰富了对瘤胃微生物的种类和多样性等方面的认识,也推动了对瘤胃壁黏附细菌的深入研究。本文主要从瘤胃壁黏附细菌的多样性、影响因素和功能3个方面对其研究进展进行综述。
20世纪,研究人员主要通过纯培养法研究瘤胃壁黏附细菌,所分离出的菌株见表1。瘤胃壁黏附细菌多为杆菌、球菌等。纯培养发现瘤胃壁黏附菌群在瘤胃里的比例较低,占总菌数的1%~2%[4]。
| 表1 纯培养所得到的瘤胃壁黏附菌与瘤胃内容物菌群 Table 1 Rumen epithelial adherent bacteria flora and rumen content bacteria flora based on isolation |
1990年,Semjén等[5]从瘤胃壁分离出1株很常见的尿素降解菌,为革兰氏阳性球菌,占瘤胃壁黏附菌群总数的11.7%~14.1%,推测它可能是葡萄球菌(Staphylococcus)或微球菌(Micrococcus)。同年Semjén等[5]从公牛瘤胃壁中分离出2株牛链球菌(Streptococcus)和1株大肠杆菌(E. coli)。Styriak等[6]从犊牛瘤胃壁上分离出6株乳酸菌(Lactobacillus)和4株牛链球菌。
纯培养发现瘤胃黏附细菌与瘤胃内容物细菌存在差异性。Cheng等[1]发现瘤胃壁黏附菌群与瘤胃内容物菌群存在很大差异,如表1所示,其中丁酸弧菌属(Butyrivibrio sp.)为瘤胃壁黏附细菌的优势菌之一,占到瘤胃壁黏附细菌总数的31.1%;拟杆菌属(Bacteroides sp.)、反刍月形单胞菌属(Selenomonas ruminantium)、溶糊精琥珀酸弧菌属(Succinivibrio dextrinosolvens)和牛链球菌属(Streptococcus)分别占瘤胃壁黏附细菌总数的22.4%、9.9%、8.7%和8.1%。而Semjén等[5]和Styriak等[6]通过纯培养分离法发现瘤胃壁黏附菌群与瘤胃内容物菌群至少有90%相似,但是还存在一定差异。
瘤胃壁黏附细菌难以分离培养,因此人们试图寻找新的方法来研究那些未培养的瘤胃壁黏附细菌。16S rDNA文库和聚合酶链式反应-变性梯度凝胶电泳(PCR-DGGE)指纹图谱法与高通量测序等为鉴定这些不能分离培养的细菌提供了技术支持。
Cho等[16]通过构建16S rDNA文库探究了瘤胃中不同生态位(瘤胃固相、瘤胃液相及瘤胃壁)中各细菌的分布状况,见表2。Sadet-Bourgeteau等[11]利用PCR-DGGE指纹图谱发现公羊瘤胃壁黏附菌可归为5个门,即厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、变形菌门(Proteobacteria)、密螺旋体门(Spirochaetes)和候选门(TM7),其中厚壁菌门和拟杆菌门是其主要细菌群(占瘤胃壁黏附细菌总数的86%),而变形菌门占瘤胃壁黏附细菌总数的13%,密螺旋体门和候选门TM7的含量均少于1%。通过该方法还发现了瘤胃壁黏附细菌中存在一些特殊的细菌——乳酸菌、嗜麦芽寡养单胞(Stenotrophomonas maltophilia)、微球菌和梭酸菌,但是没有检测到尿素分解菌[14]。在瘤胃壁黏附细菌门级分类水平上,厚壁菌门、拟杆菌门、变形菌门及未知菌是肉牛瘤胃壁黏附菌群的主要细菌,见表3。Petri等[15]通过DGGE、定量PCR和16S rRNA测序法研究8头犊牛的核心瘤胃壁黏附菌组成,如图1。其中,最新鉴定的未培养瘤胃壁黏附细菌奇异菌属(Atopobium)和欧氏菌属(Olsenella)很可能早期研究中被错误鉴定,形似微球菌(Micrococcus)、棒状杆菌(Corynebacterium)和丙酸菌(Proponibacterium)。
| 表2 16S rDNA指纹图谱法分析3个不同瘤胃生态位中各细菌分布状况 Table 2 16S rDNA fingerprint method of bacterial diversity in 3 fractions of cow rumen[16] % |
此外,瘤胃壁与瘤胃内容物细菌多样性指数略有差异,公羊瘤胃壁黏附菌群的多样性(Shannon指数=2.818)[10]略高于其瘤胃液相菌群的多样性(Shannon指数=2.546)[18]。
| 表3 瘤胃壁黏附菌与瘤胃内容物菌群比较(门级水平) Table 3 Comparison between rumen epithelial adherent bacteria and bacteria of rumen concent bacteria flora (phylum level)[18] % |
饲粮是影响瘤胃内容物菌群结构的主要因素之一[19]。然而饲粮是否也是影响瘤胃壁黏附菌群的主要因素,近些年来,人们对不同反刍动物(牛和羊)的研究结果略有差异。饲粮精粗比能影响瘤胃微生物的多样性,同样也能影响瘤胃壁黏附细菌的多样性。Sadet等[10]发现高精料和高粗料组动物个体间的瘤胃壁黏附菌多样性存在一定差异,但是不显著。随后Sadet-Bourgeteau等[11]发现高精料和高粗料组公羊瘤胃壁黏附菌的多样性差异显著。前后2次研究的结果截然不同,一方面是由于2次研究中饲粮组成和饲喂方式存在差异,另一方面可能是宿主对瘤胃壁黏附细菌的多样性影响较大,掩盖了饲粮效应。
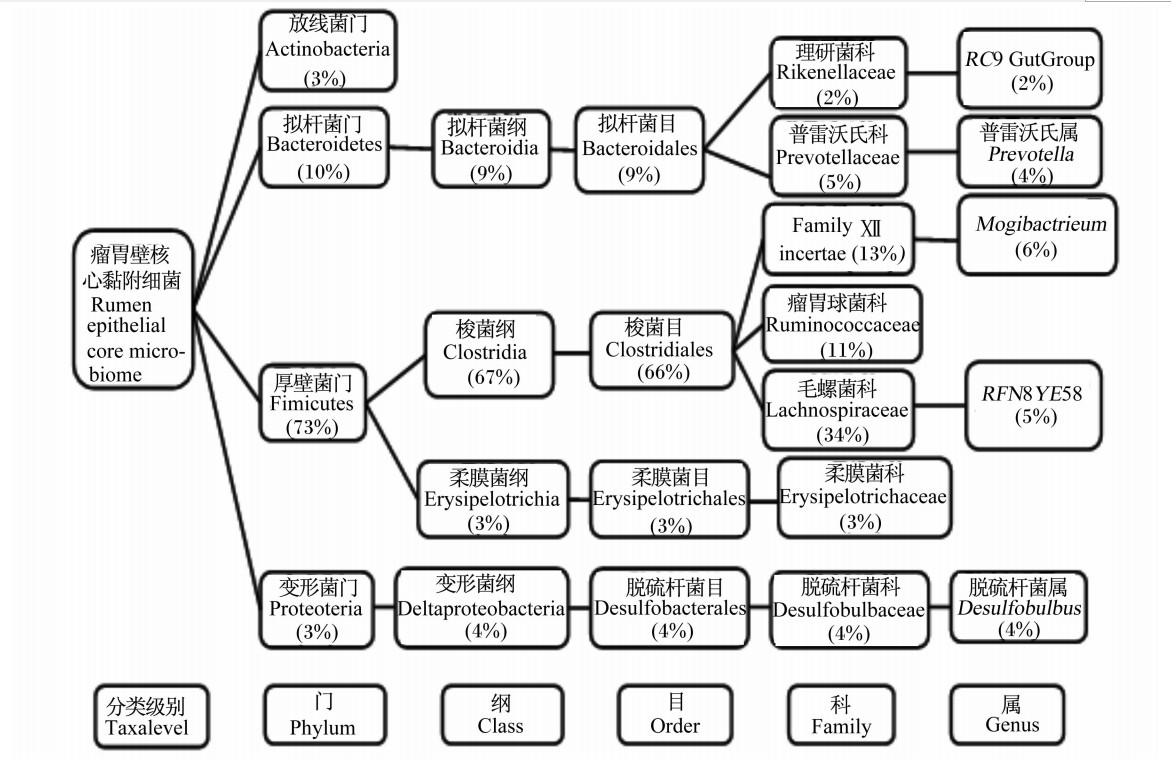 | 图1 瘤胃壁核心黏附细菌[15]
Fig.1 Rumen epithelial core adherent bacteira |
然而Chen等[17]发现:在不同精粗比饲粮转换过程中,肉牛瘤胃壁黏附菌群的多样性和浓度发生显著变化,如图2所示。随着饲粮干草含量的减少,一部分变形菌门、厚壁菌门、拟杆菌门、放线菌门及未分类菌的数量都有所减少,然而厚壁菌门细菌数量减少最多;同时还有少量的变形菌门、厚壁菌门和拟杆菌门的细菌数量有所增加。而且只在饲喂25%和7%的干草饲粮中检测到密螺旋体属、反刍杆菌属和毛螺旋菌科的细菌。这些细菌大多能分泌各种蛋白酶。二者的研究结果不相一致,其原因:一方面,两次研究中饲粮组成存在差异;另一方面,其饲粮中精粗比也不同。Sadet等[10]以小麦麸作为精料主要成分,而Chen等[17]以小麦作为精料主要成分。此外,羊和牛各自不同的物种也可能产生一定的影响。Petri等[15]发现在饲粮转换过程中,牛链球菌(Streptococcus spp.)的相对丰度未发生变化。不同饲粮组间,瘤胃球菌(Ruminococcus)和普雷沃氏菌(Prevotella spp.)没有发生变化,候选门TM7、梭菌门和无壁菌门在混合饲粮组有所增加,互养菌门增加较多,放线菌门在酸中毒时含量最多,如表4。
当饲喂高精料饲粮发生酸中毒时,Petri等[15]发现瘤胃壁黏附细菌中的奇异菌属(Atopobium)cc142、乳酸菌(Lactobacillus)、Olsenella RC39、Sharpea、Solobacterium、琥珀酸菌(Succiniclasticum)和互生球菌属(Syntrophococcus)的丰度明显增多。
宿主是影响反刍动物瘤胃壁黏附细菌的重要因素之一。Sadet等[11]研究公羊和肉犊牛瘤胃壁黏附细菌的多样性,发现其瘤胃壁黏附细菌都含有厚壁菌门、拟杆菌门、变形菌门及螺旋体门的细菌,其所属的门类几乎是一样,可能某一特定菌属或菌种存在差异。Sadet等[10]研究发现尽管给同组羊饲喂完全相同的饲粮,动物个体间的瘤胃壁黏附菌多样性仍然存在一定差异。Chen等[17]研究发现:试验组中18头犊牛中只有4头检测到无壁菌门和黏胶球形菌门的细菌;在饲喂97%干草的犊牛个体170和172中检测到厚壁杆菌门的细菌所占百分比最大,然而在饲喂25%干草的犊牛个体360中也检测到其数量最多,这些差异可能也与个体对pH变化的反应机制不同相关。Li等[18]阉牛研究中只在102个体的上皮组织样中检测到互养菌(Synergistete)。Petri等[15]发现:饲喂同样的高精料饲粮时,犊牛个体7和41中未检测到琥珀酸丝状杆菌(Fibrobacter succinogenes),犊牛个体156中琥珀酸丝状杆菌的丰度最高,Atopobacter F24-B10和U29-B03只在犊牛个体1和8中发现。还需进一步研究某特定宿主型内特殊细菌群的存在与否,这可能有助于解释所观察到个体间差异。
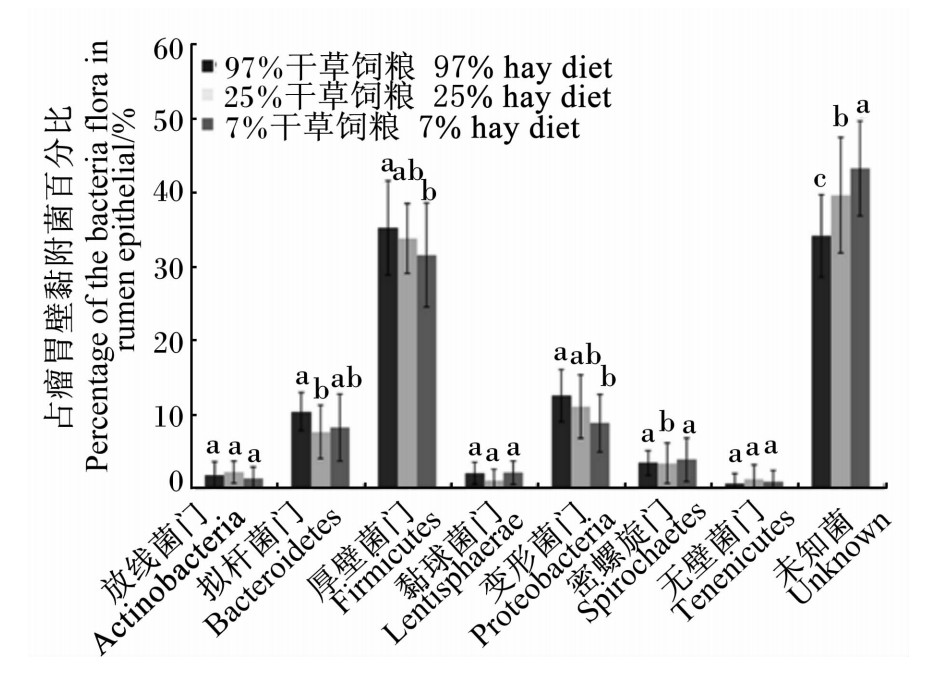 | 图2 不同饲粮组肉牛瘤胃壁黏附菌的细菌门分类比较 Fig.2 Comparison of distributions of rumen epithelial bacteria of cattle at the phylum level among different diet groups[17] |
由于瘤胃壁黏附细菌的含量较少且存在于瘤胃内容物与宿主上皮组织的交界面,因此在试验研究中采样部位可能从一定程度上影响其多样性,但是对这方面的研究很少。为了尽量减少采样引起的误差,Mead等[8]研究中从瘤胃壁上5个不同瘤胃位置(瘤胃背囊近尾部与右纵沟的连接处,瘤胃背囊近尾部,瘤胃前庭,瘤胃后庭及右纵沟处)采样来研究瘤胃细菌的多样性,发现不同部位瘤胃壁黏附细菌的数量和种类都有差异,如只有瘤胃背囊近尾部没有发现栖瘤胃拟杆菌(Bacteroides ruminicola),而且瘤胃背囊的栖瘤胃拟杆菌数量最多。Sadet等[10]采用这种取样法,但发现采样部位并不影响瘤胃壁黏附细菌的多样性。瘤胃壁黏附细菌在瘤胃壁上可能呈均匀分布;但是也可能某些特殊部位存在差异,其原因尚不清楚,需进一步研究。通过PCR-DGGE图谱分别分析犊牛和肉牛5个上皮采样部位(瘤网胃连接处、瘤胃背囊、瘤胃后庭、瘤胃腹囊、瘤胃纵沟)的瘤胃壁细菌落结构,都未发现有显著差异[14]。就目前研究来看,DGGE图谱对于鉴别瘤胃细菌的灵敏度还存在一定差异,有待于进一步提高其灵敏度。
瘤胃壁黏附细菌存在于特定的生态位(瘤胃内容物和宿主上皮组织的交界处),具有某些特殊的功能。Wallace等[13]证实,瘤胃壁黏附细菌中的微球菌和葡萄球菌能产生高活性的脲酶;此外,这些瘤胃壁黏附菌所产脲酶活性远大于瘤胃液中的脲酶(瘤胃液脲酶活性只达到16%~50%的瘤胃壁黏附菌群脲酶活性)。Mogibacterium对瘤胃氨代谢(主要是氨吸收)很重要[18]。Wells等[20]最新研究发现:羊瘤胃上皮组织内的Toll样受体(TLRs)能识别瘤胃壁黏附细菌所产生的次级代谢产物(主要是丁酸)。而Ster等[21]发现,丁酸能引发先天性免疫反应,这就能够促进上皮细胞的增值及上皮屏障功能。Chen等[22]研究发现,AR组(不易感酸中毒组)瘤胃壁黏附菌能激活先天性免疫系统,增强上皮屏障功能,以保证瘤胃上皮及瘤胃壁黏附菌群本身免受损害。新发现的一种瘤胃壁黏附细菌Azonexus,属于红环菌科的变形菌门,主要由反硝化细菌组成;放线菌(Actinobacteria)在酸中毒时含量最高,推测放线菌能产生碱性代谢产物,能中和瘤胃内过量的挥发性脂肪酸[15]。
瘤胃壁黏附菌群含量少,但结构复杂、多样,具有重要的屏障功能,与宿主上皮组织循环和氮素代谢等密切相关。瘤胃壁黏附菌群多样性的相关研究已取得了很大进展,但瘤胃黏附菌群中仍存在大量未培养菌,其精确分类和对宿主代谢的功能等问题尚未解决。今后,综合利用菌体单细胞分离和元基因组学等方法,研究瘤胃壁特殊黏附菌群结构,利用元转录组和代谢组学等方法研究瘤胃壁黏附菌群在瘤胃和瘤胃壁营养消化、吸收和代谢中的重要作用。
| 表4 实时定量PCR测定不同饲粮处理对瘤胃壁主要黏附菌种相对丰度的影响
Table 4 Effects of dietary treatment on the relative abundances of dominant rumen epithelial bacterial species determined by quantitative real-time PCR[15] %
|
| [1] | CHENG K J, MCCOWAN R P, COSTERTON J W.Adherent epithelial bacteria in ruminants and their roles in digestive tract function[J]. The American Journal of Clinical Nutrition, 1979, 32(1):139-148. ( 2) 2)
|
| [2] | MCCOWAN R P, CHENG K J, BAILEY C B, et al.Adhesion of bacteria to epithelial cell surfaces within the reticulo-rumen of cattle[J]. Applied and Environmental Microbiology, 1978, 35(1):149-155. ( 1) 1)
|
| [3] | RIEU F, FONTY G, GAILLARD B, et al.Electron microscopy study of the bacteria adherent to the rumen wall in young conventional lambs[J]. Canadian Journal of Microbiology, 1990, 36(2):140-144. ( 2) 2)
|
| [4] | RUSSELL J B, GARNER M R, FLINT J P, et al.Allisonella histaminiformans gen.nov., sp.nov:a novel bacterium that produces histamine, utilizes histidine as its sole energy source, and could play a role in bovine and equine laminitis[J]. Systematic and Applied Microbiology, 2002, 25(4):498-506. ( 1) 1)
|
| [5] | SEMJN G, GLFI P.Factors influencing the adherence of strains of Streptococcus bovis and Escherichia coli isolated from ruminal epithelium[J]. Veterinary Research Communications, 1990, 14(3):181-191. ( 3) 3)
|
| [6] | STYRIAK I, GALFI P, KMET V, et al.Adherence of ruminal Streptococcus bovis and Lactobacillus strains to primary and secondary cultures of rumen epithelium[J]. Acta Microbiologica et Immunologica Hungarica, 1992, 39(3/4):323-325. ( 2) 2)
|
| [7] | ŠTYRIAK I, GALFI P, KMET V, et al.The adherence of three Streptococcus bovis strains to cells of rumen epithelium primoculture under various conditions[J]. Arch für Tierernahr, 1994, 46(4):357-365. ( 0) 0)
|
| [8] | MEAD L J, JONES G A.Isolation and presumptive identification of adherent epithelial bacteria(‘epimural'bacteria)from the ovine rumen wall[J]. Applied and Environmental Microbiology, 1981, 41(4):1020-1028. ( 7) 7)
|
| [9] | KMET V, BOD'A K, KRAVJANSKŸ I.Adherence of bacteria to epithelial cells of the rumen wall[J]. Veterinary Medicine(Prague), 1984, 29(8):473-477. ( 1) 1)
|
| [10] | SADET S, MARTIN C, MEUNIER B, et al.PCR-DGGE analysis reveals a distinct diversity in the bacterial population attached to the rumen epithelium[J]. Animal, 2007, 1(7):939-944. ( 5) 5)
|
| [11] | SADET-BOURGETEAU S, MARTIN C, MORGAVI D P.Bacterial diversity dynamics in rumen epithelium of wethers fed forage and mixed concentrate forage diets[J]. Veterinary Microbiology, 2010, 46(1/2):98-104. ( 3) 3)
|
| [12] | GÁLFI P, NEOGRÁDY S, SEMJÉN G, et al.Attachment of different Escherichia coli strains to cultured rumen epithelial cells[J]. Veterinary Microbiology, 1998, 61(3):191-197. ( 1) 1)
|
| [13] | WALLACE R J, CHENG K J, DINSDALE D, et al.An independent microbial flora of the epithelium and its role in the ecomicrobiology of the rumen[J]. Nature, 1979, 279(5712):424-426. ( 2) 2)
|
| [14] | LUKÁS F, SIMÜNEK J, MRÁZEK J, et al.PCR-DGGE analysis of bacterial population attached to the bovine rumen wall[J]. Folia microbiologica, 2010, 55(4):345-348. ( 2) 2)
|
| [15] | PETRI R M, SCHWAIGER T, PENNER G B, et al.Changes in the rumen epimural bacterial diversity of beef cattle as affected by diet and induced ruminal acidosis[J]. Applied and Environmental Microbiology, 2013, 79(12):3744-3755. ( 7) 7)
|
| [16] | CHO S J, CHO K M, SHIN E C, et al.16S rDNA analysis of bacterial diversity in three fractions of cow rumen[J]. Microbiology and Biotechnology, 2006, 16(1):92-101. ( 2) 2)
|
| [17] | CHEN Y H, PENNER G B, LI M, et al.Changes in bacterial diversity associated with epithelial tissue in the beef cow rumen during the transition to a high-grain diet[J]. Applied and Environmental Microbiology, 2011, 77(16):5770-5781. ( 4) 4)
|
| [18] | LI M, ZHOU M, ADAMOWICZ E, et al.Characterization of bovine ruminal epithelial bacterial communities using 16S rRNA sequencing, PCR-DGGE, and qRT-PCR analysis[J]. Veterinary Microbiology, 2012, 155(1):72-80. ( 3) 3)
|
| [19] | 刘开朗, 卜登攀, 王加启, 等.六个不同品种牛的瘤胃微生物群落的比较分析[J]. 中国农业大学学报, 2009, 14(1):13-18. ( 1) 1)
|
| [20] | WELLS J M, LOONEN L M, KARCZEWSKI J M, et al.The role of innate signaling in the homeostasis of tolerance and immunity in the intestine[J]. Medical Microbiology, 2010, 300(1):41-48. ( 1) 1)
|
| [21] | STER C, LOISELLE M C, LACASSE P, et al.Effect of postcalving serum nonesterified fatty acids concentration on the functionality of bovine immune cells[J]. Journal of Dairy Science, 2012, 95(2):708-717. ( 1) 1)
|
| [22] | CHEN Y H, OBA M, GUAN L L.Variation of bacterial communities and expression of Toll-like receptor genes in the rumen of steers differing in susceptibility to subacute ruminal acidosis[J]. Veterinary Microbiology, 2012, 159(3/4):451-459. ( 1) 1)
|




