家禽肠道是一个复杂而多样的生态环境,其中有微生物超过400种,这些微生物对宿主的正常发育和营养物质的消化吸收起着重要的作用。长期以来,由于细菌形态过于简单,缺乏明显的外部特征,人们对环境中细菌菌群结构的了解不多。随着基于16S rDNA分子技术发展,更全面更深入地了解菌群结构成为可能,其中变性梯度凝胶电泳(DGGE)和温度梯度凝胶电泳(TGGE)技术被用于检测人类[1]、猪[2]、鸡[3]等的胃肠道菌群结构和多样性[4]。肠道菌群失调会减弱机体对营养物质的吸收率,降低机体免疫力,削弱肠道的屏障功能[5],影响畜禽的生长和健康,对肠道菌群失调是否会导致动物肠道功能紊乱等的相关研究的需求十分迫切。目前,对宿主与肠道菌群之间相互作用的了解非常有限,直接限制了对动物肠道菌群与肠道功能的研究。因此,本试验采用特定细菌物种通用引物对笼养的海兰褐蛋雏鸡所有肠段内容物菌群16S rDNA 高变区进行聚合酶链式反应(PCR)-DGGE指纹图谱比较分析,研究笼养雏鸡十二指肠、空肠、回肠和盲肠中内容物菌群结构和多样性的变化,旨在为研制新型微生态制剂提供理论依据。
试验动物于2013年5月选自大庆兴和牧业蛋鸡养殖场,全封闭式饲养,海兰褐蛋雏鸡采用3层笼养,舍温25 ℃,自由采食,自由饮水,采用人工光照,每日光照11~12 h,按海兰褐蛋雏鸡常规程序进行免疫。基础饲粮组成及营养水平见表1。
| 表1 基础饲粮组成及营养水平(风干基础) Table 1 Composition and nutrient levels of the basal diet (air-dry basis) % |
试验时,随机选取5只体重相近(551.74±12.45) g的8周龄雏鸡,剖杀,取全部小肠。分别取5只鸡的十二指肠、空肠、回肠和盲肠的内容物,并根据肠道部位分别混匀,按1 g/管分装至5 mL离心管中,-20 ℃保存。
采用十二烷基四乙酸二钠(SDS)高盐抽提法提取样品基因组DNA[6]。用细菌基因组DNA提取试剂盒(上海海博生物有限公司)进行过柱纯化和溶解,最终基因组DNA溶于30 μL无菌水中,-20 ℃保存。
以样品基因组DNA为模板,采用细菌通用引物GC-338F和518R扩增样品16S rDNA高变区序列[7]。
PCR扩增体系(50 μL)为:10×PCR buffer 5 μL;dNTP(2.5 mmol/L)3.2 μL;rTaq(5 U/μL)0.4 μL;GC-338F(20 mmol/L)1 μL;518R(20 mmol/L)1 μL;基因组DNA 50 ng;补双蒸水至50 μL。PCR扩增程序为:94 ℃预变性5 min;94 ℃变性1 min,55 ℃复性45 s,72 ℃延伸1 min,30个循环;最终72 ℃延伸10 min。PCR产物采用美国Omega公司DNA Gel Extraction Kit纯化回收。
PCR仪为德国Biometra公司生产的T-gradient,凝胶成像仪为美国Bio-Rad公司的Gel-Doc2000凝胶成像系统。
取10 μL PCR的产物进行DGGE分析。采用变性梯度为35%~55%、浓度为8%的聚丙烯酰胺凝胶,化学变性剂100%尿素7 mol/L和40%(v/v)的丙烯酰胺,在1×TAE缓冲液中150 V 60 ℃下电泳5 h。
| 表2 引物名称和序列
Table 2 Primer names and sequences
|
用灭菌的手术刀切下待回收DGGE条带,采用美国Omega公司Poly-Gel DNA Extraction Kit回收目的条带。以2 μL回收产物为模板,338F和518R为引物进行PCR扩增。将重新扩增的DNA片段切胶回收、纯化后,连接到PMD18-T载体上,并转化至DH5α感受态细胞中,筛选阳性克隆,菌液由北京华大基因研究中心对插入的细菌16S rDNA片段进行序列测定。
用Quantity One软件对雏鸡各肠段分离的细菌的PCR-DGGE指纹图谱进行条带计数和模拟,在GenBank中使用Blast程序进行同源性比较,获得最相似菌株。
从8周龄笼养雏鸡的十二指肠、空肠、回肠和盲肠内容物中提取的基因组DNA,经电泳分析,可见各肠段内容物基因组DNA分子质量相近,均在在2 000 bp以上(图1)。
 | 图1 基因组DNA提取结果
Fig.1 The extraction result of genome DNA |
以雏鸡各肠段内容物提取的细菌基因组DNA为模版,以GC-338F和518R为引物扩增16S rDNA序列,得到约200 bp的DNA片段(图2),后续用于DGGE分析。
 | 图2 细菌16S rDNA的PCR扩增结果 Fig.2 The result of PCR amplification of 16S rDNA of bacteria |
由图3可见,细菌通用引物扩增中,4个肠段内容物样本中均得到16S rDNA高变区片段,利用上述已得到片段16S rDNA 高变区进行PCR-DGGE指纹图谱和聚类分析。结果表明:笼养雏鸡小肠各段细菌种类丰富,不同肠段虽然有些共性条带,且菌株数量接近,但菌种类别差异很大。主要DGGE条带:十二指肠为8个,空肠为12个,回肠为8个,盲肠为9个。8周龄雏鸡,以空肠中细菌数量最多,其他3个肠段略少,且数量相近。
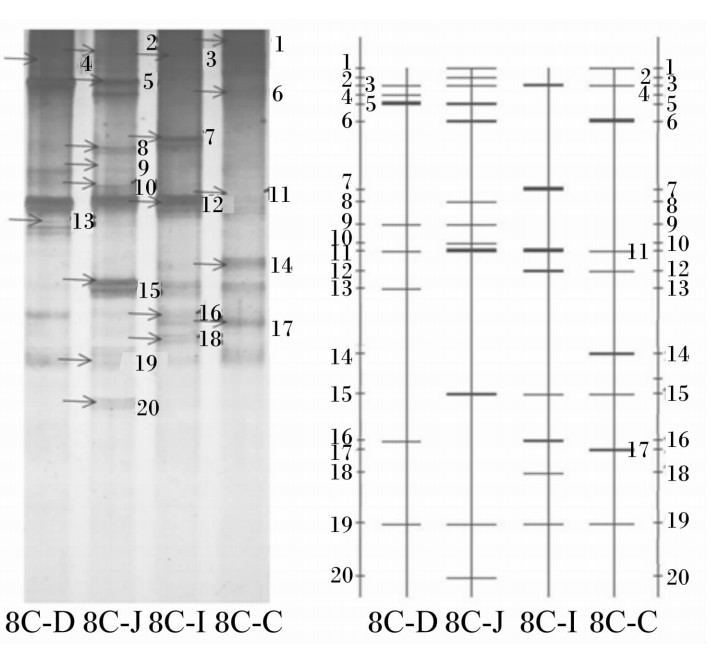 | 图3 PCR产物的DGGE结果
Fig.3 The result of DGGE of PCR products |
由表3可见,笼养雏鸡各肠段中细菌菌群的组成有很大差异,各肠段细菌菌群遗传相似指数为8.9%~43.1%;其中十二指肠与盲肠中细菌菌群组成差异性最大,相似性仅为8.9%;十二指肠与回肠中细菌菌群组成差异次之,相似性为20.1%;十二指肠与空肠细菌菌群组成相似性为34.4%;空肠与盲肠细菌菌群组成差异最小,相似性为43.1%。细菌菌群相似性系数是测量菌群间或样本间相似程度的指数,相似指数的高低说明笼养雏鸡各肠段中细菌菌群相似程度的高低,也可间接地说明共性菌群以外的菌群情况。由上述结果中可知,笼养雏鸡各肠段细菌菌群组成差别较大。
| 表3 不同肠段细菌菌群遗传相似性 Table 3 Genetic resemblance of bacteria strains in different intestinal segments % |
DGGE凝胶条带回收后,以338F/518R为引物进行PCR扩增,获得约200 bp的DNA片段(图4)。
 | 图4 主要DGGE条带PCR扩增结果
Fig.4 The result of PCR amplification of main DGGE bands |
PCR产物纯化后连接到pMD18-T载体上,转化至DH5α感受态细胞中,筛选阳性克隆测序。各主要DGGE条带分别割胶回收测序,结果见表4,测序结果与GenBank中的序列进行比对,得到与条带序列最接近的菌株。在20个条带测序结果中,与GenBank数据库中细菌的同源性绝大多数均大于98%,有的同源性甚至达到100%。
| 表4 主要DGGE条带序列Blast比对分析结果 Table 4 The result of Blast comparison analysis of sequences of main DGGE bands |
经鉴定,笼养雏鸡的小肠中乳酸菌(Lactobacillus)属的条带有7个。包括2株鸟乳酸菌(Lactobacillus aviaries)、2株棉籽糖乳球菌(Lactococcus raffinolactis)、1株敏捷乳杆菌(Lactobacillus agilis)、1株陪伴粪球菌(Coprococcus comes)和1株同代乳杆菌(Lactobacillus equigenerosi)。其中,空肠分离出5株,为1株Lactobacillus aviaries、1株Lactococcus raffinolactis、1株Lactobacillus agilis、1株Coprococcus comes和1株Lactobacillus equigenerosi;十二指肠分离出3株,为2株Lactobacillus aviaries、1株Lactobacillus equigenerosi;盲肠分离出1株,为Lactococcus raffinolactis。且各肠段有共同菌3株,为1株鲁梅利杆菌(Rummeliibacillus stabekisii)、1株Lactobacillus equigenerosi和1株阴道加德菌(Gardnerella vaginalis)。
具体分析为:十二指肠和空肠中均检测到条带5;同时在十二指肠和回肠检测到条带4;条带4和Lactobacillus aviarius相似性仅有31%,而条带5与Lactobacillus aviarius的相似性有98%,但它们与Lactobacillus aviarius的同源性均为98%,说明条带4为Lactobacillus aviarius的新种。在十二指肠、空肠和盲肠均检测到条带6,在盲肠中检测到条带17,条带6与Lactococcus raffinolactis相似性只有28%,条带17与Lactococcus raffinolactis相似性为98%,2个条带的同源性均为98%,说明条带6是Lactococcus raffinolacti的新种。在所有肠道均检测到了条带11,经分析,条带11为Lactobacillus equigenerosi,但它们的相似性只有34%,说明条带11为不能纯化培养的混合菌。在空肠检测到条带8,与Coprococcus comes相似性只有70%,且同源性为92%,说明条带8为Coprococcus comes的一个新种。
在十二指肠和空肠检测条带9,与Butyricimonas virosa相似性只有68%,且与Butyricimonas virosa同源性为92%,与不可培养菌同源性为100%,说明条带9为不能纯化培养的混合菌,且该条带代表的细菌为不可培养菌和Butyricimonas virosa的新种。
不可培养菌共发现3个条带,即条带12、14和15,同源性均在99%~100%,且盲肠中均可检出,条带15在空肠和回肠中也有分布,条带12在回肠中有分布,说明鸡群肠道中各肠段有同源不可培养菌。
在笼养雏鸡各肠段都分离得到的菌有3株,即条带3、11和19,分别为Rummeliibacillus stabekisii、Lactobacillus equigenerosi和Gardnerella vaginalis;在空肠、回肠和盲肠均有分布的菌只有1株,即条带15,不可培养菌;在空肠和盲肠都有的菌有2株,即条带1和3,为普氏梭杆菌(Flavonifractor plautii)和Rummeliibacillus stabekisii;在十二指肠和空肠都有的菌有2株,即第条带5和9,为Lactobacillus aviaries和Butyricimonas virosa;在回肠和盲肠都有的菌只有1株,即条带12,为不可培养菌;在十二指肠和回肠共有的菌只有1株,即条带16,为艰难梭菌(Clostridium difficile)。十二指肠独有的菌有2株,即条带4和13,为Lactobacillus aviaries和缓腐梭菌(Clostridium lentocellum);在空肠独有的菌有4株,即条带2、8、10和20,为蓝斑拟杆菌(Bacteroides plebeius)、Coprococcus comes、克拉副普氏菌(Paraprevotella clara)和螺旋链霉菌(Streptomyces spiralis);回肠独有的菌有2株,即条带7和18,为Lactobacillus agilis和煎盘梭菌(Clostridium sartagoforme);盲肠独有的菌有2株,即条带14和17,为不可培养菌和Lactococcus raffinolactis。
目前,微生态制剂由于其可替代部分抗生素,且无毒、无害、无残留、无耐药等特点,被用在禽类和单胃动物的饲养中。目前大多数微生态制剂菌为商业菌,饲喂动物时,由于其对胃肠道的适应性和消化液的耐受性等较差,实际应用的效果都不是太好。因此,饲喂动物的微生态制剂菌株应尽量来自动物肠道,这方面的研究也逐渐开展起来,而且由于人们普遍认识到乳酸菌对动物更有益处,近年来,乳酸菌分离研究的较多[8, 9, 10]。
由于动物种类[11]、饲养环境、温度[12]等均影响肠道菌群结构,且基于传统培养方法很难全面地反映或比较肠道菌群的结构特征,本试验采用先进且有效的DGGE技术对8周龄笼养海兰褐蛋雏鸡十二指肠、空肠、回肠和盲肠内容物中细菌组成进行研究,分析各肠段中菌群结构变化。从雏鸡整个小肠中发现共20个菌株,有乳酸菌属7株,其中有5株分布于空肠,3株分布于十二指肠,2株分布于盲肠,仅1株分布在回肠,空肠和回肠为连接在一起的肠道,但其中菌种的分布却差别很大,说明肠道中物理环境对菌的生存有很大影响。为了开发动物用益生菌制剂菌,在各个肠道均存活的菌株将作为后备菌进行研究。本试验从笼养雏鸡各肠段分离出共同菌株共3株,其中乳酸菌仅为1株,即Lactobacillus equigenerosi。目前,关于Lactobacillus equigenerosi的研究报道仅有3篇[13, 14, 15],且均是从马的肠道中分离出来或是做成微生态制剂饲喂马的,对该菌进行生物学特性研究发现,Lactobacillus equigenerosi具有高耐酸耐胆盐能力,可以在pH为3.0的环境存活,且对肠上皮细胞黏附力高达60%以上,没有致病性,且降低了马血液胆固醇和尿素含量。他们的研究结果指出,Lactobacillus equigenerosi可作为益生菌进行开发。本试验首次从笼养海兰褐蛋雏鸡肠道中分离出Lactobacillus equigenerosi,根据以上研究结果,推测Lactobacillus equigenerosi可以作为蛋鸡用微生态制剂的后备菌在未来试验中进行深入研究。
同时雏鸡各肠段还有2株非乳酸菌属共同菌,为1株Rummeliibacillus stabekisii和1株Gardnerella vaginalis。其中的Gardnerella vaginalis,有多个研究表明,该菌为致病菌[16, 17],会产生生殖道炎症,因此不可作为益生菌制剂后备菌。目前,直接研究Rummeliibacillus stabekisii的报道仅有2篇[18, 19],均是从土壤中分离并鉴定的,但是Parag等[18]分离出的Rummeliibacillus stabekisii,经测定严格需氧,耐高盐,28~32 ℃生长良好;而Her等[19]分离出的Rummeliibacillus stabekisii不耐盐(氯化钠需<1.5%),也不耐酸(pH适宜范围5~10),不具有微生态制剂菌的优良特性。因此本试验分离出的Rummeliibacillus stabekisii是否可作为微生态制剂制备菌,有待于进一步的研究确定。
此外,本试验在雏鸡肠道中还发现了其他类乳酸菌,如十二指肠和空肠中分离的Lactobacillus aviaries,在回肠中分离的Lactobacillus agilis,在空肠和盲肠中分离的Lactococcus raffinolactis,在空肠分离的Coprococcus comes。其中Lactobacillus aviaries仅在Waters等[20]的一篇综述中提到过可能作为微生态制剂菌,没有发现其他的相关研究报道。关于Lactobacillus agilis的研究有3个,最早的研究是1995年,Llanos等[21]研究发现一株Lactobacillus agilis R16能够在含有芥末籽提取物的环境中生长,并可以降解硫甙,说明该菌具有一定的生命抗性和生物活性;Baele等[22]证实Lactobacillus agilis为鸽子肠道中乳酸菌属的重要组成部分;最近的研究,Stephenson等[23]研究发现,Lactobacillus agilis为肉鸡肠道常驻乳酸菌属菌株,具有高黏附力和定植力,并具有高效表达抗菌蛋白的功能;本试验虽然只在回肠发现了Lactobacillus agilis,但通过以上研究的对比,说明该菌在禽类肠道具有很好的定植的能力和适应性,因此Lactobacillus agilis可作为微生态制剂后备菌进行进一步研究。
关于Lactococcus raffinolactis的研究都是近年的,且均为发酵牛奶中关于该菌的研究[24, 25],至今没有从禽肠道中分离鉴定该菌的研究。Meslier等[24]在文章中提到,Lactococcus raffinolactis为环境中的常见乳酸菌,而本试验在笼养雏鸡肠道中分离出该菌。根据前人研究它的促发酵作用,该菌具有微生态制剂后备菌的潜质,具体应用效果有待于进一步研究。
关于Coprococcus comes的研究,多数为20世纪90年代前的[26],最近的仅有1篇,为Graessler等[27]的研究,均说明了Coprococcus comes在肠道中数量的增多与克罗恩氏病(节段性肠炎)有关,因此该菌不可作为微生态制剂后备菌。
综上所述,本试验从笼养雏鸡肠道中分离鉴定出20株菌,其中除Lactobacillus equigenerosi已在马属动物上作为微生态制剂菌应用过,乳酸菌Lactobacillus aviaries、Lactobacillus agilis、Lactococcus raffinolactis及芽孢杆菌Rummeliibacillus stabekisii是否可作为微生态制剂后备菌,均有待于进一步的研究确定,可作为后备菌保存。本试验结果对了解笼养蛋雏鸡正常肠道细菌菌群组成提供了重要的数据支持,为微生态制剂开发奠定基础。
同龄笼养海兰褐蛋雏鸡肠道细菌菌群结构与肠道的不同部位有关,各肠段中细菌菌群的组成差异很大。
| [1] | NADAL I, DONANT E, RIBES-KONINCKX C, et al.Imbalance in the composition of the duodenal microbiota of children with coeliac disease[J]. Journal of Medical Microbiology, 2007, 56:1669-1674. ( 1) 1)
|
| [2] | KONSTANTINOV S R, ZHU W Y, WILLIAMS B A, et al.Effect of fermentable carbohydrates on piglet faecal bacterial communities as revealed by denaturing gradient gel electrophoresis analysis of 16S ribosomal DNA[J]. FEMS Microbiology Ecology, 2002, 43(2):225-235. ( 1) 1)
|
| [3] | VAN DER WIELEN P W, KEUZENKAMP D A, LIPMAN L J, et al.Spatial and temporal variation of the intestinal bacterial community in commercially raised broiler chickens during growth[J]. Microbial Ecology, 2002, 44(3):286-293. ( 1) 1)
|
| [4] | FlÓREZ A B, MAYO B.PCR-DGGE as a tool for characterizing dominant microbial populations in the Spanish blue-veined Cabrales cheese[J]. International Dairy Journal, 2006, 16(10):1205-1206. ( 1) 1)
|
| [5] | ZOETENDAL E G, COLLIER C T, KOIKE S, et al.Molecular ecological analysis of the gastrointestinal microbiota:a review[J]. The Journal of Nutrition, 2004, 134(2):464-472. ( 1) 1)
|
| [6] | TAPIA-PANIAGUA S T, CHABRILLÓN M, DÍAZ-ROSALES P, et al.Intestinal microbiota diversity of the flat fish Solea senegalensis (Kaup, 1858) following probiotic administration[J]. Microbiological Ecology, 2010, 60(2):310-319. ( 1) 1)
|
| [7] | FLIEGEROVÁ K, MRÁZEK J, KAJAN M, et al.The effect of maize silage as co-substrate for swine manure on the bacterial community structure in biogas plants[J]. Folia Microbiology, 2012, 57(4):281-284. ( 1) 1)
|
| [8] | OUWEHAND A C, SALMINEN S, ISOLAURI E.Probiotics:an overview of beneficial effects[J]. Antonie van Leeuwenhoek, 2002, 82(1/2/3/4):279-289. ( 1) 1)
|
| [9] | 步卫东, 潘裕华, 刘彦.鸽小肠粘膜乳酸菌的分离及鉴定方法研究[J]. 中国家禽, 2010, 32(4):30-33. ( 1) 1)
|
| [10] | 刘雪美, 栾淑莉, 温建新.健康家兔肠道中乳酸菌的分离鉴定[J]. 青岛农业大学学报, 2011, 28(1):36-39. ( 1) 1)
|
| [11] | LI X M, YU Y H, FENG W S, et al.Host species as a strong determinant of the intestinal microbiota of fish larvae[J]. The Journal of Microbiology, 2012, 50(1):29-37. ( 1) 1)
|
| [12] | LÜY, LI N, GONG D C, et al.The effect of temperature on the structure and function of a cellulose-degrading microbial community[J]. Applied Biochemistry and Biotechnology, 2012, 168(2):219-233. ( 1) 1)
|
| [13] | AKIHITO E, STEFAN R, EⅡCHI S, et al.Lactobacillus equigenerosi sp.nov., a coccoid species isolated from faeces of thoroughbred racehorses[J]. International Journal of Systematic and Evolutionary Microbiology, 2008, 58:914-918. ( 1) 1)
|
| [14] | MORITA H, NAKANO A, SHIMAZU M, et al.Lactobacillus hayakitensis, L.equigenerosi and L.equi, predominant lactobacilli in the intestinal flora of healthy thoroughbreds[J]. Animal Science Journal, 2009, 80(3):339-346. ( 1) 1)
|
| [15] | BOTHA M, BOTES M, LOOS B, et al.Lactobacillus equigenerosi strain Le1 invades equine epithelial cells[J]. Applied and Environmental Microbiology, 2012, 78(12):4248-4255. ( 1) 1)
|
| [16] | MACHADO A, JEFFERSON K, CERCA N.Interactions between Lactobacillus crispatus and Bacterial Vaginosis (BV)-associated bacterial species in initial attachment and biofilm formation[J]. International Journal of Molecular Sciences, 2013, 14(6):12004-12012. ( 1) 1)
|
| [17] | TOMUSIAK A, HECZKO P B, JANECZKO J, et al.Bacterial infections of the lower genital tract in fertile and infertile women from the southeastern Poland[J]. Ginekologia Polska, 2013, 84(5):352-358. ( 1) 1)
|
| [18] | VAISHAMPAYAN P, MIYASHITA M, OHNISHI A, et al.Description of RummelⅡbacillus stabekisⅡ gen.nov., sp.nov.and reclassification of Bacillus pycnus Nakamura et al.2002 as RummelⅡbacillus pycnus comb.nov[J]. International Journal of Systematic and Evolutionary Microbiology, 2009, 59:1094-1099. ( 2) 2)
|
| [19] | HER J, KIM J.RummelⅡbacillus suwonensis sp.nov., isolated from soil collected in a mountain area of South Korea[J]. Journal of Microbiology, 2013, 51(2):268-272. ( 2) 2)
|
| [20] | WATERS S M, MURPHY R A, POWER R F.Characterisation of prototype Nurmi cultures using culture-based microbiological techniques and PCR-DGGE[J]. International Journal of Food Microbiology, 2006, 110(3):268-277. ( 1) 1)
|
| [21] | PALOP M L, SMITHS J P, BRINK B T.Degradation of sinigrin by Lactobacillus agilis strain R16[J]. International Journal of Food Microbiology, 1995, 26(2):219-229. ( 1) 1)
|
| [22] | BAELE M, DEVRIESE L A, HAESEBROUCK F.Lactobacillus agilis is an important component of the pigeon crop flora[J]. Journal of Applied Microbiology, 2001, 91(3):488-491. ( 1) 1)
|
| [23] | STEPHENSON D, MOORE R J, ALLISO G E.Transformation of, and heterologous protein expression in, Lactobacillus agilis and Lactobacillus vaginalis isolates from the chicken gastrointestinal tract[J]. Applied and Environmental Microbiology, 2011, 77(1):220-228. ( 1) 1)
|
| [24] | MESLIER V, LOUX V, RENAULT P.Genome sequence of Lactococcus raffinolactis strain 4877, isolated from natural dairy starter culture[J]. Journal of Bacteriology, 2012, 194(22):6364. ( 2) 2)
|
| [25] | KIMOTO-NIRA H, AOKI R, MIZUMACHI K, et al.Interaction between Lactococcus lactis and Lactococcus raffinolactis durin growth in milk:development of a new starter culture[J]. Journal of Dairy Science, 2012, 95(4):2176-2185. ( 1) 1)
|
| [26] | BULL K, MATTHEWS N, RHODES J.Antibody response to anaerobic cooccoid rods in Crohn's disease[J]. Journal of Clinical Pathology, 1986, 39(10):1130-1134. ( 1) 1)
|
| [27] | GRAESSLER J, QIN Y, ZHONG H, et al.Metagenomic sequencing of the human gut microbiome before and after bariatric surgery in obese patients with type 2 diabetes:correlation with inflammatory and metabolic parameters[J]. Pharmacogenomics Journal, 2013, 13(6):514-522. ( 1) 1)
|




