牛是反刍动物,具有不同于其他食草动物的特殊生理结构,可食用难以被其他动物体所消化的纤维素物质,并通过自身的生物转化,将一些人类无法直接利用的营养物质合成动物性蛋白质(肉、奶),为人类所利用[1]。牛瘤胃与肠道中的微生态系统可以帮助宿主消化动物体自身难以利用的粗纤维(纤维素、半纤维素、木质素),从大量的低营养的植物纤维中摄取所需的能量,并且还具有为宿主分解代谢过程中产生的有毒物质的能力[2]。不同部位的微生物群落组成与数量都有很大的差异,如瘤胃中的优势菌群为一些纤维素降解菌,而肠道中的优势菌群则逐渐转化为可利用果糖的微生物。造成这些差异的主要原因是微生物所定植的各个部位均承担着各自不同的生理功能。因此,对牛体内共生微生物的研究不仅在牛的营养方面有指导作用,而且在牛疾病防控以及环境污染、食品安全等方面也具有指导意义。
1 瘤胃及肠道中常见微生物Hungate[3]第1个发起了对瘤胃中微生物生态系统进行系统的研究,被誉为瘤胃微生物学之父,在他研究的基础之上,越来越多的研究者加入这个行列之中,新的研究手段与技术也不断的开发与改进。瘤胃作为一个庞大的菌种资源库,其主要的微生物有细菌、古菌、原虫、真菌等。细菌的种类主要有纤维素降解菌、淀粉降解菌、半纤维素降解菌、蛋白质降解菌、脂肪降解菌、酸利用菌和乳酸菌等。
近年来的研究表明,真菌不仅在降解饲粮中的植物纤维过程中起重要作用,还与瘤胃中的细菌有着互利共生的合作关系。目前从瘤胃中分离得到的真菌共计6个属16个种之多。根据游离孢子的形态和菌丝的形成方式,可将它们划分为2种类型,即单中心类型真菌和多中心类型真菌。利用瘤胃真菌降解纤维素也是目前研究的热点,一些高效降解纤维素的厌氧真菌也作为饲料添加剂加以利用。Tuyen等[4]研究发现,真菌对瘤胃发酵的影响十分显著,真菌更偏向于利用饲粮中的木质素而非纤维素类物质。Sekhavati等[5]通过定量PCR(Q-PCR)技术对不同饮食条件及不同时间点下的瘤胃真菌进行定量研究。Asa等[6]研究了由植物乳杆菌和明串珠菌产生的一种具有蛋白酶抗性的抑菌物质PRA,该物质能减少甲烷的产量,推测可能是一种抑制产甲烷菌的抗真菌物质。
瘤胃与肠道中古菌的数量较少,产甲烷菌则为其中数量最大和多样性最复杂的古菌。Das等[7]认为甲烷的产量可以作为瘤胃对饲粮的消化率与利用率的指示剂。目前在瘤胃中已经分离到甲烷短杆菌属、甲烷细菌属、甲烷微菌属、甲烷八叠球菌属等,其中对可活动甲烷微菌、反刍兽甲烷短杆菌、巴氏甲烷八叠球菌、甲酸甲烷杆菌等研究的较多。反刍家畜应该对温室效应负有一定的责任,人们认为甲烷促使全球气候变暖,使气候发生改变。甲烷是瘤胃微生物生态系统对植物性物质发酵后终产物的一种,主要由产甲烷菌产生[8]。瘤胃发酵的顺利进行需要低氢气(H2)压环境,产甲烷菌对于维持瘤胃中的正常H2分压有着重要的意义,已有一些产甲烷菌成功的从瘤胃中分离出来[9]。 2 瘤胃及肠道中微生物多样性 2.1 瘤胃微生物多样性
提高饲料的利用率,降低肉用动物的饲养成本,减小对环境的不良影响,这些在养殖业中都是亟待解决的问题。如何提高瘤胃微生物的发酵效率,增加瘤胃细菌的发酵底物种类,目前的主要研究手段还集中在对瘤胃中的微生物进行分离鉴定以及生理功能的评估。虽然直接,但却未必能反映微生物在瘤胃中的真实发酵情况。因此,结合高通量测序技术,免去了对不可培养微生物的资源流失,还可以研究不同时间点下微生物群落的动态变化,对此,近年来国内外已有许多报道。瘤胃中已测序的菌株序列见表1。
Li等[10]通过多种分子技术联用,对瘤胃上皮细胞附着的微生物与瘤胃内容物中的微生物做了详细的对比。内容物中的细菌量多于附着于上皮细胞的细菌量,上皮细胞微生物群落中硬壁菌门为优势菌群,内容物中以拟杆菌门为优势菌群,此现象可能与瘤胃对特殊食物的降解密切相关。Jami等[11]采集了16头泌乳期的奶牛瘤胃内容物样品,通过焦磷酸测序分析,发现不同个体中有51%的细菌分类相似,如普雷沃氏菌属在不同个体中的数量相当,相反,如颤螺旋菌属的数量平均值还不到0.1%。综合多个样本,普雷沃氏菌属、丁酸弧菌属和毛螺旋菌科的数量都比较大。Pandya等[12]运用非培养方法对3头成年条苏尔蒂水牛瘤胃液中的细菌进行测序分析,最后,通过基于16S rRNA序列的系统发育分析,区分了42个操作分类单位(OTU),19个OTU属于1个仍未鉴别的种族,11个OTU属于低G+C厚壁菌门,7个OTU属于纤维黏菌属-产黄菌属-拟杆菌属(Cytophaga-Flexibacter-Bacteroides),4个OTU属于螺旋体门,1个OUT属于放线菌门,每个OUT均保证有10个以上的单克隆,94.76%16S rRNA文库覆盖率表明这些序列可以较完整的表现瘤胃中的细菌多样性。Singh等[13]采用454测序技术测定了水牛瘤胃中的宏基因组,分析了水牛瘤胃中的微生物多样性,同时鉴定出具有代表性的功能基因,如抗生素抗性基因与细菌毒素基因。Petri等[14]采用变性梯度凝胶电泳(DGGE)、Q-PCR以及焦磷酸测序等多种技术联用,对8头肉牛的瘤胃上皮黏附的微生物群落组成进行研究,检测饮食从牧草到浓缩型饲粮的转变、酸中毒及恢复过程中的微生物群落的变化。Li等[15]对牛瘤胃中的细菌V3~V5区进行宏基因组测序,最终鉴定得到了以45个核心微生物属为主的共107个细菌属。Yang等[16]通过焦磷酸测序分析了瘤胃内容物中的微生物群落结构,结果表明,溶纤维丁酸弧菌、瘤胃假丁酸弧菌等为优势菌群。
瘤胃也可以作为一个富含功能基因的克隆文库被加以利用[18]。López-Cortés等[18]从牛瘤胃微生物中鉴定出了新的瘤胃水解酶,乙酰基木聚糖酯酶,并分析了该酶的分子生物学特性。Palackal等[19]从牛瘤胃微生物中分离了复合糖基水解酶,鉴定该复合酶的酶学特性,并肯定了该复合酶潜在的工业价值。Weimer等[20]成功地从牛瘤胃微生物中分离出1株梭状芽孢杆菌,证实该菌株有产己酸的功能,并具有应用到工业生产中的巨大潜能。Liu等[21]也采用宏基因组学研究手段从荷斯坦奶牛的瘤胃宏基因组文库中获得2个新的脂肪酶基因RlipE1和RlipE2。Wang等[22]通过高通量测序技术,对牛瘤胃中的宏基因组BAC克隆文库进行测序分析,成功的筛选到了多个木聚糖酶基因,并成功的在大肠杆菌中表达。
| 表1 瘤胃中已测序的菌株 Table 1 Sequenced strains in rumen |
2.2 肠道微生物多样性
肠道是动物体消化吸收营养物质的主要场所,起到关键作用的就是其中定殖的微生物,目前国外在牛肠道微生物多样性研究方面已做了很多工作。Durso等[23]对6头肉牛胃与肠道中细菌的16S rRNA基因全长序列进行了焦磷酸测序,硬壁菌门在整个微生物群落中占主导地位,拟杆菌门和变形菌门次之,在试验的6头肉牛的胃肠道中总共发现有7个门下的细菌。Uyeno等[24]对4头新生的荷斯坦母牛从出生到12周的肠道微生物群落变化进行了分析,采用Q-PCR技术,样品为犊牛的新鲜粪便,设计了12个特异性引物探针,基本覆盖了犊牛肠道中所有主要的细菌类别,结果显示,1周龄时普雷沃氏菌属拷贝数占总16S rRNA基因的40.0%,柔嫩梭菌属占21.7%,拟球梭菌-直肠真杆菌占16.7%,奇异菌属占10.9%;在整个12周的试验中普雷沃氏菌属和拟球梭菌-直肠真杆菌都保持着优势菌群的地位,其他的奇异菌属、柔嫩梭菌属以及一些益生菌属(如乳酸菌属、双歧杆菌属)的细菌数量随着周龄的增长呈下降的趋势;断奶后,本应出现在瘤胃中黄色瘤胃球菌和纤维杆菌也从粪便中检测出来。Malmuthuge等[25]研究了犊牛料对断奶期犊牛肠道微生物群落结构与组成以及黏膜免疫功能的影响,分别从瘤胃、空肠、回肠、盲肠、结肠中直接采取样本,不同位置采取的样本中细菌的密度有所不同,饲粮不同导致微生物的构成也不同,黏膜细胞表达的免疫相关的基因也发生差异表达。Jami等[26]研究1日龄的犊牛与2岁的奶牛瘤胃微生物群落的组成及改变,发现瘤胃微生物也从最初的需氧型微生物向厌氧型与兼性厌氧型微生物群落的转变。随着犊牛饲粮的复杂度加大,其瘤胃微生物群落结构也日趋丰富。瘤胃中的细菌数量庞大,种类繁多,相互协调,共同作用于饲粮底物。Durso等[27]提取了肉牛粪便样品中的总DNA,通过高通量测序,分析了肉牛粪便中的抗生素抗性基因与细菌毒素基因,16S rDNA全长测序结果发现,这些微生物大部分都在放线菌门与变形菌门这2个主要的细菌门下。细菌毒素基因和抗生素抗性基因大约占总序列的8.4%。比之前在马尾藻海、鸡盲肠、奶牛瘤胃等微生物环境中检测到的结果要高。该研究也为研究建立抗生素抗性基因在各个微环境中的转运模型提供了一个新的视野。
目前大多研究多集中在单一组织部位,然而胃肠道作为执行消化吸收的整体,其各个部位的微生物群落必然具有共性,但也肯定存在差异。De Oliveira等[28]对健康牛胃肠道的不同部位分别采样,如前胃、小肠以及大肠。对这些部位采集的样品进行高通量测序,发现在门水平上无明显差异,大多分属于硬壁菌门和拟杆菌门,但是在科水平上就有较明显的差异,瘤胃、粪便以及胃肠道的各个部位中的微生物群落多样性作对比后发现,微生物的群落构成虽然是在同一宿主体内,但是仍然存在较大的差异,这可能与各个部位承担的生理功能不同有关。许强等[29]利用限制性片段长度多态性技术(restricted fragment length polymorphisms,RFLP)分析2周龄荷斯坦犊牛直肠中主要微生物群落的组成,腹泻组克隆阳性率达98.75%,优势菌群以乳杆菌属、肠球菌属和埃希菌属等需氧和兼性厌氧菌为主,健康组克隆阳性率达96.45%,优势菌群以梭菌属、双歧杆菌属和巨型球菌属等专性厌氧菌为主;2周龄犊牛直肠微生物群落复杂多样,并且具有自己的独特微生物群落,且腹泻时乳杆菌属、肠球菌属、埃希氏菌属等显著增加。Mao等[30]以4头荷斯坦奶牛为对象,采集粪便样品,对粪便中的微生物16S rRNA基因的V1~V3区进行焦磷酸测序分析,分析了各样本中挥发性脂肪酸的浓度差异与粪便中微生物群落结构的差异。Frey等[31]利用末端RFLP(T-RFLP)技术分析了泌乳期奶牛瘤胃与肠道各部分乃至粪便中的微生物群落结构。Romero-Pérez等[32]认为,瘤胃与直肠中的微生物群落结构较为稳定,受到外界温度等影响较小,来源不同的样本在外界环境的共同驱使下,也未形成明显的相同的进化趋势。Durso等[33]采集了6头成年肉牛的新鲜粪便样品,通过对16S rRNA基因全长进行测序,分析了牛肠道中微生物区系的主要分类。
3 导致胃肠道微生物群落变化的因素影响胃肠道微生态的因素有很多,如饲粮以及宿主基因型、健康状况、生活环境以及环境中的共生微生物之间的群体效应等[34]。肠道黏膜会分泌一种特殊的黏蛋白,并且铺满了整个肠管内表皮。黏蛋白层是肠道内容物和肠上皮细胞之间的一张物化屏障,除了具有保护作用之外,黏蛋白层也可以与共生菌或致病菌细胞膜上的受体相结合,为这些微生物提供定植的场所。因此,肠道黏膜也是影响微生物群落构成的一个原因[35]。
3.1 饲粮研究表明,饲喂高粗饲料饲粮的反刍家畜瘤胃细菌有以下特征:大部分细菌属于革兰氏阴性菌,当饲粮中高热量饲料含量增加时,革兰氏阳性菌数量增加;大部分细菌为绝对厌氧菌,其中一些对氧气十分敏感,遇到氧气就会死亡。Shanks等[36]将30头成年健康肉牛分为6个组,饲喂条件不同,采集粪便样品进行宏基因组测序分析发现各组的主要微生物群落都分属于拟杆菌门、硬壁菌门、变形菌门,且不同的饲喂条件对肠道内的微生物群落结构和分类都有影响。杨舒黎等[37]研究云南大额牛的瘤胃微生物,大额牛的特殊食物是竹子,其对干物质的消化率以及对各种粗饲草料的消化率明显高于黄牛,瘤胃中活性细菌和纤维素降解菌数分别为4.51×109和1.63×109 CFU/mL,显著高于黄牛,其优势菌主要是瘤胃纤维素降解菌。Boots等[38]从饲粮变化对瘤胃中的厌氧真菌多样性的影响方面进行了研究,Neocallimastigales assemblages的数量变化较明显,在瘤胃中,真菌与其他微生物协同发酵的假设也得到印证。Brulc等[39]对牛瘤胃中纤维表面黏附的微生物和瘤胃中的液体混合物中的微生物进行测序,比较发现,虽然饲粮相同,但是微生物群落结构、生物型预测、代谢潜能都不同。对糖苷水解酶和纤维素功能基因比对后发现瘤胃中纤维上最初定植的微生物最先利用一些复杂植物多糖的侧链,而不是那些高强度的主链,尤其是纤维素细胞壁细胞膜。与白蚁的尾肠的基因文库相比,糖苷水解酶的含量有一些本质的区别,推测是由饲粮差异所造成的。Carberry等[40]将瘤胃中剩余的食物残渣表型联合饲喂草料中的原始组分一同分析比较,旨在研究肉牛瘤胃中的微生物对饲粮的分解对草料发酵中的偏向性,并且通过DGGE与Q-PCR的联用对不同饲粮组肉牛瘤胃微生物的多样性差异进行分析,结果见图1。
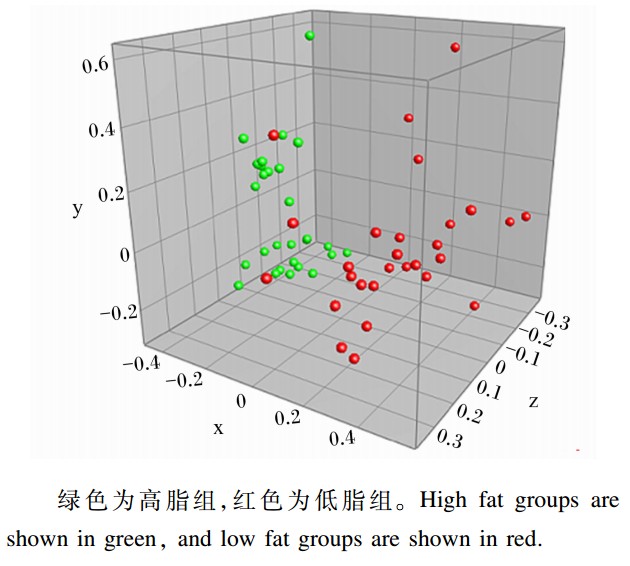 | 图1 Q-PCR与DGGE联用的多为尺度分析模型 Fig.1 MDS analytical model of Q-PCR-DGGE[40] |
Kong等[41]设计了寡核苷酸探针,用其对饲喂青贮型和干草型饲粮的奶牛的瘤胃食糜进行原位杂交,通过荧光定量技术分析了这2种不同饲喂条件下瘤胃中的微生物区系组成情况,拟杆菌门、硬壁菌门、变形菌门在2个饲喂组中均占很大比重,在细菌总数中占到31.8%~87.3%,瘤胃球菌属、产甲烷菌占细胞总数的一小部分。这证明饲喂青贮饲料的奶牛瘤胃细菌明显比饲喂干草饲粮的奶牛瘤胃细菌种类及数量更丰富。Callaway等[42]以6头健康牛为研究对象,将其分成3个组,每组饲粮中均添加不同比例的干酒糟及其可溶物,采集瘤胃与粪便样品进行测序,分析其中的微生物群落变化。结果发现,添加高水平的干酒糟及其可溶物降低了瘤胃内容物的pH,对比饲粮改变前后发现,添加干酒糟及其可溶物提高了普雷沃氏菌属和拟杆菌属的数量,降低了梭杆菌属、琥珀酸弧菌属等的数量。Vinh等[43]研究发现,尿素和石灰处理的稻草和含4%尿素的精饲料的饲粮能够提高水牛瘤胃发酵效率、瘤胃降解纤维素微生物的数量,从而改变瘤胃生态环境。Wanapat等[44]采用Q-PCR分析不同粗饲料水平下沼泽地水牛瘤胃食糜和消化液中主要纤维素降解菌种群的变化规律。Qiao等[45]研究饲粮中添加地衣芽孢杆菌和枯草芽孢杆菌的活性菌对中国荷斯坦奶牛瘤胃发酵、产奶量和乳成分的影响。研究结果表明,地衣芽孢杆菌组产奶量和乳蛋白含量显著增加,枯草芽孢杆菌的添加对瘤胃发酵、十二指肠微生物氮和瘤胃营养物质表观消化率均无显著影响。Sirohi等[46]研究了高纤维饲粮对牛瘤胃微生物群落变化的影响,采用种特异性引物进行Q-PCR,发现在高纤维饲粮的影响下,纤维素降解菌、瘤胃球菌属、黄色瘤胃球菌、丁酸弧菌属、产琥珀酸拟杆菌的基因拷贝数明显增加,细菌总量与厌氧真菌总量较改变饲粮之前均增加了近50%。González-Ortiz等[47]运用Q-PCR技术,研究了驱除原虫以及饲粮对瘤胃微生物群落的影响,发现驱除原虫后栖瘤胃普雷沃氏菌、牛链球菌和反刍兽新月单胞菌数量增加。Martin等[48]研究了在荷斯坦奶牛饲粮中添加蛋氨酸类似物对乳成分以及瘤胃微生物群落和发酵功能的影响,采用Q-PCR技术对瘤胃中的主要纤维素降解菌、产琥珀酸丝状杆菌、黄色瘤胃球菌、白色瘤胃球菌进行定量,产琥珀酸丝状杆菌、黄色瘤胃球菌数量明显增加,但是瘤胃对纤维素的水解/分解能力并未有明显的改变。橡树叶中含有大量的可水解单宁酸,可水解单宁酸是一种抗菌剂,牛食用这种橡树叶容易引起中毒,但也有一些细菌可以抵抗这种毒素,并且将单宁酸代谢生成一些对机体并无影响的产物。Belenguer等[49]研究了3头(1~3号)牛食用橡树叶后瘤胃微生物群落的变化,结果表明,在未出现中毒反应的1号牛瘤胃中的牛链球菌的相对表达量比2、3号牛低,2、3号牛均出现较严重的中毒反应,3头牛普雷沃氏菌属相对表达量均增加,而反刍兽新月单胞菌相对表达量减少;通过对细菌限制性片段长度多态性进行聚类分析发现,1号明显区别于2、3号牛,结果证明1号牛的瘤胃微生物群落与其他2头牛的微生物群落的差别可能就是1号牛未发生中毒反应的原因。González等[50]认为瘤胃酸中毒的主要原因是在饲喂过程中,饲粮中谷物颗粒含量较高,而粗饲料含量较少,长期的高精谷物饲粮使得胃中的微生物消化能力退化,微生物群落结构也发生改变。已有的大量的研究都集中在饲粮对于瘤胃微生态影响的方面,长期固定饲粮习惯,必定对瘤胃乃至肠道中的微生物定植产生导向性的作用,研究者希望能掌握微生物与饲粮结构之间的影响途径,从而对饲粮以及瘤胃微生物进行合理的优化或改造,使之朝向生产最优化方向倾斜或发展[ 51,52 ]。Chung等[53]在奶牛饲粮中添加了外源性的纤维素酶,通过对16S rRNA基因拷贝数的分析发现,嗜淀粉瘤胃杆菌数量增加,产琥珀酸丝状杆菌数量在高剂量酶组中增加,反刍兽新月单胞菌数量与酶添加剂量呈正比,牛链球菌在低剂量酶组呈下降趋势,与剂量增加呈线性增长的还有肠溶物中的甲烷产量,这些改变可能暗示着瘤胃对饲粮的降解力增强。Durso等[54]在饲粮中分别采用40%玉米和可溶解的谷物,分析20头肉牛的粪便样品的微生物多样性,测序结果显示,普雷沃氏菌属和厌氧杆菌属为出现最多的细菌,与采用玉米的饲粮相比,采用可溶解的谷物的牛粪便大肠杆菌(E.coli)O157∶ H7数量增加,提示采用该饲粮牛粪便中的微生物结构发生改变,说明饲粮对牛肠道微生态产生了影响。
3.2 宿主基因型Liggenstoffer等[55]对30个不同的草食性反刍动物与非反刍动物肠道中的厌氧真菌的系统发育多样性与群落结构等进行了研究,认为宿主的基因型是影响肠道中真菌群落结构的重要因素。Chaudhary等[56]采用Q-PCR技术,对喂食相同饲粮的印度家牛与水牛的瘤胃微生物进行对比,发现家牛瘤胃中的厌氧真菌和黄色瘤胃球菌的拷贝数明显高于二者在水牛瘤胃中,而水牛瘤胃中纤维素降解菌、产琥珀酸丝状杆菌和白色瘤胃球菌的拷贝数又明显高于其在家牛瘤胃中,再次印证了宿主的基因型也是影响瘤胃中微生物多样性的一个重要原因。Mayera等[57]比较研究了6对双胞胎犊牛与同龄相同饲养条件下的单胎犊牛的肠道微生物群落,发现双胞胎犊牛的微生物群落结构相同,因此认为这是由于宿主的基因型所决定的。
3.3 外源微生物、抗生素及环境选择压犊牛因腹泻病的高死亡率给养殖也带来了巨大的损失,超过50%的初生犊牛都是死于腹泻病,研究表明,腹泻病与肠道微生物群落有着密切的关系[58]。Lee等[59]在饲粮中添加了植物乳杆菌与枯草芽孢杆菌,对韩国本土的12头新断奶的犊牛进行了为期4周的喂养试验,之后对肠道微生物群落进行分析,发现显著提高了肠道内容物乳酸杆菌和肠杆菌的数量,并且降低了患犊牛腹泻病的几率。Signorini等[60]研究发现,饲喂全脂牛奶会提升乳酸菌在牛肠道微生物群落中的比例,降低腹泻病的发生几率,他们认为这可能是全脂牛奶提高了益生菌的活性,起到了益生元的作用,提示乳酸菌可以作为抗生素的替代品在饲粮中添加。牛的肠道微生物群落在新生牛早期种类很少,这也是犊牛易患腹泻病的一个原因。Nagashima等[61]研究发现,犊牛早期肠道内的微生物数量极少,主要为梭状芽胞杆菌,初生犊牛肠道内数量较多的是双歧杆菌和乳酸杆菌,出生15 d后数量较多的是拟杆菌,28 d之后的样本中主要微生物群落是Bacteroides,在饲粮中添加植物乳杆菌能有效地降低腹泻病的发病几率,调节肠道微生物群落结构,提高犊牛的生长速度。Michinaka等[62]利用稳定性同位素探测技术以及经优化后的T-RFLP技术对牛粪便中的益生元同化菌进行分析,牛链球菌为果糖的主要消费者,犊牛乳酸杆菌为次级果糖利用菌。梭菌属的一些微生物群落不能发酵利用果糖。此研究将传统的饲粮营养成分对肠道的微生物群落结构影响直接定位到益生元对肠道中的微生物群落影响,从而发现牛肠道中的益生菌特性,为益生菌在提高牛肠道微生物的营养能力方面提供了新的研究思路。
Grønvold等[63]对15头犊牛进行不经肠道的青霉素注射,发现虽不经过肠道,但仍旧对肠道微生物的构成产生了很大影响,对粪便中大肠杆菌的耐药性也产生了影响。Durso等[64]对牛栏地表的微生物多样性进行分析,采取土壤样本与牛粪便样本,分别进行16S rRNA基因序列分析,主要微生物群落的比例为:放线菌门42%,硬壁菌门24%,拟杆菌门24%,变形菌门9%,种类与牛粪便有明显的不同。鉴定出139个属,其中只有25个属同时出现在2个样本中。他们分析认为,形成于牛粪便中的微生物,在被排泄到环境中后,面对着新环境中多种选择压力,生存受到极大的挑战,粪便中的微生物群落结构在土壤中发生了剧烈的改变,这可能是造成牛粪便样品与牛栏表层土壤中微生物群落结果不同的主要原因。
4 小 结经过长期的选择和适应,微生物和宿主之间及微生物群落与群落之间形成了一种相互制约、相互依赖的动态平衡。这种平衡对于维持反刍动物的机体健康、提高反刍动物的生产性能、减少环境污染及保证产品安全等方面具有重要作用。围绕牛微生物多样性的研究大多集中在胃肠道环境中,由于反刍动物的特殊生理功能,可以将瘤胃微生物与口腔微生物相结合对比进行研究。分子生物学技术的广泛应用,推动了对非培养微生物的研究,但是传统的纯培养方法仍不可忽视,因为其有助于研究者获得新的功能基因以及具有优良品质的新菌种。尽管很多牛瘤胃微生物已经得到鉴定,但是,能够将完整的群落结构与其中的功能基因联系起来的技术仍旧缺乏。对牛乳房炎与犊牛腹泻病的研究中,以往多是将两者分开研究,在今后的研究中,应该将两者适当的结合在一起,探究母牛乳房炎与新生犊牛腹泻病之间是否存在某种联系。关于反刍动物的微生态研究也多集中在瘤胃细菌与肠道细菌方面,实际上在有些报道中也提到瘤胃中的真菌与古菌发挥着不可替代的作用。因而在今后的研究中,应该多关注一些真菌在反刍动物营养过程中的益生机制。目前大多研究者都将研究方向集中在对微生物群落的多样性研究,单一的定量定性研究只能让我们了解这些复杂微生物的表层信息,然而对微生物与宿主间的以及不同群体之间的物质交换、能量代谢甚至于环境是通过何种方式来改变微生物群落的,以及其中的信号传导机制等深层次研究都是一大空白。目前关于饲粮和宿主基因型哪个影响占主导地位争论也较多,虽然有大量的研究,但是,并不能确定影响微生物群落结构的主导因素是什么。所以,在动物体内共生微生物的研究领域还急需研究者进行深层次的探索,相信在不断更新的生物学新技术的帮助下,在共生微生物这个领域一定会有更大的突破。
| [1] | MIZRAHI I.Rumen symbioses[M]//The prokaryotes.Berlin Heidelberg:Springer,2013:533-544.( 1) 1)
|
| [2] | KOHL K D,WEISS R B,DALE C,et al.Diversity and novelty of the gut microbial community of an herbivorous rodent (Neotoma bryanti)[J]. Symbiosis,2011,54(1):47-54. ( 1) 1)
|
| [3] | HUNGATE R E.The rumen and its microbes[M]. New York:Academic Press,1966. ( 1) 1)
|
| [4] | TUYEN V D,CONE J W,BAARS J J P,et al.Fungal strain and incubation period affect chemical composition and nutrient availability of wheat straw for rumen fermentation[J]. Bioresource Technology,2012,111:336-342. ( 1) 1)
|
| [5] | SEKHAVATI M H,MESGARAN M D,NASSIRI M R,et al.Development and use of quantitative competitive PCR assays for relative quantifying rumen anaerobic fungal populations in both in vitro and in vivo systems[J]. Mycological Research,2009,113(10):1146-1153. ( 1) 1)
|
| [6] | ASA R,TANAKA A,UEHARA A,et al.Effects of protease-resistant antimicrobial substances produced by lactic acid bacteria on rumen methanogenesis[J]. Asian-Australasian Journal of Animal Sciences,2010,23(6):700-707. ( 1) 1)
|
| [7] | DAS K C,HUNDAL J,MAHAPATRA P S,et al.Chemical composition and in vitro gas production of fodder tree leaves and shrubs[J].Indian Veterinary Journal,2010,87:899-901. ( 1) 1)
|
| [8] | WRIGHT A D,KLIEVE A V.Does the complexity of the rumen microbial ecology preclude methane mitigation?[J]. Animal Feed Science and Technology,2011,166:248-253. ( 1) 1)
|
| [9] | ZHOU M,MCALLISTER T A,GUAN L L.Molecular identification of rumen methanogens:technologies,advances and prospects[J]. Animal Feed Science and Technology,2011,166:76-86. ( 1) 1)
|
| [10] | LI M,ZHOU M,ADAMOWICZB E,et al.Characterization of bovine ruminal epithelial bacterial communities using 16S rRNA sequencing,PCR-DGGE,and qRT-PCR analysis[J]. Veterinary Microbiology,2012,155(1):72-80. ( 1) 1)
|
| [11] | JAMI E,MIZRAHI I.Composition and similarity of bovine rumen microbiota across individual animals[J]. PLoS One,2012,7(3):e33306. ( 1) 1)
|
| [12] | PANDYA P R,SINGH K M,PARNERKAR S,et al.Bacterial diversity in the rumen of Indian Surti buffalo (Bubalus bubalis),assessed by 16S rDNA analysis[J]. Journal of Applied Genetics,2010,51(3):395-402. ( 1) 1)
|
| [13] | SINGH K M,JAKHESARA S J,KORINGA P G,et al.Metagenomic analysis of virulence-associated and antibiotic resistance genes of microbes in rumen of Indian buffalo (Bubalus bubalis)[J]. Gene,2012,507(2):146-151. ( 1) 1)
|
| [14] | PETRI R M,SCHWAIGER T,PENNER G B,et al.Changes in the rumen epimural bacterial diversity of beef cattle as affected by diet and induced ruminal acidosis[J]. Applied and Environmental Microbiology,2013,79(12):3744-3755. ( 1) 1)
|
| [15] | LI R W,CONNOR E E,LI C,et al.Characterization of the rumen microbiota of pre-ruminant calves using metagenomic tools[J]. Environmental Microbiology,2012,14(1):129-139. ( 1) 1)
|
| [16] | YANG L Y,CHEN J,CHENG X L,et al.Phylogenetic analysis of 16S rRNA gene sequences reveals rumen bacterial diversity in Yaks (Bos grunniens)[J]. Molecular Biology Reports,2010,37(1):553-562. ( 1) 1)
|
| [17] | SUENAGA H.Targeted metagenomics:a high-resolution metagenomics approach for specific gene clusters in complex microbial communities[J]. Environmental Microbiology,2012,14(1):13-22. ( 0) 0)
|
| [18] | LÓPEZ-CORTÉS N,REYES-DUARTE D,BELOQUI A,et al.Catalytic role of conserved HQGE motif in the CE6 carbohydrate esterase family[J]. FEBS Letters,2007,581(24):4657-4662. ( 1) 1)
|
| [19] | PALACKAL N,LYON C S,ZAIDI S,et al.A multifunctional hybrid glycosyl hydrolase discovered in an uncultured microbial consortium from ruminant gut[J]. Applied and Environmental Microbiology,2007,74(1):113-124. ( 1) 1)
|
| [20] | WEIMER P J,STEVENSON D M.Isolation characterization and quantification of Clostridium kluyveri from the bovine rumen[J]. Applied Microbiology and Biotechnology,2012,94(2):461-466. ( 1) 1)
|
| [21] | LIU K,WANG J,BU D,et al.Isolation and biochemical characterization of two lipases from a metagenomic library of China Holstein cow rumen[J]. Biochemical and Biophysical Research Communications,2009,385(4):605-611. ( 1) 1)
|
| [22] | WANG M,CHEN F,ZHANG S,et al.Analysis of xylanases derived from the metagenomic BAC clone library of yak rumen[J]. Acta Microbiologica Sinica,2011,51(10):1364-1373. ( 1) 1)
|
| [23] | DURSO L M,HARHAY G P,SMITH T P L,et al.Animal-to-animal variation in fecal microbial diversity among beef cattle[J]. Applied and Environmental Microbiology,2010,76(14):4858-4862. ( 1) 1)
|
| [24] | UYENO Y,SEKIGUCHI V,KAMAGATA Y.rRNA-based analysis to monitor succession of faecal bacterial communities in Holstein calves[J]. Letter in Applied Microbiology,51(5):570-577. ( 1) 1)
|
| [25] | MALMUTHUGE N,LI M,GOONEWARDENE L A,et al.Effect of calf starter feeding on gut microbial diversity and expression of genes involved in host immune responses and tight junctions in dairy calves during weaning transition[J]. Journal of Dairy Science,2013,96(5):3189-3200. ( 1) 1)
|
| [26] | JAMI E,ISR AEL A,KOTSER A,et al.Exploring the bovine rumen bacterial community from birth to adulthood[J]. The ISME Journal,2013,7(6):1069-1079. ( 1) 1)
|
| [27] | DURSO L M,HARHAY G P,BONO J L,et al.Virulence-associated and antibiotic resistance genes of microbial populations in cattle feces analyzed using a metagenomic approach[J]. Journal of Microbiological Methods,2011,84(2):278-282. ( 1) 1)
|
| [28] | DE OLIVEIRA M N V,JEWELL K A,FREITAS F S,et al.Characterizing the microbiota across the gastrointestinal tract of a Brazilian Nelore steer[J]. Veterinary Microbiology,2013,164(3/4):307-314. ( 1) 1)
|
| [29] | 许强,康立超,薄新文,等.规模化牛场犊牛直肠细菌多样性分析[J]. 微生物学报2012,52(3):304-310. ( 1) 1)
|
| [30] | MAO S Y,ZHANG R Y,WANG D S,et al.The diversity of the fecal bacterial community and its relationship with the concentration of volatile fatty acids in the feces during subacute rumen acidosis in dairy cows[J]. BMC Veterinary Research,2012,8:237. ( 1) 1)
|
| [31] | FREY J C,PELL A N,BERTHIAUME R,et al.Comparative studies of microbial populations in the rumen,duodenum,ileum and faeces of lactating dairy cows[J]. Journal of Applied Microbiology,2010,108(6):1982-1993. ( 1) 1)
|
| [32] | ROMERO-PÉREZ G A,OMINSKI K H,MCALLISTER T A,et al.Effect of environmental factors and influence of rumen and hindgut biogeography on bacterial communities in steers[J]. Applied and Environmental Microbiology,2011,77(1):258-268. ( 1) 1)
|
| [33] | DURSO L M,HARHAY G P,SMITH T P L,et al.Diversity of beef cattle fecal microbiota varies by individual[J]. Applied and Environmental Microbiology,2010,doi:10.1128/AEM.00207-10. ( 1) 1)
|
| [34] | 卢玉飞,周凌云,赵圣国,等.近10年瘤胃微生物分离培养研究进展[J]. 中国微生态学杂志,2012,24(9):856-862. ( 1) 1)
|
| [35] | JUGE N.Microbial adhesins to gastrointestinal mucus[J].Trends in Microbiology,2011,20(1):30-39. ( 1) 1)
|
| [36] | SHANKS O C,KELTY C A,ARCHIBEQUE S,et al.Community structures of fecal bacteria in cattle from different animal feeding operations[J]. Applied and Environmental Microbiology,2011,77(9):2992-3001. ( 1) 1)
|
| [37] | 杨舒黎,苟潇,冷静,等.云南大额牛的生物学特征及其瘤胃微生物特点综述[J]. 农业科学与技术,2011(12):1234-1237. ( 1) 1)
|
| [38] | BOOTS B,LILLIS L,CLIPSON N,et al.Responses of anaerobic rumen fungal diversity (phylum Neocallimastigomycota) to changes in bovine diet[J]. Journal of Applied Microbiology,2012,114:626-635. ( 1) 1)
|
| [39] | BRULC J M,ANTONOPOULOS D A,MILLERA M E B,et al.Gene-centric metagenomics of the fiber-adherent bovine rumen microbiome reveals forage specific glycoside hydrolases[J]. Proceedings of the National Academy of Sciences of the United States of America,2009,106(6):1948-1953. ( 1) 1)
|
| [40] | CARBERRY C A,KENNY D A,HAN S,et al.Effect of phenotypic residual feed intake and dietary forage content on the rumen microbial community of beef cattle[J]. Applied and Environmental Microbiology,2012,78(14):144949-144958. ( 2) 2)
|
| [41] | KONG Y,HE M,MCALISTER T,et al.Quantitative fluorescence in situ hybridization of microbial communities in the rumens of cattle fed different diets[J]. Applied and Environmental Microbiology,2010,76(20):6933-6938. ( 1) 1)
|
| [42] | CALLAWAY T R,DOWD S E,EDRINGTON T S,et al.Evaluation of bacterial diversity in the rumen and feces of cattle fed different levels of dried distillers grains plus solubles using bacterial tag-encoded FLX amplicon pyrosequencing[J]. Journal of Animal Science,2010,88(12):3977-3983. ( 1) 1)
|
| [43] | VINH N T,WANAPAT M,KHEJORNSART P,et al.Studies of diversity of rumen microorganisms and fermentation in swamp buffalo fed different diets[J]. Journal of Animal and Veterinary Advances,2011,10(4):406-414. ( 1) 1)
|
| [44] | WANAPAT M,CHERDTHONG A.Use of real-time PCR technique in studying rumen cellulolytic bacteria population as affected by level of roughage in swamp buffalo[J]. Current Microbiology,2009,58(4):294-299. ( 1) 1)
|
| [45] | QIAO G H,SHAN A S,MA N,et al.Effect of supplemental Bacillus cultures on rumen fermentation and milk yield in Chinese Holstein cows[J]. Journal of Animal Physiology and Animal Nutrition,2009,94(4):429-436. ( 1) 1)
|
| [46] | SIROHI S K,DAGAR S S,SINGH N,et al.Differential rumen microbial dynamics and fermentation parameters in cattle fed on high fibre and high concentrate diets[J]. Indian Journal of Animal Nutrition,2013,30(1):60. ( 1) 1)
|
| [47] | GONZÁLEZ-ORTIZ G,ADELANTADO C,SEGURA A G,et al.Effect of defaunation and diet on rumen microbial populations [C]//ⅩⅩⅩⅠⅩ Jornadas de Estudio,ⅩⅢ Jornadas sobre Producción Animal.Zaragoza:Asociación Interprofesional parael Desarrollo Agrario,2009:751-753. ( 1) 1)
|
| [48] | MARTIN C,MIRANDE C,MORGAVI D P,et al.Methionine analogues HMB and HMBi increase the abundance of cellulolytic bacterial representatives in the rumen of cattle with no direct effects on fibre degradation[J]. Animal Feed Science and Technology,2013,182(1/2/3/4):16-24. ( 1) 1)
|
| [49] | BELENGUER A,HERVÁS G,YÁÑEZ-RUIZ D R,et al.Preliminary study of the changes in rumen bacterial populations from cattle intoxicated with young oak (Quercus pyrenaica) leaves[J]. Animal Production Science,2010,50(3):228-234. ( 1) 1)
|
| [50] | GONZÁLEZ L A,MANTECA X,CALSAMIGLIA S,et al.Ruminal acidosis in feedlot cattle:interplay between feed ingredients,rumen function and feeding behavior (a review)[J]. Animal Feed Science and Technology,2012,172(1):66-79. ( 1) 1)
|
| [51] | CASTAÑEDA SERRANO R D,PEÑUELA SIERRA L M,CONEGLIAN S M,et al.Microbial synthesis in the rumen[J]. PUBVET,2011,5:5. ( 1) 1)
|
| [52] | WILSON A.Bacterial community dynamics and molecular ecology within the rumen of bos indicus cattle fed tropical diets [D]Ph.D.Thesis.Queensland:The University of Queensland,2010. ( 1) 1)
|
| [53] | CHUNG Y H,ZHOU M,HOLTSHAUSEN L,et al.A fibrolytic enzyme additive for lactating Holstein cow diets:ruminal fermentation,rumen microbial populations,and enteric methane emissions[J]. Journal of Dairy Science,2012,95(3):1419-1427. ( 1) 1)
|
| [54] | DURSO L M,WELLS J E,HARHAY G P,et al.Comparison of bacterial communities in faeces of beef cattle fed diets containing corn and wet distillers’ grain with solubles[J]. Letter in Applied Microbiology,55(2):109-114. ( 1) 1)
|
| [55] | LIGGENSTOFFER A S,YOUSSEF N H,COUGER M B,et al.Phylogenetic diversity and community structure of anaerobic gut fungi (phylum Neocallimastigomycota) in ruminant and non-ruminant herbivores[J]. The ISME Journal,2010,4(10):1225-1235. ( 1) 1)
|
| [56] | CHAUDHARY P P,DAGAR S S,SIROHI S K.Comparative quantification of major rumen microbial population in Indian cattle and buffalo fed on wheat straws based diet[J]. Prime Journal of Microbiology Research,2012,2(2):105-108. ( 1) 1)
|
| [57] | MAYERA M,ABENTHUMA A,MATTHESA J M,et al.Development and genetic influence of the rectal bacterial flora of newborn calves[J]. Veterinary Microbiology,2012,161(1/2):179-185. ( 1) 1)
|
| [58] | CHO Y I,KIM Y I,LIU S Y,et al.Development of a panel of multiplex real-time polymerase chain reaction assays for simultaneous detection of major agents causing calf diarrhea in feces[J]. Journal of Veterinary Diagnostic Investigation,2010,22(4):509-517. ( 1) 1)
|
| [59] | LEE Y E,KANG I J,YU E A,et al.Effect of feeding the combination with Lactobacillus plantarum and Bacillus subtilis on fecal microflora and diarrhea incidence of Korean native calves[J]. Korean Journal of Veterinary Service,2012,35(4):343-346. ( 1) 1)
|
| [60] | SIGNORINI M L,SOTO L P,ZBRUN M V,et al.Impact of probiotic administration on the health and fecal microbiota of young calves:a meta-analysis of randomized controlled trials of lactic acid bacteria[J]. Research in Veterinary Science,2012,93(1):250-258. ( 1) 1)
|
| [61] | NAGASHIMA K,YASOKAWA D,ABE K,et al.Effect of a lactobacillus species on incidence of diarrhea in calves and change of the microflora associated with growth[J]. Bioscience and Microflora,2010,29(2):97-110. ( 1) 1)
|
| [62] | MICHINAKA A,FUJII T.Efficient and direct identification of fructose fermenting and non-fermenting bacteria from calf gut microbiota using stable isotope probing and modified T-RFLP[J]. Journal of General and Applied Microbiology,2012,58(4):297. ( 1) 1)
|
| [63] | GRØNVOLD A M,MAO Y,L’ABÉE-LUND TM T M,et al.Fecal microbiota of calves in the clinical setting:effect of penicillin treatment[J]. Veterinary Microbiology,2011,153(3/4):354-360. ( 1) 1)
|
| [64] | DURSO L M,HARHAY G P,SMITH T P L,et al.Bacterial community analysis of beef cattle feedlots reveals that pen surface is distinct from feces[J]. Foodborne Pathogens and Disease,2011,8(5):647-649. ( 1) 1)
|




