2. 四川农业大学动物生物技术中心, 雅安 625014
2. Animal Biotechnology Center, Sichuan Agricultural University, Ya'an 625014, China
口腔是动物消化道的起始部分,栖息着一个复杂的微生态系统,其中有700多种细菌[1]。适宜的温度、湿度、丰富的营养为口腔内微生物的多样性提供了有利条件[2]。口腔微生物的生长、发育、衰亡及复杂的代谢活动与动物体的健康密切相关。近年来,国内外学者运用多种分子生物学方法对人类口腔微生物进行了大量研究[3,4,5],但鲜见对畜禽口腔微生物,特别是唾液微生物的报道。深入研究畜禽唾液微生物的多样性,对预防和治疗口腔疾病,保障动物健康具有重要意义。传统 的微生物分离和培养的方法,不但费时费力,而且 结果不够准确,重复性差。通过纯培养技术分离的微生物种类仅为11%左右[6],大部分信息仍处于未知状态。Muyzer等[7]最早应用聚合酶链式反应-变性梯度凝胶电泳(PCR-DGGE)技术研究微生物群落结构和多样性,该方法能较全面地反映复杂肠道微生物菌群多样性。刘怡然等[8]已成功将PCR-DGGE技术应用于研究与龋齿相关细菌的生物多样性及其动态变化。本研究拟采用PCR-DGGE技术结合聚类分析及条带的克隆和测序等,分析健康猪唾液细菌的多样性,并比较仔猪和育肥猪唾液细菌群落结构差异。
1 材料与方法 1.1 材料 1.1.1 样品的采集和处理猪唾液样品采自四川农业大学教学农场,试猪饲喂基础饲粮。饲喂前用灭菌棉球无菌操作采集健康育肥猪(145日龄)和哺乳仔猪(15日龄)唾液各5份(育肥猪样品标记为:A1~A5,仔猪样品标记为:B1~B5),置于灭菌EP管中,-20 ℃冰箱保存备用。
1.1.2 主要试剂DGGE成套试剂购自Bio-Rad公司;银染药品和试剂购自四川瑞进特科技有限公司;Gel Extraction Kit购自Omega公司;pMD 19-T Vector和Agarose Gel DNA Purification Kit购自宝生物工程(大连)有限公司;PCR引物和2×Taq MasterMix(含染料)均购自英潍捷基(上海)贸易有限公司。
1.2 唾液细菌总DNA的提取采用酚/氯仿/异戊醇法提取唾液细菌总DNA[9],用核酸浓度测定仪(NanoDrop ND-1000 UV-Vis Spectrophotometer)测定浓度后置-20 ℃冰箱保存备用。
1.3 16S rDNA V3区域PCR扩增对细菌16S rDNA V3区域片段(339~539 bp)进行PCR扩增(PCR仪为MJ Research PRC-200)。引物序列如下所示:上游引物为带GC发夹的5′-CGC CCG GGG CGC GCC CCG GGC GGG GCG GGG GCA CGG GGG GAC TCC TAC GGG AGG CAG CAG T-3′,下游引物为5′-GTA TTA CCG CGG CTG CTG GCA C-3′[10]。扩增体系25 μL:0.5 μL dNTPs(10 pmol/μL),1.25 μL引物(10 pmol/μL),1.0 μL二甲基亚砜(DMSO),2.5 μL 10×缓冲液,1.0~2.0 μL模板DNA,0.5 μL TaqDNA聚合酶(5 U/μL),最后用ddH2O补足25 μL,同时设置阴性对照组。PCR扩增程序为:94 ℃预变性4 min;94 ℃ 30 s,58 ℃ 30 s,72 ℃ 2 min,30个循环;72 ℃延伸10 min。用1.0%琼脂糖凝胶电泳检测PCR产物片段大小和浓度。
1.4 PCR-DGGE分析PCR-DGGE采用Bio-Rad Dcode Universal Detection System进行[11],凝胶电泳梯度为35%~65%(100%的变性剂包括7 mol/L尿素和40%甲酰胺),变性方向与电泳方向一致。电泳缓冲液为1×TAE,于100 V、60 ℃条件下电泳16 h。采用硝酸银染色后用Bio-Rad GS800 Calibrated Densitometer扫描凝胶。
1.5 回收共性及差异性条带并克隆测序将PCR-DGGE图谱上回收的条带浸泡于15 μL加有0.1% Triton X-100的缓冲液中4 ℃过夜。取1 μL回收的DNA用无GC夹子的引物按1.3方法扩增16S rDNA V3区域,扩增产物用2%琼脂糖凝胶电泳检测,用纯化回收试剂盒回收DNA。采用pMD 19-T Vector试剂盒,参照使用手册对目标模板PCR产物进行连接,转入大肠杆菌DH5α感受态细胞中,在含有X-Gal、异丙基-β-D-硫代半乳糖苷(IPTG)和氨苄青霉素的营养琼脂平板上培养,每个条带选取3个以上的阳性克隆送英潍捷基(上海)贸易有限公司测序。测序结果在GenBank数据库中比对,确定细菌分类信息。
1.6 数据分析将PCR-DGGE图谱数字化、标准化后用Excel表格进行整理,采用SPSS 19.0、SAS 9.1软件和NTSYS 2.1软件分别进行统计分析、主成分分析及聚类分析。
2 结 果 2.1 猪唾液细菌的16S rDNA V3区域PCR-DGGE图谱分析从PCR-DGGE图谱(图1)可看出,深色的电泳条带反映了猪口腔中的优势菌群,条带数量和位置的复杂性代表细菌菌群组成和结构的多样性。仔猪和育肥猪唾液细菌的PCR-DGGE图谱均产生了丰富的电泳条带,但条带数目和条带颜色深浅存在较大差别:仔猪唾液样品与育肥猪唾液样品相比其物种丰富度低,仔猪唾液细菌平均条带数仅为23条,且不同个体间存在较大差异,B3样品的条带数仅19条,而B1样品的条带数达29条;育肥猪唾液细菌平均条带数为28条,且5个体间无明显差异,A4样品条带数最低,为26条,A5样品条带数最高,为31条。因此,不同生长阶段的猪唾液细菌存在明显差异,随着猪的生长发育,唾液细菌的多样性增加且趋于稳定。
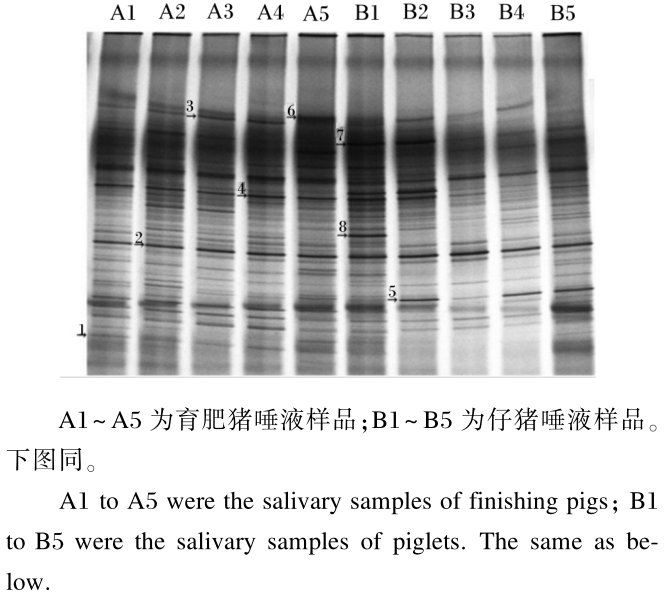 | 图1 健康猪唾液细菌的16S rDNA V3 区域PCR-DGGE图谱 Fig.1 PCR-DGGE profiles of 16S rDNA V3 region of the salivary bacteria of healthy pigs |
PCR-DGGE图谱(图1)中,各泳道具有不同 的条带数,反映不同唾液样品细菌种群的遗传多 样性;各条带具有不同的强度和迁移率,由多样性指数、丰富度和均匀度进行分析,反映不同唾液样品细菌群落物种数和个体数及其分布均匀度。由表1可知,仔猪唾液样品中细菌种群的多样性指数、均匀度和丰富度均较低,分别为3.17、0.834和23.20,而育肥猪唾液样品中细菌种群的多样性指数、均匀度和丰富度均较高,分别为3.34、0.877和28.40,表明育肥猪唾液样品细菌种群具有较高的多样性和均匀度。此结果与PCR-DGGE图谱结果相一致。
| 表1 不同样品细菌种群的多样性指数、均匀度及丰富度 Table 1 Diversity index,evenness and richness of bacteria population in difference samples |
通过NTSYS软件分析猪唾液样品的PCR-DGGE图谱,比较各个样品间的相似性。聚类分析结果(图2)显示,5头仔猪和5头育肥猪唾液样品各聚集成一族,且所有样品间相似性系数均高于0.66。仔猪唾液样品中,B2和B4间的相似性系数高达0.93,但B1和B5间的相似性系数却低至0.84;育肥猪唾液样品中,A2和A3间的相似性系数高达0.96。由此可见,不同生长阶段猪唾液样品的PCR-DGGE图谱有一定差异,幼龄阶段仔猪之间差异较大,而育肥猪之间差异较小。
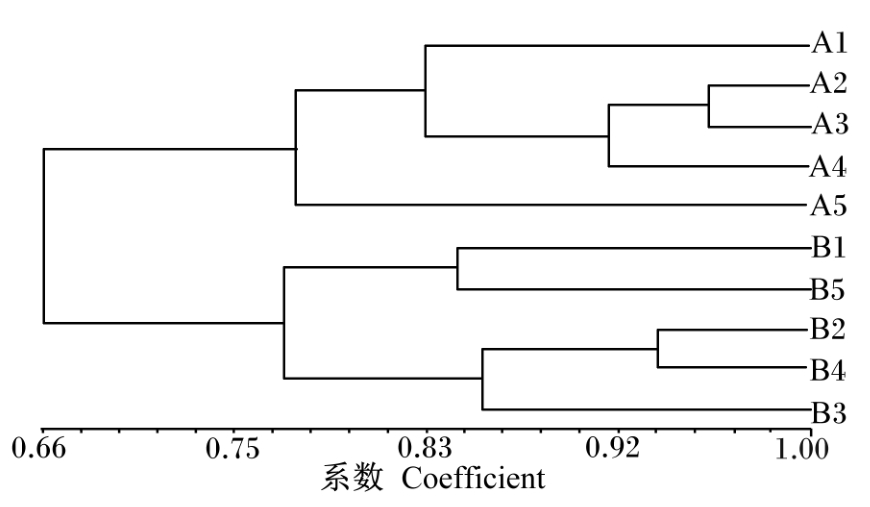 | 图2 PCR-DGGE图谱条带的聚类分析 Fig.2 Cluster analysis of PCR-DGGE profile bands |
对PCR-DGGE图谱条带的主成分分析(图3)同聚类分析的结果相一致。主成分因子1(PC1)的贡献率为33.8%,主成分因子2(PC2)的贡献率为20.1%;PC1明显地将样品分成2个部分,仔猪的唾液样品主要分布在图的左边,育肥猪的唾液样品主要分布在图的右边;PC2将B1、B5和A5猪的唾液样品同其他猪的唾液样品明显分开。
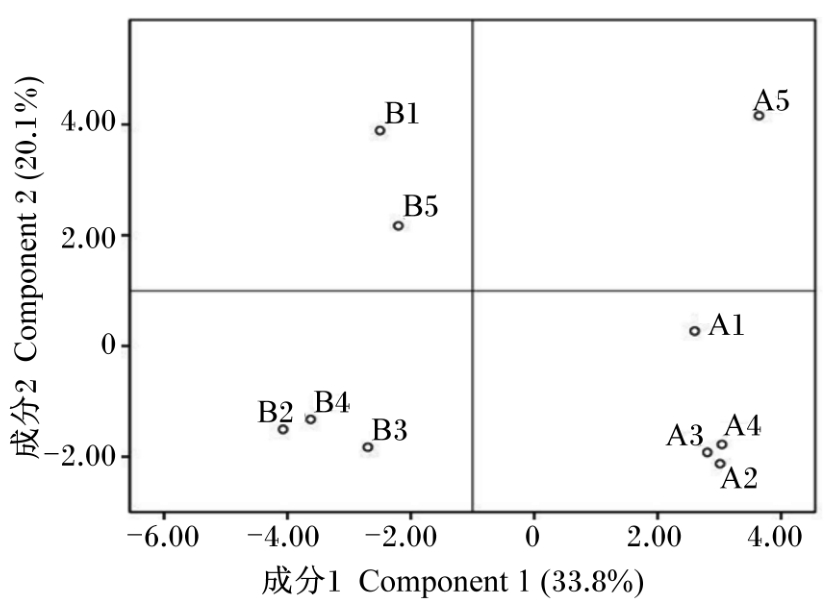 | 图3 PCR-DGGE图谱条带的聚类分析 Fig.3 Cluster analysis of PCR-DGGE profile bands |
为进一步了解猪唾液细菌的系统来源信息,对PCR-DGGE图谱中出现的5个共性条带和3个特异性条带(图1箭头所指)进行回收、克隆和测序,然后在GenBank数据库中进行序列比对分析,结果见表2。可以看出发酵乳杆菌(Lactobacillus fermentum)、大肠杆菌(Escherichia coli)、Mycobacterium arosiense、厚壁菌(Firmicutes)和猪链球菌(Streptococcus suis)是仔猪和育肥猪唾液中共同存在优势菌群。本试验回收的特异性条带Neisseria dentiae和Neisseria shayeganii来自仔猪,而葡萄球菌亚种(Staphylococcus sp.)来自育肥猪。在8个测序结果中,与GenBank数据库中细菌的同源性绝大多数都达到了100%,说明试验中所得的序列与已鉴定的微生物亲缘性高。
| 表2 PCR-DGGE共性条带和特异性条带的基因片段序列的对比结果
Table 2 Comparison results of genomic sequences in common bands and special bands of PCR-DGGE
|
本研究采用PCR-DGGE技术分析猪唾液样品的细菌群落,获得了仔猪和育肥猪唾液微生物的群落结构特征。一般来说,动物在出生时其胃肠道内存在的微生物种类和数量较少,新生动物从富含母体微生物的环境中获得胃肠道菌系,并且随着动物的生长发育而形成一个庞大的微生物群落[12]。本研究的PCR-DGGE图谱(图1)显示育肥猪的唾液细菌多样性较为丰富,多样性指数、均匀度和丰富度均比仔猪高,表明随着猪的生长发育,唾液细菌的多样性不断增加。
对PCR-DGGE图谱中的共性条带和特异性条 带的测序结果显示:仔猪和育肥猪唾液样品中均 存在大量的发酵乳杆菌、大肠杆菌、分枝杆菌、厚壁菌和猪链球菌,说明唾液样品中的优势菌多数为杆菌和变形菌。林均德等[13]研究表明,胚胎时期的动物胃肠道内存在乳杆菌、大肠杆菌、葡萄球菌、变性杆菌、微球菌和消化链球菌,它们可能是动物出生后最早定植在胃肠道内的细菌并且多数为有益菌。高侃等[14]综述了近10年间与肠道微生物相关的资料表明益生菌可通过促进黏液层的形成、分泌抗菌因子、促进肠道分泌型免疫球蛋白A(sIgA)的分泌、竞争黏附位点、增进紧密连接形成等多种方式来发挥益生作用。Goldstein等[15]研究发现,刚出生的科摩多巨蜥唾液中存在的细菌全是需氧菌,而成年科摩多巨蜥唾液中除了含有松鼠葡萄球菌(Staphylococcus sciuri)和粪肠球菌(Enterococcus faecalis)等需氧菌外,还存在大量梭状芽孢杆菌等厌氧细菌。在本研究中,Neisseria dentiae和Neisseria shayeganii是仔猪特有的细菌,其可能通过产生多糖物质或自身的荚膜对口腔中的链球菌产生凝集反应[16]。于淼等[17]研究不同龋况母子间的口腔微生物,发现链球菌和奈瑟氏球菌是高龋组儿童唾液中的优势细菌,链球菌能快速产酸,而少数奈瑟氏球菌能与变形链球菌聚集共同促进厌氧菌的生长。由此推测,奈瑟氏球菌对猪的健康可能起到非常重要的作用,但其具体作用还需要进一步研究。禹慧明等[18]研究28日龄断奶仔猪不同消化道肠段细菌数量,同样发现乳杆菌、葡萄球菌、大肠杆菌和肠球菌等优势细菌。结合本研究结果,进一步证明消化道不同部位,包括口腔,细菌的组成具有一定的相关性。且随着动物日龄的增加,唾液中的细菌多样性逐渐增加。
聚类分析(图2)及主成分分析(图3)结果表明,同一生长阶段的不同猪个体间唾液细菌的相似性更高,不同生长阶段的猪个体间的相似性较低,生长阶段不同的样品被分别聚为2大族。其原因可能是生长发育初期仔猪口腔中的细菌种类和数量均较少,而育肥猪口腔中已基本定植了种类繁多及数量巨大的细菌。本研究还发现仔猪个体间的唾液细菌存在较大差异,这可能与仔猪生长发育差异较大有关。Jami等[19]采用454焦磷酸测序技术和实时荧光定量PCR方法研究健康荷斯坦奶牛个体间瘤胃微生物的组成和相似性,发现虽然所有个体都有共同优势菌群,但个体间仍存在一定差异。本研究仅对健康仔猪和育肥猪的唾液细菌多样性和相似性进行了初步的研究,但是每种细菌的数量差异还有待进一步研究。
4 结 论① 健康仔猪和育肥猪具有共同的唾液细菌,主要是发酵乳杆菌、大肠杆菌、Mycobacterium arosiense、厚壁菌和猪链球菌;而Neisseria dentiae和Neisseria shayeganii是仔猪特有的细菌,葡萄球菌亚种是育肥猪特有的细菌。
② 随着猪的生长发育,唾液细菌多样性增加且趋于稳定。
| [1] | KAZOR C E,MITCHELL P M,LEE A M,et al.Diversity of bacterial populations on the tongue dorsa of patients with halitosis and healthy patients[J]. Journal of Clinical Microbiology,2003,41(2):558-563. ( 1) 1)
|
| [2] | PASTER B J,BOCHES S K,GALVIN J L,et al.Bacterial diversity in human subgingival plaque[J]. Journal of Bacteriology,2001,183(12):3770-3783. ( 1) 1)
|
| [3] | BONETTA S,BONETTA S,CARRARO E,et al.Microbiological characterization of Robiola di Roccaverano cheese using PCR-DGGE[J]. Food Microbiology,2008,25(6):786-792. ( 1) 1)
|
| [4] | KOLLS J K,LINDN A.Interleukin-17 family members and inflammation[J]. Immunity,2004,21(4):467-476. ( 1) 1)
|
| [5] | 吴明桃,徐晓,陈慧,等.运用DGGE分析慢性牙周炎患者唾液微生物群落结构[J]. 口腔医学,2012,32(2):65-68. ( 1) 1)
|
| [6] | EDWARDS J E,MCEWAN N R,TRAVIS A J,et al.16S rDNA library-based analysis of ruminal bacterial diversity[J]. Antonie Van Leeuwenhoek,2004,86(3):263-281. ( 1) 1)
|
| [7] | MUYZER G,DE WAAL E C,UITTERLINDEN A G.Profiling of complex microbial population by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes encoding for 16S rRNA[J]. Applied and Environmental Microbiology,1993,59(3):695-700. ( 1) 1)
|
| [8] | 刘怡然,王伟健,沈家平,等.PCR-DGGE检测早期儿童龋治疗前后口腔菌群多样性的初步研究[J]. 口腔生物医学,2012,3(4):201-204. ( 1) 1)
|
| [9] | 田晓娟,张志红,黄江丽,等.体外培养瘤胃微生物总DNA的提取与纯化[J]. 江西科学,2012,30(1):44-46,102. ( 1) 1)
|
| [10] | WALTER J,HERTEL C,TANNOCK G W,et al.Detection of Lactobacillus,Pediococcus,Leuconostoc and Weissellaspecies in human feces by using group-specific PCR primers and denaturing gradient gel electrophoresis[J]. Applied and Environmental Microbiology,2001,67(6):2578-2585. ( 1) 1)
|
| [11] | 倪学勤,GONG J,YU H,等.采用PCR-DGGE技术分析蛋鸡肠道细菌种群结构及多样性[J]. 畜牧兽医学报,2008,39(7):955-961. ( 1) 1)
|
| [12] | 张柏林,秦贵信,孙泽威,等.仔猪胃肠道微生物菌群定植规律及其功能的研究进展[J]. 中国畜牧杂志,2009,45(19):66-69. ( 1) 1)
|
| [13] | 林均德,鄂禄祥,宋连喜,等.猪和兔胚胎胃肠道菌群的初步研究[J]. 中国畜牧杂志,2007,43(10):24-26. ( 1) 1)
|
| [14] | 高侃,汪海峰,章文明,等.益生菌调节肠道上皮屏障功能及作用机制[J]. 动物营养学报,2013,25(9):1936-1945. ( 1) 1)
|
| [15] | GOLDSTEIN E J C,TYRRELL K L,CITRON D M,et al.Anaerobic and aerobic bacteriology of the saliva and gingiva from 16 captive Komodo dragons (Varanus komodoensis):new implications for the "bacteria as venom" model[J]. Journal of Zoo and Wildlife Medicine,2013,44(2):262-272. ( 1) 1)
|
| [16] | RUBY J D,SHIREY R E,GERENCSER V F,et al.Extracellular iodophilic polysaccharide synthesized by Neisseria in human dental plaque[J]. Journal of Dental Research,1982,61(5):627-631. ( 1) 1)
|
| [17] | 于淼,仪红,孟玲娜,等.不同龋状况母子间口腔微生物群落结构分析[J]. 口腔医学研究,2010,26(3):387-390. ( 1) 1)
|
| [18] | 禹慧明,廖玲,陈平洁,等.断奶仔猪肠道菌群的研究[J]. 中国微生态学杂志,2000,12(2):81-82. ( 1) 1)
|
| [19] | JAMI E,MIZRAHI I.Composition and similarity of bovine rumen microbiota across individual animals[J]. PloS One,2012,7(3):e33306. ( 1) 1)
|




