我国养猪业经过几十年的发展有了很大的进步,集约化、规模化程度越来越高。然而,在现代化养殖条件下,即使猪群个体之间具有相同的遗传背景和养殖环境,其生长性能依然存在着比较明显的差异,这给规模化养猪生产在圈舍利用、疾病防治、饲粮配制等环节的管理造成了很大的不便,增加了饲养成本。因此,揭示不同生长性能的猪在营养素消化、吸收、代谢、沉积等方面的差异规律及其机制,对于通过亚群体或个性化营养优化,提高养猪业的经济效益具有重要意义。
评价动物生长性能的主要指标有饲料转化率(feed conversion ratio,FCR)、平均日增重(average daily gain,ADG)、出栏时间等,其中,动物将饲粮转化为动物产品的效率对于集约化养殖具有重要意义。动物及人肠道内栖居着丰富的微生物菌群,与肠道环境形成一个相对动态平衡、稳定的微生态系统,对宿主的生长和健康起着重要的作用[1]。近年来的研究表明,肠道微生物对于营养素的吸收与利用具有重要影响,如Delzenne等[2]以人类和大鼠为模型研究了胃肠道菌群对肥胖症的影响,结果表明胃肠道菌群能够调节宿主的能量吸收及营养代谢;单胃动物的盲肠中,微生物可向宿主提供25%~35%因细菌降解多糖而产生的营养物质[3]。
在肠道细菌与饲料转化率的关系方面,Stanley等[4]研究发现不同饲料转化率鸡盲肠中的细菌种群存在显著差异;Singh等[5]应用高通量测序技术鉴定出了不同饲料转化率肉仔鸡粪中丰度存在显著差异的细菌种群;并应用鸟枪测序法、结合SEED数据库对不同饲料转化率肉鸡粪样中的差异微生物种群进行宏基因组学分析,发现多样性存在显著差异的细菌种群与动物对碳水化合物、氨基酸及其衍生物和蛋白质的代谢有关[6]。Hernandez-Sanabria等[7]分别给肉牛饲喂高能与低能饲粮,发现了不同饲粮条件下胃肠道内的一些特定菌群与动物的饲料转化率存在相关关系。这些研究都提示胃肠道菌群可能对动物的生长具有重要的影响。但是,对于猪肠道菌群结构与生长性能关系的研究尚未见报道。因此,本研究拟通过对不同生长性能猪不同肠段微生物的组成进行比较分析,从而在胃肠道菌群水平上揭示可能影响猪生长性能的因素。 1 材料与方法 1.1 试验材料 1.1.1 试验动物
本试验在国家饲料工程技术研究中心/农业部饲料工业中心丰宁动物试验基地进行。选取36头体重为(26.6±2.6) kg、体质健康的阉公猪(杜×长×大),称重记录耳号与栏号。试验饲粮参照NY/T 65—2004《猪饲养标准》,采用玉米-豆粕型饲粮。单栏饲养42 d,不锈钢可调式料槽,乳头式饮水器。粉料饲喂,自由采食和饮水,其他消毒、卫生等常规程序按照猪场日常管理制度进行。
1.1.2 主要试剂与仪器本试验DNA提取采用的试剂盒为德国Qiagen公司生产的QIAAMP DNA Stool Mini Kit;PCR Master Mix由Promega公司提供;琼脂糖胶浓度为1.0%;变性梯度凝胶电泳(DGGE)成套试剂均购自美国Bio-Rad公司;DGGE染色溶液用pH 7.0~8.5的缓冲液按照10 000 ∶ 1的比例稀释SYBR Green Ⅰ浓缩液混匀制成。
试验用到的仪器主要有紫外凝胶成像仪(Universal Hood Ⅱ型,美国Bio-Rad公司)、核酸浓度仪(P330型,德国Implen公司)、电泳仪(DYCP-31DN型,北京市六一仪器厂)、PCR仪(5020型,美国Thermo Fisher Scientific公司)、DGGE突变检测系统(Dcode型,美国Bio-Rad公司)。
1.2 动物分组每周以动物个体为单位记录采食量与体重,以计算每周的FCR及ADG;试验结束日计算整个试验期内的FCR以及ADG。
为了排除猪初始体重对试验结果的影响,用SAS 9.1.3软件根据初始体重与FCR和ADG的关系分别做一次回归方程,预测每头猪基于初始体重的FCR与ADG的值,并与每周实际观测值进行对比,从而得到每周FCR、ADG的残差值;试验结束日再计算整个试验期内的FCR与ADG残差值。
用Excel软件对每周FCR、ADG的残差值以及整个试验期残差值按照从高到低进行排序,最后根据总残差的排序(每周残差与整个试验期残差排序)确定不同生长速度猪的生长性能(图1)。FCR、ADG以及残差的计算公式如下:
FCR=消耗的饲料量/增加的活重;
ADG=(试验结束日重量-试验
初始日重量)/饲养天数;
残差=实际观测值-预测值。
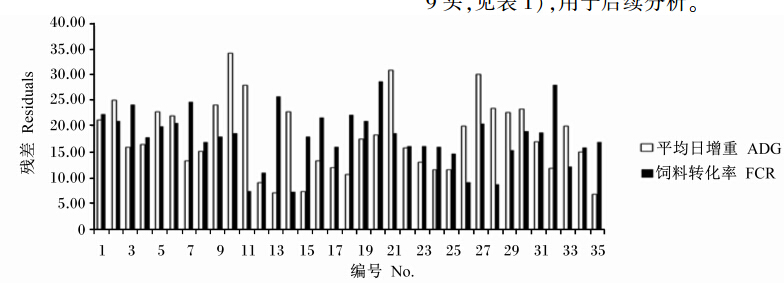 | 图1 饲料转化率及平均日增重残差总排序Fig.1 Total ranks of residuals for FCR and ADG |
该试验中,对于FCR来说,残差值越低,排序越靠后,证明实际值比预测值越小,即FCR值越小;而FCR代表消耗的饲料与增加活重的比值,其值越小表示生长性能越高;而对于ADG,其残差值越高,排序越靠前,证明实际值比预测值越大,即ADG值越大;而ADG是指试验期内动物平均每天增加的体重,其值越大表示生长性能越高。即FCR总排序越靠后、ADG总排序越靠前,表示猪生长性能越好。以FCR残差排序为主,ADG残差排序作为参考因子,将36头猪分为生长性能高、中、低3组(HP、MP和LP组),每组选择3头(共9头,见表1),用于后续分析。
| 表1 试验选取的高、中、低生长性能的9头猪的FCR与ADG残差总排序 Table 1 The total ranks of residuals for FCR and ADG of selected 9 pigs with high,middle and low growth performance |
试验最后1 d,早上将当日饲料一次性投放,从HP、MP、LP组中各选择3头屠宰后,迅速分离肠段,找到回肠、盲肠、结肠,取各肠段肠道内容物样品分装于灭菌的5 mL离心管中,并于结肠末端取粪样分装于5 mL离心管中。将样品液氮速冻,-80 ℃保存。
1.4 细菌总DNA的提取采用Qiagen公司QIAAMP DNA Stool Mini Kit提取样品细菌基因组DNA,操作方法按说明书进行。用核酸浓度测定仪测定总DNA浓度并检测其质量后,置于-20 ℃冰箱保存备用。
1.5 16S rDNA V6~V8区扩增采用16S rRNA V6~V8区引物(U968-GC-f、L1401-r)扩增细菌16S rRNA基因可变区。上游引物序列为:L1401r(5′-CGG TGT GTA CAA GAC CC-3′);下游引物设计为带GC发夹结构:U968f-GC(5′-CGC CCG GGG CGC GCC CCG GGC GGG GCG GGG GCA CGG GGG GAA CGC GAA GAA CCT TAC-3′)。反应条件参考朱伟云等[8]:上、下游引物(10 μmol/L)各1.0 μL,2×Taq Master Mix 25 μL,总DNA(模板)1 μL,PCR水补足至50 μL,同时设阴性对照管。PCR扩增条件为94 ℃预变性5 min;94 ℃变性10 s,56 ℃退火20 s,68 ℃延伸40 s,30个循环;最后68 ℃延伸7 min。用1.0%琼脂糖凝胶电泳检测PCR产物片段大小和浓度,PCR产物-20 ℃保存备用。
1.6 聚合酶链式反应-变性梯度凝胶电泳(PCR-DGGE)分析采用Bio-Rad Dcode进行DGGE。聚丙烯酰胺凝胶浓度为8%,凝胶梯度为38%~55%。采用1×TAE作电泳缓冲液,220 V预跑10 min后,在65 V电压下电泳16 h。电泳结束后用去离子水清洗胶面,放置于SYBR Green Ⅰ染色,室温条件下在摇床上摇荡15 min,在紫外凝胶成像仪下拍照。
1.7 PCR-DGGE图谱分析采用Quantity One 4.6.6软件对胶图的条带进行分析。根据文献[9, 10]从群落多样性、物种丰富程度、群落中个体分布的均匀度及优势种群的优势度等方面对微生物群落进行分析,对每头猪不同肠道菌群DGGE图谱条带数、香农指数与条带相似性指数进行计算,并将相同组的3头猪的数据做平均值用于分析。
香农指数=-∑(Ni/N)×ln(Ni/N)。
式中:Ni为单个条带的亮度,N为所有条带的亮度。
条带相似性指数=2nAB/(nA+nB)。
式中:nA代表泳道A的条带数,nB代表泳道B的条带数,nAB代表泳道A和B共有条带数。
1.8 肠道差异微生物条带割胶回收和测序对DGGE图谱上共性和差异条带进行割胶测序分析。将切下的胶带回收到装有50 μL TE缓冲液的离心管中,用枪头捣碎,4 ℃浸泡过夜后取1 μL上清液作为模板,用不带GC夹子的引物重新扩增,PCR条件同上,扩增产物采用琼脂糖凝胶电泳检测。将切割条带测序(北京擎科新业生物技术有限公司),测序结果用Blast程序与GenBank数据库中的序列进行比较。
1.9 统计分析试验结果采用SAS 9.1.3程序中的ANOVA过程分析,将每1组3头猪作为1个试验单元,P<0.05表示差异显著,P<0.01表示差异极显著。 2 结 果 2.1 肠道菌群16S rDNA V6~V8区基因片段的PCR-DGGE图谱分析
由图2可以看出,粪样以及回肠、盲肠、结肠内容物样品中微生物的16S rRNA基因片段经过DGGE分离后均得到了丰富的条带,并且后肠段内容物样品中的DGGE条带数多于前肠段。
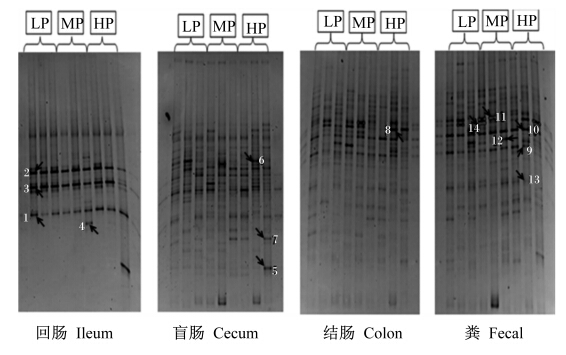 | 图2 不同生长性能猪肠道微生物群落PCR-DGGE图谱Fig.2 PCR-DGGE profiles of enteric microflora in pigs with different growth performance |
采用Quantity One 4.6.6软件对PCR-DGGE 条带进行多样性和相似性分析,结果(表2)显示不同生长性能猪各肠段菌群DGGE条带数、香农指数存在差异。与LP组相比,HP、MP组回肠样品DGGE条带数显著增加(P<0.05);结肠样品DGGE条带数,HP组与MP、LP组相比差异显著(P<0.05);盲肠与粪样DGGE条带数数值上有提高,但统计学差异不显著(P>0.05)。各肠道内容物样品中DGGE微生物图谱香农指数各组间并无显著差异(P>0.05),但是数值上由LP组到HP组呈现增加趋势。
| 表2 条带数、香农指数及条带相似性指数 Table 2 Band number,Shannon index and band similarity index |
对DGGE优势条带(图2)切胶回收测序,运用Blast程序将DGGE条带切胶测序结果与GenBank数据库中的序列进行比较,比较结果见表3。
表3显示切胶回收的条带所代表的细菌与现有数据库中的细菌有很高的相似性。基因测序结果显示,条带2与已鉴定的马犹姆贝梭菌相似性为100%,条带3、4、5、7、10分别与已鉴定的Turicibacter sanguinis、格氏链球菌、嗜酸乳杆菌、粪球菌属、梭菌属相似性为99%,条带12与已鉴定的罗氏菌属M88/1相似性为97%,条带13、14分别与已鉴定的腐败梭菌、柔嫩梭菌相似性为95%,条带6与已鉴定的罗氏菌属499相似性为90%,条带1、8、9、11属于不可培养细菌。
| 表3 PCR-DGGE分离获得的细菌16S rDNA V6~V8区序列 Table 3 Bacteria 16S rDNA V6 to V8 sequences detected by PCR-DGGE |
条带5、7分别为嗜酸乳杆菌和粪球菌属,为HP组盲肠特有条带,条带亮度较高;条带8所代表的菌群为未培养的罗氏菌属,为HP、MP组结肠特有条带,条带亮度高;其他条带所代表的菌群为HP、MP、LP组共有优势条带。 3 讨 论
胃肠道菌群在机体的各项生命活动中起着非常重要的作用,不仅对于抵御病原微生物的侵入和免疫系统发育有重要作用[11],而且可以帮助宿主消化、吸收营养物质[12],通过其代谢产物为机体提供能量[13]。近些年来的研究表明,胃肠道菌群与动物对营养素的消化、吸收、利用具有重要的关系[4,5,6,7]。本研究通过对不同生长性能猪肠道微生物组成进行比较分析发现:与生长性能低的猪相比,生长性能高的猪回肠、结肠、盲肠和粪样中DGGE条带数增加,香农指数呈现增长趋势,并且 二者盲肠、结肠中相关细菌的丰度也存在显著差异。
DGGE图谱中不同位置条带代表着不同的细菌种群,电泳条带数和位置的复杂性代表了菌群的多样性,条带信号越强表示该条带代表的相应细菌数量较多且为优势菌[14]。本试验中不同生长性能组回肠、盲肠、结肠和粪样DGGE图谱条带数均存在差异,这充分反映了肠道微生物菌群的多样性。此外,HP组回肠、结肠和盲肠DGGE条带数都高于LP组,说明生长性能高的猪肠道菌群丰度、优势菌丰度更高。DDGE条带数反映肠道微生物中的优势菌群,条带数越多,有益微生物菌群可能就越多,这对肠道屏障、免疫、营养物质消化吸收等具有重要作用[15]。
香农指数常用于评价群落的多样性,能体现群落内种群数和种群间个体分配的均匀度,并且该指数反映了群落中的常见种群[16]。肠道微生物种群数越多、分配的均匀度越高,菌种之间的相互依赖和制约能力就越强,当机体遭受应激刺激或者外界环境波动较大时菌群能更好地保持动态平衡,从而增加机体应对不良因素的能力[17,18]。本试验中HP组回肠、盲肠、结肠和粪样DGGE微生物图谱香农指数数值上均大于LP组,表示细菌群落在优势度、丰度和均匀度方面较好,证明了猪生长性能与肠道微生物群落多样性具有直接关系。
通过对切胶回收的条带进行测序及与DGGE图谱对比发现,HP组盲肠中的乳酸杆菌和粪球菌较为丰富,Wang等[19]认为乳酸杆菌可以调节仔猪肠道菌群平衡,抑制肠道不良微生物的增殖;Holdeman等[20]研究报道粪球菌属细菌可以发酵碳水化合物,利用甲酸和丙酸产生丁酸、醋酸。HP、MP组结肠中罗氏菌属细菌丰度较高,有研究报道表明,罗氏菌属细菌可以合成丁酸供机体利用,丁酸不仅可以抵抗致病微生物入侵,增强肠道的屏障功能[21,22] ,而且可以降低肠段食糜流通速率从而提高机体对于营养物质的吸收利用[23,24]。 4 结 论
本试验结果表明生长性能高的猪其肠道菌群的丰度、肠道菌群分配的均匀度、肠道菌群相似度更高,并且粪肠菌、罗氏菌属细菌以及乳酸菌在生长性能高的猪肠道中丰度也较高。
| [1] | 杭苏琴, 毛胜勇, 黄瑞华, 等.断奶仔猪饲喂甘露寡糖后粪样菌群的变化[J]. 农业生物技术学报, 2006, 14(5):701-705. ( 1) 1)
|
| [2] | DELZENNE N M, CANI P D.Interaction between obesity and the gut microbiota:relevance in nutrition[J]. Annual Review of Nutrition, 2010, 31:15-31. ( 1) 1)
|
| [3] | 张日俊.消化道微生物与宿主营养素的吸收和代谢研究[J]. 中国饲料, 2003(2):11-14. ( 1) 1)
|
| [4] | STANLEY D, DENMAN S E, HUGHES R J, et al.Intestinal microbiota associated with differential feed conversion efficiency in chickens[J]. Applied Microbiology and Biothechnology, 2012, 96(5):1361-1369. ( 2) 2)
|
| [5] | SINGH K M, SHAH T, DESHPANDE S, et al.High through put 16S rRNA gene-based pyrosequencing analysis of the fecal microbiota of high FCR and low FCR broiler growers[J]. Molecular Biology Reports, 2012, 39(12):10595-10602. ( 2) 2)
|
| [6] | SINGH K M, SHAN T M, REDDY B, et al.Taxonomic and gene-centric metagenomics of the fecal microbiome of low and high feed conversion ratio (FCR) broilers[J]. Journal of Applied Genetics, 2014, 55(1):145-154. ( 2) 2)
|
| [7] | HERNANDEZ-SANABRIA E, GOONEWARDENE L A, WANG Z, et al.Impact of feed efficiency and diet on adaptive variations in the bacterial community in the rumen fluid of cattle[J]. Applied and Environmental Microbiology, 2012, 78(4):1203-1214. ( 2) 2)
|
| [8] | 朱伟云, 姚文, 毛胜勇.变性梯度凝胶电泳法研究断奶仔猪粪样细菌区系变化[J]. 微生物学报, 2003, 43(4):503-508. ( 1) 1)
|
| [9] | GAFAN G P, LUCAS V S, ROBERTS G J, et al.Statistical analyses of complex denaturing gradient gel electrophoresis profiles[J]. Journal of Clinical Microbiology, 2005, 43(8):3971-3978. ( 1) 1)
|
| [10] | LEDDER R G, GILBERT P, HUWS S A, et al.Molecular analysis of the subgingival microbiota in health and disease[J]. Applied and Environmental Microbiology, 2007, 73(2):516-523. ( 1) 1)
|
| [11] | 杨玲, 杨卫景, 马先进, 等.仔猪消化道微生物区系变化影响因素研究[J]. 中兽医学杂志, 2009(5):41-45. ( 1) 1)
|
| [12] | CHADWICK R W, GEORGE S E, CLAXTON L D.Role of the gastrointestinal mucosa and microflora in the bioactivation of dietary and environmental mutagens or carcinogens[J]. Drug Metabolism Reviews, 1999, 24(4):425-492. ( 1) 1)
|
| [13] | SALMINEN S, BOULEY C, BOUTRON M C, et al.Functional food science and gastrointestinal physiology and function[J]. British Journal of Nutrition, 1998, 80(Suppl.1):147S-171S. ( 1) 1)
|
| [14] | MUYZER G, SMALLA K.Application of denaturing gradient gel electrophoresis (DGGE) and temperature gradient gel electrophoresis (TGGE) in microbial ecology[J]. Antonie van Leeuwenhoek, 1998, 73(1):127-141. ( 1) 1)
|
| [15] | RAKOFF-NAHOUM S, PAGLINO J, ESLAMI-VARZANEH F, et al.Recognition of commensal microflora by toll-like receptors is required for intestinal homeostasis[J]. Cell, 2004, 118(2):229-241. ( 1) 1)
|
| [16] | GREENBERH J H.The measurement of linguistic diversity[J]. Language, 1956, 32(1):105-115. ( 1) 1)
|
| [17] | HOOPER L V, MACPHERSON A J.Immune adaptations that maintain homeostasis with the intestinal microbiota[J]. Nature Reviews Immunology, 2010, 10(3):159-169. ( 1) 1)
|
| [18] | SHANAHAN F.Probiotics in perspective[J]. Gastroenterology, 2010, 139(6):1808-1812. ( 1) 1)
|
| [19] | WANG X Q, YANG F, LIU C, et al.Dietary supplementation with the probiotic Lactobacillus fermentum I5007 and the antibiotic aureomycin differentially affects the small intestinal proteomes of weanling piglets[J]. The Journal of Nutrition, 2012, 142(1):7-13. ( 1) 1)
|
| [20] | HOLDEMAN L V, MOORE W E C.New genus, Coprococcus, twelve new species, and emended descriptions of four previously described species of bacteria from human feces[J]. International Journal of Systematic Bacteriology, 1974, 24(2):260-277. ( 1) 1)
|
| [21] | RAQIB R, SARKER P, BERGMAN P, et al.Improved outcome in shigellosis associated with butyrate induction of an endogenous peptide antibiotic[J]. Proceedings of the National Academy of Sciences of the United State of America, 2006, 103(24):9178-9183. ( 1) 1)
|
| [22] | GUILLOTEAU T, MARTIN L, EECKHAUT V, et al.From the gut to the peripheral tissues:the multiple effects of butyrate[J]. Nutrition Research Reviews, 2010, 23(2):366-384. ( 1) 1)
|
| [23] | KOTUNIA A, WOLINSKI J, LAUBITZ D, et al.Effect of sodium butyrate on the small intestine development in neonatal piglets fed by artificial sow[J]. Journal of Physiology and Pharmacology, 2004, 55(Suppl.2):59-68. ( 1) 1)
|
| [24] | LE GALL M, GALLOIS M, SVE B, et al.Comparative effect of orally administered sodium butyrate before or after weaning on growth and several indices of gastrointestinal biology of piglets[J]. British Journal of Nutrition, 2009, 102(9):1285-1296. ( 1) 1)
|




