2. 乐至县天龙农牧科技有限公司, 乐至 641507
2. Lezhi County Tianlong Agriculture and Animal Husbandry Technology Co., Ltd., Lezhi 641507, China
蛋白质饲料是反刍动物饲粮中最主要的氮源,但是我国蛋白质饲料资源十分紧张,估计2020年我国蛋白质饲料的供需缺口将达到0.48亿t[1]。高效合理地利用蛋白质饲料资源可在一定程度上缓解蛋白质饲料资源紧张的局面。反刍动物是主要的粗饲料利用者,其饲粮主要依靠瘤胃微生物来消化,瘤胃中存在的细菌、真菌及原虫均具有分解利用纤维物质的能力,在纤维物质的分解利用过程中细菌和真菌发挥了80%的作用,原虫起平衡控制作用[2]。产琥珀酸丝状杆菌、黄色瘤胃球菌和白色瘤胃球菌是3种主要的瘤胃纤维素分解菌[3]。调控瘤胃微生物是提高饲料的利用率、缓解蛋白质饲料资源短缺、提高经济效益的有效途径之一。而前人对瘤胃微生物的研究中,在饲粮方面主要是针对精粗比[4, 5, 6, 7]和粗饲料[8, 9, 10]不同的研究,对精料中蛋白质饲料不同对瘤胃纤维降解微生物的影响方面的研究较少。瘤胃微生物在瘤胃内主要是存在于瘤胃液、附着于瘤胃内壁上和黏附于饲料颗粒上[11]。大量研究表明,瘤胃固相微生物数量高于瘤胃液,是瘤胃中主要的功能微生物[6, 9, 10, 12]。因此,研究不同蛋白质饲料对宣汉黄牛瘤胃固相黏附纤维降解微生物的影响显得非常重要。豆粕、菜籽粕和棉籽粕是我国经常使用的植物性蛋白质饲料。本试验选用这3种蛋白质饲料等量替代基础饲粮中15%精料来饲喂宣汉黄牛,研究瘤胃固相黏附纤维降解微生物数量的变化,旨在为更好地调控瘤胃微生物,提高饲料的利用率提供一定的参考。
1 材料与方法 1.1 试验材料与设备菜籽粕、棉籽粕、豆粕及其他饲料原料均来自四川农业大学动物营养研究所试验基地;TIANamp Stool DNA Kit、RNaseA (100 mg/mL)、DNA分子质量标准、6×DNA上样缓冲液、DNase/RNase-free ddH2O、SuperReal PreMix Plus(SYBR Green)均购自TIANGEN(北京)公司。主要设备有荧光定量PCR仪(CFX96TM,美国Bio-Rad)、凝胶成像系统(美国Syngene)、核酸蛋白检测仪(DU-800,美国Beckman)。
1.2 试验动物和试验设计选用四川农业大学动物营养研究所试验基地4头体况良好,装有永久性瘤胃瘘管的4岁去势宣汉黄牛[平均体重(455.45±18.93) kg]。采用4×4的拉丁方设计,共4期,每期10 d(预饲7 d,采样3 d)。基础饲粮参考我国《肉牛饲养标准》(NY/T 815—2004)设计,1.3倍维持需要,精粗比为4 ∶ 6,基础饲粮组成及营养水平见表1,基础组饲喂基础饲粮。试验组分别饲喂以豆粕(豆粕组)、棉籽粕(棉籽粕组)和菜籽粕(菜籽粕组)等量替换基础饲粮中的精料(替换比例为基础饲粮的15%)的试验饲粮,其营养水平见表2。基础组、豆粕组、菜籽粕组和棉籽粕组每头牛饲喂量分别为7.35、6.93、7.19和6.99 kg/d(干物质基础),平均分成2次定时(07:00和17:00)定量饲喂,拴系饲养,自由饮水。
参考张丽英[13]的方法测定饲粮干物质(DM)和粗蛋白质(CP)含量。按照Van Soest等[14]的方法测定饲粮中性洗涤纤维(NDF)、酸性洗涤纤维(ADF)和酸性洗涤木质素(ADL)含量。采用高锰酸钾法间接测定饲粮钙(Ca)含量,采用钼酸盐法[15]测定饲粮总磷(TP)含量。
| 表1 基础饲粮组成及营养水平(风干基础) Table 1 Composition and nutrient levels of the basal diet (air-dry basis) |
| 表2 试验饲粮营养水平(风干基础) Table 2 Nutrient levels of experimental diets (air-dry basis) |
3 d采样时间分别为第1天08:00和14:00,第2天10:00和16:00,第3天12:00,即采食后1、3、5、7和9 h。在每头牛的瘤胃内分别取上、下、左、右、中5个位置的瘤胃内容物约200 g,并用4层纱布过滤分离出固相样品,装入无菌封口袋中,样品于-80 ℃保存备用。
1.4 测定指标及方法 1.4.1 瘤胃固相微生物总DNA的提取将瘤胃固相样品4 ℃解冻后,参考Kang等[16]和Laure等[17]的方法洗脱固相黏附微生物。取1 g样品于灭菌10 mL离心管中,加入5 mL磷酸盐缓冲液悬浮,混匀30 s,350×g室温离心15 min,取上清于一新的灭菌离心管。再向残渣部分加入4 mL厌氧条件下制备的0.15%(V/V)吐温80,充分混匀后在冰上放置2.5 h,350×g室温离心15 min,取上清于前一离心管,10 000×g 4 ℃离心20 min,去上清留沉淀。然后使用TIANamp Stool DNA Kit试剂盒提取微生物总DNA。用核酸蛋白检测仪测定提取的总DNA的浓度及OD260 nm/OD280 nm。于-20 ℃保存。
1.4.2 瘤胃固相微生物数量的测定采用荧光定量PCR技术测定各组饲喂后1、3、5、7和9 h宣汉黄牛瘤胃固相黏附纤维降解微生物相对于瘤胃总菌的数量。引物序列及参数见表3,引物由上海华大生物技术有限公司合成。反应体系为15 μL:2×SuperReal PreMix Plus 7.5 μL,上、下游引物(10 μmol/L)各0.45 μL,总DNA溶液0.6 μL,RNase-free ddH2O 6 μL。扩增条件为:95 ℃预变性15 min,95 ℃变性10 s,相应的退火温度30 s,共40个循环。自动检测荧光强度。扩增反应的特异性通过对PCR终产物制作熔解曲线来确定,方法为:从65 ℃升温到95 ℃,每隔5 s升高0.5 ℃,仪器自动绘制熔解曲线。荧光定量PCR产物采用2%的琼脂糖凝胶电泳检测。
| 表3 引物序列及参数 Table 3 Sequences and parameters of primers |
根据Chen等[23]的方法将微生物数量表示为相对于总菌16S rDNA的百分比,公式如下:
目标微生物(%)=2-(Ct目标微生物-Ct总菌)×100。
式中:Ct为阈值循环。
试验结果用Excel 2007进行预处理,用SPSS 18.0统计软件对结果进行单因素方差分析,采用Duncan氏法多重比较检验,结果以平均值±标准差表示(P<0.05表示差异显著,P<0.01表示差异极显著)。
2 结 果 2.1 总DNA提取结果和引物特异性提取的总DNA OD260 nm/OD280 nm在1.6~1.9,说明得到的总DNA能够很好地应用于后面的荧光定量PCR扩增试验。瘤胃固相黏附纤维降解微生物荧光定量PCR产物电泳检测结果见图1,扩增条带清晰且无拖带现象,扩增片段长度与文献报道基本一致。这说明所采用的引物有很高的特异性,扩增体系没有被污染,扩增条件和参数适宜。
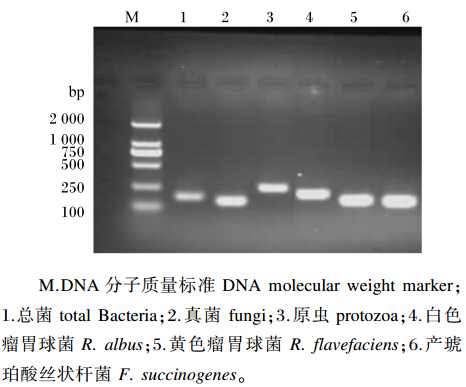 | 图1 瘤胃固相黏附纤维降解微生物荧光定量PCR产物电泳检测结果 Fig. 1 Electrophoresis results of products of fluorescent quantitation PCR of cellulosis microbes adherent to solid fractions in rumen |
由表4可知,随着采食时间的延长,基础组和菜籽粕组真菌数量变化趋势相似,均呈现降低-升高-降低-升高的趋势。各组原虫、白色瘤胃球菌、黄色瘤胃球菌、产琥珀酸丝状杆菌数量的变化趋势各不相同。
2.2.1 真菌基础组、菜籽粕组、豆粕组和棉籽粕组的真菌数量分别在采食后9、5、5和7 h时达到最大值。除采食后5 h外其余各时间点组间差异显著(P<0.05)。其中,采食后1 h,豆粕组真菌的数量显著低于其余各组(P<0.05)。采食后3和9 h,3个试验组均显著低于基础组(P<0.05)。采食后7 h,棉籽粕组显著高于菜籽粕组(P<0.05),极显著高于豆粕组和基础组(P<0.01),菜籽粕组显著高于豆粕组和基础组(P<0.05)。从各组平均值来看,基础组和棉籽粕组均显著高于菜籽粕组和豆粕组(P<0.05)。
2.2.2 原虫基础组、菜籽粕组、豆粕组和棉籽粕组的原虫数量分别在采食后3、3、7和9 h时达到最大值。采食后1和3 h,菜籽粕组原虫的数量显著或极显著高于其余3组(P<0.05或P<0.01)。采食后5和7 h,各试验组都显著高于基础组(P<0.05)。采食后9 h,棉籽粕组显著高于菜籽粕组(P<0.05),极显著高于豆粕组和基础组(P<0.01),菜籽粕组显著高于豆粕组和基础组(P<0.05)。从各组平均值来看,菜籽粕组原虫数量显著高于豆粕组和棉籽粕组(P<0.05)、极显著高于基础组(P<0.01)。
2.2.3 白色瘤胃球菌基础组和棉籽粕组的白色瘤胃球菌数量在采食后7 h时达到最大值,菜籽粕组和豆粕组在采食后5 h时达到最大值。采食后1 h,菜籽粕组白色瘤胃球菌的数量显著高于其余3组(P<0.05)。采食后3 h,棉籽粕组显著高于菜籽粕组和基础组(P<0.05)。采食后5 h,豆粕组显著高于菜籽粕组和棉籽粕组(P<0.05)。采食后7 h,棉籽粕组显著高于菜籽粕组(P<0.05),极显著高于豆粕组(P<0.01)。采食后9 h,棉籽粕组显著高于其余3组(P<0.05)。从各组平均值来看,各组间差异不显著(P>0.05),从数值上排序为棉籽粕组>基础组>豆粕组>菜籽粕组。
2.2.4 黄色瘤胃球菌基础组和菜籽粕组的黄色瘤胃球菌数量在采食后9 h时达到最大值,豆粕组和棉籽粕组在采食后7 h时达到最大值。除采食后3 h时菜籽粕组和豆粕组差异不显著(P>0.05)外,3个试验组其余4个时间点和平均值都是菜籽粕组显著或极显著高于豆粕组和棉籽粕组(P<0.05或P<0.01)。除采食后1和3 h时,豆粕组显著高于棉籽粕组(P<0.05)外,其余3个时间点和平均值豆粕组和棉籽粕组差异不显著(P>0.05)。
2.2.5 产琥珀酸丝状杆菌基础组、菜籽粕组、豆粕组和棉籽粕组的产琥珀酸丝状杆菌的数量分别在采食后3、9、7和7 h达到最大值。采食后1 h,菜籽粕组和豆粕组产琥珀酸丝状杆菌的数量显著高于棉籽粕组和基础组(P<0.05)。采食后3和7 h,3个试验组间差异均不显著(P>0.05)。采食后5 h,菜籽粕组显著高于豆粕组(P<0.05)。采食后9 h,菜籽粕组显著高于豆粕组和棉籽粕组(P<0.05)。从各组平均值来看,菜籽粕组产琥珀酸丝状杆菌数量显著高于豆粕组和棉籽粕组(P<0.05)。
| 表4 不同蛋白质饲料对瘤胃固相黏附纤维降解微生物数量的影响 Table 4 Effects of different protein feeds on the populations of cellulosis microbes adherent to solid fractions in rumen |
瘤胃真菌在纤维物质降解过程中起着积极的作用[24]。在正常饲养条件下,瘤胃微生物数量在109~1010个/mL时,真菌游动孢子数量在103~105个/mL之间[25, 26],本试验得到的真菌数量较总菌数量低4个数量级左右,与之一致。
本试验结果表明,不同蛋白质饲料显著影响瘤胃固相黏附真菌的数量,其中棉籽粕组真菌的数量平均值显著高于菜籽粕组和豆粕组。王海荣[27]对绵羊的研究得出,不同氮源饲粮对瘤胃真菌浓度和数量影响显著,棉籽粕组瘤胃真菌数量显著高于豆粕组;王贞贞[28]在绵羊上的研究得到,棉籽粕组瘤胃真菌数量最多,豆粕组数量最少。以上研究均与本试验结果一致或相似。瘤胃厌氧真菌的种群密度与饲粮中的纤维物质含量有很大关系,纤维物质含量丰富的饲粮因在瘤胃中有较长的滞留时间,通常导致瘤胃中厌氧真菌的数量较高[29, 30],本试验所用的4种饲粮,棉籽粕组和基础组的纤维含量略高于豆粕组和菜籽粕组,这与各组真菌数量的差异结果一致。
3.2 不同蛋白质饲料对瘤胃固相黏附原虫数量的影响一般将原虫分为鞭毛虫和纤毛虫,瘤胃中个体最大,数量最多,最重要的原虫是纤毛虫[31]。大多数纤毛虫具有很强的纤维降解活性,去原虫会降低纤维素的消化率[32]。已有研究表明,原虫对不同的蛋白质底物的作用不同,饲粮结构在很大程度上影响反刍动物瘤胃中纤毛虫的组成和数量[33]。王梦芝等[34]运用遗传指纹图谱研究不同的蛋白质饲料对体外培养瘤胃微生物发酵和其群体结构的影响,表明原虫区系类群结构在各组间明显不同;罗成[35]的研究也得出,不同蛋白质组成的饲粮对陕北白绒山羊瘤胃原虫蛋白质浓度有显著的影响。本试验结果说明,不同蛋白质饲料显著影响瘤胃固相黏附原虫的数量,前文提及的研究结果与本试验的结果一致。菜籽粕组原虫数量平均值显著高于豆粕组和棉籽粕组,可能是因为纤毛虫降解可溶性蛋白质,分解不溶性的蛋白质颗粒能力较强,本课题组之前的研究就表明,菜籽粕的不可利用蛋白质的含量极显著地高于豆粕和棉籽粕[36]。
正常饲喂条件下,真菌和原虫在瘤胃共存,它们在纤维物质降解中起重要作用,但他们之间存在着捕食与被捕食的关系,原虫摄取了真菌的假根和孢子囊[37],原虫对营养物质的利用导致真菌数量减少[38],而且原虫的酶可以降解真菌细胞壁,这种方式对真菌的生长有负面影响。本试验棉籽粕组和菜籽粕组在原虫和真菌的数量上刚好相反,或许正是这个原因造成的。
3.3 不同蛋白质饲料对瘤胃固相黏附纤维素分解菌数量的影响反刍动物是粗饲料的主要利用者,细胞壁降解的80%由瘤胃中细菌和真菌完成。细菌由于其在数量上的绝对优势,在纤维物质分解过程中发挥了特别重要的作用。瘤胃中的纤维素分解菌主要包括白色瘤胃球菌、黄色瘤胃球菌和产琥珀酸丝状杆菌[3]。
本试验结果表明,3种纤维素分解菌中,各组产琥珀酸丝状杆菌的数量最高,这与前人的研究结果一致[21, 27, 39],说明产琥珀酸丝状杆菌也是宣汉黄牛瘤胃内的优势纤维素分解菌。棉籽粕组白色瘤胃球菌和产琥珀酸丝状杆菌的数量均高于豆粕组,这与王海荣[27]在绵羊上研究得到的结果一致。豆粕组和菜籽粕组白色瘤胃球菌的数量均高于黄色瘤胃球菌的数量,可能是因为白色瘤胃球菌产生的细菌素抑制了黄色瘤胃球菌的生长[40]。菜籽粕组白色瘤胃球菌的数量在各试验组间最低,棉籽粕组最高,但是菜籽粕组产琥珀酸丝状杆菌的数量最高,显著高于豆粕组和棉籽粕组,这可能是因为当产琥珀酸丝状杆菌与瘤胃球菌同时存在时,产琥珀酸丝状杆菌的数量明显受到抑制[24]。当以纤维素为碳源时,黄色瘤胃球菌与产琥珀酸丝状杆菌存在着竞争关系,底物不同会影响到它们的竞争状态[41]。本试验豆粕组黄色瘤胃球菌的数量显著低于棉籽粕组,但是其产琥珀酸丝状杆菌的数量高于棉籽粕组,可能正是因为底物的不同导致竞争状态的差异。
4 结 论不同蛋白质饲料对瘤胃固相黏附纤维降解微生物的数量有显著影响,以菜籽粕对宣汉黄牛瘤胃固相黏附纤维降解微生物的促进效应最好。
| [1] | 袁涛,张伟力.我国几种蛋白质饲料资源现状[J]. 江西饲料,2004(2):25-28. ( 1) 1)
|
| [2] | SUN Y Z,MAO S Y,YAO W,et al.DGGE and 16S rDNA analysis reveals a highly diverse and rapidly colonising bacterial community on different substrates in the rumen of goats[J]. Animal,2008,2(3):391-398. ( 1) 1)
|
| [3] | STEWART C S,FLINT H J,BRYANT M P.The rumen bacteria[M]//HOBSON P N,STEWART C S.The rumen microbial ecosystem.Netherlands:Springer,1997:10-72. ( 2) 2)
|
| [4] | FERNANDO S C,PURVIS Ⅱ H T,NAJAR F Z,et al.Rumen microbial population dynamics during adaptation to a high-grain diet[J]. Applied and Environmental Microbiology,2010,76(22):7482-7490. ( 1) 1)
|
| [5] | KIM M,MORRISON M,YU Z T,et al.Phylogenetic diversity of bacterial communities in bovine rumen as affected by diets and microenvironments[J]. Folia Microbiology,2011,56(5):453-458. ( 1) 1)
|
| [6] | PETRI R M,FORSTER R J,YANG W,et al.Characterization of rumen bacterial diversity and fermentation parameters in concentrate fed cattle with and without forage[J]. Journal of Applied Microbiology,2012,112(6):1152-1162. ( 2) 2)
|
| [7] | CARBERRY C A,KENNY D A,HAN S,et al.Effect of phenotypic residual feed intake and dietary forage content on the rumen microbial community of beef cattle[J]. Applied and Environmental Microbiology,2012,78(14):4949-4958. ( 1) 1)
|
| [8] | 冯薇,王加启,刘开朗,等.运用PCR-DGGE分析比较瘤胃中不同饲料固相粘附微生物区系[J]. 畜牧兽医学报,2010,41(12):1556-1562. ( 1) 1)
|
| [9] | HUWS S A,LEE M R,MUETZEL S M,et al.Forage type and fish oil cause shifts in rumen bacterial diversity[J]. FEMS Microbiology Ecology,2010,73(2):396-407. ( 2) 2)
|
| [10] | KONG Y H,TEATHER R,FORSTER R.Composition,spatial distribution,and diversity of the bacterial communities in the rumen of cows fed different forages[J]. FEMS Microbiology Ecology,2010,74(3):612-622. ( 2) 2)
|
| [11] | MCALLISTER T A,BAE H D,JONES G A,et al.Microbial attachment and feed digestion in the rumen[J]. Journal of Animal Science,1994,72(11):3004-3018. ( 1) 1)
|
| [12] | KIM M,MORRISON M,YU Z.Status of the phylogenetic diversity census of ruminal microbiomes[J]. FEMS Microbiology Ecology,2011,76(1):49-63. ( 1) 1)
|
| [13] | 张丽英.饲料分析及饲料质量检测技术[M]. 3版.北京:中国农业大学出版社,2007. ( 1) 1)
|
| [14] | VAN SOEST P J,ROBERTSON J B,LEWIS B A.Methods for dietary fiber,neutral detergent fiber,and nonstarch polysaccharides in relation to animal nutrition[J]. Journal of Dairy Science,1991,74(10):3583-3597. ( 1) 1)
|
| [15] | HARRIS W D,POPAT P.Determination of the phosphorus content of lipids[J]. Journal of the American Oil Chemists' Society,1954,31(4):124-127. ( 1) 1)
|
| [16] | KANG S H,DENMAN S E,MORRISON M I.An efficient RNA extraction method for estimating gut microbial diversity by polymerase chain reaction[J]. Current Microbiology,2009,58(5):464-471. ( 1) 1)
|
| [17] | LARUE R,YU Z T,PARISI V A.Novel microbial diversity adherent to plant biomass in the herbivore gastrointestinal tract,as revealed by ribosomal intergenic spacer analysis and rrs gene sequencing[J]. Environmental Microbiology,2005,7(4):530-543. ( 1) 1)
|
| [18] | MUYZER G,DE WAAL E C,UITTERLINDEN A G.Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J]. Applied and Environmental Microbiology,1993,59(3):695-700. ( 1) 1)
|
| [19] | 徐爱秋,王梦芝,王洪荣,等.体外培养条件下氨基酸对瘤胃蛋白质降解菌生长限制性的影响[J]. 动物营养学报,2010,22(1):88-92. ( 1) 1)
|
| [20] | DENMAN S E,TOMKINS N W,MCSWEENEY C S.Quantitation and diversity analysis of ruminal methanogenic populations in response to the antimethanogenic compound bromochloromethane[J]. FEMS Microbiology Ecology,2007,62(3):313-322. ( 1) 1)
|
| [21] | KOIKE S,KOBAYASHI Y.Development and use of competitive PCR assays for the rumen cellulolytic bacteria:Fibrobacter succinogenes,Ruminococcus albus and Ruminococcus flavefaciens[J]. FEMS Microbiology Letters,2001,204(2):361-366. ( 2) 2)
|
| [22] | DENMAN S E,MCSWEENEY C S.Development of a real-time PCR assay for monitoring anaerobic fungal and cellulolytic bacterial populations within the rumen[J]. FEMS Microbiology Ecology,2006,58(3):572-582. ( 1) 1)
|
| [23] | CHEN X L,WANG J K,WU Y M,et al.Effects of chemical treatments of rice straw on rumen fermentation characteristics,fibrolytic enzyme activities and populations of liquid- and solid-associated ruminal microbes in vitro[J]. Animal Feed Science and Technology,2008,141(1/2):1-14. ( 1) 1)
|
| [24] | 冯仰廉.反刍动物营养学[M]. 北京:科学出版社,2004. ( 2) 2)
|
| [25] | HUNGATE R E.A roll-tube method for cultivation of strict anaerobes[M]//Methods in microbiology.London:Academic Press,1969:117-132. ( 1) 1)
|
| [26] | THEODOROU M K,GILL M,KING-SPOONER C,et al.Enumeration of anaerobic chytridiomycetes as thallus-forming units:novel method for quantification of fibrolytic fungal populations from the digestive tract ecosystem[J]. Applied and Environmental Microbiology,1990,56(4):1073-1078. ( 1) 1)
|
| [27] | 王海荣.不同日粮精粗比及氮源对绵羊瘤胃纤维降解菌群和纤维物质降解的影响[D]. 博士学位论文.呼和浩特:内蒙古农业大学,2006. ( 3) 3)
|
| [28] | 王贞贞.不同氮源日粮对绵羊瘤胃纤维降解菌群及纤维降解的影响[D]. 硕士学位论文.呼和浩特:内蒙古农业大学,2007. ( 1) 1)
|
| [29] | DEHORITY B A,ORPIN C G.Development of,and natural fluctuations in,rumen microbial populations[M]//HOBSON P N,STEWART C S.The rumen microbial ecosystem.Netherlands:Springer,1997:196-245. ( 1) 1)
|
| [30] | FONDEVILA M,DEHORITY B A.Interactions between Fibrobacter succinogenes,Prevotella ruminicola,and Ruminococcus flavefaciens in the digestion of cellulose from forages[J]. Journal of Animal Science,1996,74(3):678-684. ( 1) 1)
|
| [31] | USHIDA K,JOUANY J P.Methane production associated with rumen-ciliated protozoa and its effect on protozoan activity[J]. Letters in Applied Microbiology,1996,23(2):129-132. ( 1) 1)
|
| [32] | YANG C J M,VARGAT G A.The effects of continuous ruminal dosing with dioctyl sodium sulphosuccinate on ruminal and metabolic characteristics of lactating Holstein cows[J]. British Journal of Nutrition,1993,69(2):397-408. ( 1) 1)
|
| [33] | IVAN M,NEILL L,FORSTER R,et al.Effects of Isotricha,Dasytricha,Entodinium,and total fauna on ruminal fermentation and duodenal flow in wethers fed different diets[J]. Journal of Dairy Science,2000,83(4):776-787. ( 1) 1)
|
| [34] | 王梦芝,喻礼怀,王洪荣,等.不同蛋白质饲料对瘤胃微生物体外发酵和群体结构的影响[J]. 动物营养学报,2009,21(5):673-679. ( 1) 1)
|
| [35] | 罗成.不同蛋白质饲料对陕北白绒山羊瘤胃内环境及MCP浓度和瘤胃微生物群落的影响[D]. 硕士学位论文.杨凌:西北农林科技大学,2011. ( 1) 1)
|
| [36] | 唐春梅.不同来源蛋白饲料的养分组成差异及瘤胃降解效率的比较研究[D]. 硕士学位论文. 雅安:四川农业大学,2013. ( 1) 1)
|
| [37] | WILLIAMS A G,WITHERS S E,ORPIN C G.Effect of the carbohydrate growth substrate on polysaccharolytic enzyme formation by anaerobic fungi isolated from the foregut and hindgut of nonruminant herbivores and the forestomach of ruminants[J]. Letters in Applied Microbiology,1994,18(3):147-151. ( 1) 1)
|
| [38] | MORGAVI D P,SAKURADA M,TOMITA Y,et al.Presence in rumen bacterial and protozoal populations of enzymes capable of degrading fungal cell walls[J]. Microbiology,1994,140(3):631-636. ( 1) 1)
|
| [39] | KOIKE S,PAN J,KOBAYASHI Y,et al.Kinetics of in sacco fiber-attachment of representative ruminal cellulolytic bacteria monitored by competitive PCR[J]. Journal of Dairy Science,2003,86(4):1429-1435. ( 1) 1)
|
| [40] | ODENYO A A,MACKIE R I,STAHL D A,et al.The use of 16S rRNA-targeted oligonucleotide probes to study competition between ruminal fibrolytic bacteria:development of probes for ruminococcus species and evidence for bacteriocin production[J]. Applied and Environmental Microbiology,1994,60(10):3688-3669. ( 1) 1)
|
| [41] | ODENYO A A,MACKIE R I,STAHL D A,et al.The use of 16S rRNA-targeted oligonucleotide probes to study competition between ruminal fibrolytic bacteria:pure-culture studies with cellulose and alkaline peroxide-treated wheat straw[J]. Applied and Environmental Microbiology,1994,60(10):3697-3703. ( 1) 1)
|




