表观遗传学是研究在不涉及DNA序列改变的情况下,引起基因表达调控的可遗传变化的一门学科[1]。表观遗传学修饰在基因的表达调控中具有重要作用。DNA甲基化是最常见的一种表观遗传学修饰,可引起DNA构象、染色质结构及DNA与蛋白质相互作用方式的改变,进而影响基因表达。胚胎期是表观基因组重编程的关键时期,易受外界环境影响。研究表明,孕妇摄取叶酸可预防胎儿发生心血管疾病,而叶酸是一种可通过改变DNA甲基化来影响基因表达的甲基供体[2]。孕鼠饲粮中添加的富含甲基的营养物质,可通过改变相关基因特异位点“CpG岛”的甲基化,加重后代的过敏性哮喘[3]。刺鼠胚胎期饲粮中的甲基供体可使胎儿相关基因启动子区域甲基化而影响其表达,最终改变后代的毛皮颜色[4]。将怀孕大鼠在高剂量农药中暴露一段时间后,后代成年后会出现器官损伤,而雄性子代精子出现的异常的DNA甲基化模式至少可持续4代[5]。因此,了解鸡胚发育过程中DNA甲基化的变化规律,对于从胚胎期入手研究家禽表观遗传学具有重要意义。
目前,用于甲基化检测的方法有亚硫酸盐修饰后测序[如甲基化特异性PCR(methylation specific PCR,MSP)]、甲基敏感扩增片段多态性(methylation sensitive amplified polymorphism,MSAP)、甲基化DNA免疫沉淀(methylated DNA immunoprecipitation,MeDIP)和焦磷酸测序等。MSP法利用亚硫酸氢盐可将未甲基化的C转化为U,经PCR扩增后又转变成T,而甲基化的C则不变,测序后比较修饰前后PCR产物的序列差异可获得其中的甲基化信息[6]。此方法属于位点特异性DNA甲基化检测,只适于检测已知基因中包含若干CpG位点的某一片段的甲基化状态。MSAP法使用的一对同裂酶(HPaⅡ和MspⅠ)可识别基因组中相同的CCGG位点,产生甲基化敏感多态性片段,随后进行PCR扩增,可明确甲基化状态[7]。该方法最大的缺点是,无法识别非CCGG序列中的CG位点。MeDIP法应用5-甲基胞嘧啶抗体特异性识别并分离甲基化的DNA片段,进行高通量测序。这种方法费用昂贵,不适合多样品的基因组DNA甲基化测序。焦磷酸测序法可测定基因组中大量存在且DNA甲基化高发的重复原件Alu和LINE-1(long interspread nucleotide element-1)的甲基化率,并以此代表基因组DNA甲基化的总体水平[8],但该法得到的并非严格意义上的基因组DNA甲基化水平。高效液相色谱(HPLC)法能够快速、准确测定基因组DNA甲基化水平,并可用于高通量混合样本检测,准确显示目的片段所有CpG位点的甲基化情况[9]。
本试验改进了基因组DNA提取方法、DNA酶解体系和HPLC检测条件,通过检测科宝(Cobb)500肉鸡胚胎发育第8、11、14和17天心脏、肝脏和肌肉的基因组DNA甲基化水平,以初步了解鸡胚发育过程中DNA甲基化水平的变化规律。
1 材料与方法 1.1 仪器和试剂
蓝电孵化机(9TV-3A,北京蓝天蛟电子技术有限公司),日立D-2000 HPLC仪[天美(中国)科学仪器有限公司],Ultimate Polar-RP色谱柱(4.6 mm×250 mm,5 μm)[月旭材料科技(上海)有限公司];脱氧胞苷(deoxycytidine,dC)(Sigma)和5-甲基脱氧胞苷(deoxy-5-methylcytidine,5-mdC)标准品(TCI);RNase A,RNase T1,DNase Ⅰ,FastAP Thermosensitive Alkaline Phosphatase,Exonuclease Ⅰ(Fermentas)。
1.2 孵化管理与样品采集选择外形、重量均匀的Cobb 500肉鸡种蛋100枚孵化,孵化条件如下。
温度:前期(1~10 d)38.0~38.2 ℃,后期(11~18 d)37.8~38.0 ℃,落盘(19~21 d)37.5~37.8 ℃;相对湿度:45%~65%;翻蛋周期:120 min;翻蛋时间:180 s。
于孵化期第8、11、14和17天(分别记为E8、E11、E14和E17)分别选取发育良好的种蛋6枚,取鸡胚心脏、肝脏和肌肉放入冻存管,-80 ℃保存备用。
1.3 样品处理DNA提取:苯酚/氯仿/异戊醇(25:24:1,体积比)法提取组织DNA。第2次抽提前,加RNase A至终浓度80 μg/mL,RNase T1至终浓度1 500 U/mL,混匀后37 ℃水浴1 h。提取的DNA溶于40 μL TE缓冲液中,-20 ℃保存备用。
DNA酶解:参照前人研究[9, 10, 11, 12, 13],将DNA酶解体系设为150 μL,其中组织DNA约20 μg,醋酸铵(pH=7.5)15 μL,DNase Ⅰ 10 μL,10×DNaseⅠBuffer(MgCl2)15 μL,FastAP Thermosensitive Alkaline Phosphatase 10 μL,10×Alkaline Phosphatase Buffer 15 μL,Exonuclease Ⅰ 5 μL,10×Exonuclease Ⅰ Buffer 15 μL,超纯水补足至150 μL。37 ℃孵育4~5 h,过0.22 μm滤膜,4 ℃保存备用。
HPLC分析:色谱柱为Ultimate Polar-RP(4.6 mm×250.0 mm,5 μm);流动相为0.2%(体积分数)磷酸;流速为1.3 mL/min;检测波长为273 nm;进样量为20 μL。
1.4 DNA甲基化水平计算分别配制7种浓度(0.5、1.0、2.0、5.0、10.0、20.0和50.0 mg/L)的dC和5-mdC标准品溶液,用于绘制标准曲线。根据标准曲线计算样品色谱图中2种脱氧核苷峰面积对应的浓度(分别记为CdC和C5-mdC),则样品基因组DNA的甲基化水平(methylation level,ML)为:
ML(%)=100×C5-mdC/(C5-mdC+CdC)。
1.5 统计分析采用SPSS 18.0统计软件单因素方差分析(one-way ANOVA)过程对同一胚龄不同组织和不同胚龄同一组织的基因组DNA甲基化水平进行单因素方差分析,采用Duncan氏法对差异显著处理进行多重比较,显著水平为P<0.05。
2 结果与分析 2.1 DNA酶解效果检测
HPLC法检测基因组DNA甲基化水平的前提是DNA必须完全酶解为单个脱氧核苷。为检验样品DNA是否完全酶解为单个脱氧核苷,并比较组织DNA与酶解DNA,本试验对酶解前后的组织DNA进行琼脂糖凝胶电泳检测,结果(图1)显示,基因组DNA已酶解为单个脱氧核苷。
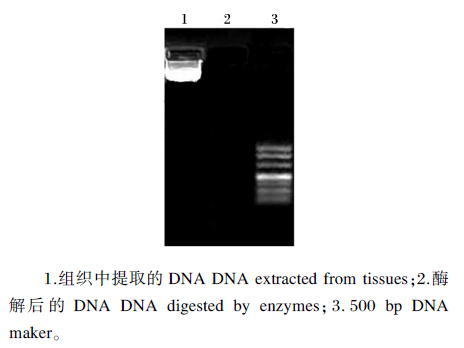 | 图1 DNA酶解效果
Fig. 1 Result of DNA digested by enzymes |
2.2 DNA甲基化水平的计算
通过2种方法确定dC和5-mdC的色谱峰位置(图2和图3):比较样品洗脱峰和标准品洗脱峰的保留时间;在样品中添加标准品,观察色谱峰的变化。根据标准曲线[相对标准偏差(RSD)=0.68%(n=7)](图4)计算样品中2种脱氧核苷的浓度,并通过公式得到样品基因组DNA甲基化水平。
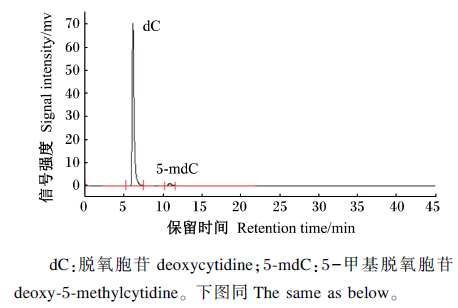 | 图2 2种脱氧核苷标准品的色谱图 Fig. 2 Chromatograms of 2 kinds of deoxynucleoside standard samples |
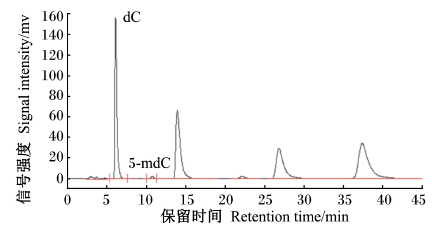 | 图3 DNA样品的色谱图 Fig. 3 Chromatogram of DNA sample |
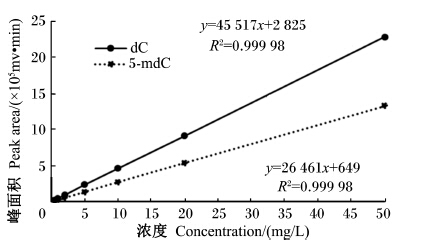 | 图4 2种脱氧核苷的标准曲线 Fig. 4 Standard curves of 2 kinds of deoxynucleoside |
以信噪比为3和10确定的dC的最低检测限(LOD)和最小定量限(LOQ)分别为0.015和0.045 mg/L,5-mdC的分别为0.097和0.323 mg/L。
分别配制5种不同浓度(1、2、5、10和20 mg/L)的dC和5-mdC标准品溶液,取已知dC和5-mdC浓度的样品进行加标回收率测定,每个标准品浓度重复3次,试验结果见表1。
| 表1 脱氧胞苷和5-甲基脱氧胞苷的平均回收率 Table 1 The average recovery of dC and 5-mdC (n=3) |
由表2可知,鸡胚心脏、肝脏和肌肉的基因组DNA甲基化水平均随胚龄增加而升高。E14和E17心脏的基因组DNA甲基化水平显著高于E8和E11(P<0.05);各胚龄肝脏的基因组DNA甲基化水平均差异显著(P<0.05);E8和E17肌肉的基因组DNA甲基化水平差异显著(P<0.05),E11和E14肌肉的基因组DNA甲基化水平分别与其他3个胚龄差异不显著(P>0.05)。
| 表2 不同胚龄鸡胚各组织基因组DNA甲基化水平 Table 2 Genome DNA methylation level of different tissues at different chick embryo ages |
由表2可知,E8、E11和E14肝脏基因组DNA甲基化水平与其他2种组织均差异不显著(P> 0.05),但E17肝脏基因组DNA甲基化水平显著 高于心脏和肌肉(P<0.05)。各胚龄心脏和肌肉基因组DNA甲基化水平均差异不显著(P>0.05)。
3 讨 论 3.1 HPLC法测定基因组DNA甲基化条件的优化及注意事项 3.1.1 HPLC法测定基因组DNA甲基化条件的优化DNA分阶段酶解[10, 13]需要至少3次酶解和水浴,操作繁琐且孵育时间长。本试验采用了尹慧等[11]的酶解体系,一次性加入DNA酶解所需的核酸外切酶、内切酶和碱性磷酸酶,只经过1次水浴,并在保证酶解效果的前提下将酶解时间缩短至4~5 h。将酶解体系增加至150 μL,使经0.22 μm滤膜的滤液更多,保证足够的滤液用于HPLC上样前的润洗和进样。
根据脱氧核苷的性质,本试验选择0.2%(体积分数)磷酸作为流动相[11]。Ultimate Polar-RP色谱柱的最佳流速为0.8 mL/min,在保证色谱柱使用寿命和色谱峰分离效果的同时,为尽量缩短采样时间,本试验将流速设为1.3 mL/min。用于DNA酶解的关键酶全部购自同一家公司,改善了dC和5-mdC色谱峰的分离效果(图2和图3)。
DNA变性是指核酸双螺旋碱基对的氢键断裂,双链变成单链,改变了核酸的天然构象和性质。将DNA样本在100 ℃加热3 min可实现DNA变性[13]。DNA甲基化水平检测需在上样检测前彻底酶解为单个脱氧核苷,因此本试验省去了DNA变性过程。终止酶解反应是为防止残存酶液对后续反应产生影响,常通过高温来实现,如65或85 ℃加热10~15 min[11, 13]。本试验中,酶解后的DNA为核酸基本单位,且直接用于HPLC检测,无后续反应,故省略。
3.1.2 HPLC法测定基因组DNA甲基化的注意事项本试验使用的HPLC法对DNA浓度和质量均有较高要求,建议用传统方法提取。经检验,本试验设置的体系最多可消化25 μg DNA。此上限内,增加用于酶解的DNA量可增加色谱峰面积,降低系统误差。
5-mdC易氧化,如果DNA放置时间过长,酶解后的HPLC色谱图中5-mdC所在峰右侧会出现与主峰混合的小杂峰,造成积分误差。因此,应尽早酶解DNA进行HPLC检测。
酶解后的DNA体系,在过0.22 μm滤膜前后进行瞬时离心,可保证较多的液体用于HPLC上样检测。
3.2 鸡胚各组织基因组DNA甲基化水平变化规律在哺乳动物上的研究表明,表观基因组于配子形成期和胚胎发育期经历2次消除和重建[14],而表观基因组水平与其稳定性和环境敏感性直接相关。传统的静态模型理论认为,表观基因组的稳定性与DNA甲基化和组蛋白修饰情况呈正比[15]。配子形成期和胚胎发育期的表观基因组处于动态变化过程,易受外界环境影响。因家禽的胚胎发育环境区别于哺乳动物,本试验研究了肉鸡的胚胎发育期,以初步了解家禽胚胎发育过程中表观基因组重编程的基本规律。
爱拔益加肉鸡和红原鸡的DNA甲基化图谱显示,DNA甲基化集中在基因体和重复序列,转录起始位点和转录终止位点的甲基化水平较低[16]。基因启动子区域的甲基化与否与基因表达密切相关:启动子甲基化抑制基因表达,去甲基化促进基因表达。当基因具有转录活性时,其启动子处于低甲基化状态,即开启基因表达。染色体存在复杂的折叠结构,影响mRNA的转录效率,而DNA甲基化可改变染色体的空间结构,舒展折叠状态,利于转录因子与基因结合,促进转录。研究表明,有活性X染色体的甲基化水平是无活性的2倍,且主要表现在基因体上,基因启动子则呈现低甲基化状态[17]。
鸡胚发育分3个阶段:孵化第1~4天发育内部器官,为发育早期;第5~15天发育外部器官发育阶段,为发育中期;第16~19天是鸡胚的生长阶段,为发育后期。实际操作中,从鸡胚发育第8天起能有效分离足够的心脏、肝脏和肌肉组织用于DNA提取,因此,本试验选取的第1个时间点为鸡胚发育第8天。试验结果表明,鸡胚发育过程中基因组DNA甲基化水平同哺乳动物一样呈逐渐升高的趋势。
鸡胚发育中期,骨骼和肌肉等功能相对单一的外部器官未完全发挥功能,DNA甲基化水平升高缓慢;鸡胚发育后期,各组织器官体积快速增大,充分发挥功能以备出壳,基因中有碍mRNA转录的结构可能通过甲基化作用得以改善,使DNA甲基化水平随之迅速升高。部分研究认为,鸡胚在孵化后期出现甲基化高峰可能与该时期的胚胎体重增加有关[18]。
研究表明,同种动物不同组织的基因组DNA甲基化模式存在差异[19, 20, 21]。本试验中,肝脏的甲基化模式明显异于心脏和肌肉,可能与肝脏系生化代谢中心,各种mRNA转录旺盛,而心脏和肌肉同属肌肉组织,功能相对单一有关。家鸡胚胎发育早期DNA甲基化的MSAP分析结果显示,家鸡胚胎甲基化呈现较高的组织特异性,其中肝脏的甲基化程度较高,肺脏较低,肾脏和胸肌的甲基化水平相近。随着鸡胚发育的进行,DNA甲基化水平呈逐渐增高的趋势[18]。本试验结果与之相同。
4 结 论
① 本试验应用HPLC法检测了鸡胚基因组DNA甲基化水平,优化后的HPLC法简化了操作步骤,缩短了酶解时间,改善了dC和5-mdC色谱峰的分离效果,为快速、准确获取各种组织基因组DNA甲基化水平奠定了基础。
② 随着鸡胚发育的进行,各组织基因组DNA甲基化水平逐渐升高,且鸡胚发育后期肝脏呈现较高的甲基化水平。
| [1] | HOLLIDAY R.The inheritance of epigenetic defects[J]. Science,1987,238(4824):163-170. ( 1) 1)
|
| [2] | ROSENQUIST T H,FINNELL R H.Genes,folate and homocysteine in embryonic development[J].Proceedings of the Nutrition Society,2001,60(1):53-61. ( 1) 1)
|
| [3] | HOLLINGWWORTH J W,MARUOKA S,BOON K,et al.In utero supplementation with methyl donors enhances allergic airway disease in mice[J]. The Journal of Clinical Investigation,2008,118(10):3462-3469. ( 1) 1)
|
| [4] | WATERLAND R A,JIRTLE R L.Transposable elements:targets for early nutritional effects on epigenetic gene regulation[J]. Molecular and Cellular Biology,2003,23(15):5293-5300. ( 1) 1)
|
| [5] | SKINNER M K,HAQUE C G B M,NILSSON E,et al.Environmentally induced transgenerational epigenetic reprogramming of primordial germ cells and the subsequent germ line[J]. PLoS One,2013,8(7):e66318. ( 1) 1)
|
| [6] | HERMAN J G,GRAFF J R,MYÖHÄNEN S,et al.Methylation-specific PCR:a novel PCR assay for methylation status of CpG islands[J]. Proceedings of the National Academy of Sciences of the United States of America,1996,93(18),9821-9826. ( 1) 1)
|
| [7] | GONZALGO M L,LIANG G N,SPRUCK Ⅲ C H,et al.Identification and characterization of differentially methylated regions of genomic DNA by methylation-sensitive arbitrarily primed PCR[J].Cancer Research,1997,57(4):594-599. ( 1) 1)
|
| [8] | YANG A S,ESTECIO M R H,DOSHI K,et al.A simple method for estimating global DNA methylation using bisulfite PCR of repetitive DNA elements[J]. Nucleic Acids Research,2004,32(3):e38. ( 1) 1)
|
| [9] | KUO K C,MCCUNE R A,GEHRKE C W,et al.Quantitative reversed-phase high performance liquid chromatographic determination of major and modified deoxyribonucleosides in DNA[J]. Nucleic Acids Research,1980,8(20):4763-4776. ( 2) 2)
|
| [10] | 彭思远,张洁,田美平,等.液相色谱-串联质谱法测定生物样本全基因组DNA甲基化[J]. 分析化学,2012,40(8):1201-1206. ( 2) 2)
|
| [11] | 尹慧,黄代新,翟仙敦,等.HPLC法检测人外周血5-mC含量及其与年龄相关性研究[J]. 中国法医学杂志,2007,22(1):8-11. ( 4) 4)
|
| [12] | CRAIN P F.Preparation and enzymatic hydrolysis of DNA and RNA for mass spectrometry[J].Methods in Enzymology,1990,193:782-790. ( 1) 1)
|
| [13] | FRISO S,CHOI S W,DOLNIKOWSKI G G,et al.A method to assess genomic DNA methylation using high-performance liquid chromatography/electrospray ionization mass spectrometry[J]. Analytical Chemistry,2002,74(17):4526-4531. ( 4) 4)
|
| [14] | REIK W,DEAN W,WALTER J.Epigenetic reprogramming in mammalian development[J]. Science,2001,293(5532):1089-1093. ( 1) 1)
|
| [15] | HEMBERGER M,PEDERSEN R.Epigenome disruptors[J]. Science,2010,330(6004):598-599. ( 1) 1)
|
| [16] | LI Q H,LI N,HU X X,et al.Genome-wide mapping of DNA methylation in chicken[J]. PLoS One,2011,6(5):e19428 ( 1) 1)
|
| [17] | HELLMAN A,CHESS A.Gene body-specific methylation on the active X chromosome[J]. Science,2007,315(5815):1141-1143. ( 1) 1)
|
| [18] | 班谦.家鸡胚胎早期发育过程中DNA甲基化的MSAP分析[D]. 硕士学位论文.石河子:石河子大学,2009. ( 2) 2)
|
| [19] | BIRD A.DNA methylation patterns and epigenetic memory[J]. Genes & Development,2002,16(1):6-21. ( 1) 1)
|
| [20] | VANYUSHIN B F.Enzymatic DNA methylation is an epigenetic control for genetic functions of the cell[J]. Biochemistry (Moscow),2005,70(5):488-499. ( 1) 1)
|
| [21] | 唐韶青,张沅,徐青,等.不同动物部分组织基因组甲基化程度的差异分析[J]. 农业生物技术学报,2006,14(4):507-511. ( 1) 1)
|




