2. 动物疫病与人类健康四川省重点实验室, 成都 611130
2. Sichuan Province Key Laboratory of Animal Disease and Human Health, Chengdu 611130, China
动物每一个暴露于外界的部位,包括皮肤、口腔、呼吸道、泌尿生殖道和消化道均有大量的微生物定植,其中消化道是最密集的微生物定植器官[1]。而在动物消化道中,产乳酸的细菌是最主要的益生菌之一。乳酸可以降低胃肠道内的pH,抑制大肠杆菌、沙门氏菌、志贺氏菌等病原菌的生长,也具有活化消化酶、促进营养物质消化吸收、促进肠道上皮生长等作用[2, 3]。在人和动物组织内只存在能够代谢L-乳酸的L-乳酸脱氢酶[4],因此,动物机体只能利用L-乳酸,不能代谢D-乳酸。目前,益生菌作为饲用抗生素的替代品越来越受到大家的重视,已有许多益生菌被筛选出来,并被广泛应用于畜牧养殖业中。通过实际生产发现,乳酸菌虽对动物具有显著的益生作用,但其效果不稳定,且不耐高温、不易保存、对外界环境敏感,这些缺点限制了其在生产实践中的应用;而L-乳酸芽孢杆菌弥补了乳酸菌的不足,并且还能给动物机体提供可消化利用的L-乳酸,可以说它兼具了芽孢杆菌与乳酸菌的优点。现在已有多种益生菌被制成微生态制剂用于促进动物的生产性能,提高饲料的利用率,增强机体的抗病能力[5]。但未见产L-乳酸芽孢杆菌对肉鸡肠道菌群影响的报道。因此,本试验拟对已分离的一株产L-乳酸芽孢杆菌BLZ01进行鉴定及乳酸消旋类型的鉴别;然后再利用动物试验,采用聚合酶链式反应-变性梯度电泳技术(PCR-DGGE)技术观察该菌株对肉鸡肠道菌群的影响,为该菌的应用提供理论依据。
1 材料与方法 1.1 菌种芽孢杆菌BLZ01由四川农业大学动物医学院发酵工程实验室分离保存。
1.2 LB培养基蛋白胨10 g,氯化钠5 g,牛肉浸膏5 g,酵母膏5 g,葡萄糖15 g,硫酸镁0.2 g,蒸馏水1 000 mL,pH 7.0~7.5,121℃灭菌30 min。如制作平板,加入2%琼脂。
1.3 试剂和仪器设备DL2000 DNA Marker,50 bp DNA Ladder,宝生物工程(大连)有限公司产品;全血乳酸试剂盒(南京建成生工)。PCR仪,Dcode DGGE仪,BIO-RAD Laboratories(美国)。
1.4 菌种活化将保存的芽孢杆菌BLZ01放入80℃水浴锅中加热10 min,在LB培养基上划线活化,37℃培养36 h,观察菌落形态、革兰氏染色和镜检。
1.5 初步鉴定参考《伯杰氏细菌鉴定手册》中芽孢杆菌属的特性,对菌株BLZ01的菌落、细菌形态和生理生化特性进行鉴定,并用全血乳酸测试盒检测菌株BLZ01培养液中的乳酸消旋类型。
1.6 16S rRNA序列分析参照文献[6]报道方法进行。
1.7 乳酸芽孢杆菌BLZ01活菌粉剂的制备乳酸芽孢杆菌BLZ01通过菌种活化、浅盘固体培养、收集菌苔、加入粉碎并40目过筛的麸皮、干燥等制成乳酸芽孢杆菌BZL01活菌制剂,测定其活芽孢数量,调整含活芽孢数为2.0×109 CFU/g,4℃冰箱中保存备用。
1.8 动物分组及饲养1日龄的艾维茵肉鸡预喂至7日龄,选取健康的20羽,随机分成2组,即对照组和试验组,每组10只。对照组饲喂不含抗生素的基础饲粮,试验组饲喂基础饲粮+0.05 g/kg的乳酸芽孢杆菌BLZ01制剂,基础饲粮组成参照文献[7]。2组鸡分别养于不同的鸡笼内,自由采食,自由饮水。
1.9 PCR-DGGE分析肉鸡肠道菌群多样性肉鸡饲养到42日龄时,停料12 h,每组随机取5只,颈动脉放血处死,取肉鸡回肠和盲肠段,准确称取1 g肠道内容物,置于10 mL灭菌离心管内。按照张保卫等[8]的方法提取内容物中的总DNA,提取物定溶于20 μL TE溶液中。以等量的每组鸡肠道细菌DNA提取物进行混合,并以之为模板,然后按文献[9, 10]的方法进行DGGE电泳。将得到的DGGE指纹图谱用Quantity One软件进行指纹图谱分析和多样性分析。
2 结 果 2.1 菌落及细菌形态特征菌株BLZ01在培养基上培养24 h,菌落呈乳白色而有光泽,表面湿润,边缘整齐,中央隆起;在生长后期菌落表面有皱褶,表面干燥,无光泽。革兰氏阳性,显微镜下细胞呈直杆状,常以成对或链状排列,芽孢椭圆中生,液体培养时为需氧型。
2.2 生理生化特征参考《伯杰细菌鉴定手册》,乳酸芽孢杆菌BLZ01的生理生化特征见表1。根据菌落特征和显微镜下菌体的形态特征,结合生理生化鉴定结果和全血乳酸测试盒(南京建成生物工程研究所)对培养液中乳酸类型的鉴定结果,初步确定菌株BLZ01为一株产L-乳酸的巨大芽孢杆菌( Bacillus megaterium)。
| 表1 菌株BLZ01生理生化特征 Table 1 Physiological and biochemical property of strain BLZ01 |
PCR产物经琼脂糖凝胶电泳检测,扩增产物是一条大小约1.5 kb的特异性条带。将PCR产物回收并测序,测得16S rRNA序列长度为1 463 bp。将得到的16S rRNA序列提交到NCBI核酸数据库中进行BLAT在线分析,结果见表2。在NCBI获得的登录号为KP343685。
| 表2 菌株BLZ01 16S rRNA序列与GenBank中细菌菌株同源性比较 Table 2 Compare the 16S rRNA sequence of strain BLZ01 with strains in GenBank |
通过16S rRNA序列同源性比对,菌株BLZ01与GenBank中前100个序列相似性均为99%,其中确切命名为巨大芽孢杆菌的就有46条序列。同时,在GenBank里搜索芽孢杆菌科内9个不同属的16S rRNA序列(登录号可见进化树),应用ClustalX 1.81软件进行多重序列比对,再用TreeView和Phylodraw软件进行系统发育分析,构建无根进化树,结果见图1。
图1中除菌株BLZ01、Bacillus cereus(JQ806056)、Bacillus thurinqiensis(KF935650)及4株Bacillus megaterium之外,其他菌株分别为芽孢杆菌科内9种属的模式菌株,其中Bacillus cereus、Bacillus thurinqiensis、Bacillus subtili、Bacillus megaterium均属于芽孢杆菌属。从图1中可以看出,菌株BLZ01与4株Bacillus megaterium距离最近,且都在同一个分支中,说明它们的亲缘关系最近。再结合前面的菌落及细菌形态、生理生化特征以及相似性比较结果,最终确定菌株BLZ01是产L-乳酸的巨大芽孢杆菌(Bacillus megaterium)。
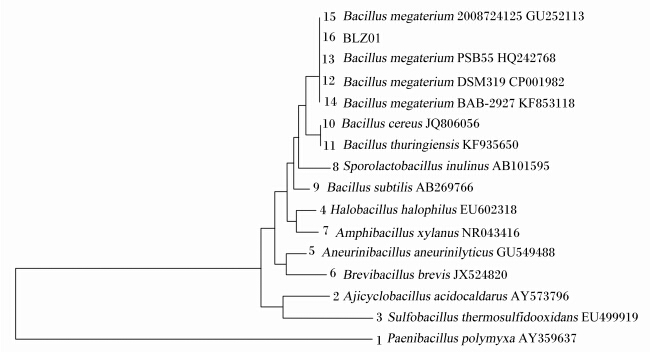 | 图1 BLZ01的系统进化树 Fig. 1 The phylogenetic tree of BLZ01 |
图2为DGGE指纹图谱,Marker为本实验室保存的条带数较多的肠道菌群样品,CI和CC分别为对照组的回肠和盲肠样品,TI和TC分别为试验组的回肠和盲肠样品。通过该图可以看出,对照组及试验组盲肠条带数均多于回肠,且亮条带也较多;肉鸡饲喂巨大芽孢杆菌BLZ01后,试验组的回肠、盲肠条带数均多于对照组,且都出现了特异性条带,如图2中的4号和9号条带;说明肉鸡饲喂巨大芽孢杆菌BLZ01后肠道菌群多样性及种群密度均有所增加。
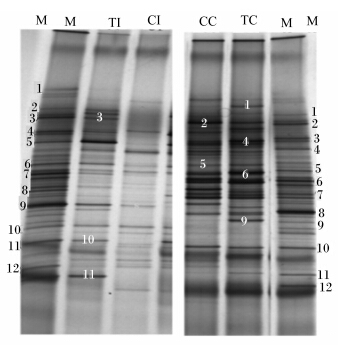 | M:分子质量标准 Marker; CI:对照组的回肠样品 Ileum sample in control group; CC:对照组的盲肠样品 Appendix sample in control group; TI:试验组的回肠样品 Ileum sample in experiment group; TC:试验组盲肠样品 Appendix sample in experiment group. 下图同。The same as below. 图2 肉鸡肠道菌群PCR-DGGE指纹图谱(泳道内数字为回收条带) Fig. 2 PCR-DGGE fingerprint of intestinal microflora of broilers (the marked bands had been recovered) |
利用Quantity One软件对图2的DGGE指纹图谱进行定量分析,得到泳道/条带识别图(图3)、相似性矩阵图(图4)和系统树(图5)。
 | 图3 泳道/条带识别图 Fig. 3 Compare lane images |
 | 图4 相似性矩阵图 Fig. 4 Similarity matrix of DGGE patterns |
图3中显示共有40类条带,22号、23号等9个条带在每个样品中均有出现,同时每个样品均有自己的特异性条带,10号、14号、37号和40号条带也被视为特征性条带,它们在某些样品中的泳道中较粗,均被回收、纯化和测序。通过图3可以看出每个样品的条带数都不相同,TI有32个条带,CI有22个条带,CC有32个条带,TC有35个条带。再根据条带比对结果,运用戴斯系数Cs法计算出各样品的相似性,并得出相似性矩阵图(图4)。样品间相似度最大的为59.6%(TI和CI),样品间相似度最小的为27.6%(TI和TC)。利用UPGMA(unweighted pair group method with arithmetic averages)算法进行聚类分析,并构建系统树(图5)。系统树图5显示4个样品可分为两大类,分别为TI和CI,CC和TC。
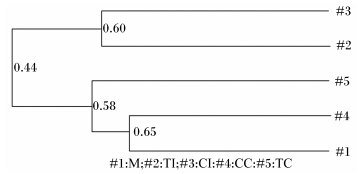 | 图5 系统树图 Fig. 5 Clustering analysis of DGGE patterns |
分别选取对照组和试验组的DGGE图谱上的7个共性条带和4个试验组的特异性条带,见图2,共11条特征性条带割胶回收,进行测序,在NCBI 中进行BLAST,结果见表3。回收的条带中,第2、3、4号均为乳杆菌;第6号条带为链球菌,也是乳酸菌类型;其余条带序列均为未培养的细菌。试验组条带种类与对照组相比,多出了第1、3、9、11号特异性条带,这说明试验组回肠、盲肠的菌群种类及数量发生了一定的变化。
3 讨 论 3.1 菌株BLZ01的鉴定结果NCBI数据库中,关于巨大芽孢杆菌的基因序列有很多,只有少数序列涵盖了巨大芽孢杆菌的完整基因,而不同菌种之间的相似性差异又很小,故仅通过16S rRNA基因序列相似性和系统进化树分析的说服力不强。传统的菌种鉴定包括菌落、菌体形态特征观察和生理生化反应特征,由于鉴定内容多,很多性状都是基于感官来判断的,主观干扰很容易被引入,给鉴定工作带来误差[11]。因此本试验先通过传统的细菌形态特观察和生理生化反应,结合培养液中乳酸类型的分析结果,可以初步判定菌株BLZ01为一株产L-乳酸的巨大芽孢杆菌。再通过16S rRNA基因序列相似性分析和构建系统发育树可以看出,该菌株在分类地位上的定位与巨大芽孢杆菌的形态特征、生理生化特征相符,且与其他4株巨大芽孢杆菌的亲缘关系最近,同源性达到了99%,且与NCBI中Bacillus megaterium DSM319相似性达到了99%,比对总分值达到了29 109。因此,本试验可以确定菌株BLZ01为一株产L-乳酸的巨大芽孢杆菌。
| 表3 DGGE图谱对应的单个条带DNA序列 Table 3 Sequences analysis of DNA recovered from single band in DGGE fingerprints |
动物肠道寄居着复杂的微生物,这些微生物具有菌群结构多样性的特性。目前,很多研究成果已证实肠道菌群结构和宿主健康之间存在紧密的联系[12],这种联系引起了人们对肠道微生物菌群如何构建以及它们如何影响宿主健康方面的强烈兴趣。由于许多因素影响鸡肠道菌群组成,包括饲喂含有益生菌的饲料,故本试验采用PCR-DGGE技术来分析肉鸡饲喂巨大芽孢杆菌BLZ01后,用Quantity One定量分析回肠、盲肠细菌菌群结构的变化及差异。
由于DGGE指纹图谱中的一个条带代表的是一种菌群,观察得到的指纹图谱,可以发现不同的条带在不同的样品中粗细度不一样,条带粗的,则对应的浓度较大,条带细的,则对应的浓度较小,因此不同菌群在对照组与试验组的不同肠道中的浓度存在差异,说明肉鸡饲喂巨大芽孢杆菌BLZ01可以引起肠道内菌群的浓度发生变化。运用Quantity One软件对DGGE指纹图谱进行泳道/条带分析发现,对照组回肠、盲肠与Marker的相似度分别为43.2%和64.8%,试验组回肠、盲肠与Marker的相似度分别为61.4%和60.5%,可见所有样品与Marker相似度均比较低,这说明对照组与试验组的不同肠道中的细菌群落结构差异比较大。但样品间也存在较多的共性条带,如图3中的22号、23号条带,说明对照组与试验组的不同肠道中也存在共同的菌群。将对照组和试验组的电泳条带进行聚类分析,两两对比可以发现,对照组回肠与试验组回肠的相似度最大,为59.6%;其次对照组盲肠与试验组盲肠相似度为55.6%;试验组回肠与盲肠的相似度最小,为27.6%。进一步再构建系统树,系统树中将对照组和试验组样品分为两大类,即回肠为一类,盲肠为一类,这与相似度分析结果一致。这就表示,肉鸡回肠、盲肠细菌菌群结构差别比较突出,即使饲喂益生菌巨大芽孢杆菌BLZ01也不能改变这种差异。这与王秋菊等[13]研究的关于雏鸡肠道菌群的PCR-DGGE分析结果相同。但是将相同肠道进行比较,回肠的相似度为59.6%,盲肠的相似度为55.6%,表明试验组的回肠、盲肠与对照组回肠、盲肠的菌群结构差异比较明显,这就证实巨大芽孢杆菌BLZ01具有改变肉鸡肠道内菌群组成结构的作用。同时,根据图谱可以发现,试验组的条带数均多于对照组,与吴小盼等[13]报道的在肉鸡饲粮中添加益生菌后,利用PCR-DGGE技术检测不同肠段菌群多样性的结果一致。且盲肠多于回肠,大部分盲肠条带比回肠条带较亮,与倪学勤[14]、Gong等[15]采用PCR-DGGE检测蛋鸡的肠道菌群DNA扩增结果相同。这就说明巨大芽孢杆菌BLZ01可以丰富肉鸡肠道菌群种类。
研究表明,随着鸡肠道成熟化,肠道菌群从短暂而单一的构成向复杂逐渐演化[16]。鸡回肠微生物菌群构建大概需要2周,而盲肠的构建大概需要6~7周,随后回肠和盲肠各自具有自己唯一的微生物菌群且是一种相对稳定的微生态系统[17]。故本试验选择了42日龄肉鸡,并对其DGGE分离的7个共性条带和4个试验组的特异性条带进行克隆测序。结果表明,肉鸡肠道内具有很多未培养出的细菌,已培养出的细菌是以乳杆菌属为主,说明巨大芽孢杆菌BLZ01引起了肉鸡肠道菌群结构的变化。
4 结 论① 肉鸡回肠、盲肠的肠道菌群存在较大差异,且以乳杆菌和未培养的细菌为主。
② 肉鸡饲喂芽孢杆菌BLZ01后,通过改变肠道内菌群浓度和丰富菌群种类的方式,达到了改变肉鸡肠道菌群结构的作用。因此我们认为,巨大芽孢杆菌BLZ01具有明显提高肉鸡肠道菌群的多样性和种群密度的特点。
| [1] | GERRITSEN J,SMIDT H,RIJKERS G T,et al.Intestinal microbiota in human health and disease:the impact of probiotics[J]. Genes and Nutrition,2011,6(3):209-240. ( 1) 1)
|
| [2] | SAMUEL BS,SHAITO A,MOTOIKE T,et al.Effects of the gut microbiota on host adiposity are modulated by the short-chain fatty-acid binding G protein-coupled receptor,Gpr41[J]. Proceedings of the National Academy of Sciences of the United States of America,2008,105(43):16767-16772. ( 1) 1)
|
| [3] | CERF-BENSUSSAN N,GABORIAU-ROUTHIAU V.The immune system and the gut microbiota:friends or foes?[J]. Nature Reviews Immunology,2010,10(10):735-744. ( 1) 1)
|
| [4] | 高江婧.产L-乳酸菌种的选育和发酵条件的研究[D]. 硕士学位论文.无锡:江南大学,2009:31-32. ( 1) 1)
|
| [5] | 杨汉博,潘康成.不同剂量益生芽孢杆菌对肉鸡免疫功能的影响[J]. 兽药与饲料添加剂,2003,8(4):8-10. ( 1) 1)
|
| [6] | 潘康成,冯兴,崔恒敏,等.利用16S rDNA序列对两种芽孢杆菌的鉴定[J]. 中国兽医科学,2009,39(6):550-554. ( 1) 1)
|
| [7] | 唐小波.产植酸酶芽孢杆菌的筛选及对肉鸡生长性能的影响[D]. 硕士学位论文.雅安:四川农业大学,2013:7-8. ( 1) 1)
|
| [8] | 张保卫,魏辅文,李明,等.大熊猫和小熊猫粪便DNA提取的简易方法[J]. 动物学报,2004,50(3):452-458. ( 1) 1)
|
| [9] | 佘跃惠,张凡,向延生,等.PCR-DGGE方法分析原油储层微生物群落结构及种群多样性[J]. 生态学报,2005,25(2):237-242. ( 1) 1)
|
| [10] | 潘康成,陈正礼,崔恒敏,等.利用ERIC-PCR和PCR-DGGE技术分析喂服枯草芽孢杆菌肉鸡肠道菌群的多样性[J]. 动物营养学报,2010,22(4):985-991. ( 1) 1)
|
| [11] | 潘康成,高传庆,张均利,等.三株益生芽孢杆菌的16S rDNA测序鉴定[J]. 饲料工业,2010,31(8):24-27. ( 1) 1)
|
| [12] | 王秋菊,崔一喆,刘胜军,等.笼养海兰褐蛋雏鸡肠道菌群的聚合酶链式反应-变性梯度凝胶电泳分析[J]. 动物营养学报,2014,26(2):504-512. ( 1) 1)
|
| [13] | 吴晓盼,朱年华,张日俊.日粮添加益生菌和抗生素对肉鸡不同肠道菌群多样性的研究[C]//中国畜牧兽医学会动物微生态学分会第四届第九次学术研讨会暨饲料和动物源食品安全战略论坛论文集.新乡:中国畜牧兽医学会动物微生态学分,2008:469-474. ( 2) 2)
|
| [14] | 倪学勤,GONG J,YU H,等.采用PCR-DGGE技术分析蛋鸡肠道细菌种群结构及多样性[J]. 畜牧兽医学报,2008,39(7):955-961. ( 1) 1)
|
| [15] | GONG J H,SI W D,FORSTER R J,et al.16S rRNA gene-based analysis of mucosa-associated bacterial community and phylogeny in the chicken gastrointestinal tracts:from crops to ceca[J]. FEMS Microbiology Ecology,2007,59(1):147-157. ( 1) 1)
|
| [16] | ZHU X Y,ZHONG T Y,PANDYA Y,et al.16S rRNA-based analysis of microbiota from the cecum of broiler chickens[J]. Applied and Environmental Microbiology,2002,68(1):124-137. ( 1) 1)
|
| [17] | 王丽凤,张家超,马晨,等.鸡肠道微生物研究进展[J]. 动物营养学报,2013,25(3):494-502. ( 1) 1)
|




