长期以来,使用抗生素一直作为预防和治疗仔猪腹泻、促进生长的最主要手段,但使用抗生素带来的负面影响及禁用抗生素的呼声高涨使得养猪生产上不得不选择替代抗生素发挥抑制病原菌,促进仔猪生长,且无毒、无害、无残留的添加剂,而益生菌制剂由于具备上述特点而成为当前研究的热点。乳杆菌是世界上公认安全的、最具潜力代替抗生素添加于饲料中的有益菌类,已在畜禽养殖生产上广泛应用,但由于其种属繁多,特性各异,添加时间、剂量等不同,导致使用效果参差不齐,究其原因是乳杆菌在肠道中发挥其作用的机制较为复杂,尤其是乳杆菌与宿主肠道上皮细胞(IECs)的相互作用及其对肠道黏膜免疫系统的调节机制仍需加以深入研究。本文结合仔猪肠道的生理特点及乳杆菌的特性,综述乳杆菌调节仔猪肠道黏膜免疫系统的分子机制,为今后仔猪肠道黏膜免疫系统的调节的研究提供依据。
1 乳杆菌在仔猪肠道中发挥的作用仔猪肠道免疫系统的发育与肠道微生态系统发育相辅相成,相互促进,维持仔猪肠道健康。乳杆菌作为肠道内重要的益生菌,对肠道健康发挥有益作用的一个机制是促进肠道非免疫防御屏障,表现为调节肠道自身微生物丛的平衡;另一个机制是改善肠道免疫屏障功能[1]。Ahrne等[2]认为,乳杆菌通过增加黏蛋白分泌、调节免疫、抑制病原菌以及影响整个微生物丛,从而促进肠道屏障功能完整性。Konstantinov等[3]发现饲喂乳杆菌L. sobrius可减少仔猪肠中大肠杆菌F4的数量,维持肠道菌群平衡,促进受感染仔猪生长。Zhang等[4]应用肠毒性大肠埃希杆菌(ETEC)K88诱导的仔猪腹泻模型研究发现,鼠李糖乳杆菌(LGG)有效减缓了ETEC K88诱导的断奶仔猪腹泻发生,并认为其可能的机制是通过调控肠道微生物菌群、增强肠道抗体防御蛋白[分泌型免疫球蛋白A(sIgA)]以及调节系统性炎症细胞因子[白细胞介素-6(IL-6)、肿瘤坏死因子-α(TNF-α)和白细胞介素-1β(IL-1β)]的生成。国内外大量试验也证明了乳杆菌对仔猪肠道健康有重要的免疫调控作用。但到目前为止,其作用机制仍不十分明确,大大限制了乳杆菌在仔猪生产上的应用。突出的问题主要表现在:乳杆菌与IECs、抗原递呈细胞[如树突状细胞(DCs)]和免疫相关淋巴细胞之间的相互识别机制不清楚。由于乳杆菌作用于肠道,其在肠道中的识别、黏附能力往往作为判定乳杆菌发挥免疫调节功能的一个重要前提[5]。因此,仔猪IECs受体对乳杆菌的识别作用及其介导的免疫调控机制引起了人们的关注,成为近年来研究的热点。
2 乳杆菌与仔猪肠道细胞相互识别机制仔猪肠道黏膜免疫系统是防御细菌和病毒感染的第1道防线,IECs、DCs通过模式受体(PRRs)识别抗原,是识别肠道微生物的2种主要途径,在维持肠道正常生理功能及防止病原菌入侵等方面起重要作用。Toll样受体(TLRs)是目前发现的最重要PRRs之一,在先天免疫系统识别微生物过程中发挥关键作用。TLRs信号通过髓样分化因子88(MyD88)依赖途径或非依赖途径[β干扰素TIR结构域衔接蛋白(TRIF)途径]2条主要途径诱导炎症因子的表达。除Toll样受体3(TLR3)外,所有TLRs均利用MyD88依赖途径,在MyD88募集白细胞介素-1(IL-1)受体相关激酶(IRAKs)、活化肿瘤坏死因子受体相关因子6(TRAF6)之后,通过TRAF6-核转录因子-κB(NF-κB)抑制剂(IκB)激酶(IKK)-NF-κB、TRAF6-丝裂原活化蛋白激酶(MAPKs)或TRAF6-干扰调节因子5(IRF5)等信号通路调控炎症细胞因子的表达。TLR3和Toll样受体4(TLR4)还可通过MyD88非依赖途径,通过TLRs-TRIF-MAPKs或TLRs-TRIF-NF-κB等信号通路诱导炎症因子的表达。TLRs的激活可诱导很强的免疫反应,有利于机体抵抗病原体感染或组织损伤,但是过度的免疫反应也会带来不利影响,如产生内毒素休克、自身免疫性疾病等[6]。
仔猪肠道黏膜系统也是乳杆菌发挥作用的关键部位,乳杆菌表面的微生物相关分子模式(MAMPs)有肽聚糖(PG)、多糖、胞壁磷壁酸(WTA)和脂磷壁酸(LTA)等[7]。
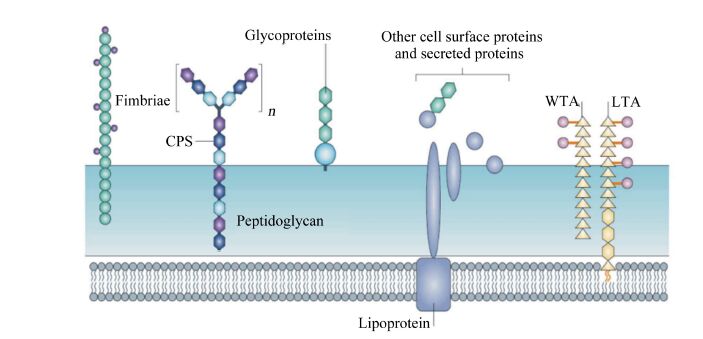 | Fimbriae:菌毛;Peptidoglycan:肽聚糖;Glycoproteins:糖蛋白;WTA:胞壁磷壁酸 wall teichoic acids;LTA:脂磷壁酸 lipoteichoic acid;Other cell surface proteins and secreted proteins:其他细胞表面蛋白及分泌蛋白;Lipoprotein:脂蛋白;CPS:细胞壁相关多糖 cell wall associated polysaccharide。
图1 乳杆菌表面分子模式
Fig. 1 Surface molecular pattern of Lactobacillus[7] |
这些分子模式可被许多免疫细胞表面的TLRs识别。如PG主要被Toll样受体2(TLR2)识别[8],多糖主要被TLR2及Toll样受体6(TLR6)所识别[9],LTA主要被TLR2所识别[10]。研究发现,来自约氏乳杆菌、植物乳杆菌和金黄色葡萄球菌的PG通过与TLR2受体结合,抑制了干酪乳杆菌诱导细胞因子白细胞介素-12(IL-12)的生成[11],间接证明PG通过TLR2受体发挥免疫调节作用。近年来TLRs尤其是TLR2的研究是国际上免疫研究中的热点,它与TLR4的配体己经涵盖了自然界绝大多数对人体致病的微生物类别,因此,二者在TLRs家族中最为重要。产肠毒素大肠杆菌属于革兰氏阴性菌,与宿主细胞的识别主要依赖于TLR4激活MyD88依赖性NF-κB信号通路或MAPKs信号通路[12]。乳杆菌与宿主细胞的识别主要依赖于TLR2,并提高TLR2基因表达,但是TLR2发挥作用依赖于与Toll样受体1(TLR1)或TLR6结合。TLR2被激活后还激活蛋白激酶C(PKC),引起紧密连接蛋白ZO-l向细胞顶部紧密连接处转移,从而增强肠道黏膜屏障的完整性[13]。此外,乳杆菌诱导产生的锌指蛋白A20、B细胞淋巴瘤蛋白3(Bcl-3)和丝裂原活化蛋白激酶-1(MAPK-1)也有助于抑制NF-κB活性[12]。也有报道表明,乳杆菌对NF-κB有较弱的激活作用,这种弱的激活作用并未引起炎症因子[TNF-α、IL-6和白细胞介素-8(IL-8)]的释放,而是降低了IL-8和IL-6的水平,并通过激活抗凋亡的蛋白激酶B(PKB),抑制TNF-α、IL-1和干扰素(IFN)对促凋亡的p38 MAPKs的活化,从而抑制炎症的发生[14]。由此推测,乳杆菌可能通过以TLR2为主的受体介导抑制MAPKs途径或者抑制NF-κB转录后的基因调控。但也有研究表明,乳杆菌与TLR2结合,刺激产生前炎症因子如IL-6、IL-12等,发挥较弱的促炎作用。推测这与乳杆菌的种类及宿主细胞的识别状态有关。不同的乳杆菌细胞壁表面LTA结构不同,也会导致乳杆菌发挥的作用不同。Grangette等[15]通过改变具有促炎作用的1株乳杆菌菌株细胞壁表面LTA分子结构,改变后的乳杆菌具有明显抗炎作用。上述结果说明不同乳杆菌发挥作用不同与本身的分子结构不同有关。对于乳杆菌与TLR2之间的相互作用,在仔猪肠道中也得到证实[16]。
除了TLRs之外,还有NOD样受体(NLRs)。NLRs是胞质受体,配体需进入细胞质才能与之相结合,激活下游信号通路。典型的NLRs主要有Ⅱ类分子反式激活因子(CⅡTA)、神经元凋亡抑制蛋白(NALP)、白介素转化酶激活因子(IPAF)、神经元凋亡抑制蛋白(NAIP)、NOD1、NOD2等。研究最详细的是NOD1和NOD2,它们主要识别细菌表面PG成分,如胞壁酰二肽(MDP)和二氨基庚二酸(DAP)(NLRs的2个常见配体)。NOD1在肠道上皮细胞表达,NOD2只在单核细胞和巨噬细胞及肠道潘氏细胞表达。这些受体与细胞表面配体结合后激活NF-κB和MAPKs通路。目前NLRs与肽聚糖序列结合的模式还不清楚,尚未证实NLRs与PG序列是直接结合还是间接结合。有关细胞壁表面PG成分是如何进入细胞质内与NLRs结合的,仍有待进一步研究。
3 乳杆菌调控仔猪IECs的免疫分子机制研究表明,乳杆菌被仔猪IECs识别后,不会对宿主细胞产生毒性作用,而是促进抗炎因子和分泌型抗体的产生,而且宿主IECs识别乳杆菌后会将相应的信号传递给其他的免疫细胞,进而影响整个免疫系统的功能。DCs是抗原提呈能力最强的肠道相关免疫细胞,位于上皮层DCs的突触,可延长至肠腔内捕获肠腔中的抗原,并把信号传递给邻近淋巴结组织中的初始T细胞,调节适应性免疫应答。因此,只有黏附的乳杆菌才能增加与仔猪DCs细胞相互作用的强度,这对提高肠道内稳态结构具有重要意义。
NF-κB信号通路在大肠杆菌诱导的炎症反应中异常活跃。NF-κB不仅是激活免疫反应的关键信号传导通路,而且也是乳杆菌与IECs作用抑制炎症信号通路重点作用对象。NF-κB的许多组成元件的活化可被乳杆菌阻断并影响下游细胞因子的分泌。Zhang等[4]在仔猪IECs中研究活性或热灭活的鼠李糖乳杆菌时发现乳杆菌能减少IκB降解,进而抑制NF-κB转位进入细胞核内,导致IL-8表达水平下降。乳杆菌是通过什么途径降解IκB从而抑制NF-κB信号途径的,目前未见详细研究报道。
DCs在益生菌发挥免疫调控功能、抑制炎症过程中起关键作用。DCs表面也有许多TLRs,目前还不清楚DCs与乳杆菌作用的信号途径与IECs是否一致,DCs与IECs之间的互作机制也少见报道。近年来,DCs的免疫调节功能集中在诱导T细胞的分化方面,乳杆菌通过DCs表面受体传导信号,一方面介导CD4+T细胞向CD4+Foxp3+调节性T细胞(Treg)分化,并在转录因子Foxp3的特异性调节下诱导产生转化生长因子-β(TGF-β)、IL-4和白细胞介素-10(IL-10),刺激机体产生口服耐受;另一方面促进Treg分泌TGF-β,进一步促进肠道sIgA的产生,有助于肠道黏膜免疫系统的建立[17]。此外,乳杆菌与受体结合后还会分泌一些蛋白,如细胞因子信号传导抑制因子3(SOCS3)蛋白,能抑制辅助性T细胞17(Th17)的分化,促进Treg的发育,抑制p38 MAPKs激活的关键环节;而细胞因子信号传导抑制因子1(SOCS1)蛋白破坏p65/RelA蛋白的稳定性,从而抑制了NF-κB的信号通路[18],也抑制连接蛋白/信号转导子和转录激活子(JAK/STAT)信号通路和IRAKs活性。尽管人们已经清楚乳杆菌诱导DCs产生一些细胞因子抑制炎症的产生,但是乳杆菌与DCs之间的相互识别并发挥免疫调节作用的途径不清楚,DCs如何识别致病性和非致病性的抗原也不清楚,需要进一步研究。
4 乳杆菌通过自身代谢产物发挥免疫调控作用的机理乳杆菌被仔猪IECs受体识别后,不仅释放抗炎因子以提高宿主的免疫功能,改善肠道健康状况,还能分泌乳酸,而乳酸又进一步促进了乳杆菌在仔猪肠道中的定植。乳杆菌通过分泌乳酸来螯合金属离子等以及改变革兰氏阳性菌细胞膜通透性来发挥作用,乳酸还能降低肠道pH,抑制大肠杆菌、梭菌等生长,例如,由于乳酸的积累,鼠李糖乳杆菌GG对鼠伤寒沙门氏菌有很强的抗菌活性;干酪乳杆菌、嗜酸乳杆菌YIT0070和短乳杆菌YIT0076产生的非解离乳酸能改变革兰氏阳性菌细胞膜通透性,从而有效地抑制大肠杆菌生长;加氏乳杆菌CRLl421产生的乳酸能使金黄色葡萄球菌细胞膜瓦解,使其细胞内容物渗出,从而对其繁殖起到抑制作用。此外,乳杆菌通过仔猪肠道膜识别受体感受环境的变化并作出适应性调整,通过乳酸介导,乳杆菌充分利用碳水化合物进行生长繁殖及黏附[19],更好地发挥其免疫调控作用,其作用机制的研究对设计营养配方提高仔猪肠道免疫、保持仔猪肠道健康具有重要指导作用。但是Paszti-Gere等[20]研究表明,乳杆菌对仔猪IECs的保护作用及对炎症因子的抑制作用不是通过分泌的乳酸发挥作用,可能是通过乳杆菌肽发挥作用。此外,乳杆菌还产生另一类抑制物质细菌素-乳链球菌素(Nisin),它具有广谱的抑制范围,如可以抑制链球菌、葡萄球菌、梭状芽孢杆菌等致病菌的繁殖。
研究表明,乳杆菌菌体及代谢物可以抑制细胞TLRs的表达,进而影响TLRs-NF-κB信号通路[21],但是具体的作用机制还有待进一步研究。Menard等[22]研究表明,乳杆菌代谢产物可能会干扰脂多糖(LPS)信号传导,同时也会阻碍LPS与CD14受体的结合,导致NF-κB活化水平下降。例如,发酵乳杆菌DSMZ 20052的分泌物通过抑制NF-κB活性降低小肠结肠炎;干酪乳杆菌可通过增加IL-10的水平抑制促炎症反应。
5 小 结乳杆菌通过其本身的黏附作用、细胞壁多种成分及其代谢物调控宿主肠道黏膜免疫系统,对改善仔猪肠道健康具有十分重要的作用,但目前的研究多集中在体外细胞培养方面,动物试验还相对较少,对其分子机制的探讨也限于已知的信号通路的变化,不同的动物试验研究的结果也有很多的不确定性,需要从基因、蛋白质网络或者代谢组学的角度进一步研究。乳杆菌被作为益生菌添加剂广泛用于食品、饲料和医药行业,被认为是安全的食品级微生物。目前我国没有专门制定关于乳杆菌安全性评价的法规标准。为了更加安全有效使用乳杆菌,相关部门应制定相关安全评价标准,有利于乳杆菌在生产中的推广与应用。
| [1] | ZHANG J,DENG J,WANG Z,et al.Modulatory effects of Lactobacillus salivarius on intestinal mucosal immunity of piglets[J]. Current Microbiology,2011,62(5):1623-1631. ( 1) 1)
|
| [2] | AHRNE S,HAGSLATT M L.Effect of Lactobacilli on paracellular permeability in the gut [J]. Nutrients,2011,3(1):104-117. ( 1) 1)
|
| [3] | KONSTANTINOV S R,SMIDT H,MENGHERI E.Commensal microbiota is required for the normal development and function of the porcine host immune system and physiology[J]. Nutrition and Immunity,2006(1):23-38. ( 1) 1)
|
| [4] | ZHANG L,XU Y Q,LIU H Y,et al.Evaluation of Lactobacillus rhamnosus GG using an Escherichia coli K88 model of piglet diarrhoea:effects on diarrhoea incidence,faecal microflora and immune responses[J]. Veterinary Microbiology,2010,141(1/2):142-148. ( 2) 2)
|
| [5] | KILIÇ G B,KULEASAN H,SÖMER V F,et al.Determining potential probiotic properties of human originated Lactobacillus plantarum strains[J]. Biotechnology and Bioprocess Engineering,2013,18(3):479-485.10 ( 1) 1)
|
| [6] | KAWAI T,AKIRA S.The role of pattern-recognition receptors in innate immunity:update on Toll-like receptors[J]. Nature Immunology,2010,11(5):373-384. ( 1) 1)
|
| [7] | LEBEER S,VANDERLEYDEN J,DE KEERSMAECKER S C.Host interactions of probiotic bacterial surface molecules:comparison with commensals and pathogens[J]. Nature Reviews Microbiology,2010,8(3):171-184. ( 1) 1)
|
| [8] | SASAKI T,FUKAMI S,NAMIOKA S.Enhanced resistance of mice to Eschefichia coli infection induced by administration of peptidoglycan derived from Bifidobacterium thermophilum[J]. Journal of Veterinary Medical Science,1994,56(3):433-437. ( 1) 1)
|
| [9] | NAGAOKA M,HASHIMOTO S,WATANABE T,et a1.Anti-ulcer effects of lactic acid bacteria and their cell wall polysaccharides[J]. Biological & Pharmaceutical Bulletin,1994,17(8):1012-1017. ( 1) 1)
|
| [10] | MATSUGUCHI T,TAKAGI A,MATSUZAKI T,et a1.Lipoteichoicacids from Lactobacillus strains elicit strongtumor necrosis factoralpha-indacingactivities inmacrophages through Toll-like receptor 2[J]. Clinical and Diagnostic Laboratory Immunology,2003,10(2):259-266. ( 1) 1)
|
| [11] | SHIDA K,KIYOSHIMA-SHIBATA J,KAJI R,et a1.Peptidoglycanfrom Lactobacilli inhibits interleukin 12 production by macrophagesinducedby Lactobacillus casei through Toll-like receptor 2 dependent and inde-pendent mechanisms [J]. Immunology,2009,128:858-869. ( 1) 1)
|
| [12] | SHIMAZU T.Immunobiotic Lactobacillus jensenii elicit anti-inflammatory activity in porcine intestinal epithelial cells by modulating negative regulators of the Toll-like receptor signaling pathway[J]. Infection and Immunity,2012,80:276-288. ( 2) 2)
|
| [13] | CARIO E,GERKEN G,PODOLSKY D K,et al.Toll-like receptor 2 enhances ZO-1 assoeiated intestinal epithelial barrier integrity via protein kinase C [J]. Gastroenterology,2004,127(1):224-238. ( 1) 1)
|
| [14] | KARLSSON M,SCHERBAK N,REID G,et al.Lactobacillus rhamnosus GR-1 enhances NF-kappaB activation in Escherichia coli-stimulated urinary bladder cells through TLR4[J]. BMC Microbiology,2012,12:15. ( 1) 1)
|
| [15] | GRANGETTE C,NUTTEN S,PALUMBO E,et al.Enhanced antiinflammatory capacity of a Lactobacillus plantarum mutant synthesizing modified teichoic acids[J]. Proceedings of the National Academy of Sciences of the United States of America,2005,102(29):10321-10326. ( 1) 1)
|
| [16] | 杨宽民.植物乳杆菌对感染大肠杆菌K88幼龄仔猪肠道健康的影响[D]. 博士学位论文.中山:中山大学,2013. ( 1) 1)
|
| [17] | SHAH M M,SAIO M,YAMASHITA H,et al.Lactobacillus acidophilus strain L-92 induces CD4+CD25+Foxp3+regulatory T cells and suppresses allergic contact dermatitis[J]. Biological and Pharmaceutical Bulletin,2012,35(4):612-616. ( 1) 1)
|
| [18] | RYO A,SUIZU F,YOSHIDA Y,et al.Regulation of NF-κB signaling by Pin1-dependent prolyl isomerization and ubiquitin-mediated proteolysis of p65/RelA[J]. Molecular Cell,2003,12:1413-1426. ( 1) 1)
|
| [19] | DALY K,DARBY A C,HALL N,et al.Dietary supplementation with lactose or artificial sweetener enhances swine gut Lactobacillus population abundance[J]. British Journal of Nutrition,2014,2:1-6. ( 1) 1)
|
| [20] | PASZTI-GERE E,SZEKER K,CSIBRIK-NEMETH E,et al.Metabolites of Lactobacillus plantarum 2142 prevent oxidative stress-induced overexpression of proinflammatory cytokines in IPEC-J2 cell line[J]. Inflammation,2012,35(4):1487-99. ( 1) 1)
|
| [21] | CHON H,CHOI B,JEONG G,et al.Suppression of proinflammatory cytokine production by specific metabolites of Lactobacillus plantarum 10hk2 via inhibiting NF-kappaB and p38 MAPK expressions[J]. Comparative Immunology,Microbiology and Infectious Diseases,2010,33(6):e41-49. ( 1) 1)
|
| [22] | MENARD S,CANDALH C,BAMBOU J C,et al.Lactic acid bacteria secrete metabolites retaining anti-inflammatory properties after intestinal transport[J]. Gut,2004,53(6):821-828. ( 1) 1)
|




