良好的胃肠道功能对于维持反刍动物的健康、保证生产潜能的发挥至关重要。微生物群落是维系胃肠道正常功能的一个重要组分,反刍动物胃肠道的微生态平衡与宿主的健康[1]和营养物质的消化吸收[2]息息相关,特别是瘤胃微生态系统可以帮助宿主消化饲粮中纤维、淀粉、蛋白质等养分。因此,人们常通过检测瘤胃微生物的结构与组成评估宿主的健康和营养状态。目前获取瘤胃内容物样品主要采用胃管采集、瘤胃穿刺、安装瘤胃瘘管等方法,对于放养的反刍动物此类方法显然不适用。此外,这些方法会给动物造成一定的生理伤害,且安装瘘管成本较高,同时这些方法受到动物福利相关规定的限制。而粪便样品易采集,有研究表明无论用瘤胃内容物还是新鲜粪便进行体外发酵试验,各种饲料的消化率与其在体内的消化率都呈正相关[3],这也说明研究粪便微生物具有一定意义,而用粪便代替瘤胃内容物来测定饲料消化率的可行性很大程度上取决于微生物群落。关于反刍动物粪便和瘤胃微生物群落的研究报道相对较多,但以往这些研究主要采用的是传统的培养技术[4]以及以克隆/测序为主要手段的分子生物学技术[5]。培养技术仅能研究体外可生长的微生物[6],这部分微生物仅占胃肠道微生物的1%左右[7],那些能在培养基表面生长的微生物,其重要性会被过高估计,且克隆/测序技术费时费力分辨率低[8]。高通量测序(high-throughput sequencing)技术是一种研究微生物生态学的全新技术手段,能全面地反应样品微生物的结构与组成[8]。目前,应用高通量测序技术研究反刍动物瘤胃和粪便微生物的报道主要是有关牛的[9, 10, 11],缺乏有关山羊瘤胃和粪便微生物的研究。因此,本试验旨在应用高通量测序技术研究山羊瘤胃和粪便微生物结构与组成,并对比二者之间的差异。
1 材料与方法 1.1 试验动物和饲粮试验动物为6只健康已阉割的波尔山羊,均为10月龄,平均体重为(20.00±1.51) kg。饲粮精粗比为3 ∶ 7,单笼饲养,自由饮水,每日定时饲喂2次(10:00和16:00),饲喂量按体重4%供给,饲粮 组成及营养水平见表1。饲粮营养水平测定参考 张丽英[12]的方法进行。
1.2 样品采集与试验设计稳定饲喂饲粮14 d后,在第15天晨饲前采集每只羊的瘤胃液和粪便样品,分记作R组和F组,每组6个重复,每个样品为1个重复。瘤胃液采集方法:将10 mm直径塑料管连接到一个真空泵,用开口器打开羊的口腔,将塑料管从羊口腔缓慢插入,直至瘤胃,抽取瘤胃内容物约50 mL装入充满氮气的样品袋中,置于冰上。反复拍打样品袋以确保固相微生物充分进入液相,然后用4层纱布过滤得到瘤胃液,编号分装后迅速投入液氮中保存;粪便采集方法:手戴一次性无菌手套,将食指伸入肛门,在直肠处掏取直肠粪便约20 g,编号分装后迅速投入液氮中保存。所有样品立即带回实验室转移至-80 ℃冰箱保存,备用。
| 表 1 饲粮组成及营养水平(风干基础) Table 1 Composition and nutrient levels of the diet (air-dry basis) |
取200 mg粪便或瘤胃液样品于5 mL离心管中,加入1 mL ASL缓冲液,振荡混匀,用QIAampH DNA提取试剂盒(Qiagen,德国)提取样品总DNA,具体操作步骤参照试剂盒说明书进行。为提高DNA获得率,在步骤1中需加入0.5 g皓微珠。提取的总DNA于-20 ℃保存,备用。
1.4 16S rRNA的PCR扩增和Illumina MiSeq测序以样品中菌群总DNA为模板,用古菌/细菌通用引物,针对细菌16S rRNA的V4~V5区域进行PCR扩增。引物为:515F(5′-GTGYCAGCMGCCGCGGTAA-3′)和806R (5′-GGACTACCAGGGTATCTAAT-3′)[13]。PCR采用20 μL反应体系,包括0.4 μL FastPfu Polymerase(北京全式金生物技术有限公司),4 μL 5×FastPfu Buffer,引物浓度均为5 μmol/L,各0.4 μL,2.5 mmol/L dNTP 2 μL,总DNA 10 ng,补ddH2O至20 μL。PCR过程在ABI Gene Amp 9700上进行,反应条件为:95 ℃预变性2 min;随后变性循环30次(95 ℃,30 s;55 ℃,30 s;72 ℃,30 s为1个循环);72 ℃延伸5 min,10 ℃冷却。PCR产物用含溴化乙锭的2%琼脂糖凝胶电泳鉴定并用PCR纯化试剂盒(Qiagen,德国)纯化和回收,回收的PCR产物用QuantiFlourTM-ST fluorometer[普洛麦格(北京)生物技术有限公司)]定量,并按测序要求进行混合。然后送至上海美吉生物医药科技有限公司,利用Illumina MiSeq测序平台进行高通量测序。
1.5 数据分析原始数据以Fastq文件格式保存,首先利用QIIME 1.8.0软件[14]对原始数据进行初次质控,去除序列平均碱基质量值<Q20、未知碱基数>6及同聚物太大的序列,只保留240~500 bp长度的序列。去除Barcode和引物序列,然后在Mothur[15]软件中拼接质控后的序列;再在QIIME软件中,使用Uclust法[16]将序列按照97%相似性进行聚类运算分类单位(operational taxonomic units,OTU),并挑选每个OTU中丰度最高的序列作为代表序列。继而采用PyNAST法[17],将所有代表序列与Greengenes参考数据库(http://greengenes.lbl.gov/)进行序列比对,并使用RDP分类器[18]对代表序列从门到属进行物种注释,并构建OTU表。继而分别剔除rep_set.fasta、OTU table和rep_set.tree中的嵌合体和Singletons。对各个样品进行α多样性分析,计算样品间Shannon-Wiener指数、ChaoⅠ指数、The_observed_species及PD_whole_tree指数,并进行差异显著性分析(采用SPSS 18.0软件进行配对t检验)。结合各个样品的OTU种类及其丰度进行计算,获得样品间Unweighted Unifrac距离矩阵,并利用非加权组平均法(UPGMA)进行聚类分析,然后用MEGA5.0软件绘制PCoA聚类图。组间各菌群相对丰度差异显著性分析采用SPSS 18.0软件进行配对t检验,结果以平均值±标准差表示。
2 结果与分析 2.1 样品测序深度和α多样性分析本次试验共获得227 729条有效序列(clean data),平均每个样品含有(18 977±5 569)条序列。基于相似度大于97%的原则,将获得的有效序列进行OTU聚类,共获得13 601个OTU,其中F组8 382个,R组5 777个,2组间共享的OTU为558个(图1)。不考虑处理效应,每个样品OTU平均值为(1 927±735)个,其中F组OTU平均值为(2 197±862)个,R组OUT平均值为(1 658±520)个,组间差异不显著(P>0.05)。
样品的稀释曲线见图2。由图2可见,在本试验的测序深度下,除了样品F3、F4,其余各样品稀疏曲线最终均趋于平缓,说明本试验的测序量可以覆盖各样品的大多数微生物。
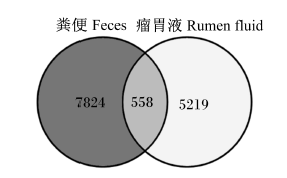 | 图1 OTU维恩图
Fig. 1 OTU venn diagram |
在取样深度为10 200时,比较粪便和瘤胃的α多样性指数可知,ChaoⅠ、The_observed_species、PD_whole_tree和Shannon-Wiener指数组间差异均不显著(P>0.05)(表2)。
2.2 不同分类水平上的物种组成分析将本试验所得有效序列在不同分类水平上进行物种注释和统计,结果表明,在界水平上,古菌占0.220%,未分类微生物占0.443%,而细菌(bacteria)的相对丰度最高,达到99.337%。各样品在门水平上的物种组成见图3,2组样品在古菌界下均只检测到一个门——广古菌门(Euryarchaeota),F组广古菌门的相对丰度为(0.130±0.033)%,而R组为(0.309±0.143)%。在细菌界中,F组共检测到17个门,而R组为21个门。其中,F组相对丰度最高的4个优势菌门从高到低依次分别为:厚壁菌门[Firmicutes,(65.400±8.090)%]、拟杆菌门[Bacteroidetes,(26.620±5.303)%]、变形菌门[Proteobacteria,(3.047±2.103)%]、未分类的细菌[(1.228±0.549)%],而R组为拟杆菌门[(60.188±4.864)%]、厚壁菌门[(21.674±5.118)%]、变形菌门[(5.827±2.312)%]和未分类的细菌[(4.649±3.385)%]。
在科水平上对比粪便和瘤胃液的物种组成,从所有12个样品中共检测到129个科,其中F组100个,R组105个。组间相对丰度差异达显著水平(P<0.05)的共有22个科,差异极显著(P<0.01)的有10个科,详见表3。组间差异显著或极显著的科中,F组相对丰度最高的科为厚壁菌门下的瘤胃球菌科(Ruminococcaceae),其相对丰度高达37.705%,极显著高于R组(P<0.01),其次为厚壁菌门下的毛螺旋菌科(Lachnospiraceae,13.802%),显著高于R组(P<0.05);R组中,相对丰度最高的为拟杆菌门下的普雷沃氏菌科(Prevotellaceae,29.959%),极显著高于F组(P<0.01),其次为拟杆菌门下未分类的拟杆菌门(11.375%),显著高于F组(P<0.05)。
 | 图2 样品稀释曲线
Fig. 2 Rarefaction curves of samples |
所有样品共检测到258个属,表4分别列出F组和R组相对丰度最高的20个属。其中F组有12个属于厚壁菌门,5个属于拟杆菌门,属于变形菌门、未分类细菌、纤维杆菌门的各有1个;R组有8个属于厚壁菌门,4个属于拟杆菌门,4属于变形菌门,属于未分类细菌、软壁菌门(Tenericutes)、黏胶球形菌门(Lentisphaerae)、疣微菌门(Verrucomicrobia)的各有1个。F组相对丰度大于1%的属有18个,而R组有15个。F组相对丰度最高的3个属分别为厚壁菌门瘤胃球菌科未分类的属[(26.914±5.639)%]、拟杆菌门的拟杆菌属[Bacteroides,(11.252±4.244)%]、厚壁菌门毛螺菌科未分类的属[(10.128±4.548)%];R组相对丰度最高的3个属均源于拟杆菌门,分别为普雷沃菌属[Prevotella,(28.621±8.389)%]、拟杆菌门未分类的属[(11.375±6.476)%]、拟杆菌目未分类的属[(11.299±4.132)%]。
| 表 2 取样深度为10 200时,瘤胃和粪便微生物α多样性指数对比 Table 2 Comparison of α diversity indices of microbial communities between rumen and eces calculated at a sample depth of 10 200 |
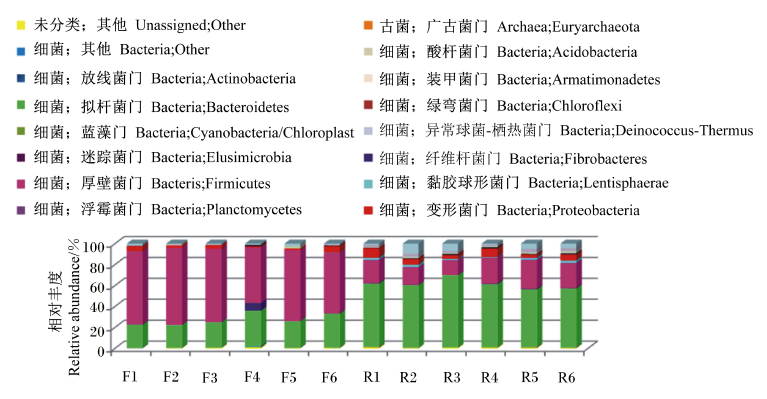 | 图3 门水平上菌群组成
Fig. 3 Composition of microbial community at phylum level |
| 表3 瘤胃和粪便差异显著和极显著的科
Table 3 The families that were significantly different or extremely significantly different between rumen and feces (n=6)
|
| 表4 粪便和瘤胃中序列相对丰度最高的20个属
Table 4 Twenty genera with the highest sequence relative abundance in rumen and feces (n=6)
|
结合样本的OTU的种类及其相对丰度,计算样本间加权遗传距离矩阵(weighted unifrac distance),利用距离矩阵绘制PCoA聚类图,分析其组间相似性(图4)。结果表明,F组与R组间相似性为(36.61±4.88)%。各样品依据所处组别的不同而相互聚类到一起,表明组内相似性比组间相似度高。此外,F组个体的聚集程度比R组高,说明F组组内的相似度高于R组。
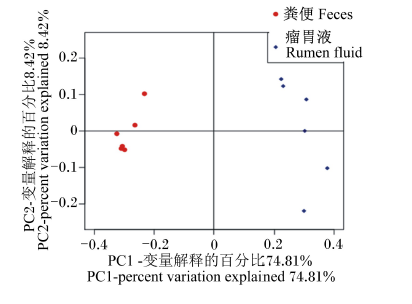 | 图4 PCoA聚类分析
Fig. 4 Cluster analysis by principal co-ordinate analysis |
2.4 共享属分析
F组和R组组内及组间的共享属见表5。在F组内共发现了22个共享属,共来源于7个门,其中13个属源于厚壁菌门,拟杆菌属[(11.252±4.244)%]为其相对丰度最高的共享属;R组内共发现了30个共享属,共来源于12个门,其中有12个属源于厚壁菌门,普雷沃菌属[(28.621±8.389)%]为其相对丰度最高的共享属。所有12个样品间共发现了14个共享属,其中有9个属于厚壁菌门,1个属于古菌界广古菌门,其他4个分别属于不同的门,相对丰度最高的为厚壁菌门的梭菌属4[Clostridium_Ⅳ,(1.748±1.330)%]。
3 讨 论反刍动物胃肠道微生物可帮助宿主从饲料中获取养分,并抵御病原微生物感染,其瘤胃和粪便微生物的结构与组成常被用于评估宿主的健康状况[19, 20, 21, 22]。目前,有关利用高通量测序技术研究山羊瘤胃和粪便微生物的报道较少[23, 24],有关二者组成和结构差异的研究还未见报道。为使人们进一步认识山羊胃肠道微生物,本试验用高通量测序技术研究了山羊瘤胃和粪便微生物的物种组成及其丰度,并比较了两者的差异。
Kim等[25]对GenBank数据库中的所有可利用瘤胃微生物序列进行了总结,结果表明瘤胃相对丰度最高的微生物为厚壁菌门,其次为拟杆菌门。但本试验发现,山羊瘤胃中相对丰度最高的是拟杆菌门,其次为厚壁菌门。出现该差异可能是因为检测方法不同,本试验所采用的为高通量测序技术,而Kim等[25]所总结的序列主要是通过变性梯度凝胶电泳、限制酶切片段多态性等技术得到的;此外,也可能是因为Kim等[25]所研究的对象主要为牛和绵羊的瘤胃微生物序列,与山羊瘤胃微生物序列存在一定的种间差异。除了研究方法、动物品种,饲粮和日龄的不同也会影响瘤胃微生物[23, 26]。Huo等[23]利用高通量测序法研究山羊瘤胃微生物时发现,饲喂高粗料饲粮条件下瘤胃固相微生物中拟杆菌门丰度最高,液相微生物中拟杆菌门和厚壁菌门丰度相当,但饲喂高精料饲粮时无论是瘤胃固相微生物还是液相微生物,相对丰度最高的门均为厚壁菌门。Jami等[26]发现1~3日龄的奶牛犊牛瘤胃中丰度从高到低依次为变形菌门、厚壁菌门和拟杆菌门,但随着年龄的增长,2、6以及24月龄牛瘤胃中,拟杆菌门相对丰度均超过50%。
在属水平上,本研究发现山羊瘤胃液中相对丰度最高的菌属为普雷沃菌属。这与以往在其他反刍动物如奶牛[27]、肉牛[11]和绵羊[28]上的研究结果一致,这可能意味着普雷沃菌属为所有反刍动物瘤胃微生物的优势菌属。普雷沃菌属能在瘤胃中处于优势地位,可能与该属微生物种型多,遗传多样性丰富,底物宽泛等因素有关。目前可分离培养的普雷沃菌属有栖瘤胃普雷沃菌(Prevotella ruminicola)、短普雷沃氏菌(Prevotella brevis)、布氏普雷沃氏菌(Prevotella bryantii)和Prevotella albensis[29]。其中,栖瘤胃普雷沃菌和布氏普雷沃氏菌均有降解寡糖、蛋白质、淀粉、木聚糖等营养物质的能力[30];大部分可分离培养的短普雷沃氏菌能够发酵纤维二糖、果糖、葡萄糖、菊糖、乳糖、麦芽糖、甘露糖、蜜二糖、蜜三糖、蔗糖、多肽[29];有研究表明大多数可分离培养的Prevotella albensis能够发酵葡萄糖、 木糖、水杨苷和多肽,此外还能产生很弱的木羧甲基纤维素酶活性[29]。由此可见,除脂质外,普雷沃菌属几乎可以单独完成或参与瘤胃内所有饲粮成分的降解,因此其在瘤胃生态中扮演着重要角色。基于培养和非培养的分子技术方法的研究表明,普雷沃菌属在山羊瘤胃的相对丰度分别为60%和56%[28]。但可能由于研究技术不同,本试验发现瘤胃液中普雷沃氏菌的相对丰度为28.621%,数值上低于前人的研究结果。
| 表5 瘤胃和粪便或所有样品的共享属
Table 5 The shared genera between rumen and feces or all samples
|
瘤胃球菌属以往被认为在瘤胃降解饲料养分中发挥着重要作用,因为该属含有2种功能强大的纤维降解菌——白色瘤胃球菌(Ruminococcus albus)和黄色瘤胃球菌(Ruminococcus flavefaciens),这2种菌能够产生大量的纤维素酶和半纤维素酶[31, 32],是瘤胃中主要的纤维降解菌。但本研究发现瘤胃球菌属在瘤胃中丰度仅有0.669%,Girija等[33]关于牛瘤胃微生物的研究也表明瘤胃球菌属丰度仅有2%,表明瘤胃球菌属在瘤胃中仅占很小的比例。这说明以往依靠传统的分离培养技术得到的试验结果,可能严重夸大了瘤胃球菌属在瘤胃中的作用。
本研究结果表明,山羊粪便中相对丰度最高的微生物为厚壁菌门(65.400%),其次为拟杆菌门(26.620%)、变形菌门(3.047%),这与前人用高通量测序方法研究牛粪便微生物组成的结果相一致[11]。De Jesùs-Laboy等[34]利用基因芯片技术研究了圈养山羊和野山羊粪便的微生物组成,发现相对丰度排在前2位的也是厚壁菌门和拟杆菌门,而排在第3位的为放线菌门,与本试验的结果稍有差异。但Min等[24]的研究却发现,山羊粪便中相对丰度最高的为变形菌门,拟杆菌门和厚壁菌分别位于第2和3位,这与本研究结果差异较大,这可能与试验动物品种、试验动物饲粮、试验条件等综合因素有关。本试验得出,在属水平上山羊粪便相对丰度最高的为瘤胃球菌科未分类的属。Gu等[35]利用高通量测序技术研究小鼠胃肠道各个部位的微生物多样性,结果表明瘤胃球菌科是小鼠大肠以及粪便中的优势菌群,且其在粪便中的相对丰度高于其他胃肠道部位。这与本试验的结果一致,本试验也发现瘤胃球菌科在山羊粪便中的丰度极显著高于瘤胃。瘤胃球菌科中有些菌种已被培养鉴定,有的已被全基因测序,这些菌种被证明能够产生纤维素酶、淀粉酶等碳水化合物降解酶[36],说明瘤胃球菌科微生物具有广泛的碳水化合物降解活性。其在粪便中相对丰度较高可能表明山羊后肠碳水化合物的发酵很活跃,但也有可能与动物体内炎症有关,因为瘤胃球菌科的一些种类是促炎症细菌,据报道其在发炎性肠道疾病的病人体内有很高的丰度[37]。此外,瘤胃球菌科的部分菌可参与不饱和脂肪酸的生物氢化作用[38],有的能够降解芳香族化合物、肉桂酸和巴豆酸酯[39]。
本试验将所得有效序列进行OTU聚类后,共获得13 601个OUT,但粪便和瘤胃液共享的OUT仅有558个,仅占OUT总数的4.1%,这说明山羊粪便和瘤胃微生物的菌群结构差异较大。比较2组的α多样性指数,虽然反映样品中物种丰富度(richness)和/或均匀度(eveness)的ChaoⅠ、The_observed_species、PD_whole_tree和Shannon-Wiener指数组间差异均不显著,但在数值上,F组的各项指数都大于R组,说明山羊粪便微生物的多样性高于瘤胃微生物,而差异不显著可能是由于组内标准差过大。在科水平上,F组和R组组间差异显著的微生物共有22个科,差异极显著的微生物有10个科。在属水平上, F组和R组相对丰度最高的共享属都属于拟杆菌门,分别为拟杆菌属和普雷沃菌属。但拟杆菌属不是R组共享属,普雷沃菌属也是F组的共享属。饲料纤维绝大部分都在瘤胃中被降解,许多已知在纤维分解中有重要作用的属如丁酸弧菌属(Butyrivibrio)、纤维杆菌属(Fibrobacter),以及降解半纤维素的属如琥珀酸弧菌属(Succinivibrio)、螺旋体属(Spirochaeta)、密螺旋体属(Treponema)仅为R组的共享属。而F组部分样本也可检测到少量纤维降解菌,主要是因为后肠段存在一定的纤维发酵活动。
瘤胃微生物主要可分为固相微生物和液相微生物,液相微生物存在于瘤胃液中,而固相微生物主要疏松或紧密附着在饲料颗粒表面或存在于饲料颗粒内部,在粪便瘤胃固相和液相微生物不再存在。De Oliveira等[11]用454焦磷酸测序方法研究了阉牛不同胃肠道部位的微生物分布以及发育规律,结果表明,粪便与结肠微生物组成最接近,而与瘤胃差异最大,瘤胃与粪便微生物共享OTU仅有66个。Frey等[40]也报道,当食糜从胃肠道的一个部位进入另一个部位时,其内的微生物会发生显著变化。从本试验结果中也可知,相比于瘤胃,粪便微生物菌群分布变化很大。这主要是因为瘤胃微生物随食糜流向小肠时,肠道内酸性环境使得微生物细胞被溶解,微生物蛋白质连同饲料非降解蛋白质组成小肠蛋白质,供宿主吸收利用[41]。因此,到达后肠的瘤胃微生物很少。粪便菌群结构与瘤胃相比有较大变化也可能与食糜在后肠的停留时间较短有关,且一些微生物生长需要特定的生长环境[42]。瘤胃内古菌主要以产甲烷菌为主,产甲烷菌虽然数量远小于细菌,但其能利用瘤胃发酵产生的氢气将二氧化碳还原最终生成甲烷,该过程会消耗2%~15%的饲料总能。瘤胃内产甲烷菌最主要的种类为甲烷短杆菌(Methanobrevibacter),本试验发现在所有瘤胃和粪便古菌种类中,甲烷短杆菌的相对丰度均为最高,且在所有粪便和瘤胃样品中均被检测到。瘤胃甲烷短杆菌属的相对丰度为0.072%,占瘤胃所有古菌的23%;粪便甲烷短杆菌属的相对丰度为0.075%,但其占粪便所有古菌的比例达到58%。这说明后肠中也存在一定数量的产甲烷菌,虽然粪便中古菌数量有所减少,但甲烷短杆菌属的相对丰度并未有较大变化。这预示着也许可通过检测粪便中的产甲烷菌(特别是甲烷短杆菌属)来反映瘤胃内产甲烷菌的结构与组成、评估反刍动物的产甲烷能力或预测反刍动物的甲烷产量,而这需要更多研究来进一步确定二者之间的关系。本研究还发现粪便和瘤胃中未分类的微生物相对丰度均大于1%,且各个分类水平上均发现较多未能被分类的微生物,这表明在瘤胃和粪便中还有许多微生物未被人们所清楚认识,而这些微生物可能在动物生理活动中扮演者举足轻重的作用,有待深入研究。
4 结 论① 瘤胃内容物及粪便中的微生物群落存在较大的差异,瘤胃微生物主要以拟杆菌门为主,厚壁菌门次之,相对丰度最高的属为普雷沃菌属;而粪便中的微生物以厚壁菌门为主,拟杆菌门次之,相对丰度最高的属为瘤胃球菌科下未分类的属。
② 瘤胃中还有许多未被分类鉴定且相对丰度较高的微生物,其结构和功能有待深入研究。
| [1] | MACKIE R I,WHITE B A.Recent advances in rumen microbial ecology and metabolism:potential impact on nutrient output[J]. Journal of Dairy Science,1990,73(10):2971-2995. ( 1) 1)
|
| [2] | RUSSELL J B,RYCHLIK J L.Factors that alter rumen microbial ecology[J]. Science,2001,292(5519):1119-1122. ( 1) 1)
|
| [3] | EL-MEADAWAY A,MIR Z,MIR P S,et al.Relative efficacy of inocula from rumen fluid or faecal solution for determining in vitro digestibility and gas production[J]. Canadian Journal of Animal Science,1998,78(4):673-679. ( 1) 1)
|
| [4] | HESPELL R B,AKIN D E,DEHORITY B A.Bacteria,fungi,and protozoa of the rumen[C]//MACKIE R I,WHITE B A,ISAACSON R E.Gastrointestinal microbiology.New York:Chapman and Hall,1997. ( 1) 1)
|
| [5] | YANG L Y,CHEN J,CHENG X L,et al.Phylogenetic analysis of 16S rRNA gene sequences reveals rumen bacterial diversity in Yaks(Bos grunniens)[J]. Molecular Biology Reports,2010,37(1):553-562. ( 1) 1)
|
| [6] | PACE N R.A molecular view of microbial diversity and the biosphere[J]. Science,1997,276(5313):734-740. ( 1) 1)
|
| [7] | EDWARDS J E,HUWS S A,KIM E J,et al.Characterization of the dynamics of initial bacterial colonization of nonconserved forage in the bovine rumen[J]. FEMS Microbiology Ecology,2007,62(3):323-335. ( 1) 1)
|
| [8] | HIGHLANDER S K.High throughput sequencing methods for microbiome profiling:application to food animal systems[J]. Animal Health Research Reviews,2012,13(01):40-53. ( 2) 2)
|
| [9] | SANDRI M,MANFRIN C,PALLAVICINI A,et al.Microbial biodiversity of the liquid fraction of rumen content from lactating cows[J]. Animal,2014,8(04):572-579. ( 1) 1)
|
| [10] | REY M,ENJALBERT F,COMBES S,et al.Establishment of ruminal bacterial community in dairy calves from birth to weaning is sequential[J]. Journal of Applied Microbiology,2014,116(2):245-257. ( 1) 1)
|
| [11] | DE OLIVEIRA M N V,JEWELL K A,FREITAS F S,et al.Characterizing the microbiota across the gastrointestinal tract of a Brazilian Nelore steer[J]. Veterinary Microbiology,2013,164(3):307-314. ( 4) 4)
|
| [12] | 张丽英.饲料分析及饲料质量检测技术[M]. 北京:中国农业大学出版社,2003. ( 1) 1)
|
| [13] | CAPORASO J G,LAUBER C L,WALTERS W A,et al.Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample[J]. Proceedings of the National Academy of Sciences,2011,108(Suppl.1):4516-4522. ( 1) 1)
|
| [14] | CAPORASO J G,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J]. Nature Methods,2010,7(5):335-336. ( 1) 1)
|
| [15] | SCHLOSS P D,WESTCOTT S L,RYABIN T,et al.Introducing mothur:open-source,platform-independent,community-supported software for describing and comparing microbial communities[J]. Applied and Environmental Microbiology,2009,75(23):7537-7541. ( 1) 1)
|
| [16] | EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics,2010,26(19):2460-2461. ( 1) 1)
|
| [17] | CAPORASO J G,BITTINGER K,BUSHMAN F D,et al.PyNAST:a flexible tool for aligning sequences to a template alignment[J]. Bioinformatics,2010,26(2):266-267. ( 1) 1)
|
| [18] | COLE J R,WANG Q,CARDENAS E,et al.The Ribosomal Database Project:improved alignments and new tools for rRNA analysis[J]. Nucleic Acids Research,2009,37(suppl.1):D141-D145. ( 1) 1)
|
| [19] | DOWD S E,CALLAWAY T R,WOLCOTT R D,et al.Evaluation of the bacterial diversity in the feces of cattle using 16S rDNA bacterial tag-encoded FLX amplicon pyrosequencing(bTEFAP)[J]. BMC Microbiology,2008,8(1):125. ( 1) 1)
|
| [20] | LETTAT A,NOZIÈRE P,SILBERBERG M,et al.Rumen microbial and fermentation characteristics are affected differently by bacterial probiotic supplementation during induced lactic and subacute acidosis in sheep[J]. BMC Microbiology,2012,12(1):142. ( 1) 1)
|
| [21] | VAN BAALE M J,SARGEANT J M,Gnad D P,et al.Effect of forage or grain diets with or without monensin on ruminal persistence and fecal Escherichia coli O157:H7 in cattle[J]. Applied and Environmental Microbiology,2004,70(9):5336-5342. ( 1) 1)
|
| [22] | VAN DONKERSGOED J,GRAHAM T,GANNON V.The prevalence of verotoxins,Escherichia coli O157:H7,and Salmonella in the feces and rumen of cattle at processing[J]. The Canadian Veterinary Journal,1999,40(5):332. ( 1) 1)
|
| [23] | HUO W,ZHU W,MAO S.Impact of subacute ruminal acidosis on the diversity of liquid and solid-associated bacteria in the rumen of goats[J]. World Journal of Microbiology and Biotechnology,2014,30(2):669-680. ( 2) 2)
|
| [24] | MIN B R,SOLAIMAN S,SHANGE R,et al.Gastrointestinal bacterial and methanogenic archaea diversity dynamics associated with condensed tannin-containing pine bark diet in goats using 16S rDNA amplicon pyrosequencing[J/OL].International Journal of Microbiology,2014.http://dx.doi.org/10.1155/2014/141909. ( 2) 2)
|
| [25] | KIM M,MORRISON M,YU Z.Status of the phylogenetic diversity census of ruminal microbiomes[J]. FEMS Microbiology Ecology,2011,76(1):49-63. ( 2) 2)
|
| [26] | JAMI E,MIZRAHI I.Composition and similarity of bovine rumen microbiota across individual animals[J]. PloS One,2012,7(3):e33306. ( 2) 2)
|
| [27] | STEVENSON D M,WEIMER P J.Dominance of Prevotella and low abundance of classical ruminal bacterial species in the bovine rumen revealed by relative quantification real-time PCR[J]. Applied Microbiology and Biotechnology,2007,75(1):165-174. ( 1) 1)
|
| [28] | BEKELE A Z,KOIKE S,KOBAYASHI Y.Genetic diversity and diet specificity of ruminal Prevotella revealed by 16S rRNA gene-based analysis[J]. FEMS Microbiology Letters,2010,305(1):49-57. ( 2) 2)
|
| [29] | AVGUTIN G,WALLACE R J,FLINT H J.Phenotypic diversity among ruminal isolates of Prevotella ruminicola:proposal of Prevotella brevis sp.nov.,Prevotella bryantii sp.nov.,and Prevotella albensis sp.nov.and redefinition of Prevotella ruminicola[J]. International Journal of Systematic Bacteriology,1997,47(2):284-288. ( 2) 2)
|
| [30] | PURUSHE J,FOUTS D E,MORRISON M,et al.Comparative genome analysis of Prevotella ruminicola and Prevotella bryantii:insights into their environmental niche[J]. Microbial Ecology,2010,60(4):721-729. ( 1) 1)
|
| [31] | WOOD T M,WILSON C A,STEWART C S.Preparation of the cellulase from the cellulolytic anaerobic rumen bacterium Ruminococcus albus and its release from the bacterial cell wall[J]. Biochemical Journal,1982,205:129-137. ( 1) 1)
|
| [32] | DOERNER K C,WHITE B A.Assessment of the endo-1,4-beta-glucanase components of Ruminococcus flavefaciens FD-1[J].Applied and Environmental Microbiology,1990,56(6):1844-1850. ( 1) 1)
|
| [33] | GIRIJA D,DEEPA K,XAVIER F,et al.Analysis of cow dung microbiota-a metagenomic approach[J].Indian Journal of Biochemistry,2013,12:372-378. ( 1) 1)
|
| [34] | DE JESÙS-LABOY K M,GODOY-VITORINO F,PICENO Y M,et al.Comparison of the fecal microbiota in feral and domestic goats[J]. Genes,2011,3(1):1-18. ( 1) 1)
|
| [35] | GU S,CHEN D,ZHANG J N,et al.Bacterial community mapping of the mouse gastrointestinal tract[J]. PloS One,2013,8(10):e74957. ( 1) 1)
|
| [36] | SUEN G,STEVENSON D M,BRUCE D C,et al.Complete genome of the cellulolytic ruminal bacterium Ruminococcus albus 7[J]. Journal of Bacteriology,2011,193(19):5574-5575. ( 1) 1)
|
| [37] | FRANK D N,AMAND A L S,FELDMAN R A,et al.Molecular-phylogenetic characterization of microbial community imbalances in human inflammatory bowel diseases[J]. Proceedings of the National Academy of Sciences,2007,104(34):13780-13785. ( 1) 1)
|
| [38] | HUWS S A,KIM E J,LEE M R F,et al.As yet uncultured bacteria phylogenetically classified as Prevotella,Lachnospiraceae incertae sedis and unclassified Bacteroidales,Clostridiales and Ruminococcaceae may play a predominant role in ruminal biohydrogenation[J]. Environmental Microbiology,2011,13(6):1500-1512. ( 1) 1)
|
| [39] | DEFNOUN S,AMBROSIO M,GARCIA J L,et al.Degradation of cinnamate via β-oxidation to benzoate by a defined,syntrophic consortium of anaerobic bacteria[J]. Current Microbiology,2003,46(1):0047-0052. ( 1) 1)
|
| [40] | FREY J C,PELL A N,Berthiaume R,et al.Comparative studies of microbial populations in the rumen,duodenum,ileum and faeces of lactating dairy cows[J]. Journal of Applied Microbiology,2010,108(6):1982-1993. ( 1) 1)
|
| [41] | 冯仰廉.反刍动物营养学[M]. 北京:科学出版社,2004. ( 1) 1)
|
| [42] | WILLIAMS A G,COLEMAN G S.The rumen protozoa[M]. New York:Springer-Verlag; 1992. ( 1) 1)
|




