家兔是单胃草食动物,其盲肠非常发达,占整个消化道容积的49%,其中微生物数量达到1010~1012个/g。这些微生物可利用前肠道未消化吸收的物质合成维生素等营养物质,并以软粪的形式被采食和利用[1,2]。同时,盲肠中的正常菌群也是其末端免疫器官即蚓突发育和成熟所必需的[3]。因此,促进幼兔盲肠微生物区系的建立及维持其正常菌群的平衡对营养物质的消化吸收及肠道健康都非常重要。幼兔断奶后,尤其在断奶后2~3周内,由于失去兔乳中中链脂肪酸的保护,以及食物和环境的改变、自身消化系统和肠道免疫系统发育不完善等原因,消化道中尤其是盲肠中的微生物区系很容易发生紊乱,并因此引起腹泻甚至死亡[4,5]。通过检测断奶后健康幼兔盲肠微生物群落多样性,可了解断奶家兔盲肠优势菌组成及断奶后不同日龄微生物群落的变化,为研究开发断奶幼兔特异的益生菌或有利于盲肠优势菌增殖的益生元等提供理论依据。
早期主要通过微生物培养的方式研究动物肠道菌群的结构,但该方法仅能检测出1%~10%的肠道菌种[6,7]。近年来,分子生物学技术被广泛应用于微生物群落多样性研究,包括变性梯度凝胶电泳和单链构象多态性等方法[8,9],但这些方法有各自的局限性,尤其是不能真实地反映内容物中细菌种类和丰度。Illumina MiSeq测序技术克服了传统检测方法的缺陷,能全面和客观地反映动物肠道菌群结构[10,11]。此外,从目前检测家兔盲肠微生物多样性的研究报道看,试验饲粮中均添加了抗生素,而抗生素会影响肠道菌群的结构[9,12]。鉴于此,本试验旨在采用Illumina MiSeq测序技术,检测采食无抗生素饲粮健康幼兔盲肠微生物组成,以研究断奶幼兔盲肠微生物群落多样性及菌群发育情况,为后续断奶幼兔益生菌开发及肠道微生物疾病防治等提供依据。
1 材料与方法center 1.1 试验动物及饲粮center选取30窝同日出生、胎次相同和平均窝重差异不显著(P>0.05)的健康新生仔兔,每窝8只,于14日龄开始补饲未添加抗生素的基础饲粮。整个哺乳期母仔分离饲养,每天07:00哺乳1次。30日龄断奶时,从30窝中选择体重差异不显著(P>0.05)的48只健康幼兔作为试验兔,断奶后每只幼兔单笼饲养,且继续饲喂未添加抗生素的基础饲粮。断奶前对兔舍进行严格消毒。试验期间兔舍自然采光和通风,幼兔限饲但自由饮水,每天清粪1次,并观察和记录幼兔粪便变化、采食及精神状态等健康情况。对有腹泻等异常情况的幼兔立即隔离饲养且不再作为试验兔。
参考NRC(1977)和AEC(1993)标准,结合本地区饲料资源状况设计基础饲粮[13],制成直径为4~6 mm的颗粒饲料。基础饲粮组成及营养水平见表1。
| 表1 基础饲粮组成及营养水平(风干基础) Table 1 Composition and nutrient levels of the basal diet (air-dry basis) |
分别于断奶当日及断奶后1、2和3周分别随机选取6只健康幼兔进行屠宰,解剖后观察幼兔肠道及内容物,选取肠道无鼓胀、内容物无水状、无黏液的3只健康幼兔的盲肠内容物作为后续测序样本,装入灭菌离心管中,经液氮速冻后,迅速转移到-80 ℃冰箱保存备用。
1.3 盲肠内容物微生物基因组总DNA的提取center盲肠内容物基因组总DNA采用改进的氯仿异戊醇法[14]提取。所提的总DNA溶于无菌TE缓冲液,用微量核酸测定仪(NANO DROP 2000)测定DNA浓度及质量,-20 ℃贮存。
1.4 Illumina MiSeq测序center对微生物基因组16S rDNA V4高变区进行扩增,引物为F515(5’-GTGCCAGCMGCCGC GGTAA-3’)和R806(5’-GGACTACVSGGGTATCTAAT-3’)。每个样本的引物5’端加barcode序列。用Takara ExTag Hotstart酶对每个样品做3个50 μL的平行PCR反应,并将产物使用Omega试剂盒进行胶回收,再用Invitrogen Qubit dsDNA BR试剂盒对纯化的PCR产物进行准确定量。多个样品进行等量混合,构建2个鸟枪法文库。由Macrogen公司采用Illumina MiSeq 250PE测序。
1.5 生物信息分析与数据统计center采用CASAVA v1.8.2和FastQC软件,按Illumina标准流程进行base calling及数据产出统计[15]。使用QIIME软件,将拼接之后的序列按照barcode标签区分,去除与barcode不符的序列、带有N的序列和含有连续3个质量值低于20的碱基的序列,要求质量值大于20且连续碱基长度必须大于序列全长的75%[16]。最后去除barcode及引物序列,使用UCLUST软件,按照97%相似性进行操作分类单位(operational taxonomic units,OTU)聚类,挑选出每个OTU的代表序列[17]。用RDP Classifier软件,置信度0.9对过滤后的OTU进行物种注释,参考物种数据库为RDP[18]。盲肠微生物构成的稀疏曲线图、热图和主坐标分析(principle coordinate analysis,PCoA)图均是由R软件制作,而样本间相似度比对的非加权组平均法(unweighted pair-group method with arithmetic means,UPGMA)聚类图是由Mega软件制作而成。采用SPSS 19.0统计软件对不同日龄幼兔盲肠微生物OTU总数、ACE值、Chao1值、Shannnon值和相对丰度进行单因素方差分析(one-way ANOVA),Duncan氏法多重比较检验,显著水平为P<0.05。
2 结 果center 2.1 测序结果的质量分析center 2.1.1 样品测序数据样本数目为12份,经MiSeq 250PE测序,得到1 175 992条长度约324 bp的序列,总碱基数381 021 408 bp。
2.1.2 稀疏曲线
模拟输入序列数目(小于总的样本序列条数)与OTU个数产出间的相互关系,绘制稀疏曲线。根据曲线是否达到平台期或者不再有升高的趋势阐明测序数据是否足以覆盖微生物群落全部的多样性。由图1可知,所有样品曲线均趋于平缓达到平台期,说明实际测序量足以覆盖群落物种组成,能够真实反映群落各物种间的相对比例关系。
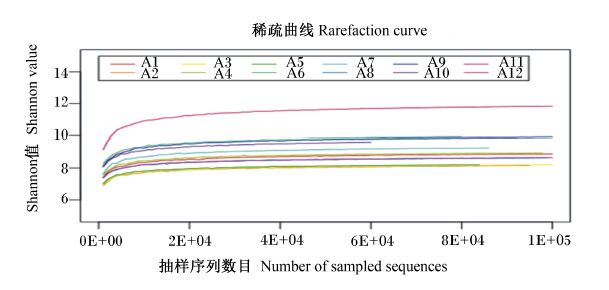 | A1~A12为样本名称。图2、图4同。A1 to A12 were names of samples.The same as Fig.2 and Fig.4.图1 12只健康幼兔盲肠微生物的Shannon值与稀疏曲线 Fig. 1 Shannon value and rarefaction curve of caecal microbiota in 12 healthy young rabbits |
α多样性是对单个样品中物种多样性的分析。样品的Chao1值、ACE值和Shannon指数这几个指标数值越大,说明样品中的物种越丰富。由表2可知,Chao1值(P=0.062)、ACE值(P=0.051)和Shannon指数(P=0.094)随日龄的增加趋于升高,说明随着年龄的增长,幼兔盲肠微生物种类趋于复杂。
| 表2 断奶后不同周数健康幼兔盲肠微生物α多样性指标均值 Table 2 Mean values of caecal microbiota alpha diversity indices in healthy young rabbits at different weeks after weaning |
由表3可知,12只健康幼兔的盲肠微生物均主要由厚壁菌门、拟杆菌门、未分类菌门、变形菌门、疣微菌门、绿弯菌门、酸杆菌门和放线菌门组成。在断奶当天以及断奶后1、2和3周这几种菌门共占盲肠微生物的比例分别达到97.34%、96.03%、97.87%和95.00%,且断奶后不同周数幼兔的各菌门相对丰度差异均不显著(P>0.05)。断奶当天及断奶后1~3周幼兔盲肠微生物中相对丰度最大的优势菌门为厚壁菌门。
| 表3 断奶后不同周数健康幼兔盲肠微生物群落在门水平上的分布 Table 3 Distribution of caecal microbiota in healthy young rabbits by phylum level at different weeks after weaning |
图2是在科水平上12只健康幼兔盲肠菌群构成的热图,热图中每小格代表所在样品中某种菌的相对丰度,颜色越红代表相对丰度越高。由图可知,在科水平上,各日龄幼兔盲肠微生物中毛螺旋菌科(9.90%~20.03%)、瘤胃球菌科(12.60%~15.50%)、拟杆菌科(4.20%~20.13%)和紫单胞菌科(3.77%~5.97%)相对丰度较高。检测结果显示,家兔盲肠中的厚壁菌门主要由毛螺旋菌科和瘤胃球菌科组成,拟杆菌门主要由拟杆菌科、紫单胞菌科和普雷沃氏菌科组成。断奶当天幼兔盲肠中拟杆菌科相对丰度显著高于其他日龄组(P<0.05),肠杆菌科相对丰度在断奶当天到断奶后2周期间有升高的趋势,断奶后2周到断奶后3周期间有降低的趋势(P=0.058),其他菌科相对丰度随着日龄的增长变化不显著(P>0.05)。
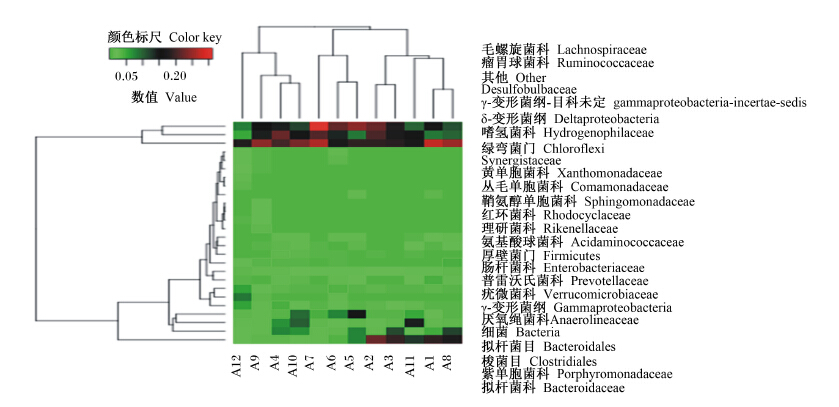 | 图2 在科分类水平上12只健康幼兔盲肠菌群构成的热图 Fig. 2 Heatmap of caecal bacterial constitution in 12 healthy young rabbits at family level |
以丰度大于1%作为优势菌属的划分依据[19]。如表4所示,断奶当天及断奶后1、2和3周幼兔盲肠内优势菌属分别为10、13、12和13种。此外,拟杆菌属、Barnesiella、Akkermansia、普雷沃氏菌属和4种未知菌属在不同日龄幼兔盲肠微生物中丰度均大于1%,因此,在断奶至断奶后3周期间,健康幼兔盲肠中优势菌属均为拟杆菌属、Barnesiella、Akkermansia、普雷沃氏菌属和4种未知菌属。
| 表4 断奶后不同周数健康幼兔盲肠微生物群落在属水平上的分布 Table 4 Distribution of caecal microbiota in healthy young rabbits by genera level at different weeks after weaning |
属水平上确定的纤维分解菌是醋弧菌属和瘤胃球菌属。醋弧菌属在不同日龄幼兔盲肠微生物 中的相对丰度差异不显著(P>0.05),随着年龄的增长,瘤胃球菌属的相对丰度有降低趋势(P=0.070)。不同日龄健康幼兔盲肠中乳杆菌属相对丰度均小于1%,断奶当天及断奶后1、2和3周分别为0.43%、0.40%、0.53%和0.47%,且年龄对其相对丰度无显著影响(P>0.05)。断奶当天幼兔盲肠中拟杆菌属和Parabacteroides相对丰度显著高于其他日龄组(P<0.05),在断奶后1、2和3周,拟杆菌属相对丰度分别降低了79.14%、66.56%和73.03%,Parabacteroides相对丰度分别降低了57.92%、92.90%和63.39%,其他菌属相对丰度断奶前与断奶后无显著的变化(P>0.05)。
2.3.4 断奶幼兔盲肠中乳杆菌属和拟杆菌属的组成分析分别对12只幼兔盲肠乳杆菌属中相对丰度最高OTU的16S rDNA V4区序列进行比对分析,发现其与耐酸乳杆菌、同型腐酒乳杆菌和食果糖乳杆菌的16S rDNA V4区序列相似性大于97%。由表4可知,各日龄幼兔盲肠微生物相对丰度最高的菌属是拟杆菌属,通过对12只幼兔盲肠中拟杆菌属中相对丰度最高OTU的16S rDNA V4区序列进行比对分析,发现其与Bacteroides dorei、单形拟杆菌、Bacteroides rodentium和Bacteroides xylanisolvens的16S rDNA V4区序列相似性大于97%。对拟杆菌属其他OTU进行16S rDNA V4区序列比对分析发现,家兔盲肠中还包含Bacteroides sartorii、Bacteroides chinchillae、多形拟杆菌、粪拟杆菌和溃疡拟杆菌等拟杆菌属的菌种。
2.4 β多样性分析center图3是断奶后不同周数健康幼兔盲肠微生物群落构成的PCoA,通过图中样品点距离的远近,可观察个体或群体间的差异,样品点越相近说明样品间微生物群落构成越相似。由图可知,A9和A12与同日龄以及其他日龄样品均距离较远,说明A9和A12与其他样品微生物群落构成差异较大。从图4可以看出,除A9和A12外,其他样品均在同一分支上,说明日龄对微生物群落结构影响不大。
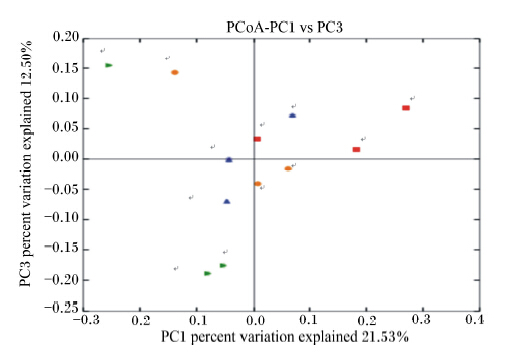 | 红色正方形:30 日龄;蓝色三角形37 日龄;橘色圆形:44 日龄:51日龄。 Red squre: 30days of age; bule triangle: 37 days of age; orange circle: 44 days of age; green triangle: 51 days of age.图3 断奶后不同周数健康幼兔盲肠微生物群落构成的PCoA比较 Fig. 3 Comparison of caecal microbiota composition in healthy young rabbits at different weeks after weaning using PCoA |
 | 图4 样本间相似度比对UPGMA聚类图 Fig. 4 UPGMA dendrogram of similarity among all samples |
家兔断奶后,失去了母乳中中链脂肪酸的抗菌保护作用[4],同时由于断奶应激、饲料转换应激以及消化系统和免疫系统尚未建立等原因,极易造成断奶幼兔腹泻,从而使生长受阻,甚至引起死亡,严重影响兔场效益[5]。幼兔断奶后盲肠中正常菌群的建立与动态平衡的维持有利于抵御病原菌的入侵,降低其腹泻率。
白秀娟等[9]和Combes等[8]分别利用聚合酶链式反应-变性梯度凝胶电泳(PCR-DGGE)和毛细管电泳-单链构象多态性(CE-SSCP)方法研究了不同日龄的家兔肠道微生物组成,均发现日龄对细菌种群结构和多样性有显著影响,随着日龄增加,断奶幼兔盲肠微生物组成更加复杂。但本试验检测的结果显示,断奶后日龄对家兔盲肠微生物群落多样性无显著影响,只是趋于复杂化。饲粮为动物肠道生态系统提供营养物质和能量,是影响成年动物肠道微生物平衡的主要因素[20]。本研究整个试验期饲粮稳定可能是盲肠微生物群落结构保持相对稳定的重要原因。这也提示,断奶后一定时间内饲喂补饲料有利于维持断奶后幼兔肠道微生物群落的稳定。此外,通过聚类分析发现同日龄幼兔样品不能聚到一簇,这表明动物个体间肠道微生物群落结构存在差异,Combes等[8]也证实了这一点。
本研究发现,30~51日龄幼兔盲肠微生物中相对丰度最高的菌门是厚壁菌门,且其中瘤胃球菌科和毛螺旋菌科数量最多,这与Buerl等[21]对40日龄家兔盲肠微生物以及Eshar等[10]对66~81日龄家兔粪便微生物的研究结果相同。分析发现,试验期间健康幼兔盲肠内纤维分解菌主要为瘤胃球菌科的醋弧菌属和瘤胃球菌属,且醋弧菌属相对丰度保持稳定,而瘤胃球菌属相对丰度在后期趋于下降。Bennegadi-Laurent等[22]报道,家兔盲肠纤溶能力随日龄的增长保持稳定后期趋于下降,这种变化趋势可能与瘤胃球菌属丰度随日龄变化的趋势有关。本试验结果显示,断奶后毛螺旋菌科丰度最高,而毛螺旋菌科中多个菌属为丁酸生成菌属[23]。家兔盲肠发酵产物中丁酸的含量高于丙酸[24],这应该与家兔盲肠中高丰度的毛螺旋菌科有关。
本试验检测结果显示,在30~51日龄期间,拟杆菌门中的拟杆菌属、Barnesiellae、普雷沃氏菌属是家兔盲肠中的优势菌属。有研究发现,拟杆菌属的脆弱拟杆菌能促进蚓突及整个肠道免疫系统的发育[25]。本试验显示,断奶后幼兔盲肠中拟杆菌属与Parabacteroides属相对丰度均显著下降,说明它们易受到断奶应激的抑制,这也可能是导致断奶幼兔容易腹泻的重要原因。白秀娟等[9]发现家兔65日龄时盲肠中拟杆菌含量高于55日龄组,说明断奶后随着幼兔对饲粮和环境的适应,盲肠中拟杆菌的数量又会逐渐升高并趋于平衡。因此,如何维持或尽快恢复断奶后肠道中拟杆菌属与Parabacteroides的稳定将是我们后续需要研究的内容。
Gouet等[26]报道,利用纯培养方法没有在家兔肠道中检测到乳杆菌属,而Zhu等[13]及张煜坤[14]利用实时定量PCR检验的结果表明家兔盲肠中存在乳杆菌属,本试验也检测到家兔盲肠中存在乳杆菌属。同Zhu等[13]研究结果的一致性证实了Illumina MiSeq测序技术在检测低丰度菌及反映肠道菌群结构上的可靠性,同时也佐证了通过培养方法检测肠道菌群结构的局限性。乳酸菌是猪、家禽及人类肠道中的优势菌群,多种乳酸菌被开发为益生菌[27,28]。菌属丰度分析表明,乳杆菌属不是家兔肠道中优势菌属,但其在每个个体中均存在,且相对丰度在不同周龄保持稳定(0.40%~0.53%)。乳酸杆菌对家兔肠道功能的影响,以及是否可开发为家兔的益生菌有待进一步研究。
本研究结果显示,家兔盲肠的优势菌属中包括4种未知菌属,它们在盲肠中的相对丰度较高(4种菌属相对丰度总和在24.37%~31.40%),是盲肠中正常菌群的重要组成部分,序列比对发现,在瘤胃及小鼠和人的粪便中也存在与这4种未知菌属高度相似的序列,对这几种未知菌属的鉴定及功能进行深入研究具有重要意义。
4 结 论center① 幼兔盲肠微生物区系在断奶时已基本形成,且以拟杆菌属相对丰度最大,但在断奶至断奶后3周龄期间随日龄的增长种类趋于复杂。
② 断奶至断奶后3周期间幼兔盲肠内优势菌门为厚壁菌门>拟杆菌门>变形菌门>疣微菌门;优势菌科为毛螺旋菌科>瘤胃球菌科>拟杆菌科>紫单胞菌科>普雷沃氏菌科>疣微菌科;断奶后优势菌属为未知菌属、拟杆菌属、Barnesiella、Akkermansia和普雷沃氏菌属。
③ 幼兔盲肠内存在乳杆菌属,且其相对丰度保持稳定。
| [1] | 卡卢金.家兔营养生理学[M]. 文光华,译.北京:农业出版社,1984. ( 1) 1)
|
| [2] | DI MAURO A,NEU J,RIEZZO G,et al.Gastrointestinal function development and microbiota[J]. Italian Journal of Pediatrics,2013,39:15. ( 1) 1)
|
| [3] | GUARMER F,MALAGELADA J R.Gut flora in health and disease[J]. The Lancet,2003,361(9356):512-519. ( 1) 1)
|
| [4] | SKŘIVANOVÁ E,MOLATOVÁ Z,SKŘIVANOVÁ V,et al.Inhibitory activity of rabbit milk and medium-chain fatty acids against enteropathogenic Escherichia coli O128[J]. Veterinary Microbiology,2009,135(3/4):358-362. ( 2) 2)
|
| [5] | HOTCHKISS C E,MERRITT A M.Evaluation of cecal ligation as a model of mucoid enteropathy in specific-pathogen-free rabbits[J]. Laboratory Animal Science,1996,46(2):174-178. ( 2) 2)
|
| [6] | FONTY G,GOUET PH.Fibre-degrading microorganisms in the monogastric digestive tract[J]. Animal Feed Science and Technology,1989,23(1/2/3):91-107. ( 1) 1)
|
| [7] | HARASZTHY V I,ZAMBON J J,SREENIVASAN P K,et al.Identification of oral bacterial species associated with halitosis[J]. The Journal of the American Dental Association,2007,138(8):1113-1120. ( 1) 1)
|
| [8] | COMBES S,MICHELLAND R J,MONTEILS V,et al.Postnatal development of the rabbit caecal microbiota composition and activity[J]. FEMS Microbiology Ecology,2011,77(3):680-689. ( 3) 3)
|
| [9] | 白秀娟,刘诚刚,杜智恒,等.PCR-DGGE技术分析断奶仔兔肠道微生物菌群结构及多样性[J]. 东北农业大学学报,2012,43(9):64-69. ( 4) 4)
|
| [10] | ESHAR D,WEESE J S.Molecular analysis of the microbiota in hard feces from healthy rabbits (Oryctolagus cuniculus) medicated with long term oral meloxicam[J]. BMC Veterinary Research,2014,10:62. ( 2) 2)
|
| [11] | HUYBENS N,HOUEIX J,LICOIS D,et al.Pyrosequencing of epizootic rabbit enteropathy inocula and rabbit caecal samples[J]. The Veterinary Journal,2013,196(1):109-110. ( 1) 1)
|
| [12] | MONTEILS V,CAUQUIL L,COMBES S,et al.Potential core species and satellite species in the bacterial community within the rabbit caecum[J]. FEMS Microbiology Ecology,2008,66(3):620-629. ( 1) 1)
|
| [13] | ZHU K H,XU X R,SUN D F,et al.Effects of drinking water acidification by organic acidifier on growth performance,digestive enzyme activity and caecal bacteria in growing rabbits[J]. Animal Feed Science and Technology,2014,190:87-94. ( 3) 3)
|
| [14] | 张煜坤.脆弱拟杆菌与枯草芽孢杆菌对仔兔肠道微生物区系及免疫的影响[D]. 硕士学位论文.杨凌:西北农林科技大学,2014. ( 2) 2)
|
| [15] | COCK P J A,FIELDS C J,GOTO N,et al.The Sanger FASTQ file format for sequences with quality scores,and the Solexa/Illumina FASTQ variants[J]. Nucleic Acids Research,2010,38(6):1767-1771. ( 1) 1)
|
| [16] | CAPORASO J G,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J]. Nature Methods,2010,7(5):335-336. ( 1) 1)
|
| [17] | EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics,2010,26(19):2460-2461. ( 1) 1)
|
| [18] | WANG Q,GARRITY G M,TIEDJE J M,et al.Nave Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied and Environmental Microbiology,2007,73(16):5261-5267. ( 1) 1)
|
| [19] | 朱岩丽.日粮纤维/淀粉对生长肉兔生长发育、免疫应答、肠道菌群的影响及肠道蛋白质组学初探[D]. 博士学位论文.泰安:山东农业大学,2013. ( 1) 1)
|
| [20] | FONTY G,CHAUCHEYRAS-DURAND F.Les Ècosystèmes Digestifs[M]. Pairs:Tech & Doc Publishing,2007. ( 1) 1)
|
| [21] | BÄUERL C,COLLADO M C,ŹÑIGA M,et al.Changes in cecal microbiota and mucosal gene expression revealed new aspects of epizootic rabbit enteropathy[J]. PLoS One,2014,9(8):e105707. ( 1) 1)
|
| [22] | BENNEGADI-LAURENT N,GIDENNE T,LICOIS D.Nutritional and sanitary statuses alter postweaning development of caecal microbial activity in the rabbit[J]. Comparative Biochemistry and Physiology Part A:Molecular & Intergrative Physiology,2004,139(3):293-300. ( 1) 1)
|
| [23] | PRYDE S E,DUNCAN S H,HOLD G L,et al.The microbiology of butyrate formation in the human colon[J]. FEMS Microbiology Letters,2002,217(2):133-139. ( 1) 1)
|
| [24] | MICHELLAND R J,COMBES S,MONTEILS V,et al.Rapid adaptation of the bacterial community in the growing rabbit caecum after a change in dietary fibre supply[J]. Animal,2011,5(11):1761-1768. ( 1) 1)
|
| [25] | RHEE K J,SETHUPATH P,DRUKS A,et al.Role of commensal bacteria in development of gut-associated lymphoid tissues and preimmune antibody repertoire[J]. The Journal of Immunology,2004,172(2):1118-1124. ( 1) 1)
|
| [26] | GOUET PH,FONTY G.Changes in the digestive microflora of holoxenic rabbits from birth until adulthood[J]. Annales de Biologie Animale Biochimie Biophysique,1979,19(3A):553-566. ( 1) 1)
|
| [27] | 楚青惠,曾勇庆,汪官保,等.饲喂不同剂量乳酸菌对保育仔猪生产性能及血清生化指标的影响[J]. 中国畜牧杂志,2014,50(15):59-62. ( 1) 1)
|
| [28] | LIÉVIN-LE MOAL V,SERVIN A L.Anti-infective activities of lactobacillus strains in the human intestinal microbiota:from probiotics to gastrointestinal anti-infectious biotherapeutic agents[J]. Clinical Microbiology Reviews,2014,27(2):167-199. ( 1) 1)
|




