2. 牛低碳养殖与安全生产四川省高校重点实验室, 雅安 625014
2. Key Laboratory for Low-Carbon Farming and Safety Production in Cattle of Sichuan Province, Ya'an 625014, China
随着人们生活水平的提高,人们对畜产品需求量也不断增加,以及世界气象组织对气候变化和生态环境变化的重视,要求畜牧业在不降低动物生产效率的同时,尽可能地减少温室气体排放和保护生态环境。在我国,肉牛养殖成本从2001年的1 500元/头增加到2009年的4 500元/头,其中饲养成本对养殖成本增加的贡献率为27%[1],而2014年的饲养成本已高达5 500元/头。在肉牛生产体系中,饲料成本是肉牛生产成本中变化最大的可调控成本,因此通过提高动物饲料效率,降低肉牛饲料成本,提高整个肉牛生产体系的效益,为肉牛养殖的可持续发展提供保障。
国内外通常采用采食量与体增重的比值——料重比(feed intake/gain,F/G)来评定饲料效率,但是,料重比不能解释不同体型和不同生长速率的动物之间饲料效率的固有差异。早在1963年Koch等[2]提出用剩余采食量(residual feed intake,RFI)来更有效地评定肉牛饲料效率,RFI是指在特定测量期间每头动物的真实采食量与通过维持需要和生产需求所得的预期采食量之间的差值。Herd等[3]指出可用RFI比较不同生产水平动物的个体差异,尤其是在低利润率和高饲料成本阶段,可有效改进饲粮配方和饲养管理。瘤胃是反刍动物特有的消化器官,在消化代谢中起着极其重要的作用,是反刍动物健康高效养殖的基础。瘤胃内栖息着复杂多样的非致病微生物,一方面,动物品种、年龄、健康状况、饲粮,以及抗生素的使用等均会影响瘤胃微生物菌群结构;另一方面瘤胃微生物菌群结构又通过影响瘤胃内环境,并影响饲料的降解利用效率[4]。因此本文就对最近几年国外采用PCR-变性梯度凝胶电泳(DGGE)、文库克隆测序、实时定量PCR和高通量测序技术研究肉牛RFI表型与瘤胃中的微生物菌群结构之间的关系所取得的进展进行综述。
1 RFI的定义及主要预测模型 1.1 RFI的定义RFI被定义为动物的实际采食量与根据其维持和生产(生长、泌乳和产仔等)所计算的预期采食量的之间差值,是用来衡量动物饲料效率的有效指标[5]。在测定肉牛RFI时,通过持续饲喂肉牛63~84 d,在饲养期间准确记录每头/组牛每天的干物质采食量(DMI)和平均日增重(ADG)[6]。饲养试验结束后,利用回归模型计算出肉牛的预期采食量,实际记录的采食量减去预期采食量即为RFI。RFI是一个具有中等遗传力的负向选择性状,其遗传力为0.18~0.43[7],饲料效率越高的肉牛,其RFI越低。已有研究表明,RFI与饲料转化率和DMI 2个重要经济性状显著相关,而与ADG相关性不显著[8, 9],说明RFI是独立于动物的体增重和生长速度。
一般根据动物的RFI对动物进行分组,Nkrumah等[10]根据动物的RFI平均值和标准差(SD),将动物的RFI>平均值+0.5SD定为高RFI(H-RFI)动物,即低饲料效率动物;将RFI<平均值-0.5SD定为低RFI(L-RFI)动物,即高饲料效率动物。而Ramos等[11]是将高于平均值1倍标准差的动物定义为H-RFI(RFI>平均值+1SD),L-RFI动物则是RFI<平均值-1SD。虽然其划分标准略有不同,但都能涵盖最低和最高RFI的动物,不同的划分标准可能会使H-RFI和L-RFI动物在某些层面上达到显著的水平不同。
1.2 RFI的主要预测模型用于估测RFI的模型较多,不同品种动物会采用不同的模型,目前主要的RFI估测模型有以下4种:第1种是Koch等[2]1963年提出的RFI估测模型,即RFI1963。Yi=β0+β1(ADGi)+β2MWTi+ei。式中,Yi为动物i的采食量,β0为回归截距,β1为采食量对ADG的偏回归系数,MWTi为动物i的平均中期体重,β2为采食量对MWTi的偏回归系数,ei为动物i的采食量残差,即RFI。第2种是目前澳大利亚用于估测肉牛RFI的模型,即RFIB。Yi=β0+β1ADGi+β2MMWTi+ei。式中,Yi、β0、β1和ei代表的意义同RFI1963模型,MMWTi为平均中期代谢体重(W0.73),β2为采食量对MMWTi的偏回归系数。第3种模型是澳大利亚提出的,其中计算饲料干物质预测采食量所采用的公式是澳大利亚农业常务委员会(Standing Committee on Agriculture,SCA)1990年公布的通用公式,由于总的能量需要是维持和增重的能量需要之和,其中增重需要(Egain,MJ/d)={EBG×[(20.3-R)/1+exp(2.4-6P)]+(6.7+R)}/Kg。式中,EBG为空腹增重,按0.92×ADG(kg/d)计算,R为校正体重增加或减少的量,按250×EBG/(SRW0.75)-1计算(SRW为标准参考体重),P为生长阶段,按ALW/SRW计算(ALW为测试期的平均活体重),Kg为用于增重的代谢能(ME)利用效率;维持需要(Emaint,MJ/d)=K×0.28×ALW0.75×exp(-0.03A)/(Km+0.1×Egain)。式中,对于普通牛K=1.4,贝尔蒙红牛和圣达牛K=1.3,婆罗门牛K=1.2,A为年龄,Km为用于维持的ME利用效率[12],然后根据总的能量需要和饲料能量浓度,计算出饲料干物质预测采食量,RFI就是平均实际日采食量与根据维持需要和增重需要预测的采食量之间的差值,标记为RFISCA。而第4种模型是包含体组成的模型,即RFIF。Yi=β0+β1ADGi+β2W(mid-test)i+β3FD+β4MD+ei[13]。式中:Yi、β0、β1和ei代表的意义同RFI1963模型,W(mid-test)i为动物i试验中期体重,β2为采食量对W(mid-test)i的偏回归系数,FD为背膘厚度,β3采食量对FD的偏回归系数,MD为眼肌厚度,β4为采食量对MD的偏回归系数。
Knott等[14]用这4种RFI模型估测肉羊的RFI,尽管每个模型的估测的RFI略有不同,但各个模型与动物表型和排序的相关性表明模型之间是近似的。
2 RFI与瘤胃微生物菌群变化反刍动物瘤胃内栖息着种类繁多、数量庞大的微生物,主要包括瘤胃厌氧菌(1010~1011个/mL)、单细胞纤毛原虫(104~106个/mL)、真菌(游动孢子103~105个/mL)和噬菌体(108~109个/mL)[15],以及产甲烷菌等古菌(占胃肠道总菌0.3%~3.3%)[16]。这些瘤胃微生物具有高度特异性,并执行特定的代谢功能,适宜的微生物菌群组成有利于饲料在瘤胃中发酵降解,瘤胃发酵产生的挥发性脂肪酸(VFA)可为反刍动物提供约70%的能量,对反刍动物生长发育和营养健康起着关键作用[17]。而宿主基因型、健康状况、生活环境和饲粮组成又在一定程度上影响瘤胃微生物的菌群结构及其代谢活性[4]。Nkrumah等[10]和Hegarty等[18]的研究表明,不同RFI肉牛的能量代谢显著不同,而且反刍动物消化和瘤胃发酵过程可解释19%的RFI变化[3],这些表明瘤胃微生物种群组成可能与肉牛的RFI表型存在一定关系。
2.1 RFI与瘤胃菌群Guan等[19]运用PCR-DGGE技术首次考察了18头不同RFI肉牛在饲喂高能量饲粮(ME 12.18 MJ/kg)时瘤胃中的细菌组成差异,发现在L-RFI组中聚集的细菌菌谱相对紧凑,且与H-RFI组中的菌谱相分隔,且L-RFI组中肉牛的瘤胃细菌组成的相似百分率(91%)高于H-RFI组 (71%),表明某些特定细菌可能只出现在L-RFI 或H-RFI肉牛瘤胃中。通过比较9头不同RFI安格斯肉牛的瘤胃细菌组成可见,H-RFI和L-RFI肉牛的菌谱明显不同(图1)。
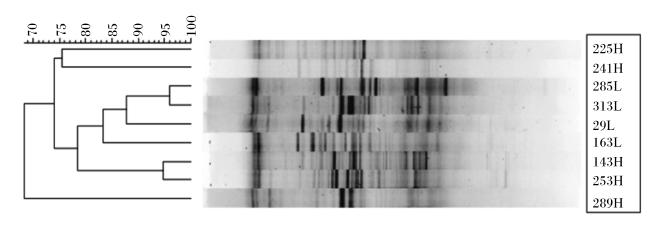 | 图1 9头安格斯牛瘤胃液DNA的PCR-DGGE菌谱 Fig. 1 PCR-DGGE profiles generated from ruminal fluid DNA from 9 Angus steers[19] |
Hernandez-Sanabria等[20]采用PCR-DGGE和克隆测序技术考察了58头海福特安格斯肉牛在饲喂低能饲粮(ME 10.88 MJ/kg)时瘤胃中细菌群落变化。结果显示,PCR-DGGE菌谱条带出现频率有2个集中区域,其中未培养的毛螺菌科杆菌属、栖瘤胃普雷沃菌、未培养的普氏菌sp. T0505克隆,瘤胃球菌属bromii YE282菌株和2种未被成功鉴定的菌只出现在H-RFI肉牛中,而瘤胃球菌属gauvreauii CCRI菌株和1种未被成功鉴定的菌只出现在L-RFI肉牛中,表明瘤胃中的这些细菌可能与肉牛的RFI表型,即饲料效率相关。Hernandez-Sanabria等[21]先给55头海福特安格斯公牛饲喂90 d低能饲粮(ME 10.88 MJ/kg),然后再提供90 d的高能饲粮(ME 12.18 MJ/kg),发现在2种饲粮下,肉牛根据RFI的排序有所不同。其PCR-DGGE菌谱显示,在高能饲粮下,肉牛瘤胃中的菌落倾向于根据其RFI表型集群,通过对条带进行克隆测序和实时定量PCR分析发现,琥珀酸弧菌属(Succinivibrio sp.)、优杆菌属(Eubacterium sp.)和Robinsoniella sp.这3个特定菌落与RFI相关。琥珀酸弧菌属的主要发酵产物是乙酸和琥珀酸(丙酸的前体物)[4],它可能在丙酸生成中起重要作用,通过瘤胃壁吸收后进入肝脏的丙酸是糖异生主要前提物质,进而调节宿主的能量代谢,所以在低能量饲粮下,L-RFI组(饲料效率高)的肉牛瘤胃中琥珀酸弧菌拷贝数高于H-RFI组(饲料效率低)肉牛。优杆菌属是目前只从人和猪肠道中分离到的,可利用乙酸,并生成丁酸[22]。当L-RFI组肉牛从低能饲粮转变为高能饲粮时,优杆菌属是最主要的细菌,可能这种菌可以承受较低的pH[23]。因此,在L-RFI肉牛中,优杆菌属利用的乙酸可能与琥珀酸弧菌属生成的乙酸相互作用,这个过程需要消耗更多的氢,从而降低甲烷生成量,提高饲料效率。而Robinsoniella sp.的功能目前尚不清楚,Cotta等[24]揭示乙酸和琥珀酸是其主要的代谢终产物,也生成少量的甲酸和乳酸盐。
Carberry等[25]给28头不同RFI肉牛(H-RFI,n=14;L-RFI,n=14)饲喂44 d低能高粗料(HF)饲粮后,经过56 d放牧饲养,随后饲喂35 d高能低粗料(LF)饲粮,用PCR-DGGE技术考察不同RFI肉牛在不同粗料水平下的瘤胃菌群组成。发现饲喂HF饲粮时,L-RFI和H-RFI肉牛的PCR-DGGE细菌谱带存在2个相分隔的独立区系,而在LF饲粮下不存在,所以在HF饲粮下,肉牛瘤胃中的细菌菌落倾向于根据其RFI表型进行分区集群。采用实时定量PCR发现,L-RFI组普雷沃菌属(Prevotella)的相对丰度低于H-RFI组。在HF饲粮下,L-RFI组白色瘤胃球菌(R. albus)的含量高于H-RFI组,表明给肉牛提供高粗料饲粮时,L-RFI肉牛对纤维饲料的利用率可能高于H-RFI肉牛;而在LF饲粮下,差异不显著,RFI表型和饲粮对R. albus有显著的互作效应。McCann等[26]利用454焦磷酸测序技术考察肉牛RFI表型在不同载畜量牧场饲养条件下其瘤胃细菌菌群组成,发现普雷沃菌属是最主要的优势菌属,H-RFI组中普雷沃菌属的相对丰度显著高于L-RFI;而在L-RFI组中有1种未被鉴定的拟杆菌科的相对丰度显著高于H-RFI组。Hernandez-Sanabria等[21]也发现未培养的普雷沃菌sp. T0505克隆只出现在H-RFI肉牛中,表明普雷沃菌丰度在不同RFI肉牛中可能存在遗传差异。目前瘤胃中的普雷沃菌属包括栖瘤胃普雷沃氏菌(P. ruminicola)、短普雷沃氏菌(P. brevis)、布氏普雷沃氏菌(P. bryantii)和P. albensis 4种菌[27],这些菌可以利用淀粉、非纤维多糖(木聚糖和果胶)和单糖为能量来源,以多肽和氨为氮源进行生长繁殖,其发酵产物包括乙酸、琥珀酸和丙酸,其中琥珀酸是最主要的发酵终产物[28]。在饲喂粗料或精料的动物中,普雷沃氏菌都是瘤胃中的主要优势菌,H-RFI肉牛瘤胃中的普雷沃菌属的相对丰度高于L-RFI动物,暗示普雷沃菌属中不同菌种在降解代谢饲粮中的淀粉、蛋白质、肽类、半纤维素和果胶时,其活性和发酵途径可能存在差异,可能在瘤胃微生物遗传和代谢多样性方面起重要作用。
2.2 RFI与瘤胃甲烷菌群瘤胃中的甲烷菌可利用氢气(H2)和碳底物,主要是二氧化碳(CO2)、乙酸、甲酸、乙醇或甲醇,生成甲烷。这个过程一方面可维持瘤胃的低氢压,促进瘤胃产氢菌对饲料的降解,另一方面却引起饲粮能量损失(2%~12%)[29],且排放的甲烷对全球温室气体“贡献率”达13%~19%[30],是畜牧业方面影响全球变暖主要因素。研究发现,L-RFI牛只的甲烷排放量比H-RFI肉牛减少15%~30%,粪污产量减少15%~20%[10, 18],推断瘤胃中的甲烷菌群组成与肉牛RFI表型间可能存在一定关系。
Zhou等[31]通过16S rRNA克隆文库测序技术考察了L-RFI组(n=29)和H-RFI组(n=29)肉牛在采食低能量饲粮时瘤胃中甲烷菌菌群差异,结果2组动物共享18个分类操作单元(OTU,占OTU总数的58.06%),L-RFI组和H-RFI组分别独占4个和9个OTU,可见H-RFI组甲烷菌种别多样性高于L-RFI组,而反刍兽甲烷短杆菌(Methanobrevibacter ruminantium)是2组动物的主要优势甲烷菌。用实时定量PCR测得H-RFI组中的Methanosphaera stadtmanae和Methanobrevibacter sp. AbM4菌株的拷贝数均显著高于L-RFI组,但总甲烷菌拷贝数差异不显著。Zhou等[32]先给56头肉牛饲喂90 d的低能饲粮,根据其RFI将肉牛分为H-RFI组(n=20)和L-RFI组(n=22)。接着又饲喂90 d的高能饲粮,将动物重新划分为H-RFI组(n=14)和L-RFI组(n=19)。用PCR-DGGE技术分析发现从L-RFI组转变到H-RFI组肉牛,出现Methanobrevibacter smithii PS、Methanobrevibacter sp. AbM4和Methanobrevibacter smithii SM9 3种之前没有的甲烷菌;从H-RFI组转变到L-RFI组肉牛中出现了Methanobrevibacter thaueri strain CW。另外,有3种菌只出现在L-RFI组,2种菌只出现在H-RFI组,其中已鉴定的1种是Methanobrevibacter smithii PS,可见该菌株与肉牛的饲料效率相关的。由于多条DGGE带代表同一种菌,表明甲烷菌在菌种、菌株和基因型水平上的结构差异可能与宿主饲料效率相关。所以在考虑甲烷菌对肉牛瘤胃功能影响时,不仅要考虑饲粮结构差异,还要考虑甲烷菌的菌株和基因型的多样性。
Carberry等[33]通过16S rRNA文库克隆测序技术研究不同RFI肉牛在高、低粗料饲粮条件下瘤胃中的甲烷菌组成,结果发现Methanobrevibacter是瘤胃中的优势菌属,主要包括Methanobrevibacter smithii、Methanobrevibacter ruminantium和Methanosphaera stadtmanae。又用454焦磷酸测序技术比较分析7个最高RFI和7个最低RFI肉牛瘤胃中的甲烷菌群组成,结果显示2个表型组共享所有OTU,表明2组动物的甲烷菌菌群具有高度相似性,但H-RFI组中Methanobrevibacter smithii的2个基因型丰度显著高于L-RFI组。另外Methanobrevibacter smithii和Methanosphaera stadtmanae的基因型丰度在高粗料和低粗料饲粮下也不同。尽管核心甲烷菌群的OTU在不同RFI的动物和不同饲粮下均存在,但这些OTU的基因型分布存在显著差异,所以肉牛瘤胃中这些基因型丰度的变化可能导致不同RFI动物的甲烷排放量不同。
因此不同RFI肉牛瘤胃中的甲烷菌在菌种、菌株和基因型,甚至基因亚型水平上的多样性和丰度差异可能会导致其能量利用和甲烷排放量不同。在H-RFI动物瘤胃中的Methanobrevibacter smithii、Methanosphaera stadtmanae和Methanobrevibacter sp. AbM4基因型丰度有高于L-RFI动物的倾向,由于Methanobrevibacter smithii可以利用CO2-H2和/或甲酸生成甲烷,还包含参与甲醇-乙醇途径的相关酶[34],Methanosphaera stadtmanae只能通过H2还原甲醇生成甲烷,甲醇是原虫和具有酯化酶活性的细菌水解果胶的产物[35]。Methanobrevibacter sp. AbM4生成甲烷所偏好底物还不清楚,由于其拷贝数与乙酸浓度存在显著的的负相关关系,表明它可能利用乙酸生成甲烷[32],暗示这些甲烷菌以有机物为底物的甲烷生成途径也可能导致动物的饲料效率不同。瘤胃中的甲酸、乙酸、甲醇、乙醇等有机物浓度与瘤胃中的某些细菌组成和活性息息相关,如琥珀酸弧菌、优杆菌、普雷沃菌均可产生乙酸,琥珀酸弧菌还能生成甲酸,普雷沃菌可利用果胶生成甲醇,所以不同基因型的甲烷菌与瘤胃中的主要微生物群间互作关系也可能与肉牛的RFI表型相关。
3 小 结RFI高低影响了瘤胃微生物菌群组成结构,以及瘤胃发酵模式和VFA生成,采用合适的RFI预测模型指导生产,有助于促进肉牛精准饲养。瘤胃中的普雷沃菌、琥珀酸弧菌、优杆菌通过调节宿主的能量代谢影响其饲料利用效率,甲烷菌在菌株和基因型丰度上差异也可能与宿主饲料效率和甲烷排放量相关。应进一步分离培养普雷沃菌属、琥珀酸弧菌属、优杆菌属中菌株,在全基因组测序基础上研究菌种组成与肉牛饲料效率间的关系。另外有必要进一步了解不同基因型的甲烷菌与瘤胃中的主要微生物群间互作关系,探清甲烷菌的基因型在饲料效率和甲烷生成方面扮演的角色,为寻找有效的甲烷减排措施提供理论基础。
| [1] | 韩洁,曹建民.新时期农户肉牛养殖成本与利润变动及政策研究[J]. 中国畜牧杂志,2012,48(4):58-62. ( 1) 1)
|
| [2] | KOCH R M,SWIGER L A,CHAMBERS D,et al.Efficiency of feed use in beef cattle[J]. Journal of Animal Science,1963,22(2):486-494. ( 2) 2)
|
| [3] | HERD R M,ARTHUR P F.Physiological basis for residual feed intake[J]. Journal of Animal Science,2009,87(14S):E64-E71. ( 2) 2)
|
| [4] | STEWART C S,FLINT H J,BRYANT M P.The rumen bacteria[M]//HOBSON P N,STEWART C S.The Rumen Microbial Ecosystem.Netherlands:Springer,1997:10-72 ( 3) 3)
|
| [5] | BASARAB J A,PRICE M A,AALHUS J L,et al.Residual feed intake and body composition in young growing cattle[J]. Canadian Journal of Animal Science,2003,83(2):189-204. ( 1) 1)
|
| [6] | WANG Z,NKRUMAH J D,LI C,et al.Test duration for growth,feed intake,and feed efficiency in beef cattle using the Growsafe System[J]. Journal of Animal Science,2006,84(9):2289-2298. ( 1) 1)
|
| [7] | CROWLEY J J,MCGEE M,KENNY D A,et al.Phenotypic and genetic parameters for different measures of feed efficiency in different breeds of irish performance-tested beef bulls[J]. Journal of Animal Science,2010,88(3):885-894. ( 1) 1)
|
| [8] | BAKER S D,SZASZ J I,KLEIN T A,et al.Residual feed intake of purebred angus steers:effects on meat quality and palatability[J]. Journal of Animal Science,2006,84(4):938-945. ( 1) 1)
|
| [9] | KELLY A K,MCGEE M,CREWS D H,Jr.,et al.Effect of divergence in residual feed intake on feeding behavior,blood metabolic variables,and body composition traits in growing beef heifers[J]. Journal of Animal Science,2010,88(1):109-123. ( 1) 1)
|
| [10] | NKRUMAH J D,OKINE E K,MATHISON G W,et al.Relationships of feedlot feed efficiency,performance,and feeding behavior with metabolic rate,methane production,and energy partitioning in beef cattle[J]. Journal of Animal Science,2006,84(1):145-153. ( 3) 3)
|
| [11] | RAMOS M H,KERLEY M S.Mitochondrial complex Ⅰ protein differs among residual feed intake phenotype in beef cattle[J]. Journal of Animal Science,2013,91(7):3299-3304. ( 1) 1)
|
| [12] | ROBINSON D L,ODDY V H.Genetic parameters for feed efficiency,fatness,muscle area and feeding behaviour of feedlot finished beef cattle[J]. Livestock Production Science,2004,90(2/3):255-270. ( 1) 1)
|
| [13] | FRANÇOIS D,BIBÉ B,BOUIX J,et al.Genetic parameters of feeding traits in meat sheep[C]//Proceedings of the 7th world congress on genetics applied to livestock production.Montpellier,France:Institut National dela Recherche Agronomique(INRA),2002.( 1) 1)
|
| [14] | KNOTT S A,CUMMINS L J,DUNSHEA F R,et al.The use of different models for the estimation of residual feed intake (RFI) as a measure of feed efficiency in meat sheep[J]. Animal Feed Science and Technology,2008,143(1/2/3/4):242-255. ( 1) 1)
|
| [15] | KAMRA D.Rumen microbial ecosystem[J]. Current Science,2005,89(1):124-135. ( 1) 1)
|
| [16] | JANSSEN P H,KIRS M.Structure of the archaeal community of the rumen[J]. Applied and Environmental Microbiology,2008,74(12):3619. ( 1) 1)
|
| [17] | MORGAVI D P,FORANO E,MARTIN C,et al.Microbial ecosystem and methanogenesis in ruminants[J]. Animal,2010,4(7):1024-1036. ( 1) 1)
|
| [18] | HEGARTY R S,GOOPY J P,HERD R M,et al.Cattle selected for lower residual feed intake have reduced daily methane production[J]. Journal of Animal Science,2007,85(6):1479-1486. ( 2) 2)
|
| [19] | GUAN L L,NKRUMAH J D,BASARAB J A,et al.Linkage of microbial ecology to phenotype:correlation of rumen microbial ecology to cattle's feed efficiency[J]. FEMS Microbiology Letters,2008,288(1):85-91. ( 2) 2)
|
| [20] | HERNANDEZ-SANABRIA E,GUAN L L,GOONEWARDENE L A,et al.Correlation of particular bacterial PCR-denaturing gradient gel electrophoresis patterns with bovine ruminal fermentation parameters and feed efficiency traits[J]. Applied and Environmental Microbiology,2010,76(19):6338-6350. ( 1) 1)
|
| [21] | HERNANDEZ-SANABRIA E,GOONEWARDENE L A,WANG Z Q,et al.Impact of feed efficiency and diet on adaptive variations in the bacterial community in the rumen fluid of cattle[J]. Applied and Environmental Microbiology,2012,78(4):1203-1214. ( 2) 2)
|
| [22] | FLINT H J,DUNCAN S H,SCOTT K P,et al.Interactions and competition within the microbial community of the human colon:links between diet and health[J]. Environmental Microbiology,2007,9(5):1101-1111. ( 1) 1)
|
| [23] | DUNCAN S H,LOUIS P,THOMSON J M,et al.The role of pH in determining the species composition of the human colonic microbiota[J]. Environmental Microbiology,2009,11(8):2112-2122. ( 1) 1)
|
| [24] | COTTA M A,WHITEHEAD T R,FALSEN E,et al.Robinsoniella peoriensis gen. nov.,sp. nov.,isolated from a swine-manure storage pit and a human clinical source[J]. International Journal of Systematic and Evolutionary Microbiology,2009,59(1):150-155. ( 1) 1)
|
| [25] | CARBERRY C A,KENNY D A,HAN S,et al.Effect of phenotypic residual feed intake and dietary forage content on the rumen microbial community of beef cattle[J]. Applied and Environmental Microbiology,2012,78(14):4949-4958. ( 1) 1)
|
| [26] | MCCANN J C,WILEY L M,FORBES T D,et al.Relationship between the rumen microbiome and residual feed intake-efficiency of brahman bulls stocked on bermudagrass pastures[J]. PLoS One,2014,9(3):e91864. ( 1) 1)
|
| [27] | AVGUŠTIN G,WALLACE R J,FLINT H J.Phenotypic diversity among ruminal isolates of Prevotella ruminicola:proposal of Prevotella brevis sp. nov.,Prevotella bryantii sp. nov.,and Prevotella albensis sp. nov. and redefinition of Prevotella ruminicola[J]. International Journal of Systematic Bacteriology,1997,47(2):284-288. ( 1) 1)
|
| [28] | PURUSHE J,FOUTS D E,MORRISON M,et al.Comparative genome analysis of Prevotella ruminicola and Prevotella bryantii:insights into their environmental niche[J]. Microbial Ecology,2010,60(4):721-729. ( 1) 1)
|
| [29] | JOHNSON K A,JOHNSON D E.Methane emissions from cattle[J]. Journal of Animal Science,1995,73(8):2483-2492. ( 1) 1)
|
| [30] | LASSEY K R,ULYATT M J,MARTIN R J,et al.Methane emissions measured directly from grazing livestock in New Zealand[J]. Atmospheric Environment,1997,31(18):2905-2914. ( 1) 1)
|
| [31] | ZHOU M,HERNANDEZ-SANABRIA E,GUAN L L.Assessment of the microbial ecology of ruminal methanogens in cattle with different feed efficiencies[J]. Applied and Environmental Microbiology,2009,75(20):6524-6533. ( 1) 1)
|
| [32] | ZHOU M,HERNANDEZ-SANABRIA E,GUAN L L.Characterization of variation in rumen methanogenic communities under different dietary and host feed efficiency conditions,as determined by PCR-denaturing gradient gel electrophoresis analysis[J]. Applied and Environmental Microbiology,2010,76(12):3776-3786. ( 2) 2)
|
| [33] | CARBERRY C A,WATERS S M,KENNY D A,et al.Rumen methanogenic genotypes differ in abundance according to host residual feed intake phenotype and diet type[J]. Applied and Environmental Microbiology,2014,80(2):586-594. ( 1) 1)
|
| [34] | SAMUEL B S,HANSEN E E,MANCHESTER J K,et al.Genomic and metabolic adaptations of Methanobrevibacter smithii to the human gut[J]. Proceedings of the National Academy of Sciences of the United States of America,2007,104(25):10643-10648. ( 1) 1)
|
| [35] | MILLER T L,WOLIN M J.Methanosphaera stadtmaniae gen. nov.,sp. nov.:a species that forms methane by reducing methanol with hydrogen[J]. Archives of Microbiology,1985,141(2):116-122. ( 1) 1)
|




