2. 动物疫病与人类健康四川省重点实验室, 雅安 625014
2. Key Laboratory of Animal Disease and Human Health of Sichuan Province, Ya'an 625014, China
反刍动物瘤胃中栖息着大量细菌、原虫、真菌和古菌,其中,瘤胃细菌能有效地利用纤维素等物质并转换成动物产品而成为研究热点[1]。然而,因瘤胃液相和固相内容物的化学组成不同[2]而使其中的微生物数量和种类存在一定的差异。目前,尽管少数研究学者已应用PCR-变性梯度凝胶电泳(DGGE)技术和实时定量PCR(real-time PCR)技术研究了反刍动物瘤胃液相和固相内容物细菌的差异[3, 4],但研究技术的局限使获得的信息量少且不够深入。因此,深入而详尽地研究瘤胃液相和固相细菌具有非常重要的意义。
近年来,以Roche公司的454[5]、ABI公司的SOLiD[6]和Illumina公司的Solexa[7]为代表的第2代测序技术在微生物的研究中已得到广泛的应用,但这些方法仍存在测序数据量少且价格昂贵等缺点。2013年出现的Illumina Miseq测序平台测序通量高、数据完整性好、经济高效且快速而备受研究者们的青睐[8]。因此,本研究应用Illumina Miseq测序平台全面评估蒙古羊瘤胃液相和固相内容物菌群多样性,为今后反刍动物胃肠道微生物的研究提供有价值的理论指导。
1 材料与方法 1.1 试验动物在四川省雅安市名山县屠宰场选择同批次屠宰的5只(48.16±1.48) kg放牧成年雄性蒙古羊作为采样对象(记为羊1、2、3、4和5)。
1.2 试验材料 1.2.1 样品的采集无菌操作采集蒙古羊的瘤胃新鲜内容物,用4层灭菌纱布过滤获得液相样品(LAB)和固相样品(SAB),来自羊1~5的样品分别编号为LAB-1~LAB-5、SAB-1~SAB-5,每份采集5 mL或5 g至液氮速冻,置-80 ℃保存。
1.2.2 主要试剂和仪器Omega D40-15粪便基因组提取试剂盒(美国Omega公司);凝胶成像系统(美国Bio-Rad公司);高速冷冻离心机(美国Bio-Rad公司);NanoDrop ND-1000核酸蛋白浓度测定仪(美国Thermo公司);凝胶成像系统(美国Bio-Rad公司);高速冷冻离心机(美国Bio-Rad公司);Illumina MiSeq测序仪(美国Illumina公司);Pico Green核酸染料(美国Life Technologies公司);DNA胶回收试剂盒(美国Omega公司);P7589PicoGreen dsDNA定量试剂盒(美国Invitrogen公司);BioTek酶标仪(美国Bio-Rad公司);MiSeq测序试剂盒V2(深圳华大基因科技服务有限公司)。
1.3 试验方法 1.3.1 细菌总DNA的提取样品的预处理参照Zhou等[9]方法,用Omega D40-15试剂盒提取细菌总DNA,并用核酸浓度测定仪测定DNA浓度,置-20 ℃保存。
1.3.2 细菌基因组文库构建及上机测序细菌基因组文库构建及上机测序均由深圳华大基因科技服务有限公司完成。具体操作步骤如下:检测合格的DNA片段经电泳回收后,用T4 DNA Polymerase、Kleneow DNA Polymerase和T4 PNK将打断形成的黏性末端修复成平末端,再通过3′端加碱基“A”,使得DNA片段能与3'端带有“T”碱基的特殊接头连接。参照Caporaso等[10]的方法对样品细菌的16S rRNA基因V4区扩增DNA文库,用Agilent 2100对PCR富集片段进行质量控制,等摩尔混合构建文库并验证DNA文库的片段大小。最后将质控的测序文库用Illumina MiSeq平台进行Paired-end测序,测序读长为2×250 bp。
1.4 数据分析原始测序数据经质量控制(去除15 bp的接头污染reads、50 bp的低质量reads、含“N”及某个碱基连续出现的长度大于10 bp的低复杂度的序列)后获得纯净测序数据,通过COPE(connecting overlapped pair-end,v1.2.1)软件对序列进行拼接并去除上、下游引物[11];利用Usearch(http://www.drive5.com/usearch/)计算预聚类后的序列(Tags)在0.03(或97%的相似度)距下利用UPARSE-操作分类单元(operational taxonomic units,OTU)算法进行OTU的聚类[12],并统计获得OTU的数量;通过QIIME软件和mothur(v1.31.2)软件对OTU进行物种注释及丰度统计,并用R(v2.15.3)软件绘制主成分分析(PCA)图[13],同时计算alpha多样性,包括物种观察指数(observed species index)、赵氏指数(Chao index)、艾斯指数(ACE index)、香农指数(Shannon index)及辛普森指数(Simpson index),公式参考http://www.mothur.org/wiki/Calculators;经UniFrac算法利用系统进化的信息来比较样品间的物种群落差异,进行beta多样性(beta diversity)分析;通过QIIME(v1.50)软件,采用迭代算法对样品进行物种聚类,方法为UPGMA(unweighted pair group method with arithmetic mean);使用Metastats软件(http://metastats.cbcb.umd.edu/)进行组间显著性差异分析。
2 结果与分析 2.1 OTU聚类分析DNA质控后舍弃LAB-3、SAB-2和SAB-3。本研究测序样品数为7个,共获得91 971条序列,平均每个样品13 138条。序列经拼接优化后在0.97相似度条件下共获得15 321个物种分类的OTU,平均每个样品2 189个OTU。LAB-1、LAB-2、LAB-4、LAB-5的OTU分别为1 446、1 289、1 961和2 499个;SAB-1、SAB-4、SAB-5的OTU分别为3 319、1 988和2 819个。所有样品的平均读长均为252 bp。
图1显示,来自同一动物个体的液相和固相样品聚类在一起。PCA获得主成分1(PC1)的贡献率为29.75%,主成分2(PC2)的贡献率为28.59%;门水平分类上的丰度热图显示液相和固相样品的主要菌群来自拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、螺旋体门(Spirochaetes)、纤维杆菌门(Fibrobacteres)、变形菌门(Proteobacteria)、广古菌门(Euryarchaeota)、放线菌门(Actinobacteria)、软壁菌门(Tenericutes)、未分类门(unclassified)、互养菌门(Synergistetes)、疣微菌门(Verrucomicrobia)、黏胶球形菌门(Lentisphaerae)、蓝藻门(Cyanobacteria)和迷踪菌门(Elusimicrobia)。其中,拟杆菌门和厚壁菌门为优势菌群。
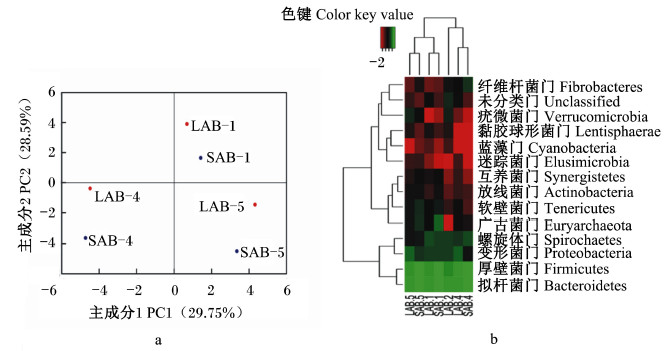 |
色键红至绿表示丰度低至高。 The change of red to green of color key indicate that the abundance change of low to high. 图1 瘤胃样品细菌OTU的主成分分析图(a)及其门水平分类上的热图(b) Fig. 1 PCA profile (a) and heatmap in phylum (b) of the liquid and solid samples from the rumen |
瘤胃液相和固相样品细菌的alpha多样性结果见图2。alpha多样性反映了单个样品内部物种的多样性,物种观察指数、赵氏指数、艾斯指数反映样品中群落的丰富度(species richness);香农指数以及辛普森指数反映群落的多样性。与液相样品相比,固相样品的物种观察指数、赵氏指数、艾斯指数和香农指数均较高,而辛普森指数较低。因此,固相样品具有更高的细菌多样性。
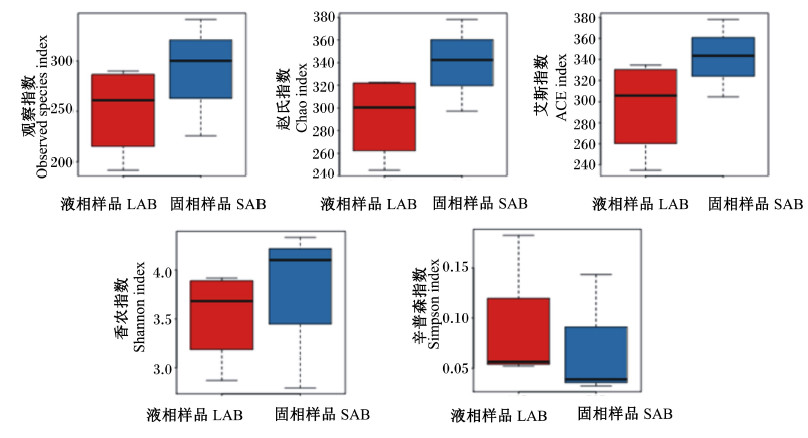 | 图2 alpha多样性指数的箱图 Fig. 2 The boxplots of alpha diversity indexes |
beta多样性分析结果展示菌群之间的相似度。瘤胃液相和固相样品细菌的beta多样性的主坐标分析(PCoA)见图3,来自液相和固相的样品聚类在一起,这与PCA结果一致。
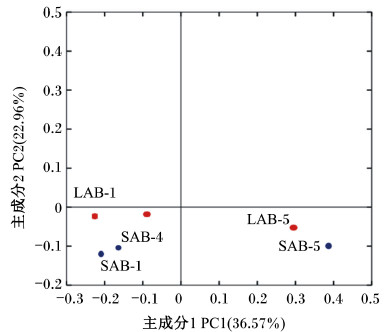 | 图3 beta多样性的PCoA图 Fig. 3 The PCoA profile of beta diversity |
蒙古羊瘤胃液相和固相样品的差异菌群统计结果见表2。在门、纲、目、科、属和种水平分类上分别有3、3、3、3、3和1种存在显著性差异的细菌菌群(P < 0.05)。门分类水平上是拟杆菌门、厚壁菌门和变形菌门,液相和固相样品中的拟杆菌门和厚壁菌门为优势菌群,但拟杆菌门在液相样品中的比例(67%)较高,而在固相样品中的比例(45%)低。属水平分类上,液相样品中存在显著性差异的菌群主要来自普氏菌属(Prevotella)(P < 0.05),所占比例为53%;而固相样品中存在显著性差异的菌群主要来1种未鉴定(unknown)的属(P < 0.05),所占比例为32%。种水平分类上,存在显著性差异的是栖瘤胃普雷沃菌(Prevotella ruminicola)(P < 0.05),其在液相和固相样品中所占的比例分别为41%和11%。因此,来自瘤胃液相和固相样品的细菌菌群多样性存在较大的差异。
| 表2 瘤胃液相和固相样品的差异菌群统计结果 Table 2 The statistical results of different bacteria between the samples of liquid and solid phases of rumen |
本研究在Illumina Miseq测序平台上,通过16S rRNA基因扩增子技术成功地检测了蒙古羊瘤胃液相和固相细菌菌群多样性,获得了大量、全面且深入的菌群信息。
研究表明,反刍动物瘤胃中主要的细菌菌群来自拟杆菌门和厚壁菌门[14, 15],这与本研究的结果一致,通过OTU聚类后在门的分类水平上共检测出13个已鉴的门类和1个未分类的门类,且优势菌群为拟杆菌门和厚壁菌门。其中,拟杆菌门在瘤胃液相样品中的比例较高,为67%,而在瘤胃固相样品中仅为45%,这可能与液相和固相样品的化学组成不同相关[2]。样品经组间显著性分析后显示,属水平分类上,普氏菌属和1个未鉴定的菌属在液相和固相样品间呈现出显著差异,普氏菌属在反刍动物对非纤维素植物多糖的利用和蛋白质降解等方面发挥着重要的作用[16]。与固相样品相比,普氏菌属在瘤胃液相样品中所占比例高达53%;与液相样品相比,未鉴定菌属在瘤胃固相样品中所占的比例高达32%,这充分显示出瘤胃固相样品中还有很多有价值的菌群信息需要深入挖掘。种水平分类上,栖瘤胃普雷沃菌是液相和固相样品间具有显著差异的菌,并且该菌在液相中的比例较高。研究表明,栖瘤胃普雷沃菌具有纤维素酶基因[17]。Osborne等[18]对细菌协同消化利用饲料纤维素的研究结果表明,栖瘤胃普雷沃菌和产琥珀酸丝状杆菌共同培养后,可有效提高鸭茅(Dactylis glomerata)的半纤维素和果胶的消化率。同样,Singh等[19]研究不同比例的纤维饲料对水牛瘤胃细菌的影响,结果表明栖瘤胃普雷沃菌在精粗比为0 ∶ 100的组中检测出的数量最高,每毫升瘤胃液中为2.52×107个。以上均说明栖瘤胃普雷沃菌在反刍动物瘤胃中具有非常重要的作用,但引起其在液相和固相样品中存在数量差异的原因还有待进一步研究。
本研究中alpha多样性指数结果显示液相和固相样品均具有复杂的细菌菌群多样性,这与Fouts等[20]研究结果一致,在其研究中,采用454测序平台检测12头奶牛的瘤胃液相和固相样品中细菌和真菌多样性,结果发现瘤胃固相和液相样品中的细菌菌群存在一定的差异。本研究中,与液相样品相比,固相样品的alpha多样性较好,而与De Menezes等[21]研究结果相反,其研究对饲喂了牧草或全混合日粮的奶牛瘤胃液相和固相样品进行了深入测序后,发现瘤胃液相具有较高的细菌多样性。此结果与本研究的结果存在差异,可能是由动物品种或饲粮不同而引起的。尽管如 此,本研究中来自同一动物个体的瘤胃液相和固相样品聚类在一起,这表明相邻的物理空间采样对瘤胃细菌的影响大于动物个体。Jami等[14]研究也表明,即使样品来自同一品种的荷斯坦牛,动物个体间仍存在差异菌群。
4 结论① 共获得91 971条序列和15 321个OTU,平均每个样品有13 138条序列和2 189个OTU,平均测序读长为252 bp。
② 蒙古羊瘤胃液相和固相样品具有共同的优势细菌菌群,存在显著性差异的菌群为:门水平分类上拟杆菌门和厚壁菌门;属水平分类上普氏菌属和1种未鉴定的属;种水平分类上栖瘤胃普雷沃菌。
③ 与液相样品相比,固相样品的alpha多样性较好;PCA和PCoA显示来自同一动物个体的液相和固相样品聚类在一起。
| [1] | KEBREAB E,DIJKSTRA J,BANNINK A,et al.Recent advances in modeling nutrient utilization in ruminants[J]. Journal of Animal Science,2009,87(14):E111-E122. ( 1) 1)
|
| [2] | VOLDEN H,MYDLAND L T,HARSTAD O M.Chemical composition of protozoal and bacterial fractions isolated from ruminal contents of dairy cows fed diets differing in nitrogen supplementation[J]. Acta Agriculturae Scandinavica,Section A:Animal Science,1999,49(4):235-244. ( 2) 2)
|
| [3] | MOSONI P,MARTIN C,FORANO E,et al.Long-term defaunation increases the abundance of cellulolytic ruminococci and methanogens but does not affect the bacterial and methanogen diversity in the rumen of sheep[J]. Journal of Animal Science,2011,89(3):783-791. ( 1) 1)
|
| [4] | ST-PIERRE B,WRIGHT A D G.Molecular analysis of methanogenic archaea in the forestomach of the alpaca (Vicugna pacos)[J]. BMC Microbiology,2012,12(1):1. ( 1) 1)
|
| [5] | MEYER M,STENZEL U,HOFREITER M.Parallel tagged sequencing on the 454 platform[J]. Nature Protocols,2008,3(2):267-278. ( 1) 1)
|
| [6] | MARDIS E R.The impact of next-generation sequencing technology on genetics[J]. Trends in Genetics,2008,24(3):133-141. ( 1) 1)
|
| [7] | QUAIL M A,KOZAREWA I,SMITH F,et al.A large genome center's improvements to the Illumina sequencing system[J]. Nature Methods,2008,5(12):1005-1010. ( 1) 1)
|
| [8] | WILLIAMS S T,FOSTER P G,LITTLEWOOD D T J.The complete mitochondrial genome of a turbinid vetigastropod from MiSeq Illumina sequencing of genomic DNA and steps towards a resolved gastropod phylogeny[J]. Gene,2014,533(1):38-47. ( 1) 1)
|
| [9] | ZHOU J Z,BRUNS M A,TIEMDJE J M.DNA recovery from soils of diverse composition[J]. Applied and Environmental Microbiology,1996,62(2):316-322. ( 1) 1)
|
| [10] | CAPORASO J G,LAUBER C L,WALTERS W A,et al.Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms[J]. The ISME Journal,2012,6(8):1621-1624. ( 1) 1)
|
| [11] | LIU B H,YUAN J Y,YIU S M,et al.COPE:an accurate k-mer-based pair-end reads connection tool to facilitate genome assembly[J]. Bioinformatics,2012,28(22):2870-2874. ( 1) 1)
|
| [12] | EDGAR R C.UPARSE:highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods,2013,10(10):996-998. ( 1) 1)
|
| [13] | DRAY S,DUFOUR A B.The ade4 package:implementing the duality diagram for ecologists[J]. Journal of Statistical Software,2007,22(4):1-20. ( 1) 1)
|
| [14] | JAMI E,MIZRAHI I.Composition and similarity of bovine rumen microbiota across individual animals [J]. PLoS ONE,2012,7(3):e33306. ( 2) 2)
|
| [15] | KITTELMANN S,SEEDORF H,WALTERS W A,et al.Simultaneous amplicon sequencing to explore co-occurrence patterns of bacterial,archaeal and eukaryotic microorganisms in rumen microbial communities[J]. PLoS One,2013,8(2):e47879. ( 1) 1)
|
| [16] | KABEL M A,YEOMAN C J,HAN Y J,et al.Biochemical characterization and relative expression levels of multiple carbohydrate esterases of the xylanolytic rumen bacterium Prevotella ruminicola 23 grown on an ester-enriched substrate[J]. Applied and Environmental Microbiology,2011,77(16):5671-5681. ( 1) 1)
|
| [17] | PURUSHE J,FOUTS D E,MORRISON M,et al.Comparative genome analysis of Prevotella ruminicola and Prevotella bryantii:insights into their environmental niche[J]. Microbial Ecology,2010,60(4):721-729. ( 1) 1)
|
| [18] | OSBORNE J M,DEHORITY B A.Synergism in degradation and utilization of intact forage cellulose,hemicellulose,and pectin by three pure cultures of ruminal bacteria[J]. Applied and Environmental Microbiology,1989,55(9):2247-2250. ( 1) 1)
|
| [19] | SINGH K M,PANDYA P R,TRIPATHI A K,et al.Study of rumen metagenome community using qPCR under different diets[J]. Meta Gene,2014,2:191-199. ( 1) 1)
|
| [20] | FOUTS D E,SZPAKOWSKI S,PURUSHE J,et al.Next generation sequencing to define prokaryotic and fungal diversity in the bovine rumen[J]. PLoS One,2012,7(11):e48289. ( 1) 1)
|
| [21] | DE MENEZES A B,LEWIS E,O'DONOVAN M,et al.Microbiome analysis of dairy cows fed pasture or total mixed ration diets[J]. FEMS Microbiology Ecology,2011,78(2):256-265. ( 1) 1)
|




