2. 北京科为博生物科技有限公司, 北京 100086
2. Beijing Creat-Value Biological Technology Co., Ltd., Beijing 100086, China
在动物胃肠道寄居着种类繁多的微生物,这些微生物菌群与寄居宿主之间形成了一个相互依赖和相互制约的微生态系统[1]。这个微生态系统存在细菌、古生菌、真菌、原虫、噬菌体等多种微生物及其分泌的代谢产物[2]。这几个相互作用和影响的集团之间任何一方发生改变,整个微生态系统都会随之做出相应的变化和调整[3]。肠道微生态系统的平衡对动物养分代谢和机体免疫等方面起着重要作用。研究动物胃肠道微生态系统有益于揭开胃肠道消化代谢体系的作用机理[4, 5]。调节动物胃肠道微生态系统可以促进动物健康及营养物质利用[6]。地衣芽孢杆菌(Bacillus licheniformis)在自然界分布广泛,是一种较为常见的革兰氏阳性菌。属于芽孢杆菌属,其具有耐热、产酶丰富且产酶量高等优良性状。不仅安全而且在其代谢过程中还可以产生许多有益活性物质[7, 8]。杨汉博[9]研究表明,向黄鸡饲粮中添加地衣芽孢杆菌可以有效改善其肠道微生态平衡。屎肠球菌(Enterococcus faecium)是一类得到广泛应用的兼性厌氧乳酸菌,其在动物肠道中可以分泌有机酸等代谢产物,提高机体抵御有害微生物的能力从而达到益生作用[10, 11]。龚琪[12]研究表明,屎肠球菌的添加可以促进肠道菌群有效完成过渡期,并在稳定期保持稳定。丁酸梭菌(Clostridium butyricum)别名酪酸菌,属于厌氧革兰氏阳性菌,进入动物肠道后不仅可以分泌具有益生作用的代谢产物还可以抵抗胃酸等消化液的作用,作为新一代的益生菌制剂,具有非常广泛的应用前景[13, 14]。马洪庆等[15]研究表明,饲粮中添加丁酸梭菌可以提高麻羽肉鸡的增重率及有效降低了其料重比。本研究在基础饲粮中分别添加地衣芽孢杆菌、屎肠球菌和丁酸梭菌,采用PCR-变性梯度凝胶电泳(DGGE)和实时荧光定量PCR(qPCR)技术分析肉鸡空肠食糜、回肠食糜和盲肠食糜中微生物菌群种类和数量差异,获取益生菌对肠道中微生物区系多样性影响的信息,阐明不同益生菌对动物肠道菌群结构影响的差异性,为益生菌的产业开发和应用研究提供理论指导。
1 材料与方法 1.1 试验材料地衣芽孢杆菌、屎肠球菌(微胶囊,纯度大于90%)购自北京挑战生物技术有限公司,丁酸梭菌(液态,纯度大于90%)购自北京科为博生物科技有限公司,爱拔益加(AA)肉鸡购自北京华都肉鸡育种有限公司。
1.2 试验设计饲养试验选取432只1日龄的肉公鸡,称量初始体重后分别随机分成4个组,每个组9个重复,每个重复12只鸡。组1饲喂基础饲粮,组2饲喂基础饲粮+地衣芽孢杆菌(前期4.8×106 CFU/g饲粮;后期4.7×106 CFU/g饲粮),组3饲喂基础饲粮+屎肠球菌(前期1.0×106 CFU/g饲粮;后期1.2×106 CFU/g饲粮),组4饲喂基础饲粮+丁酸梭菌(前期1.3×106 CFU/g饲粮;后期1.2×106 CFU/g饲粮)。试验期为6周,分1~3周龄和4~6周龄2个阶段饲养,基础饲粮组成及营养水平见表1。
| 表1 基础饲粮组成及营养水平(风干基础) Table 1 Composition and nutrient levels of basal diets (air-dry basis) |
饲养试验在中国农业科学院饲料研究所昌平区南口中试基地鸡舍进行,采用立体笼养方式。自由采食饲粮,自由饮水,按常规免疫程序免疫。鸡舍光照第1~7天为23 h,7 d以后20 h。鸡舍温度第1~3天为33~35 ℃,从第4天开始逐步降温,到第28天降到20 ℃,在饲养后期一直保持20 ℃。鸡舍湿度在第1周保持60%~70%,第2~6周保持50%~60%。
1.4 样品采集饲养试验结束后,每个重复中随机抽取3只鸡,称体重后,静脉放血处死,剖开腹腔,在无菌条件下分别采集空肠、盲肠、回肠的食糜置于冻存管中,迅速投入液氮后转至-70 ℃保存,备用。
1.5 基因组DNA的提取与PCR扩增 1.5.1 基因组DNA的提取应用天根生物技术有限公司粪便基因组提取试剂盒(DP328-02)提取肠道食糜基因组DNA,应用OMEGA真菌基因组提取试剂盒(DP307-02)提取PCR校正工程菌株的基因组DNA。
1.5.2 PCR扩增为了比较分析在基础饲粮中添加益生菌后空肠食糜、回肠食糜和盲肠食糜中微生物群落的变化,根据大肠杆菌的基因组[16]设计了引物338-F及543-R用来特异性扩增16S rDNA V3区基因片段,片段大小193 bp,在338-F引物的5’端带有1个40 bp GC夹子。在PCR扩增中以纯化过的基因组DNA为模板,进行细菌16S rDNA的PCR扩增。
| 表2 细菌16S rDNA PCR扩增引物 Table 2 Primers used in 16S rDNA PCR for bacteria |
按照DcodeTM Universal Mutation Detection Systerm的操作说明对细菌16S rDNA进行DGGE指纹图谱分析。变性物质为甲酰胺和尿素,变性梯度为40%~60%。先在80 V恒定电压下快速电泳1 h,然后在60 V恒定电压下电泳16 h;电泳结束后,将凝胶用GelRed染色液染色20 min,再用1×TAE漂洗10 min,然后在Viber凝胶成像扫描系统中获取胶图。
1.7 16S rDNA基因序列的克隆和测序将比较重要的条带按照从上到下的顺序依次切取编号,纯化后用引物338-F(不带GC夹)和543-R进行PCR扩增,将PCR产物与pEASY-T3载体连接,然后将阳性克隆子菌液送至北京睿博新科生物技术有限公司测序。将获得的测序用BLAST工具进行序列比对,选择16S rRNA数据库进行同源性搜索。选择同源性最高的序列的细菌株系为参考细菌物种。
1.8 细菌qPCR为了检测益生菌对肠道中细菌菌群数量的影 响,采用qPCR技术检测不同肠段食糜中细菌菌群数量。引物为GB-F(5’-CGGCAACGAGCGCAACCC-3’)和GB-R(5’-CCATTGTAGCACGTGTGTAGCC-3’),内标基因RFP引物为RFP-F(5’-CAGGACGGCTGCTTCATCTACAAGG-3’)和RFP-R(5’-CTTGGCCATGTAGATGGACTTGAACTCC-3’),所用引物都经过qPCR溶解曲线分析和琼脂糖凝胶电泳检测。将16S rDNA片段和RFP片段连接到pGEM-T载体上,然后转化到TransⅠ-T1宿主中,提取重组质粒之后将其进行梯度稀释,使其浓度为10~107 mol/μL,最后利用质粒为模版制作标准曲线。利用IQ5 qPCR仪检测不同肠段食糜样品中的细菌菌群数量。
1.9 数据分析所有数据经Excel 2013初步整理后,采用SPSS 16.0对数据进行单因素方差分析(one-way ANOVA)分析,DGGE图像用Quantity One软件处理分析。
2 结果分析 2.1 不同益生菌对肉鸡不同肠段PCR-DGGE指纹图谱条带数的影响由表3可知,饲粮中添加3种不同益生菌均影响了42日龄肉鸡空肠、回肠及盲肠食糜细菌16S rDNA V3区基因PCR-DGGE指纹图谱(图1~图3)。通过软件Quantity One分析条带数可知,PCR-DGGE指纹图谱的条带数发生了不同变化。肉鸡空肠:对照组、屎肠球菌组、地衣芽孢杆菌组 和丁酸梭菌组的条带数分别为15.33、18.00、 16.67和14.00。其中屎肠球菌组条带数显著高于对照组(P<0.05);而地衣芽孢杆菌和丁酸羧菌组与对照组无显著差异(P>0.05)。肉鸡回肠:对照组、屎肠球菌组、地衣芽孢杆菌组和丁酸梭菌组的条带数分别为18.67、25.33、22.00和14.33。其中屎肠球菌组条带数显著高于对照组(P<0.05);地衣芽孢杆菌和丁酸羧菌组与对照组无显著差异(P>0.05)。肉鸡盲肠:对照组、屎肠球菌组、地衣芽孢杆菌组和丁酸梭菌组的条带数分别为32.00、32.00、34.33和31.33,变化均不显著(P>0.05)。
| 表3 PCR-DGGE指纹图谱条带数 Table 3 The number of bands in PCR-DGGE fingerprints |
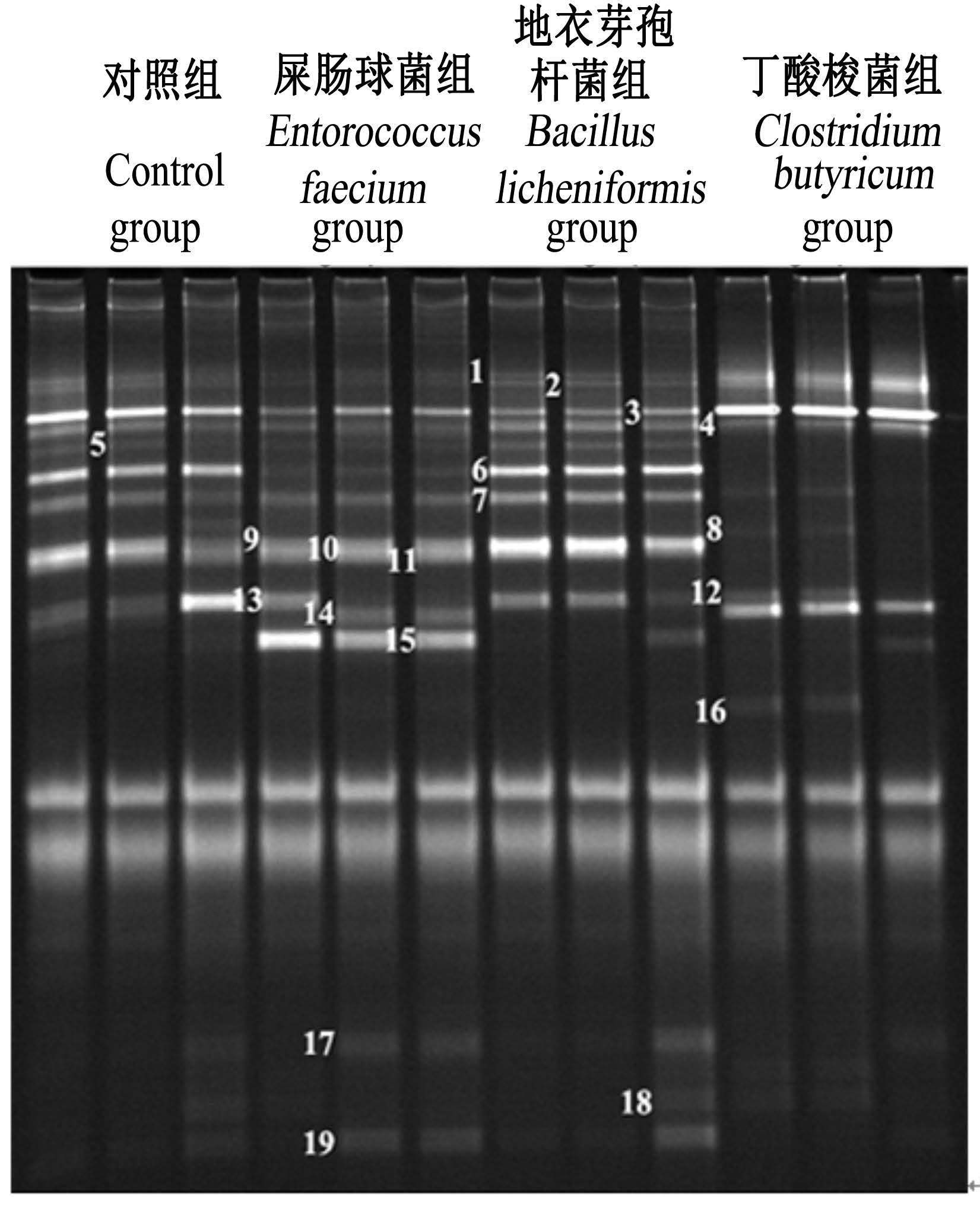 | 图1 肉鸡空肠食糜细菌16S rDNA V3区基因的 PCR-DGGE指纹图谱 Fig. 1 PCR-DGGE fingerprints genes of the V3 region genes of 16S rDNA amplified from bacteria of jejunal chyme of broilers |
 | 图2 肉鸡回肠食糜细菌16S rDNA V3区基因的PCR-DGGE指纹图谱 Fig. 2 PCR-DGGE fingerprints of the V3 region genes of 16S rDNA amplified from bacteria of ileal chyme of broilers |
 | 图3 肉鸡盲肠食糜细菌16S rDNA V3 区基因的PCR-DGGE指纹图谱 Fig. 3 PCR-DGGE fingerprints genes of the V3 region genes of 16S rDNA amplified from bacteria of cecal chyme of broilers |
饲粮中添加3种不同益生菌均显著影响了42日龄肉鸡空肠食糜细菌16S rDNA V3区基因PCR-DGGE指纹图谱(图1)。由表4可知,从该指纹图谱中选取了19条特征条带进行测序,将得到的序列在GenBank中进行了BLAST相似性分析。结果显示,这些序列与8个乳酸菌具有很高的相似性(L. equi、L. aviaries、L. hayakitensis、L. salivarius、L. ingluviei、L. gasseri ATCC33323、L. saerimneri、L. agilis)。与对照组相比,屎肠球菌组具有1条明显的独有的条带,而在对照组中没有出现,从测序结果来看与L. ingluviei最为相似,说明屎肠球菌特异的促进了该菌的生长繁殖。与对照组相比,地衣芽孢杆菌组在条带种类上无显著差异(P>0.05),但L. agilis和L. salivarius的生长得到了促进。与对照组相比,丁酸梭菌组中条带数无显著差异(P>0.05),但有多条条带的亮度变弱,甚至消失,说明丁酸梭菌抑制了这些菌的生长繁殖(L. ingluviei、L. gasseri ATCC33323、L. saerimneri、L. agilis),而条带3(L. aviaries)在丁酸梭菌组中亮度明显高于对照组,说明丁酸梭菌促进了其生长繁殖。
| 表4 肉鸡空肠食糜细菌16S rDNA V3区基因PCR-DGGE优势条带测序结果 Table 4 Sequencing results of the predominant bands cut from the 16S rDNA V3 region genes PCR-DGGE of bacteria of the jejunal chyme of broilers |
饲粮中添加3种不同益生菌均显著影响了42日龄肉鸡回肠食糜细菌16S rDNA V3区基因PCR-DGGE指纹图谱(图2)。由表5可知,从指纹图谱中选取了16条特征条带进行DNA测序,并将得到的序列在GenBank中进行了BLAST相似性分析,结果表明这些序列与7个乳酸菌(L. agilis、L. equi、L. versmoldensis、L. satsumensis、L. gasseri ATCC33323、L. salivarius、L. hayakitensis),3个屎肠球菌(E. faecium、E. thailandicus、E. cecorum),3个梭菌(C. bartlettii、 C. irregulare、C. hiranonis DSM 13275)以及1个粪肠球菌(C. eutactus)具有很高的相似性。与对照组相比,屎肠球菌组食糜中E. faecium和E. thailandicus明显增多,说明饲粮中添加的屎肠球菌在回肠食糜中发生了生长繁殖。与对照组相比,地衣芽孢杆菌组在条带种类上无显著差异(P>0.05),但C. eutactus和C. irregulare的生长受到了抑制。与对照组相比,丁酸梭菌组中条带数无显著差异(P>0.05),但L. satsumensis、L. gasseri ATCC33323、E. cecorum、L. salivarius、C. eutactus、C. irregulare生长受到了抑制,而L. versmoldensis在丁酸梭菌组中条带亮度明显高于对照组,说明丁酸梭菌促进了该菌的生长和繁殖。
| 表5 肉鸡回肠食糜细菌16S rDNA V3区基因PCR-DGGE优势条带测序结果 Table 5 Sequencing results of the predominant bands cut from the 16S rDNA V3 region genes PCR-DGGE of bacteria of the ileal chyme of broilers |
图3是不同组42日龄肉鸡盲肠食糜细菌16S rDNA V3区基因的PCR-DGGE指纹图谱。由表6可知,从该指纹图谱中选取了43条特征条带,对其进行了DNA测序,将得到的序列在GenBank中进行了BLAST相似性分析,结果表明盲肠的菌群种类非常的丰富,主要有粪肠球菌、瘤胃球菌、梭菌、拟杆菌、乳酸菌等。从整体上来看,在盲肠中的菌群种类更加丰富多样,表明在本试验条件下,肉鸡个体之间盲肠菌群结构差异较大。
| 表6 肉鸡盲肠食糜细菌16S rDNA V3区基因PCR-DGGE优势条带测序结果 Table 6 Sequencing results of the predominant bands cut from the 16S rDNA V3 region genes PCR-DGGE of bacteria of the cecal chyme of broilers |
由图4可知,饲粮添加地衣芽孢杆菌和屎肠球菌显著提高肉鸡空肠食糜中细菌的菌群数量(P<0.05),而添加丁酸梭菌对肉鸡空肠细菌的菌群数量无显著影响(P>0.05)。从回肠食糜的影响来看,添加地衣芽孢杆菌对细菌的菌群数量无显著影响(P>0.05),而饲粮添加屎肠球菌和丁酸梭菌显著增加了细菌的菌群数量(P<0.05)。饲粮添加地衣芽孢杆菌显著增加了盲肠食糜中细菌的菌群数量(P<0.05),而添加屎肠球菌和丁酸梭菌显著降低了盲肠食糜中细菌的菌群数量(P<0.05)。
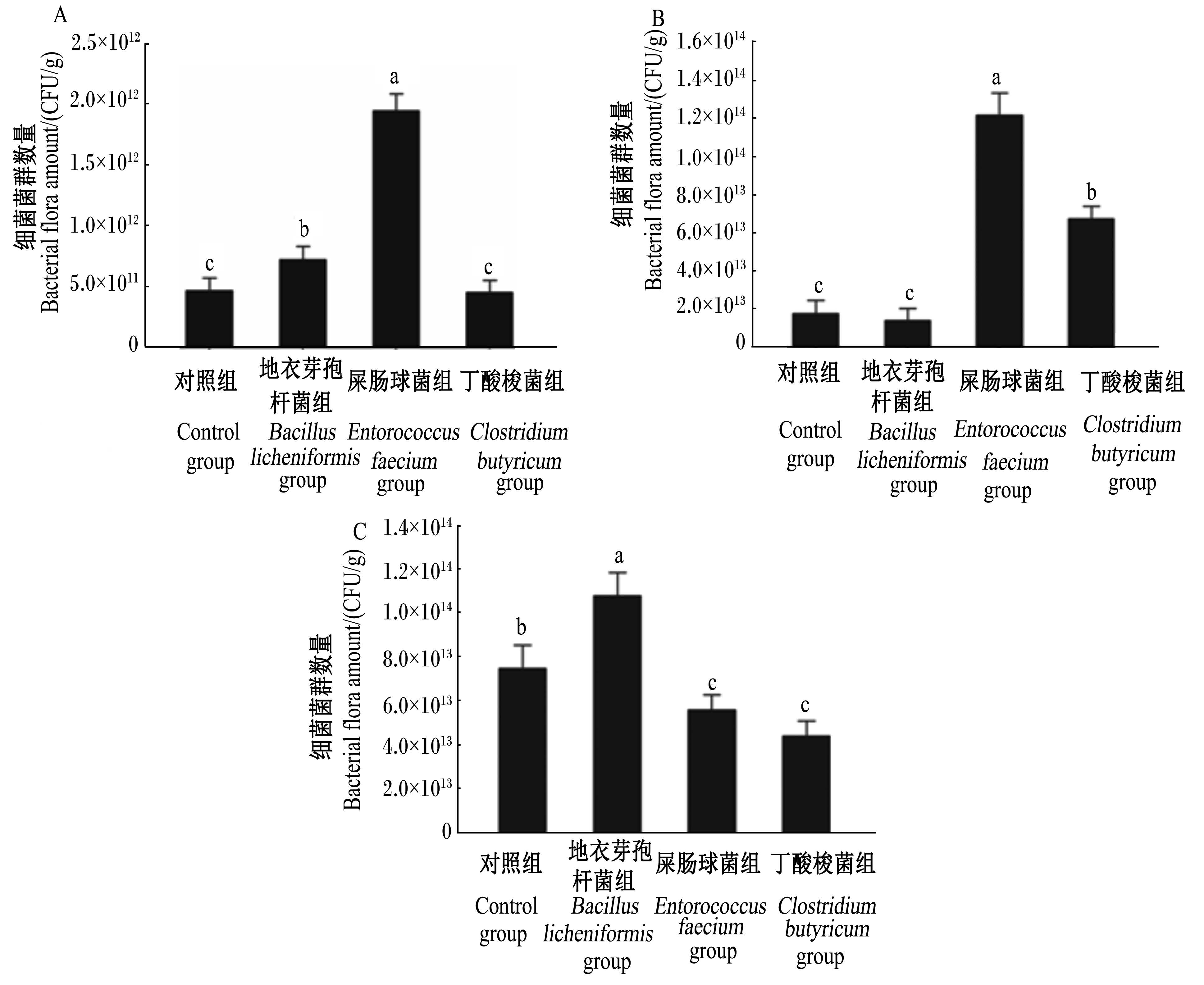 | A:空肠jejunum;B:回肠ileum;C:盲肠cecum。 数据柱上标不同字母表示差异显著(P<0.05)。Value columns with different letter superscripts mean significant difference (P<0.05).图4 不同组42日龄肉鸡空肠、回肠和盲肠中微生物数量比较分析 Fig. 4 Comparing microbial populations of jejunum,ileum,cecum between different groups of 42-day-old broilers |
肠道微生物是动物机体进行生命活动的一个重要组成单元,随着动物机体的生长、发育、疾病和衰老时刻发生着动态的变化,它对动物机体的影响是自始至终的[17]。胃肠道微生物的菌群组成与动物的基因组、营养和生活环境息息相关,它们积极参与宿主物质代谢的调控、信号传导、免疫防御,研究表明其对宿主的胃肠道、肝脏和脑具有重要的影响[18, 19, 20]。同时,胃肠道微生物菌群的密度和数量也影响着机体对营养物质的消化吸收能力以及抵抗疾病的能力[21]。本试验中,在饲粮中添加3种不同益生菌,肉鸡空肠、回肠和盲肠食糜PCR-DGGE指纹图谱的条带数及条带明亮程度发生了改变,且3种益生菌的作用效果不同。
与对照组相比,屎肠球菌组肉鸡空肠、回肠食糜细菌16S rDNA V3区基因PCR-DGGE指纹图谱的条带数显著增加。且饲粮中添加屎肠球菌促进了L. ingluviei在空肠及E. faecium和E. thailandicus在回肠中的生长繁殖且促进了乳杆菌这种益生菌的生长。
与对照组相比,地衣芽孢杆菌组肉鸡空肠、回肠及盲肠食糜细菌16S rDNA V3区基因PCR-DGGE指纹图谱的条带数变化不显著。但饲粮中添加地衣芽孢杆菌促进了L. agilis和L. salivarius在空肠内的生长,抑制了C. eutactus和C. irregulare在回肠中的生长。结果表明,地衣芽孢杆菌的添加促进了乳杆菌的生长繁殖,这与他人试验结果相同[22, 23]。
与对照组相比,丁酸梭菌组肉鸡空肠、回肠及盲肠食糜细菌16S rDNA V3区基因PCR-DGGE指纹图谱的条带数变化不显著。但饲粮中添加丁酸梭菌促进了L. aviaries在空肠及L. versmoldensis在回肠的生长繁殖;抑制了L. ingluviei、L. gasseri ATCC33323、L. saerimneri、L. agilis在空肠及L. satsumensis、L. gasseri ATCC33323、E. cecorum、L. salivarius、C. eutactus,C. irregulare在回肠的生长繁殖。结果表明,饲粮中添加丁酸梭菌,促进了2类乳杆菌的生长繁殖,而抑制了大部分其他菌,其中也有乳杆菌,这说明丁酸梭菌在肠道中产生的代谢产物有着比较专一的作用菌群。 3.2 不同益生菌对肉鸡不同肠段菌群数量的影响
由于PCR-DGGE的方法只能检测肠道中数量比较丰富的细菌,一般为总细菌数量的1%或者更多的菌群才能被检测。因此,同时采用qPCR技术检测肉鸡不同肠段中细菌菌群的数量变化情况与趋势。且qPCR方法计算细菌菌群数量不需要传统的细菌培养,省时省力的同时有效的提高了检测的灵敏度。利用2种互补研究技术的优势,综合分析了不同益生菌对肉鸡胃肠道菌群结构的影响差异。本试验中应用qPCR技术对不同肠段内细菌菌群数量分析发现饲粮添加不同益生菌产生了不同的影响效果。与对照组相比,屎肠球菌显著增加了空肠和回肠细菌的菌群数量,地衣芽孢杆菌显著增加了空肠和盲肠的细菌菌群数量;丁酸梭菌显著增加了回肠的细菌菌群数量。说明肉鸡在采食屎肠球菌后其空肠和回肠内细菌菌群数量得到增加,在采食地衣芽孢杆菌后其空肠和盲肠的细菌菌群数量得到增加,在采食丁酸梭菌后其回肠的细菌菌群数量增加。而这种增加可能是屎肠球菌、地衣芽孢杆菌和丁酸梭菌在肉鸡肠道内产生了有益该类细菌生长繁殖的代谢产物所致[24]。
饲粮中添加不同的益生菌无论是对菌群种类还是菌群数量都产生了不同程度的影响,这与菌群种类以及宿主肠道的营养物质代谢都息息相关[25]。除此之外,肠道菌群作为异源物质,其与肠道黏膜免疫系统之间也存在着复杂的相互作用[26]。各种菌群之间的相互竞争、共生关系也对最终动物机体的变化起着至关重要的作用,这都需要我们进一步的探索。
4 结 论① 饲粮添加不同益生菌对肉鸡不同肠段菌群结构产生了不同的影响:其中添加屎肠球菌后回肠、空肠样品PCR-DGGE指纹图谱的条带数增多,并促进了乳杆菌生长;地衣芽孢杆菌的添加也促进了乳杆菌的生长繁殖,而抑制了C. eutactus和C. irregulare的生长;丁酸梭菌的添加促进了L. aviaries在空肠及L. versmoldensis在回肠的生长繁殖。
② 饲粮添加不同益生菌对肉鸡不同肠段菌群数量产生了不同的影响:其中添加屎肠球菌增加了空肠和回肠细菌的菌群数量;地衣芽孢杆菌增加了空肠和盲肠的细菌菌群数量;丁酸梭菌增加了回肠的细菌菌群数量。
③ 综上所述,饲粮添加屎肠球菌、地衣芽孢杆菌和丁酸梭菌均不同程度地改善了肉鸡肠道菌群结构。
| [1] | HOOPER L V, LITTMAN D R, MACPHERSON A J.Interactions between the microbiota and the immune system[J]. Science, 2012, 336(6086):1268-1273. ( 1) 1)
|
| [2] | NICHOLSON J K, HOLMES E, KINROSS J, et al.Host-gut microbiota metabolic interactions[J]. Science, 2012, 336(6086):1262-1267. ( 1) 1)
|
| [3] | QUIGLEY E M M.Prebiotics and probiotics;modifying and mining the microbiota[J]. Pharmacological Research, 2010, 61(3):213-218. ( 1) 1)
|
| [4] | PURCHIARONI F, TORTORA A, GABRIELLI M, et al.The role of intestinal microbiota and the immune system[J]. European Review for Medical and Pharmacological Sciences, 2013, 17(3):323-333. ( 1) 1)
|
| [5] | VANBELLE M, TELLER E, FOCANT M.Probiotics in animal nutrition:a review[J]. Arch für Tierernaehrung, 1990, 40(7):543-567. ( 1) 1)
|
| [6] | VAN DER WIELEN P W J J, VAN KNAPEN F, BIESTERVELD S.Effect of administration of Lactobacillus crispatus, Clostridium lactatifermentans and dietary lactose on the development of the normal microflora and volatile fatty acids in the caeca of broiler chicks[J]. British Poultry Science, 2002, 43(4):545-550. ( 1) 1)
|
| [7] | 杨阳, 张付云, 苍桂璐, 等.地衣芽孢杆菌生物活性物质应用研究进展[J]. 生物技术进展, 2013, 3(1):22-26. ( 1) 1)
|
| [8] | LARSEN N, THORSEN L, KPIKPI E N, et al.Characterization of Bacillus spp. strains for use as probiotic additives in pig feed[J]. Applied Microbiology and Biotechnology, 2014, 98(3):1105-1118. ( 1) 1)
|
| [9] | 杨汉博.地衣芽孢杆菌BL15和BL19对黄鸡的营养、微生态效应研究[D]. 博士学位论文.雅安:四川农业大学, 2002. ( 1) 1)
|
| [10] | 葛龙, 李波.屎肠球菌在饲用微生态制剂中的研究与应用[J]. 饲料与畜牧, 2013(6):57-59. ( 1) 1)
|
| [11] | FRANZ C M A P, HUCH M, ABRIOUEL H, et al.Enterococci as probiotics and their implications in food safety[J]. International Journal of Food Microbiology, 2011, 151(2):125-140. ( 1) 1)
|
| [12] | 龚琪.益生性屎肠球菌HDRsEf1对雏鸡盲肠菌群发育的影响[D]. 硕士学位论文.武汉:华中农业大学, 2014. ( 1) 1)
|
| [13] | 张善亭, 史燕, 张淑丽, 等.丁酸梭菌的研究应用进展[J]. 生物技术通报, 2013(9):27-33. ( 1) 1)
|
| [14] | PECK M W.Biology and genomic analysis of Clostridium botulinum[J]. Advances in Microbial Physiology, 2009, 55:183-265, 320. ( 1) 1)
|
| [15] | 马洪庆, 马恺.丁酸梭菌三联制剂对麻羽肉鸡生长性能的影响[J]. 饲料研究, 2014(21):69-70, 85. ( 1) 1)
|
| [16] | BROSIUS J, DULL T J, SLEETER D D, et al.Gene organization and primary structure of a ribosomal RNA operon from Escherichia coli[J]. Journal of Molecular Biology, 1981, 148(2):107-127. ( 2) 2)
|
| [17] | KANAUCHI O, MITSUYAMA K, ARAKI Y, et al.Modification of intestinal flora in the treatment of inflammatory bowel disease[J]. Current Pharmaceutical Design, 2003, 9(4):333-346. ( 1) 1)
|
| [18] | CARABOTTI M, SCIROCCO A, MASELLI M A, et al.The gut-brain axis:interactions between enteric microbiota, central and enteric nervous systems[J]. Annals Gastroenteroloy, 2015, 28(2):203-209. ( 1) 1)
|
| [19] | MINEMURA M, SHIMIZU Y.Gut microbiota and liver diseases[J]. World Journal of Gastroenterol, 2015, 21(6):1691-1702. ( 1) 1)
|
| [20] | COOK M T, TZORTZIS G, CHARALAMPOPOULOS D, et al.Microencapsulation of probiotics for gastrointestinal delivery[J]. Journal of Controlled Release, 2012, 162(1):56-67. ( 1) 1)
|
| [21] | SMITH J M.A review of avian probiotics[J]. Journal of Avian Medicine and Surgery, 2014, 28(2):87-94. ( 1) 1)
|
| [22] | HOSOI T, AMETANI A, KIUCHI K.Changes in fecal microflora induced by intubation of mice with Bacillus suttilis (notta) spores are dependent upon dietary components[J]. Canadian Journal of Microbiology, 1999, 45(1):59-66. ( 1) 1)
|
| [23] | GUO X H, LI D F, LU W Q, et al.Screening of Bacillus strains as potential probiotics and subsequent confirmation of the in vivo effectiveness of Bacillus subtiis MA139 in pigs[J]. Antonie van Leeuwenhoek, 2006, 90(2):139-146. ( 1) 1)
|
| [24] | 何昭阳, 王增辉, 吴延春, 等.雏鸡消化道主要正常菌群定植规律的研究[J]. 畜牧兽医学报, 2000, 31(1):41-48. ( 1) 1)
|
| [25] | 刘彩虹, 张和平.肠道菌群与肠道内营养物质代谢的相互作用[J]. 中国乳品工业, 2014, 42(5):33-36. ( 1) 1)
|
| [26] | 王爱丽, 武庆斌, 孙庆林.肠道菌群与肠道黏膜免疫系统的相互作用机制[J]. 中国微生态学杂志, 2009, 21(4):382-384. ( 1) 1)
|




