随着家禽集约化养殖程度的越来越高,饲养密度成为家禽养殖中普遍存在的应激因素[1]。王龙[2]研究了不同饲养密度对层叠笼养蛋鸡生产性能及福利的影响,结果显示随着饲养密度增加,鸡群受到的应激刺激增大,鸡群羽毛覆盖程度差,血液皮质酮水平增高,入舍鸡产蛋数降低,鸡群死亡率升高,高密度饲养严重影响蛋鸡的生产性能。应激条件下,除了影响家禽外在的羽毛状态和生产性能外,对其内部肠道健康也有重要的影响[3]。肠道微生物区系是评价肠道健康的重要指标,肠道微生物对宿主的营养物质消化、吸收、免疫等均起重要作用[4]。小肠是肠道中重要的吸收部位,90%的食物在小肠中被吸收;而十二指肠是小肠中管径最大、位置最深、最为固定的小肠段,且胰管与胆总管均开口于十二指肠,因此十二指肠的健康直接影响动物机体的健康。研究饲养密度对蛋鸡十二指肠细菌菌群结构的影响,对探究家禽机体内部影响蛋鸡健康状况的作用机理至关重要。关于肠道细菌结构的研究,以往的微生物纯培养方法只能体外培养10%~60%的细菌[5],该方法不能完整、准确的描述肠道菌群结构,因此逐渐被淘汰;变性梯度凝胶电泳(DGGE)和聚合酶链式反应(PCR)等基于16S核糖体RNA(rRNA)基因为基础的分子技术的发展,加快了对肠道微生物区系的检测和认知[6, 7],因此目前多采用更加快速和细致的PCR-DGGE方法分析肠道菌群结构。关于DGGE方法检测胃肠道细菌结构的应用已非常广泛,但该技术在家禽肠道菌群结构分析的应用仍在初期,关于蛋鸡肠道菌群变化的研究很少,研究饲养密度应激对蛋鸡十二指肠肠道菌群结构的影响未见报道。因此,本研究通过采用PCR-DGGE技术分析不同饲养密度对笼养蛋鸡十二指肠肠道菌群结构的影响,从家禽肠道健康角度来反映饲养密度对蛋鸡产生的应激,为蛋鸡肠道微生物区系的全面分析奠定基础。
1 材料与方法 1.1 试验材料 1.1.1 试验动物及设计随机选取1 250只11周龄海兰灰(Hy-Line Gray)蛋鸡(由黑龙江兴和生物科技有限公司提供),饲养笼尺寸(长×宽×高)为600 mm×450 mm×430 mm,按照不同饲养密度随机分为5个组:A组900 cm2/只(3只/笼)、B组675 cm2/只(4只/笼)、C组540 cm2/只(5只/笼)、D组450 cm2/只(6只/笼)、E组380 cm2/只(7只/笼),每个组内50个重复,所有鸡只饲养在同一层笼中,即距离地面第2层。整个试验周期5个组均在同一环境和饲养管理模式下进行饲养。
1.1.2 试验样品采集与处理于16、26、50周龄时进行采样。即每个组的每15个重复随机挑选1~2个重复,每个重复选取5只鸡,进行采样。对于密度小的组(3、4只/笼),选6个重复,每2个重复选取5只鸡。将选取的试验鸡,剪断颈静脉处死,无菌操作,剖腹分离十二指肠。将每个重复5只鸡的十二指肠内容物均匀混合后装入1个15 mL离心管内,编号,-80 ℃保存备用。表1为十二指肠内容物样品编号。
|
|
表 1 十二指肠内容物样品编号 Table 1 Number of the contents of duodenum |
采用CTAB手提法进行各内容物样品的DNA 提取,以样品基因组DNA为模板,采用细菌通用 引物GC-338F和518R扩增样品16S rDNA高变区序列,引物信息见表2。
|
|
表 2 引物信息 Table 2 Primer information |
PCR扩增体系(50 μL)为:10×PCR buffer 5 μL;dNTP(2.5 mmol/L)3.2 μL;rTaq(5 U/μL)0.4 μL;GC-338F(20 μmol/L)1 μL;518R(20 μmol/L)1 μL;模板DNA 50 ng;补ddH2O至50 μL。
PCR扩增程序为:94 ℃预变性5 min;94 ℃变性1 min,55 ℃复性45 s,72 ℃延伸1 min,30个循环;最终72 ℃延伸10 min。
1.3 PCR产物的DGGE分析取10 μL PCR的产物进行DGGE分析。采用变性梯度为35%~55%、浓度为7%的聚丙烯酰胺凝胶在1×TAE缓冲液中150 V、60 ℃下电泳5 h。DGGE完毕后采用银染法染色。
1.4 DGGE图谱中优势条带的回收与测序用灭菌的手术刀切下待回收DGGE条带,采用OMEGA公司Poly-Gel DNA Extraction Kit回收目的条带。以2 μL回收产物为模板,338F/518R为引物进行PCR扩增。
将重新扩增的DNA片段切胶回收、纯化后,连接到PMD18-T载体上,并转化至DH5α感受态细胞中,筛选阳性克隆,菌液由华大基因对插入的细菌16S rDNA片段进行序列测定。
1.5 数据分析在GenBank中使用Blast程序进行同源性比较,获得最相似典型菌株的16S rDNA序列。采用MEGA 5软件,Neighbor-joining法构建系统发育树,自展数(bootstrap)为1 000。
细菌多样性指数是研究群落物种数和个体数以及均匀度的综合指标。根据电泳图谱中样品条带数目及每个条带的强度(灰度),对各样品中细菌多样性指数(H)、均匀度(E)和丰富度(S)等指标进行分析。
2 结果与分析 2.1 细菌16S rDNA的PCR扩增以GC-338F和518R为引物扩增16S rDNA序列,各条带均获得250 bp左右的DNA片段(图1),可以用于DGGE分析。
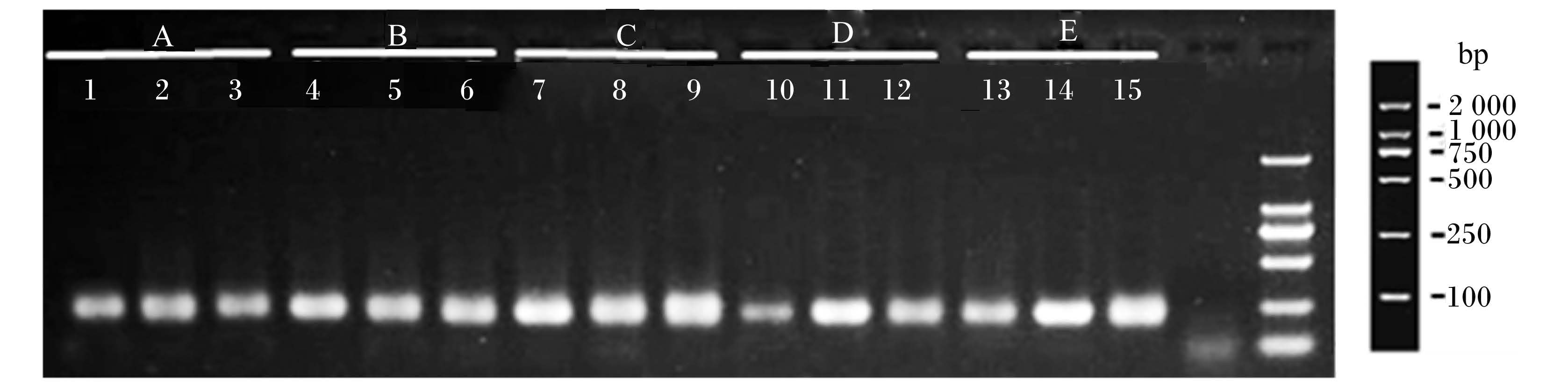
|
图 1 PCR扩增结果 Fig. 1 PCR amplification results |
样品16S rDNA PCR产物的DGGE分析结果见图2。图2所示的每条泳道的样品均是5只鸡十二指肠肠道内容物的混合物,所以它反映的是不同组别之间十二指肠肠道菌群的平均状态。图2中不同位置的条带代表不同的细菌;条带的数量反映的是十二指肠肠道细菌种群的数量,条带的数量越多,表示种群的数量越多,反之亦然;某一条带的亮度和粗细反映的是十二指肠肠道某一种群细菌的数量,越粗越亮,表示该种群细菌的数量越多,反之亦然。图中不同泳道的条带位置、数量、亮度及粗细的差异表明同一周龄下不同饲养密度之间和同一饲养密度下不同周龄之间蛋鸡十二指肠肠道菌群的结构组成均存在差异。

|
图 2 电泳分析结果(图谱中条带旁边数字为切胶编号) Fig. 2 Electrophoresis analysis results (the data besides band represent the gel extraction number) |
图3下方的百分比表示以1组样品为标准,其他各组样品与其整体的相似度。其中,3组样品与1组样品的整体相似度最高为48.2%,14组样品与1组样品的整体相似度最低为19.3%,说明各组之间菌群相似度较低,差异较大。由图3可知,1~15组样品中含有的条带数目分别是17条、14条、22条、22条、15条、22条、14条、15条、18条、12条、17条、22条、24条、14条、21条。1~15组样品没有完全共有的条带,说明菌群结构存在差异。
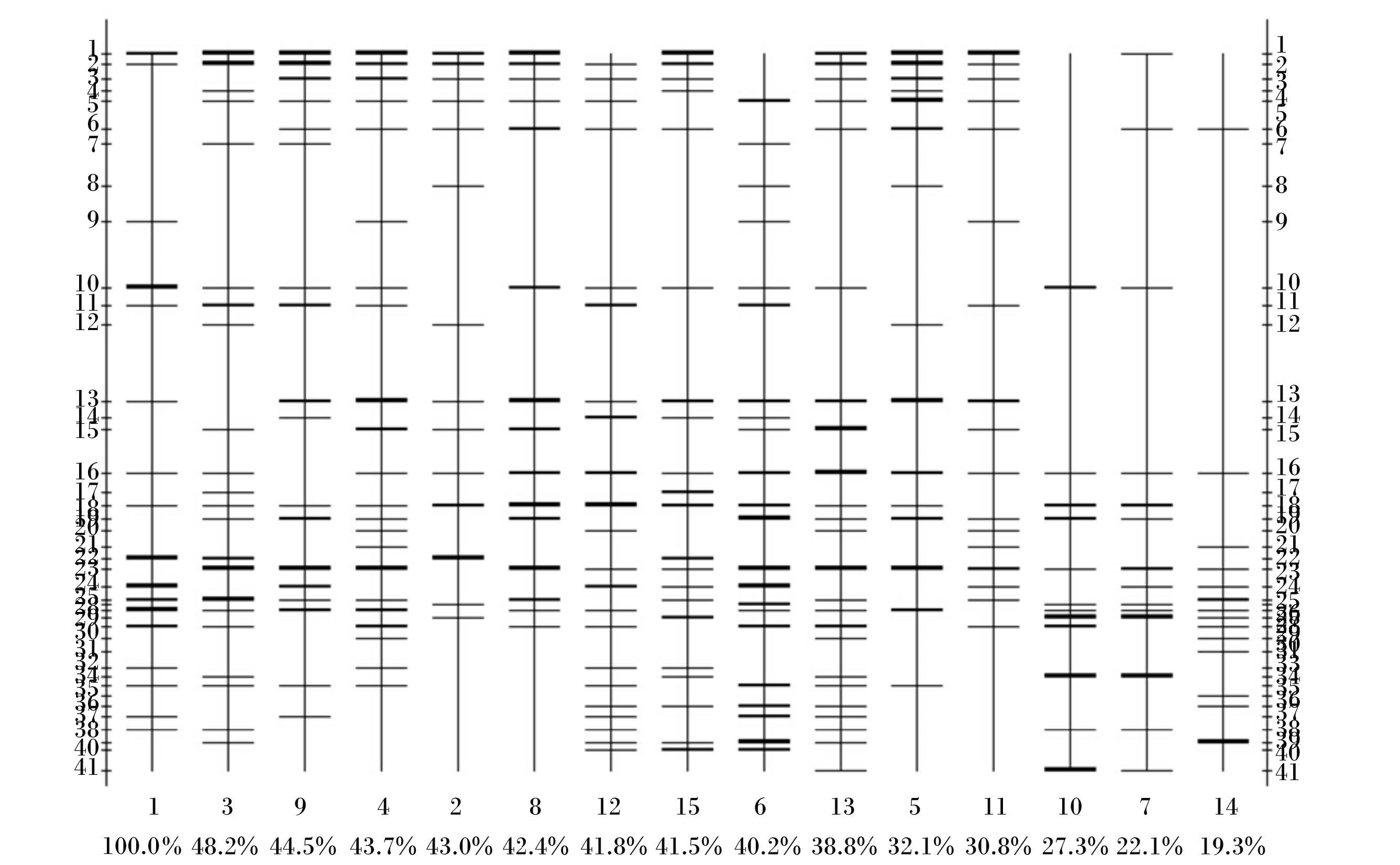
|
图 3 电泳分析结果模式图 Fig. 3 Model of electrophoresis analysis results |
由表3可知,同一饲养密度条件下不同周龄内笼养蛋鸡十二指肠菌群结构相似指数在12.1%~64.5%之间,其中6只/笼的试验组16与26周龄的菌群结构相似性最低,相似系数仅为12.1%;4只/笼的试验组16与26周龄的菌群结构相似性最高,相似系数为64.5%。整体来看,在同一个饲养密度内,随着饲养周龄的增加笼养蛋鸡十二指肠肠道菌群结构相似系数逐渐减小,肠道菌群结构差异逐渐增加,即饲养周龄对蛋鸡十二指肠肠道菌群结构影响很大。
|
|
表 3 PCR-DGGE图谱的相似性系数 Table 3 Resemblance coefficient of PCR-DGGE map |
同一周龄条件下不同饲养密度内笼养蛋鸡十二指肠菌群结构相似指数在6.0%~70.3%之间,其中26周龄时B组与E组的菌群结构相似性最低,相似系数为5.4%;16周龄时B组与E组的菌群相似性最高,相似系数为70.3%。总体来看,16周龄时高密度组与低密度组之间十二指肠肠道菌群结构相似性很高,即肠道菌群结构差异较小;26和50周龄时高密度组与低密度组之间十二指肠肠道菌群结构相似性很低,即肠道菌群结构差异很大。
2.4 主要电泳条带的序列测定DGGE凝胶条带回收后,以338F/518R为引物进行PCR扩增,获得目的DNA片段。PCR产物纯化后连接到pMD18-T载体上,转化至DH5α感受态细胞中,筛选阳性克隆测序。
测序结果与GenBank中的序列进行比对,得到条带所代表的细菌类型。每个回收条带选取3个克隆进行了序列测定,结果见表4。32个测序结果中,微生物的同源性在84%~100%之间,大多数均大于98%,10个条带的同源性达到了100%,仅6个条带的同源性小于98%,其中条带30与数据库中Pediococcus siamensis菌株的相似度仅为84%,因此该序列代表的微生物可能为厚壁菌门(Firmicutes)新种。各组样品中检测到26种厚壁菌门细菌、5种蛋白菌门(Proteobacteria)细菌、1种放线菌门(Actinobacteria)细菌,由此可以看出,蛋鸡十二指肠肠道内的优势菌群为厚壁菌门。第28号Pseudomonas japonica细菌仅第14组所特有;第16号Lactobacillus equi细菌是除第9组之外其他各组的共有菌;第7号Romboutsia ilealis细菌、第8号Lactobacillus gastricus细菌、第9号Lactobacillus vaginalis细菌、第10号Lactobacillus alvi细菌随着饲养密度的增加逐渐消失;第17号Lactobacillus equi细菌和第19号Enterococcus cecorum细菌在最高密度组中和最低密度组中均检测到。
|
|
表 4 序列比对分析结果 Table 4 Sequence bipartition analytic results |
采用MEGA 5软件,Neighbor-joining法构建系统发育树,自展数(bootstrap)为1 000,系统发育树如图4。
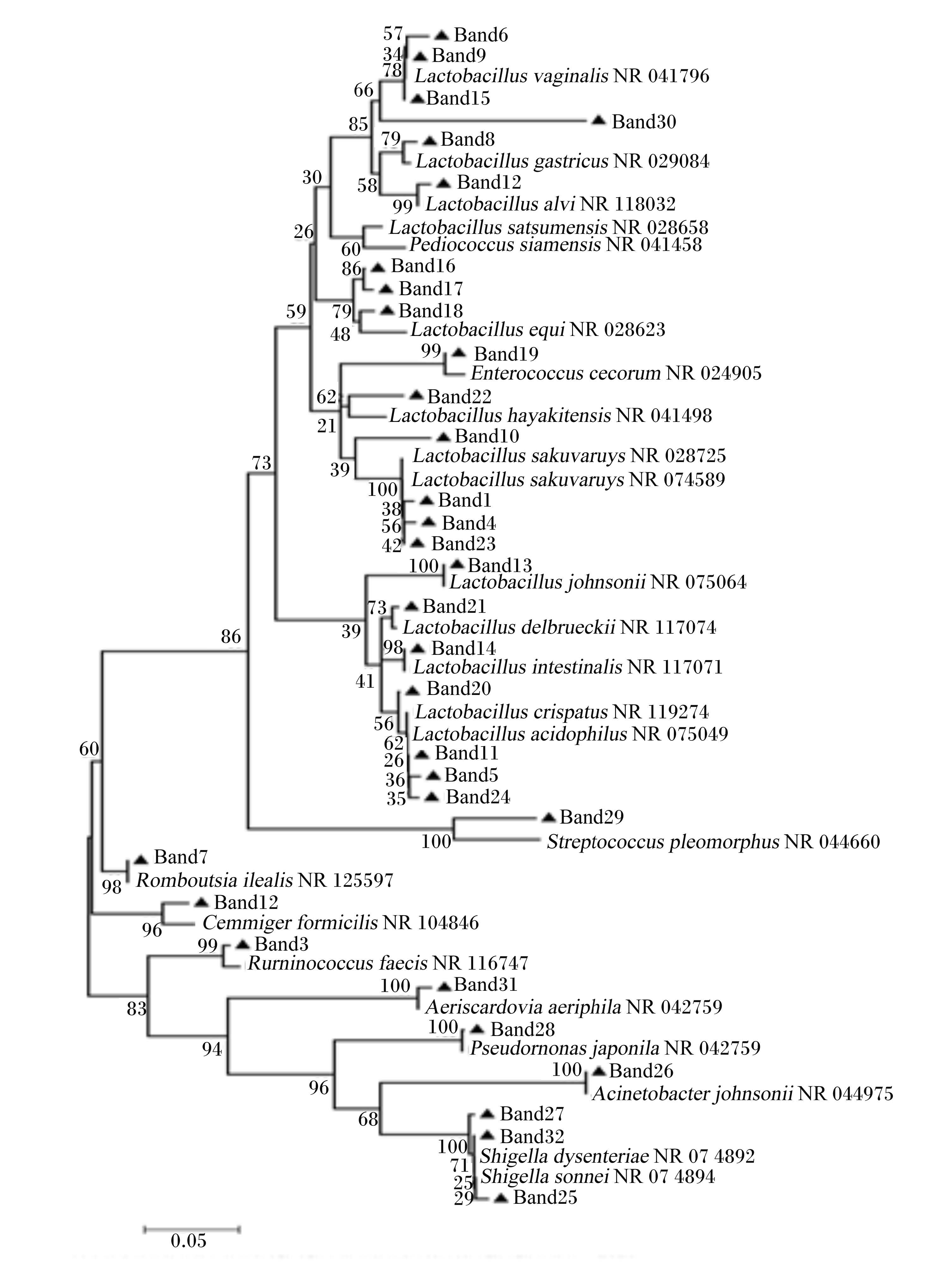
|
图 4 系统发育树 Fig. 4Phylogenetic trees |
家禽生长发育过程中,肠道微生物菌群会产生各种对机体有利或有害的代谢产物,菌群和胃肠道上皮细胞之间的相互作用导致消化道各种结构和功能的改变,菌群不仅可以影响脂肪的消化和改变碳水化合物、蛋白质的消化,还能够增加能量和蛋白质的需要量,但对维生素的消化吸收具有负面的影响,此外,有益菌可以保护机体免受病原菌的侵入,是肠道免疫系统的一部分,所以说,肠道微生物区系对肠道功能起着至关重要的作用[8]。
肠道微生物的组成复杂,数量和种类因家禽的种类、年龄[9]、饲料和环境[10]而异,即使在相同饲养环境下的不同个体肠道菌群结构也存在差异,为了减小个体差异对试验的影响,本试验在每个组中都随机选取3~6个重复,每个重复选取5只笼养蛋鸡,提取十二指肠内容物进行检测,使试 验数据更准确,提高PCR-DGGE图谱的相似性。 然而本试验不同饲养密度蛋鸡十二指肠肠道菌群的共有菌仅1株,即第16号Lactobacillus equi细菌,由此可见,饲养环境即使相同,鸡只肠道菌群仍存在很大差异,分析原因可能与本试验鸡群是大群饲养(30 000只,5层笼)、饲养空间的大小和鸡群群体大小对肠道菌群都产生影响有关。但各鸡十二指肠的共同菌即第16号Lactobacillus equi细菌曾作为日本赛马粪便中的优势菌群被报道[11, 12],说明该细菌为十二指肠肠道菌群的优势菌,并且表明该优势菌群不受饲养周龄和饲养环境的影响。第19号Enterococcus cecorum细菌是一种致病菌,大量的试验表明该细菌可以导致家禽产生关节炎及骨髓炎[13, 14, 15],本试验中在最低密度A组和最高密度E组中均检测到该细菌,说明过高或过低的密度对十二指肠肠道菌群结构均有影响。大量试验研究指出,第8号Lactobacillus gastricus细菌[16, 17]和第10号Lactobacillus alvi细菌[18]是可以维护肠道平衡,防止病原菌入侵的有益菌,但在本试验中这2种细菌均随着饲养密度的增加而消失,说明饲养密度对十二指肠肠道菌群有影响,过高的饲养密度对其有不利的影响。
有研究显示,肠道中的有益菌可以在保证鸡蛋蛋重不变的情况下增加蛋壳的重量并提高蛋壳强度[19]。有益菌也可以通过改变有害菌的受体,从而阻断其在消化道中的生长,总体而言,饲养密度或热应激的增加会加大有害菌对有益菌的损害[10]。倪学勤等[20]采用PCR-DGGE技术分析蛋鸡年龄和肠段部位对微生物群落结构和多样性的影响,结果表明蛋鸡肠道部位决定细菌群落的结构和多样性,蛋鸡的周龄影响肠道细菌多样性;倪学勤等[21]通过PCR-DGGE技术分析蛋鸡主要组织相容性复合体(MHC)基因对肠道细菌种群结构的影响,得出蛋鸡盲肠细菌的多样性最高,其次是回肠和空肠,嗉囊和十二指肠的细菌多样性比较低,且随着蛋鸡周龄增加,各肠段细菌多样性呈上升趋势;李永洙等[22]通过PCR-DGGE技术分析不同品种、饲养阶段的健康和不良鸡群对盲肠细菌种群结构和多样性的影响,结果显示,不同品种、饲养阶段的鸡群,其盲肠细菌群落的组成差异显著,并且细菌种群结构对鸡群的生长发育影响较大;王秋菊等[23]采用PCR-DGGE技术分析笼养海兰褐蛋雏鸡肠道菌群,得出同龄笼养海兰褐蛋雏鸡肠道细菌菌群结构与肠道的不同部位有关,各肠段中细菌菌群的组成差异很大。在正常蛋鸡饲养过程中,开产前与开产后蛋鸡饲粮配方不同,可能会影响蛋鸡肠道细菌组成。但本试验结果显示,16周龄时,不同饲养密度下笼养蛋鸡十二指肠肠道菌群结构差异不显著;饲喂同一饲粮的26和50周龄蛋鸡,不同饲养密度下笼养蛋鸡十二指肠肠道菌群结构差异显著,说明日龄对蛋鸡肠道菌群产生影响;而且同一个饲养密度下,随着饲养时间的延长,笼养蛋鸡十二指肠肠道菌群结构相似系数逐渐减小,肠道菌群结构差异逐渐增加,即饲养时间对蛋鸡十二指肠肠道菌群结构影响很大。
4 结 论① 饲养密度对笼养蛋鸡十二指肠肠道菌群产生影响,随着饲养密度增加,即饲养密度高于450 cm2/只时,笼养蛋鸡十二指肠中有益菌(Lactobacillus gastricus和Lactobacillus alvi)消失,对肠道菌群平衡产生不利影响。
② 在同一个饲养密度下,随着饲养时间的延长,笼养蛋鸡十二指肠肠道菌群结构相似系数逐渐减小,肠道菌群结构差异逐渐增加,即随着饲养周龄增加,蛋鸡十二指肠肠道菌群结构发生差异变化。
| [1] | ONBASILAR E E,AKSOY F T.Stress parameters and immune response of layers under different cage floor and density conditions[J]. Livestock Production Science,2005,95(3):255-263. ( 1) 1)
|
| [2] | 王龙.饲养密度对层叠笼养蛋鸡生产性能以及福利影响[D]. 硕士学位论文.大庆:黑龙江八一农垦大学,2015:25-29. ( 1) 1)
|
| [3] | SOHAIL M U,HUME M E,BYRD J A,et al.Molecular analysis of the caecal and tracheal microbiome of heat-stressed broilers supplemented with prebiotic and probiotic[J]. Avian Pathology,2015,44(2):67-74. ( 1) 1)
|
| [4] | ROBERTS T,WILSON J,GUTHRIE A,et al.New issues and science in broiler chicken intestinal health:intestinal microbial composition,shifts,and impacts[J]. World's Poultry Science Journal,2015,71(2):259-270. ( 1) 1)
|
| [5] | GONG J H,SI W D,FORSTER R J,et al.16S rRNA gene-based analysis of mucosa-associated bacterial community and phylogeny in the chicken gastrointestinal tracts:from crops to ceca[J]. FEMS Microbiology Ecology,2007,59(1):147-157. ( 1) 1)
|
| [6] | JANCZYK P,HALLE B,SOUFFRANT W.Microbial community composition of the crop and ceca contents of laying hens fed diets supplemented with Chlorella vulgaris[J]. Poultry Science,2009,88(11):2324-2332. ( 1) 1)
|
| [7] | KIM G B,SEO Y M,KIM C H,et al.Effect of dietary prebiotic supplementation on the performance,intestinal microflora,and immune response of broilers[J]. Poultry Science,2011,90(1):75-82. ( 1) 1)
|
| [8] | GABRIEL I,LESSIRE M,MALLET S,et al.Microflora of the digestive tract:critical factors and consequences for poultry[J]. World's Poultry Science Journal,2006,62(3):499-511. ( 1) 1)
|
| [9] | KNARREBORG A,SIMON M A,ENGBERG R M,et al.Effects of dietary fat source and subtherapeutic levels of antibiotic on the bacterial community in the ileum of broiler chickens at various ages[J]. Applied and Environmental Microbiology,2002,68(12):5918-5924. ( 1) 1)
|
| [10] | SUZUKI K,KODAMA Y,MITSUOKA T.Stress and intestinal flora[J]. Bifidobacteria and Microflora,1989,8(1):23-38. ( 2) 2)
|
| [11] | ENDO A,OKADA S,MORITA H.Molecular profiling of Lactobacillus,Streptococcus,and Bifidobacterium species in feces of active racehorses[J]. Journal of General and Applied Microbiology,2007,53(3):191-200. ( 1) 1)
|
| [12] | ENDO A,ROOS S,SATOH E,et al.Lactobacillus equigenerosi sp.nov.,a coccoid species isolated from faeces of thoroughbred racehorses[J]. International Journal of Systematic and Evolutionary Microbiology,2008,58(4):914-918. ( 1) 1)
|
| [13] | BORST L B, SUYEMOTO M M,SHIVARAMU K,et al.A chicken embryo lethality assay for pathogenic Enterococcus cecorum[J]. Avian Diseases,2014,58(2):244-248. ( 1) 1)
|
| [14] | KENSE M J,LANDMAN W J M.Enterococcus cecorum infections in broiler breeders and their offspring:molecular epidemiology[J]. Avian Pathology,2011,40(6):603-612. ( 1) 1)
|
| [15] | BOERLIN P,NICHOLSON V,BRASH M,et al.Diversity of Enterococcus cecorum from chickens[J]. Veterinary Microbiology,2012,157(3/4):405-411. ( 1) 1)
|
| [16] | CÁRDENAS N,MARTÍN V,DELGADO S,et al.Characterisation of Lactobacillus gastricus strains isolated from human milk[J]. International Dairy Journal,2014,39(1):167-177. ( 1) 1)
|
| [17] | MARTÍN V,CÁRDENAS N,JIMÉNEZ E,et al.Genome sequence of Lactobacillus gastricus PS3,a strain isolated from human milk[J]. Genome Announcements,2013,1(4): e00489-13,doi: 10.1128/genomeA.00489-13.. ( 1) 1)
|
| [18] | KIM H J,EOM S J,PARK S J,et al.Lactobacillus alvi sp.nov.,isolated from the intestinal tract of chicken[J]. FEMS Microbiology Letters,2011,323(1):83-87. ( 1) 1)
|
| [19] | PANDA A K,REDDY M R,RAMARAO S V,et al.Effect of dietary supplementation of probiotic on performance and immune response of layers in the decline phase of production[J]. Indian Journal of Poultry Science,2000,35(1):102-104. ( 1) 1)
|
| [20] | 倪学勤,GONG J,YU H,等.采用PCR-DGGE技术分析蛋鸡肠道细菌种群结构及多样性[J]. 畜牧兽医学报,2008,39(7):955-961. ( 1) 1)
|
| [21] | 倪学勤,GONG J,YU H,等.PCR-DGGE技术分析蛋鸡MHC基因对肠道细菌种群结构的影响[J]. 中国农业科学,2009,42(7):2564-2571. ( 1) 1)
|
| [22] | 李永洙,CUI Y Q.利用PCR-DGGE方法分析不同鸡群的盲肠微生物菌群结构变化[J]. 生态学报,2011,31(21):6513-6521. ( 1) 1)
|
| [23] | 王秋菊,崔一喆,刘胜军,等.笼养海兰褐蛋雏鸡肠道菌群的聚合酶链式反应—变性梯度凝胶电泳分析[J]. 动物营养学报,2014,26(2):504-512. ( 1) 1)
|




