2. 浙江大学动物科学学院, 杭州 310029
2. College of Animal Sciences, Zhejiang University, Hangzhou 310029, China
蛋白质的磷酸化修饰是广泛存在于生物体内各种组织细胞中的一种蛋白质翻译后修饰方式,机体大约30%的蛋白质可发生不同形式的磷酸化[1],它参与和调控生物体内的许多生命活动。生物体通过蛋白质的磷酸化与去磷酸化,调控信号转导、基因表达、细胞周期等诸多细胞过程。研究表明,植物体内某些蛋白质的磷酸化与温度胁迫存在一定的关系,比如叶绿体D1蛋白和光系统Ⅱ蛋白等。因此,研究蛋白质的磷酸化不仅可以了解其对蛋白质功能的影响,而且还可以帮助我们更加深入地理解生命系统在分子水平的调控状况[2]。
30K蛋白是一类在氨基酸组成及免疫学活性上相似的蛋白质,理论等电点在6.1~8.4之间,分子质量约为30 ku[3]。在家蚕(Bombyx mori)体中目前至少有35种分子质量约为30 ku的脂蛋白登录在美国国立生物技术信息中心(NCBI)上,有人推测其多样化可能与蛋白质的磷酸化有关。30K蛋白在五龄蚕幼虫和蚕蛹的血液中大量积累,由脂肪体合成分泌,表达量受保幼激素的调节[4]。30K蛋白是家蚕生长发育过程中极其重要的储藏蛋白,也有研究指出30K蛋白中的6G1和19G1可以特异性的与葡萄糖及葡聚糖结合,参与昆虫的免疫反应[5];而且,19G1还可能是家蚕血液中抗细胞凋亡最强的成分[6]。尽管人们通过试验的手段掌握了一些30K蛋白的功能,但是还有大量30K蛋白的功能处于未知状态[7]。比如,30K蛋白是否参与热激应答,其磷酸化是否与热激有关等,还需要进一步研究。
家蚕是重要的经济昆虫,同时也是鳞翅目昆虫的代表和模式种[8]。温度对家蚕尤其是壮蚕期的生长发育至关重要,高温往往是造成夏秋季蚕茧减产和茧质下降的主要原因。鉴于30K蛋白在五龄家蚕血液中的地位和角色以及蛋白质磷酸化在生物活动中发挥的重要作用,本试验利用高温刺激家蚕,借助双向电泳、Pro-Q Diamond荧光染色以及质谱技术探讨家蚕血液中30K蛋白的功能,分析其表达变化和磷酸化现象,进一步研究它们在环境胁迫中发挥的作用。
1 材料与方法 1.1 试验材料供试家蚕品种为秋丰(Q),由浙江大学动物科学学院蚕桑遗传育种实验室提供。家蚕幼虫均用新鲜桑叶按照常规条件饲育至五龄第3天。试验设对照组和热激组,每组随机选取30头五龄第3天家蚕幼虫(雌雄各占1/2),放在40 ℃的培养箱中分别热激0、10 min后,剪尾角取血,血液样品经液氮冷冻后于-80 ℃保存备用。
1.2 血液蛋白制备取出血液样品,放在冰上低温缓慢解冻,然后按1 μL血液加10 μL裂解缓冲液[8 mol/L尿素、2 mol/L硫脲、4%(V/V)3-环乙胺-1-丙磺酸、2%(V/V)载体两性电解质(pH 3~10)和30 mmol/L二硫苏糖醇]的比例加入裂解缓冲液,25 ℃振荡混匀,并在30 ℃摇床上摇动,每隔10 min振荡混匀1次。室温下放置10 min,超声波处理30 s,冰浴30 s,重复4次,以使样品充分裂解。于4 ℃下15 000 r/min离心2次,每次15 min,吸取上清液。血液蛋白定量方法采用Bradford[9]的方法,在595 nm处测定标准蛋白和样品的吸光度,采用标准曲线法测定计算样品蛋白浓度。最后,根据所需蛋白量分装至离心管中,-20 ℃下保存备用。
1.3 双向电泳及染色双向电泳参考Zhou等[10]的报道,采用Ettan IPGphor3等电聚焦和聚丙烯酰胺凝胶电泳垂直电泳系统Ettan Daltsix (Amersham Biosciences),胶条采用pH为3~10的24 cm线性干胶条。双向电泳完成后,小心取出凝胶,清洗后进行硝酸银和Pro-Q Diamond荧光染色。
1.4 图谱扫描和分析银染胶采用Image Scanner 2D扫描仪(Amersham Bioscience)进行扫描。通过Image Master 2D platinum分析软件进行图像分析,检测蛋白斑点,计算蛋白点百分体积,并预测其分子质量和等电点。
1.5 30K蛋白质谱鉴定和表达分析挖取Pro-Q Diamond荧光染色胶中对应的30K蛋白点,送交浙江理工大学进行质谱鉴定。胶内酶解方法参照Gharahdaghi等[11]的报道。质谱鉴定采用文献[12]中的方法和参数,质谱仪为ABI公司生产的4700 Proteomics Analyzer。数据库选用NCBInr,搜库软件为GPS ExploreTM software。
1.6 鉴定蛋白磷酸化位点预测首先登录NCBI数据库进行比对,得到鉴定蛋白的Fasta序列。下载保存后,利用KinasePhos(http://kinasephos.mbc.nctu.edu.tw/)在线提交。预测参数的设定如下:预测位点选择酪氨酸(Y)、丝氨酸(S)与苏氨酸(T)3种氨基酸,激酶选择非特定,特异性为100%,进行磷酸化位点的预测[13]。
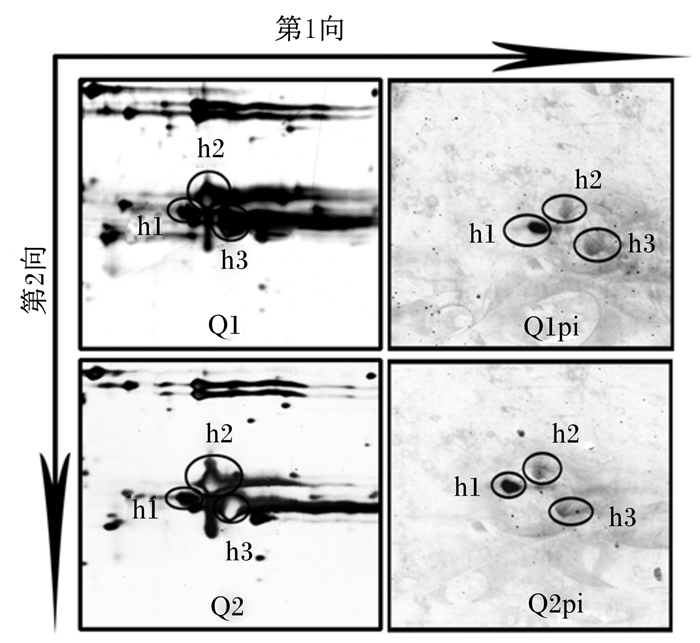
2 结果与分析 2.1 血液磷酸化蛋白图谱分析图 1为Pro-Q Diamond染色后不同热激时间五龄第3天家蚕血液磷酸化蛋白的双向电泳图谱。从图中可以看出,对照组(Q1pi)和热激组(Q2pi)中各发现磷酸化蛋白点5个(h1、h2、h3、h4和h5)。通过分析分子质量和等电点,结果发现,h4和h5的分子质量在45 ku左右,等电点在4.6左右,因此可以推测它们是30K蛋白的可能性极小。另外,从着色深浅判断,h1、h4和h5的颜色明显比h2和h3重,说明这3个蛋白的表达量较高,而且h1在性质上可能与h2和h3有一定的差异。图 2为30K蛋白银染图和Pro-Q Diamond染色图。从银染图中可以看出,h1、h2和h3这3个蛋白与其他蛋白处于胶着状态,表明五龄家蚕血液中累积了大量的30K蛋白,可能在蚕体脂质运输方面承担着重要的角色。

|
图1 家蚕血液磷酸化蛋白双向电泳图谱 Fig. 1 Two-dimensional electrophoresis map of phosphorylation proteins from silkworm haemolymph |
|
图2 30K蛋白银染图(左)和 Pro-Q Diamond染色图(右) Fig. 2 Silver stain map (L) and Pro-Q Diamond dye map (R) for 30K proteins |
通过多次取样和重复测定,成功鉴定了h1、h2和h3这3个蛋白(表 1),它们分别是30K蛋白中的PBMHPC-6 (h1)、PBMHPC-19 (h2)和PBMHPC-12 (h3)。
|
|
表 1 家蚕血液中30K蛋白的质谱鉴定 Table 1 MS identification of 30K proteins in silkworm haemolymph |
质谱鉴定结果表明(表 1),3个蛋白的序列覆盖度都在42.5%以上,蛋白得分最低为80分,匹配肽段数都在11个之上,等电点、分子质量和理论值相差不大,说明了鉴定结果的可靠性。
2.3 30K蛋白差异分析及磷酸化位点预测从图 2左侧的银染图可以看出,3个蛋白点几乎聚集在一起,和周围蛋白呈现胶着状态,所以无法进行表达差异分析。Pro-Q Diamond荧光染色解决了这个问题,从图 2右侧的Pro-Q Diamond染色图上看,3个蛋白得到了很好的分离,尤其是h1着色明显,表达量大。通过Image Master 2D platinum分析软件计算蛋白点百分体积,结果发现,热激处理10 min后3个蛋白(PBMHPC-6、PBMHPC-19和PBMHPC-12)的百分体积分别是对照组的2.15、1.86和1.81倍。根据惯例,把蛋白百分体积比值大于1.80或小于0.55作为差异显著的判定标准,这说明3个30K蛋白的表达量在热激处理后显著上调,意味着它们很可能参与热激应答。
采用KinasePhos软件预测3个蛋白的磷酸化位点,发现PBMHPC-6和PBMHPC-12各有6个位点可能会发生磷酸化,PBMHPC-19有4个位点(表 2)。以PBMHPC-6为例,6个位点共预测出10个可选修饰。根据E值和隐马尔科夫得分剔除假阳性率高的修饰,得到了第34、76、140、169、217和239位氨基酸上的可能修饰,以及5种磷酸化蛋白激酶(表 3)。
|
|
表 2 3个30K蛋白磷酸化预测位点 Table 2 Predicted phosphorylated sites of three 30K proteins |
|
|
表 3 PBMHPC-6磷酸化预测位点 Table 3 Predicted phosphorylated sites of PBMHP-6 |
30K蛋白是家蚕血液中丰度比较高的一类蛋白质,其表达具有时空特异性[14]。半定量PCR结果显示,家蚕大部分的30K蛋白基因活性在五龄第3天起上升,五龄第4天开始到吐丝期活性最高[15]。Sun等[14]对家蚕血液进行了双向电泳和质谱分析,结果发现5个不同的30K蛋白被多次检测到,推测其多样化可能与蛋白质的磷酸化有关。随后,他们用SignalP程序对30K蛋白的氨基酸序列信号肽进行了预测,结果发现它们的基因都含有信号肽,说明它们具有分泌性蛋白的属性。因为30K蛋白序列同源性较高,加之分子质量大小相似,所以其分离纯化是一个复杂的过程。从本试验的结果来看,30K蛋白聚集在一起呈现胶着状态(图 2),很难通过Imagemaster软件对银染图进行定量分析。而本试验借鉴前人的研究,采用Pro-Q Diamond染料对这些蛋白进行特异性染色,既保证了试验较高的灵敏性,还可以与质谱鉴定兼容[15]。本试验正是借助了Pro-Q Diamond荧光染色法的优点使得30K蛋白得以分开。
Sun等[14]基于家蚕基因组图谱和30K蛋白基因序列整合了10个30K蛋白的基因,发现5个基因Bmlp1、Bmlp4、Bmlp7、Bmlp8和Bmlp10都有1个核转录因子κB(NF-κB)结合位点,该因子的主要功能是调节与免疫应答相关基因的表达。Yang等[15]对家蚕血浆30K蛋白进行了天然分离、重组表达和结构解析,认为Bmlp7的N段区域(NTD)可能具有结合脂的能力,而C段区域(CTD)存在结合糖的潜在位点,因而可能间接的参与昆虫的天然免疫反应。通过本试验对荧光图谱的分析,发现30K蛋白在短时间的热激之后都呈现表达显著上调的趋势。这是否暗示着30K蛋白的功能可能与胁迫应答有关?有研究发现温度胁迫可以引起甘蓝22.9K蛋白的磷酸化现象,而且在逆境温度处理10 min内,磷酸化蛋白含量升高[16]。这个结果在本试验中得到了进一步的验证,暗示了蛋白质磷酸化在逆境胁迫中的作用。另外,由于蛋白质的磷酸化和激酶有关,而且磷酸化位点主要发生于酪氨酸、丝氨酸与苏氨酸残基上[17],因此大量的预测磷酸化位点的工具得到了开发。本试验借助KinasePhos软件对3个30K蛋白(PBMHPC-6、PBMHPC-19和PBMHPC-12)的磷酸化位点进行了预测,为进一步研究它们的磷酸化提供了参考,但是具体位点和作用激酶仍需要经过试验的验证。
4 结 论① 40 ℃热激家蚕10 min后有3个30K蛋白(PBMHPC-6、PBMHPC-19和PBMHPC-12)发生了磷酸化。这3个30K蛋白热激后表达量显著上调,说明30K蛋白很可能参与热激应答。
② KinasePhos软件预测发现30K蛋白PBMHPC-6和PBMHPC-12各有6个位点可能会发生磷酸化,而PBMHPC-19仅有4个位点可能会发生磷酸化。
| [1] |
RAGGIASCHI R, GOTTA S, TERSTAPPEN G C. Phosphoproteome analysis[J].
Bioscience Reports, 2005, 25(1/2): 33–44.
( 0) 0)
|
| [2] |
杨策, 王正国, 朱佩芳. 蛋白质组中蛋白质磷酸化研究进展[J].
生理科学进展, 2004, 35(2): 119–124.
( 0) 0)
|
| [3] |
KIM E J, PARK H J, PARK T H. Inhibition of apoptosis by recombinant 30K protein originating from silkworm hemolymph[J].
Biochemical and Biophysical Research Communications, 2003, 308(3): 523–528.
( 0) 0)
|
| [4] |
SAKAI N, MORI S, IZUMI S, et al. Structures and expression of mRNAs coding for major plasma proteins of Bombyx mori[J].
Biochimica et Biophysica Acta:Gene Structure and Expression, 1988, 949(2): 224–232.
( 0) 0)
|
| [5] |
UJITA M, KIMURA A, NISHINO D, et al. Specific binding of silkworm Bombyx mori 30-kDa lipoproteins to carbohydrates containing glucose[J].
Bioscience,Biochnology,and Biochemistry, 2002, 66(10): 2264–2266.
( 0) 0)
|
| [6] |
KIM E J, RHEE W J, PARK T H. Isolation and characterization of an apoptosis-inhibiting component from the hemolymph of Bombyx mori[J].
Biochemical and Biophysical Research Communications, 2001, 285(2): 224–228.
( 0) 0)
|
| [7] |
PIETRZYK A J, BUJACZ A, ŁOCHYÑSKA M, et al. Isolation,purification,crystallization and preliminary X-ray studies of two 30 kDa proteins from silkworm haemolymph[J].
Acta Crystallographica Section F, 2011, 67(3): 372–376.
( 0) 0)
|
| [8] |
GOLDSMITH M R, SHIMADA T, ABE H. The genetics and genomics of the silkworm,Bombyx mori[J].
Annual Review of Entomology, 2005, 50: 71–100.
( 0) 0)
|
| [9] |
BRADFORD M M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J].
Analytical Biochemistry, 1976, 72(1/2): 248–254.
( 0) 0)
|
| [10] |
ZHOU Z H, YANG H J, CHEN M, et al. Comparative proteomic analysis between the domesticated silkworm (Bombyx mori) reared on fresh mulberry leaves and on artificial diet[J].
Journal of Proteome Research, 2008, 7(12): 5103–5111.
( 0) 0)
|
| [11] |
GHARAHDAGHI F, WEINBERG C R, MEAGHER D A, et al. Mass spectrometric identification of proteins from silver-stained polyacrylamide gel:a method for the removal of silver ions to enhance sensitivity[J].
Electrophoresis, 1999, 20(3): 601–605.
( 0) 0)
|
| [12] |
LI J S, YE L P, LAN T Y, et al. Comparative proteomic and phosphoproteomic analysis of the silkworm (Bombyx mori) posterior silk gland under high temperature treatment[J].
Molecular Biology Reports, 2012, 39(8): 8447–8456.
( 0) 0)
|
| [13] |
HUANG H D, LEE T Y, TSENG S W, et al. KinasePhos:a web tool for identifying protein kinase-specific phosphorylation sites[J].
Nucleic Acids Research, 2005, 33(Suppl.2): W226–W229.
( 0) 0)
|
| [14] |
SUN Q, ZHAO P, LIN Y, et al. Analysis of the structure and expression of the 30K protein genes in silkworm,Bombyx mori[J].
Insect Science, 2007, 14(1): 5–14.
( 0) 0)
|
| [15] |
YANG J P, MA X X, HE Y X, et al. Crystal structure of the 30 K protein from the silkworm Bombyx mori reveals a new member of the β-trefoil superfamily[J].
Journal of Structural Biology, 2011, 175(1): 97–103.
( 0) 0)
|
| [16] |
吴能表, 朱利泉, 王小佳. 温度胁迫对甘蓝蛋白质磷酸化的影响[J].
西南农业大学学报:自然科学版, 2004, 26(2): 198–200.
( 0) 0)
|
| [17] |
WU H Y, TSENG V S M, LIAO P C. Mining phosphopep tide signals in liquid chromatography-mass spectrometry data for protein phosphorylation analysis[J].
Journal of Proteome Research, 2007, 6(5): 1812–1821.
( 0) 0)
|










