近年来,有4种非编码的单链短序列RNA分子被相继发现,分别为小干扰RNA(small interfering RNA, siRNA)、与Piwi蛋白相作用的RNA(piRNA)、微小RNA(microRNA, miRNA)及其他小RNA(eiRNA),均是通过转录后水平调控细胞的功能及肿瘤发生等机体生命发生过程。随着基因表达研究的不断深入,miRNA受到研究者们的普遍关注。这是一类大家族非编码RNA,可以介导RNA分子进行基因的沉默,并通过基因转录、翻译、表达来调节机体的生命活动。本文针对miRNA在反刍动物中的鉴定、表达和功能的研究进展进行综述。
1 miRNA的来源及作用机制miRNA是一类22个核苷酸左右的小分子单链非编码RNA,通过转录后调控抑制基因的表达。miRNA最初是在新杆状线虫中发现,因为其抑制蛋白编码基因lin-14的表达而被称为lin-4。随后多种动植物中的上百种miRNA被发现,并且发现它们在多种生物学过程中发挥作用,包括细胞增殖、分化、凋亡和发育。
在真核生物中,miRNA大多是由自身编码产生的,具有很高的保守性。miRNA是由一系列核酶参与加工形成。首先是miRNA的转录启动,是由RNA聚合酶Ⅱ在miRNA前体(pre-miRNA)发夹环上的结合开始的。之后原始前体转录物在微处理前体的作用下形成70~80个核苷酸并且包含茎环结构的前体。随后发生了一个限速反应过程,即pre-miRNA通过核转运体Exportin-5运送到细胞质,最后在核糖核酸酶Ⅲ(RNaseⅢ)的作用下,去除茎环,保留长度在20~22个核苷酸左右的双链miRNA复合体,最后RNaseⅢ-封闭肽复合物将双链miRNA复合体裂解为2条单链。由于稳定性不同使得稳定性较低的miRNA单链与RNA诱导基因沉默复合物(RISC)结合,形成成熟的miRNA,而另一条miRNA单链则被核酸酶降解消除,如图 1所示。
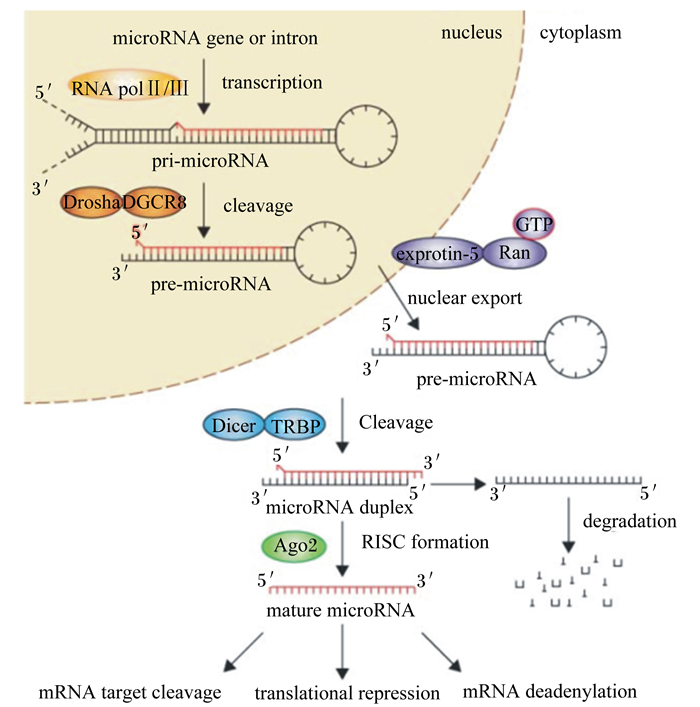
|
nucleus:细胞核;cytoplasm:细胞质;miRNA gene or intron:miRNA基因或内含子;RNA polⅡ/Ⅲ:RNA聚合酶Ⅱ/Ⅲ;exportin-5:输出蛋白5;microRNA duplex:microRNA复合体;RISC formation:RNA诱导基因沉默复合物形成;transcription:转录;cleavage:分裂;DGCR8:DiGeorge综合征相关蛋白8 DiGeorge syndrome critical region 8;TRBP:艾滋病病毒1型反应式激活RNA结合蛋白HIV-1 transactivating response RNA binding protein;mature miRNA:成熟miRNA;degradation:降解;mRNA target cleavage:mRNA靶分裂;translation repression:翻译阻抑;mRNA deadenylation:mRNA脱腺苷化。 图1 miRNA的生物合成 Fig. 1 Biosynthesis of miRNA[1] |
miRNA靶基因可以通过计算方法进行预测。这些方法主要是通过序列互补的原理来预测的,如miRanda和TargetScan法;而PicTar、DIANAmicroT、RNAhybrid、microInspector、RNA22和SVMicrO等方法则是通过不同的计算原理来实现的[2]。很难评价这些预测方法的准确性, 但多种预测方法共同预测,找出同样结果的靶基因,这种方法的准确率要远远高于单一软件的预测。miRNA的靶基因预测结果需要进一步的试验验证,常见的验证试验包括miRNA的过表达或沉默以及后续采用实时定量PCR(qRT-PCR)和荧光素酶报告试验对预测靶基因的鉴定。miRNA沉默通常是通过反义寡核苷酸(ASO)来实现的,这些反义序列与miRNA互补结合,致使碱基无法与miRNA的靶基因结合。ASO的化学修饰有2种方式:2′-O-甲基反义寡核苷酸(AMO)和锁核酸(LNA)。与AMO相比,LNA具有较高的抑制效率。另一种ASO是miRNA消除,RNA序列从表达载体转录起始,含有多种与miRNA结合的串联结合位点。miRNA消除竞争性地与miRNA结合,因此阻止了与其余靶基因的结合[3]。大量miRNA靶基因的预测工作可以通过蛋白质组学的方法完成。如选择反应检测(SRM)和同位素编码标记定量法可从秀丽隐杆线虫野生型和miR-let7突变型提取出上百个miR-let7靶蛋白,161个蛋白可以定量,其中29个在野生型和突变型之间发生了显著变化。SRM结果与一些研究者们的下游试验分析结果一致,如遗传互作、多聚核糖体分析以及荧光素酶检测。以此说明以SRM为基础的miRNA靶基因鉴定是高效的[4]。
3 miRNA在反刍动物中的研究进展 3.1 牛在反刍动物中对牛的miRNA研究较多,已有766种前体miRNA加工成的755个成熟的miRNA通过多种标记技术被鉴定出来[5]。有研究者已从牛胚胎、胸腺、小肠、淋巴结等器官鉴定了129种牛的miRNA。从牛脂肪组织和乳腺组织中发现了59种miRNA,从11种不同品种牛体组织中发现了101种牛的miRNA。也有科研人员采用计算机软件预测方法从牛的基因组中预测出496种miRNA靶基因。近些年,采用深度测序技术检测牛视网膜微血管表皮细胞中的miRNA,鉴定出255种已知的牛miRNA。更有多种新的miRNA成员相继被发现,如miR-2284和-2285家族新成员,此家族成员在血管发生中起到重要作用[6]。
牛miRNA的功能研究主要集中在脂肪、骨骼肌、卵母细胞以及早期胚胎发育方面, 如表 1所示。Jin等[14]比较研究了背部脂肪不同厚度的3种杂交品种脂肪组织中89种牛miRNA的表达情况,发现了42种miRNA的差异表达,miR-378表达量差异最为显著。miR-378在大鼠脂肪形成中起到重要的作用[15]。因此,miR-378可能参与了牛皮下脂肪的生长。在牛肌肉前体脂肪细胞分化为成熟的脂肪细胞过程中miR-143表达上调。miR-143互补序列的转染抑制了牛肌内脂肪前体细胞的分化[16]。这些研究说明miR-143在牛肌肉脂肪发育过程中具有明显的作用。Romao等[17]发现8种miRNA在脂肪组织中的表达,包括miR-19a、-92a、-92b、-101、-103、-106、-142-5p和-296,这些miRNA与高脂饮食具有相关性。因此,这些miRNA可能参与牛高脂饮食诱导的脂肪生成的基因调控网络。
|
|
表 1 反刍动物中miRNA所行使的不同功能 Table 1 Different functions of miRNAs in ruminants |
miR-1和miR-206作为2种肌肉特异性表达的miRNA在肌肉细胞分化中起重要的作用[18]。研究者发现在皮埃蒙特牛和黑白花奶牛骨骼肌细胞中miR-1和miR-206表达并因为肌肉抑制素基因的点突变产生了骨骼肌双肌性状。miR-1的表达量在2个品种或性别中无显著差别,而miR-206在皮埃蒙特母牛中的表达量显著高于黑白花奶牛[19],说明miR-206表达上调可能对皮埃蒙特母牛肌肉肥大有影响。急性应激(瘤胃瘘管安装)后的安格斯牛中,miR-181b在肌肉中表达上调,意味着其在肌细胞生成中可能具有重要的作用[20]。郭云涛等[21]采用RNA-Seq法研究推断bta-miR-320a易受到SP1等多种转录因子调控, 其可能通过靶基因TP53、丝裂原活化蛋白激酶1(mitogen-activated protein kinase 1,MAPK1)等的抑制作用调控牛脂肪细胞分化,从而对牛的脂肪沉积产生影响。孙加节[22]发现miR-10020能通过抑制Pax7基因表达,延迟早期肌细胞分化过程或抑制肌卫星细胞分化。
卵母细胞中富集有6种miRNA,包括miR-205、-150、-122、-96、-146a和-146b-5p,研究发现在卵母细胞成熟的0~22 h内表达量显著下降[23]。可能说明这些miRNA在牛卵母细胞成熟过程中起到相应的作用。miR-106a在牛卵母细胞中的表达量高于卵丘-卵母细胞复合体(COCs)中的表达量,而miR-106a靶基因WEE1同源基因WEE1A的表达量却低于COCs中的表达量[24]。这些表达量的差异说明miR-106a通过下调WEE1A基因在牛卵母细胞发育过程中起到关键的作用。同样miR-196a和miR-181a通过靶向调节NOBOX和NPM2基因调节牛卵母细胞发育,而这2种基因在卵泡生成和核与核仁组织生成中具有重要的作用[9-10]。牛胚胎从1细胞到8细胞阶段miR-21和miR-130a的表达量增加,这2种miRNA可以对牛早期胚胎发育产生影响[25]。靳丽军[26]发现miRNA及信号通路的变化与转基因克隆牛胎盘的异常发育有关。
3.2 羊miR-431、-433、-127、-434、-432和-136是首次在双肌臀羊中鉴定出来的几种miRNA,揭示其在肌肉肥大表型羊中的作用。采用深度测序的方法在双肌臀羊骨骼肌细胞中有747个miRNA、472个miRNA前体被鉴定出来[27]。Sheng等[28]采用小RNA克隆的方法鉴定出31种羊的miRNA。特赛尔羊MSTN基因3′端非翻译区(3′-UTR)存在1个G到A的突变,进一步研究发现这个突变成为了miR-1和miR-206的靶位点,因此抑制MSTN蛋白的表达引起特赛尔羊肌肉肥大。王霄鹏等[29]采用茎环-qRT-PCR法检测了羊背最长肌3种miRNA的表达,发现6月龄雄性湖羊背最长肌中oar-miR-299-5p表达量极显著高于6月龄雄性杜泊羊×湖羊杂交羊, 说明oar-miR-299-5p可能与湖羊早期背最长肌的生长发育相关。宋广超等[30]检测了绵羊miR-133前体序列的单核苷酸多态性(SNP)位点,说明其可能对miR-133前体的加工成熟具有重要影响,并影响绵羊的产肉性能。
采用miRNA芯片的方法在羊体侧皮肤和耳皮肤中可以检测出159种miRNA的表达,并发现在体侧皮肤中有19种miRNA特异性表达或者显著富集。这个结论进一步证实了这些miRNA在羊毛生长过程中的作用。孙武[31]对不同肤色山羊胎儿皮肤进行鉴定发现miR-381、-543、-129和-544可能参与了黑色素生成过程。miR-let7a在绒山羊毛囊发育生长期与退行期中均有表达,是调控绒山羊毛囊发育的重要因子,以靶基因Cmyc和FGF5在毛囊发育中行驶功能[32]。袁超[33]发现miR-125b通过抑制FGF5基因的表达对毛囊周期性变化相关基因具有一定调控作用,同时miR-125b过表达影响毛囊周期性变化相关基因mRNA的表达水平。杨姗姗等[34]发现lpa-miR-nov-66通过与靶基因sGC结合, 通过参与环单磷酸腺苷(cAMP)路径, 也可以调控黑色素细胞内黑色素的合成。同样,姬凯元等[35]的研究报道进一步印证了上述结论。
最近,McBride等[36]通过小RNA库测序的方法在羊不同生殖阶段的卵巢卵泡和黄体中鉴定出212种羊miRNA。Torley等[11]通过qRT-PCR方法比较了羊妊娠期42和75 d卵巢和睾丸中128种miRNA的表达,发现在妊娠42 d时24种miRNA在卵巢和睾丸中差异表达,而妊娠75 d时43种miRNA差异表达。采用原位杂交的方法则发现miR-22定位于睾丸支持细胞,说明miR-22在胎儿睾丸发育过程中通过抑制雌激素信号通路而产生作用。胎儿期睾酮处理引起卵巢发生病理学变化。睾酮处理的妊娠母羊的胎儿卵巢中,miR-497和miR-15b表达上调,说明这2种miRNA在睾酮诱导的卵巢病理学变化中可能产生作用[37]。
4 小结miRNA在机体生物学过程中具有重要的调节作用。目前已有数百种miRNA在各种动物中被陆续发现。这些miRNA都是以一种时空特异性的形式表达存在,说明miRNA在特异组织类型或特异发育阶段具有重要的作用。虽然反刍动物miRNA的功能研究存在许多限制因素,但是miRNA靶位点的寻找仍然是其研究的一个突破口,mRNA-miRNA之间的相互作用将是研究机体生理学过程的必经之路。近期全基因组关联研究指出复杂性状通常与非编码的SNP相关。miRNA在基因表达中的作用也可能是因为与这些非编码SNP之间的关联效应产生的,也就是说相关SNP是部分miRNA的结合位点或者miRNA序列。因此miRNA的进一步研究可以从鉴定SNP与miRNA结合位点或miRNA序列的重叠与反刍动物的生长或繁殖性状的关系入手研究。这样的研究将会为反刍动物的优良繁殖性能或生长性状的选择提供遗传学依据。
| [1] |
WINTER J, JUNG S, KELLER S, et al. Many roads to maturity:microRNA biogenesis pathways and their regulation[J].
Nature Cell Biology, 2009, 11(3): 228–234.
( 0) 0)
|
| [2] |
LIU H, YUE D, CHEN Y D, et al. Improving performance of mammalian microRNA target prediction[J].
BMC Bioinformatics, 2010, 11(1): 476.
( 0) 0)
|
| [3] |
EBERT M S, SHARP P A. MicroRNA sponges:progress and possibilities[J].
RNA, 2010, 16(11): 2043–2050.
( 0) 0)
|
| [4] |
JOVANOVIC M, REITER L, PICOTTI P, et al. A quantitative targeted proteomics approach to validate predicted microRNA targets in C.Elegans[J].
Nature Methods, 2010, 7(10): 837–842.
( 0) 0)
|
| [5] |
KOZOMARA A, GRIFFITHS-JONES S. miRBase:integrating microRNA annotation and deep-sequencing data[J].
Nucleic Acids Research, 2011, 39(Suppl. 1): D152–D157.
( 0) 0)
|
| [6] |
GUDURIC-FUCHS J, O'CONNOR A, CULLEN A, et al. Deep sequencing reveals predominant expression of miR-21 amongst the small non-coding RNAs in retinal micro vascular endothelial cells[J].
Journal of Cellular Biochemistry, 2012, 113(6): 2098–2111.
( 0) 0)
|
| [7] |
LI G X, LI Y J, LI X J, et al. MicroRNA identity and abundance in developing swine adipose tissue as determined by Solexa sequencing[J].
Journal of Cellular Biochemistry, 2011, 112(5): 1318–1328.
( 0) 0)
|
| [8] |
MA T H, JIANGI, GAO Y, et al. Microarray analysis of differentially expressed microRNAsin non-regressed and regressed bovine corpus luteum tissuemicroRNA-378 may suppress luteal cell apoptosisby targeting the interferon gamma receptor 1 gene[J].
Journal of Applied Genetics, 2011, 52: 481–486.
( 0) 0)
|
| [9] |
TRIPURANI S K, LEE K B, WEE G, et al. MicroRNA-196a regulates bovine newborn ovary homeobox gene (NOBOX) expression during early embryogenesis[J].
BMC Developmental Biology, 2011, 11(1): 25.
( 0) 0)
|
| [10] |
LINGENFELTER B M, TRIPURANI S K, TEJOMURTULA J, et al. Molecular cloning and expression of bovine nucleoplasmin 2 (NPM2):a maternal effect gene regulated by miR-181a[J].
Reproductive Biology and Endocrinology, 2011, 9(1): 40.
( 0) 0)
|
| [11] |
TORLEY K J, DA SILVEIRA J C, SMITH P, et al. Expression of miRNAs in ovine fetal gonads:potential role in gonadal differentiation[J].
Reproductive Biology and Endocrinology, 2011, 9(1): 2.
( 0) 0)
|
| [12] |
CLOP A, MARCQ F, TAKEDA H, et al. A mutation creating a potential illegitimate microRNA target site in the myostatin gene affects muscularity[J].
Nature Genetics, 2006, 38: 813–818.
( 0) 0)
|
| [13] |
TAKEDA H, SHIMADA A. The art of medaka genetics and genomics; what makes them so unique[J].
Annual Review of Genetics, 2010, 44: 217–241.
( 0) 0)
|
| [14] |
JIN W W, DODSON M V, MOORE S S, et al. Characterization of microRNA expression in bovine adipose tissues:a potential regulatory mechanism of subcutaneous adipose tissue development[J].
BMC Molecular Biology, 2010, 11(1): 29.
( 0) 0)
|
| [15] |
GERIN I, BOMMER G T, MCCOIN C S, et al. Roles for miRNA-378/378* in adipocyte gene expression and lipogenesis[J].
American Journal of Physiology Endocrinology and Metabolism, 2010, 299(2): E198–E206.
( 0) 0)
|
| [16] |
LI H X, ZHANG Z, ZHOU X, et al. Effects of microRNA-143 in the differentiation and proliferation of bovine intramuscular preadipocytes[J].
Molecular Biology Reports, 2011, 38(7): 4273–4280.
( 0) 0)
|
| [17] |
ROMAO J M, JIN W W, HE M L, et al. Altered microRNA expression in bovine subcutaneous and visceral adipose tissues from cattle under different diet[J].
PLoS One, 2012, 7(7): e40605.
( 0) 0)
|
| [18] |
TOWNLEY-TILSON W H D, CALLIS T E, WANG D Z. MicroRNAs 1, 133, and 206:critical factors of skeletal and cardiac muscle development, function, and disease[J].
The International Journal of Biochemistry & Cell Biology, 2010, 42(8): 1252–1255.
( 0) 0)
|
| [19] |
MIRETTI S, MARTIGNANI E, TAULLI R, et al. Differential expression of microRNA-206 in skeletal muscle of female Piedmontese and Friesian cattle[J].
The Veterinary Journal, 2011, 190(3): 412–413.
( 0) 0)
|
| [20] |
ZHAO C P, TIAN F, YU Y, et al. miRNA-dysregulation associated with tenderness variation induced by acute stress in Angus cattle[J].
Journal of Animal Science and Biotechnology, 2012, 3(1): 12.
( 0) 0)
|
| [21] |
郭云涛, 张秀秀, 苗向阳. 牛miR-320a的靶基因预测及生物信息学分析[J].
畜牧兽医学报, 2015, 46(11): 1952–1960.
( 0) 0)
|
| [22] |
孙加节.秦川牛肌肉与脂肪组织发育相关miRNA鉴定及miR-10020调控机制解析[D].博士学位论文.杨凌:西北农林科技大学, 2015.
( 0) 0)
|
| [23] |
ABD EL NABY W S, HAGOS T H, HOSSAIN M M, et al. Expression analysis of regulatory microRNAs in bovine cumulus oocyte complex and preimplantation embryos[J].
Zygote, 2013, 21(1): 31–51.
( 0) 0)
|
| [24] |
MILES J R, MCDANELD T G, WIEDMANN R T, et al. MicroRNA expression profile in bovine cumulus-oocyte complexes:possible role of let-7 and miR-106a in the development of bovine oocytes[J].
Animal Reproduction Science, 2012, 130(1/2): 16–26.
( 0) 0)
|
| [25] |
MONDOU E, DUFORT I, GOHIN M, et al. Analysis of microRNAs and their precursors in bovine early embryonic development[J].
Molecular Human Reproduction, 2012, 18(9): 425–434.
( 0) 0)
|
| [26] |
靳丽军.转基因克隆牛胎盘中的差异miRNAs和bta-miR-708慢病毒表达载体的构建[D].硕士学位论文.洛阳:河南科技大学, 2014.
( 0) 0)
|
| [27] |
CAIMENT F, CHARLIER C, HADFIELD T, et al. Assessing the effect of the CLPG mutation on the microRNA catalog of skeletal muscle using high-throughput sequencing[J].
Genome Research, 2010, 20(12): 1651–1662.
( 0) 0)
|
| [28] |
SHENG X H, SONG X M, YU Y, et al. Characterization of microRNAs from sheep (Ovis aries)using computational and experimental analyses[J].
Molecular Biology Reports, 2011, 38(5): 3161–3171.
( 0) 0)
|
| [29] |
王霄鹏, 宋雪梅, 姜俊芳, 等. 茎-环RT-qPCR法定量检测羊背最长肌3种miRNA的表达[J].
畜牧与兽医, 2015, 47(12): 31–35.
( 0) 0)
|
| [30] |
宋广超, 张伟, 许瑞霞, 等. 绵羊骨骼肌特异表达miR-133前体在不同绵羊群体中的多态检测[J].
家畜生态学报, 2015, 36(1): 18–23.
( 0) 0)
|
| [31] |
孙武.山羊胎儿皮肤黑色素生成有关miRNA的筛选与鉴定[D].硕士学位论文.重庆:西南大学, 2015.
( 0) 0)
|
| [32] |
江倩, 李建平, 曲海娥, 等. miR-let7a及其靶基因IGF-1R在绒山羊毛囊发育周期中的表达[J].
中国兽医学报, 2014, 34(10): 1622–1626.
( 0) 0)
|
| [33] |
袁超.绒山羊次级毛囊周期性变化相关microRNA的鉴定及miR-125b在毛乳头细胞中的功能研究[D].博士学位论文.杨凌:西北农林科技大学, 2014.
( 0) 0)
|
| [34] |
杨姗姗, 白俊明, 范瑞文, 等. 内皮素3在不同毛色绵羊皮肤中的差异表达[J].
畜牧兽医学报, 2015, 46(11): 2111–2116.
( 0) 0)
|
| [35] |
姬凯元, 石占全, 刘彧, 等. lpa-miR-nov-66通过调控cAMP路径抑制α-MSH介导的羊驼黑色素生成[J].
中国生物化学与分子生物学报, 2015, 31(11): 1206–1212.
( 0) 0)
|
| [36] |
MCBRIDE D, CARRÉ W, SONTAKKE S D, et al. Identification of miRNAs associated with the follicular-luteal transition in the ruminant ovary[J].
Reproduction, 2012, 144(2): 221–233.
( 0) 0)
|
| [37] |
LUENSE L J, VEIGA-LOPEZ A, PADMANABHAN V, et al. Developmental programming:gestational testosterone treatment alters fetal ovarian gene expression[J].
Endocrinology, 2011, 152(12): 4974–4983.
( 0) 0)
|




