鸡的肠道是一个巨大的生态系统,肠道微生物对鸡的营养、免疫等功能有着重要的影响。鸡肠道的不同部位具有不同的作用,其中回肠是营养物质吸收的关键部位,且微生物多样性较高被人们广泛关注[1]。鸡肠道微生物多样性的研究方法有很多,早期主要采用传统培养的方法[2],但肠道中厌氧细菌居多,因此大量微生物未被分离培养。随着分子指纹图谱方法的出现,如末端限制性片段长度多态性(terminal-restriction fragment length polymorphism, T-RFLP)分析[3]、变性梯度凝胶电泳[4](denaturing gradient gel electrophoresis, DGGE)和温度梯度凝胶电泳[5](temperature gradient gel electrophoresis, TTGE),使一些厌氧细菌能够在RNA水平上被检测,但研究技术的局限使获 得的信息量少且不够深入。近年来,高通量测序技术因其准确性高、快速和信息量大等优点,被广泛的应用于人[6]、林麝[7]、兔子[8]等的微生物研究中。但是,利用高通量测序技术对健康鸡回肠微生物进行研究,在国内鲜有报道。本试验通过Illumina MiSeq平台进行Paired-end测序,快速、全面地测定健康鸡回肠微生物并通过KEGG(kyoto encyclopedia of genes and genomes)数据库进行PICRUSt(phylogenetic investigation of communities by reconstruction of unobserved states)基因预测,揭示肠道菌群在回肠中的作用,为进一步研究鸡回肠微生物的结构与其在肠道中的功能提供参考依据。
1 材料与方法 1.1 试验动物与饲养管理选取15只同日孵化的1日龄健康科宝500肉鸡(购于成都温江正大有限公司),饲养21 d。饲养条件为24 h光照,自由采食和饮水,试验饲粮由玉米(51.64%)、豆粕(39.60%)、菜籽油(4.30%)、磷酸氢钙(1.85%)、碳酸钙(1.30%)、DL-蛋氨酸(0.20%)、食盐(0.40%)、胆碱(0.18%)、多维(0.03%)和微量元素预混料(0.50%)组成,每天观察鸡群的采食、饮水与健康情况。
1.2 样品采集试验第22天,将15只鸡处死,取鸡回肠内容物,分别编号为H1~H15,迅速放置液氮中保存,然后放入-80 ℃冰箱中冻存。
1.3 细菌总DNA的提取取鸡回肠内容物样品(100±10) mg,用OmegaD40-15粪样试剂盒,按操作手册要求提取总DNA,存放于-30 ℃冰箱中备用。
1.4 基于16S rRNA的V4区细菌样品检测、文库建立及上机测序DNA样品送至中国科学院成都生物研究所检验、测序,利用Illumina MiSeq平台进行Paired-end测序,对样品16S rRNA的V4区建立文库并上机测序。具体操作如下:选择16S rDNA的V4区作为目的扩增区域,用引物对515F/806R目的片段进行扩增。根据PCR产物浓度进行等量混样,充分混匀后使用2%的琼脂糖凝胶电泳检测PCR产物。对目的条带使用E.Z.N.A.TM公司的Gel Extraction Kit凝胶回收试剂盒切胶回收。用纯化后的样品基于Illumina MiSeq平台Paired-end测序技术建立文库,并进行测序。
1.5 数据分析测序数据利用FastQC软件评价测序的数量和质量,通过perl脚本去除连续低于20的碱基以及顺序掉转的reads,在Mothur 1.31.2中将处理后的数据合并成一条一条完整的目的片段;利用Usearch 7.0.1001去掉纯化后数据的嵌合体[9],利用Qiime 1.8.0合并去掉嵌合体的数据,并将数据按照uclust算法将序列相似性≥97%的操作分类单元(operational taxonomic units,OTUs),进行OTUs聚类[10],同时去掉序列数少的OTUs(OTUs中序列数<2)。
通过Qiime 1.8.0对每个样品进行物种分类,并统计信息[11]。基于样品OTUs数量与测试的深度变化的关系,通过不同生物信息学算法计算alpha多样性(样品内部的生物多样性),并绘画稀释曲线。进行物种注释和核心菌群分析,统计各水平信息,按照样品规定最低比例与OTUs数目之间的关系制作核心菌群数量变化曲线图,通过软件制作门水平所有样品共同含有的核心菌门百分含量热图,在属水平制作所有样品共同含有的核心菌属柱状图。通过KEGG数据库进行PICRUSt基因预测,利用软件R 3.1.1根据数量排列前20的预测结果制作基因预测热图。通过SPSS 19.0进行标准误计算。
2 结果与分析 2.1 各样品序列及OTUs信息本研究基于Illumina高通量测序技术对15只健康鸡的回肠内容物菌群V4区进行测序,按照97%的相似度对OTUs进行归类,去除序列数量少的OTUs(OTUs中序列数<2)后,共获得424 062条tags,被归类于1 002个OTUs,样品平均tags为(28 271±8 855)条,平均OTUs为(261±82)个。其中,tags数量最高的是样品H7,最低的是H8,相差35 100条tags。OTUs含量最高的是H1,最低的是H8,相差328个OTUs。
2.2 Alpha多样性分析采用alpha多样性指数对各样品的丰富度和多样性进行比较。从图 1可以看出,物种观察指数稀释曲线随着测试深度的不断加深逐渐增加,但没有达到饱和,说明虽然大多数菌群被覆盖,但仍有少量细菌未被测出。Alpha多样性指数分析结果(表 1)表明,样品H1的物种丰富度和多样性相对较高。
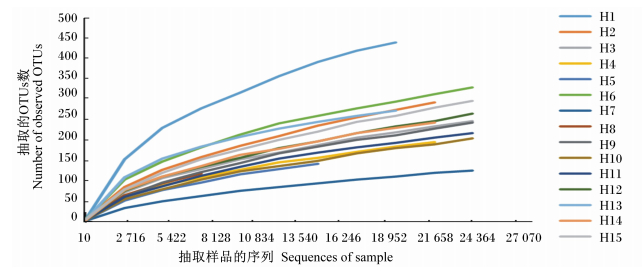
|
图 1 观察指数稀释曲线 Figure 1 Rarefaction curves of observed species |
|
|
表 1 Alpha多样指数 Table 1 Alpha diversity indices |
在门水平和属水平对样品进行物种注释分析。15个样品共获得20个菌门,160个菌属。样品中微生物主要为细菌,占所有微生物的99.54%,其余为古生菌和未被归类的微生物。
门水平含量大于0.1%有5个菌门,分别为:厚壁菌门(Firmicutes),平均含量为89.83%;变形菌门(Proteobacteria),平均含量为6.28%;蓝细菌门(Cyanobacteria),平均含量为2.16%;拟杆菌门(Bacteroidetes),平均含量为1.08%;放线菌门(Actinobacteria),平均含量为0.16%。
属水平含量大于1%的菌属共有8个,乳杆菌属(Lactobacillus)平均含量为76.77%,链球菌属(Streptococcus)平均含量为2.29%,乳酸乳球菌(Lactococcus)平均含量为2.14%,肠球菌属(Enterococcus)平均含量为1.36%,Solibacillus平均含量为1.07%。其余3种在属水平未被命名,分别归属于肠杆菌科(Enterobacteriaceae)(平均含量为5.22%)、消化链球菌科(Peptostreptococcaceae)(平均含量为2.83%)、Streptophyta(平均含量为2.15%)。
2.4 核心菌群分析核心菌群是基于OTUs通过Qiime 1.8.0软件筛选、统计,获得存在于样品规定最低百分含量中所含的OTUs,然后按照各分类水品进行分类。表 2为样品规定最低比例与OTUs数目之间的关系表。从表中可以看出,随着样品规定最低比例的增加,共同OTUs的数目呈阶梯形减少。当样品规定最低百分含量为50%时,共含有146个OTUs。规定最低百分含量为100%时,OTUs为30个。
|
|
表 2 最低百分含量与OTUs数目之间的关系 Table 2 Relationship between the minimum percentage and the number of OTUs |
在门水平上,共享核心菌门共有4个,按含量从高到底分别为:厚壁菌门(Firmicutes),平均含量为85.35%;变形菌门(Proteobacteria),平均含量为6.09%;蓝细菌门(Cyanobacteria),平均含量为2.15%;放线菌门(Actinobacteria),平均含量为0.16%。通过表 3可以看出,除样品H13中的厚壁菌门、变形菌门和样品H11中厚壁菌门外,样品在门水平的差异性较小。
|
|
表 3 共享核心菌门百分含量 Table 3 The percentage of shared core microbiome phylum |
在属水平上,共享核心菌属共有19个,分别为乳杆菌属(Lactobacillus)平均含量为76.77%,链球菌属(Streptococcus)平均含量为2.29%,乳酸乳球菌(Lactococcus)平均含量为2.14%,肠球菌属(Enterococcus)平均含量为1.36%,Solibacillus属平均含量为1.07%,芽孢杆菌属(Bacillus)平均含量为0.96%,假单孢菌属(Pseudomonas)平均含量为0.33%,杆菌属(Lysinibacillus)平均含量为 0.10%,Ewingella属平均含量为0.05%,Brochothrix 属平均含量为0.04%,明串珠菌属(Leuconostoc)平均含量为0.04%,Enhydrobacter属平均含量为0.03%,其余7种细菌在属水平未被分类,其总共占样品微生物的含量为8.57%。共享核心菌属占样品总微生物的93.75%。共享核心菌属物种注释柱状图(图 2)表明,虽然这些菌属是各样品共同含有,但其百分含量在各样品中差异较大,其中样品H13与其他样品差异最明显。
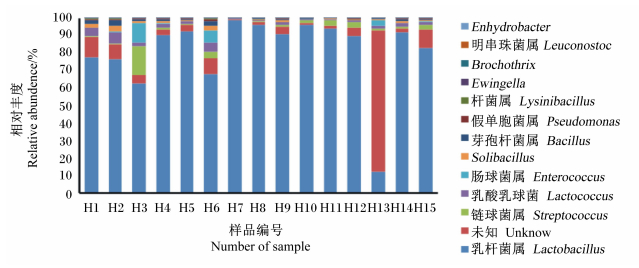
|
图 2 核心菌属柱状图 Figure 2 Core microbiome genus histogram |
PICRUSt是通过微生物群落的丰富度与数据库比,从而在不可观测的情况下推测出生物群落的功能信息。结果获得328个预测的功能,数量前20的功能分别为转运因子、一般功能预测、ABC转运体、DNA修复和重组蛋白、核糖体、嘌呤代谢、蛋白酶、嘧啶代谢、转录因子、染色体、氨基糖和核苷酸糖代谢、未知功能、双组分系统、核糖体生物合成、糖酵解/糖异生、其他离子偶联转运体、氨酰tRNA生物合成、磷酸转移酶系统、DNA复制蛋白和分泌系统。PICRUSt基因预测热图(图 3)结果表明,各样品的效能存在一定差异。
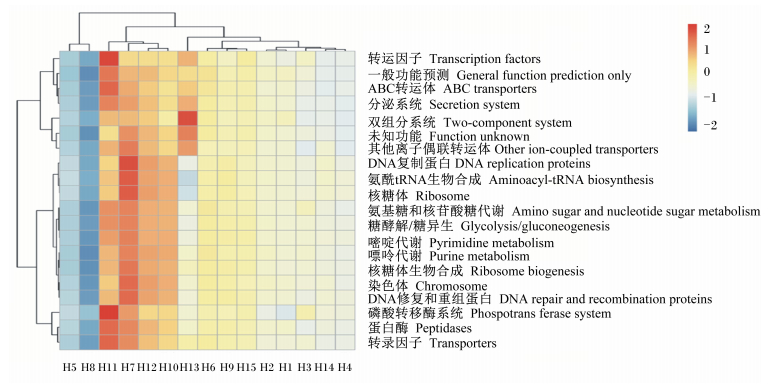
|
图 3 PICRUSt基因预测聚类热图 Figure 3 Heatmap of PICRUSt gene predicted function |
本试验通过16S rRNA V4区基因扩增子技术,在Illumina Miseq测序平台上对健康鸡的回肠菌群进行高通量测序。与以往的肠道微生物检验方法相比,高通量测序具有检测数据完整性好、测量快速、测定信息量大等优点[12-13]。
15只健康鸡回肠微生物的种类和数量不完全相同,可能由个体身体状况、饮食量等试验样品本身差异造成。Alpha稀释曲线是否达到平台期,可以反映测试样品是否达到了测试深度,即样品测试是否足以覆盖所有微生物,通过alpha稀释曲线可以看出高通量测序并没有完全覆盖所有菌群,测试结果具有一定的随机性。研究表明,肠道微生物的种类和数量与刚出生的环境有关,出生环境也可能是造成差异的原因[14]。即使严格控制饲养环境做重复试验,鸡的肠道微生物测试结果仍然会出现差异[15]。本次试验重复数多达15个,且检测结果覆盖了大多数的菌种,所以本次试验具有较好的代表性。
共享核心菌群与物种注释相比,其归类依据在物种分类水平上更高,所以在统计样品微生物百分含量时更加准确,代表性更强。通过物种注释可以看出,含量排在前五的门分别是厚壁菌门、变形菌门、蓝细菌门、拟杆菌门和放线菌门,与Shaufi等[13]人的研究结果相似。共享核心菌群比较,除蓝细菌门外,其他菌门都是共享核心菌门。从菌门百分含量可以看出,共享核心菌门中菌门百分含量小于物种注释中的百分含量。以厚壁菌门为例,厚壁菌门是健康鸡的回肠中含量最多的菌群。厚壁菌门在肠道或粪便中作为主要菌门在猫[16]、狗[17]、马[18]等动物中也有报道。据报道,厚壁菌门与粗饲料的消化有关,鸡作为以植物为主要食物禽类,厚壁菌门对其营养消化吸收有帮助作用。物种注释中显示厚壁菌门含量高达89.83%,核心菌群中显示其含量为85.35%,两者相差4.48%。造成差异的原因是物种注释中归于厚壁菌门的OTUs并非所有样品都含有。由此可见,利用共享核心菌群分析百分含量其更准确,但受个体差异影响更大。通过共享核心菌门百分含量表可知,不同个体门水平微生物百分比差异较小。在属水平上,物种注释和共享核心菌属在含量高的菌属中差异不大,因为两者归类参照的物种分类水平更相似。但定量上看,物种注释在属水上显示共含有160个属,而共享核心菌属仅有19个,造成差异的原因是样品个体差异影响。但共同含有的核心菌属在样品微生物总量的93.75%,已经表现了样品的大多数微生物。本次试验测得到的共享核心菌种与周小娟等[19]研究北京油鸡和艾拔益加肉鸡回肠优势菌种的结果存在差异,大肠埃希菌和粪链球菌并不是优势菌种。造成差异的原因除饲养环境、肉鸡品种和研究方法不同外,也可能是饲粮配方不同造成的差异。研究表明,饲粮的种类不仅对鸡的健康和代谢有影响[20],而且可以改变鸡的肠道微生物[21-22]。从核心菌属柱状图可以看出,样品之间菌属含量差异较大,此结果与Buffie等[23]、Gulino等[24]研究动物肠道菌群的结果相似。
肠道中微生物的对免疫功能、营养物质的吸收、甚至酶的代谢作用等有一定的影响[25]。研究表明,人类肠道中微生物异常可以导致过敏、肥胖和糖尿病等[25]。由此可见,肠道微生物对机体的生理功能有一定的影响。PICRUSt将不可视的微生物,通过与数据库比对和生物功能联系起来。PICRUSt基因预测表明,鸡回肠微生物与分子和蛋白质的生成和代谢等基础生命功能有关。PICRUSt基因预测热图显示,相同功能各样品间热图颜色有变化,说明各样品虽然都具有相同的功能,但是由于每个样品中所含微生物的种类和数量有区别,所以各样品的效能存在差异。这与测定样品tags和OTUs的结果吻合。
4 结 论① 15只健康鸡回肠共获得424 062条tags和1 002个非单OTUs,样品平均tags为(28 271±8 855)条,平均OTUs为(261±82)个。
② 通过物种注释和核心菌群比较可知,两者在种类和含量上存在差异。
③ 样品个体间菌群百分含量在门水平差异不大,但在属水平存在一定差异。
④ 各样品的效能差异,可能与样品菌群含量和种类有关。
| [1] |
倪学勤, GONGJ, YUH, 等. 采用PCR-DGGE技术分析蛋鸡肠道细菌种群结构及多样性[J].
倪学勤, GONGJ, YUH, 等. 采用PCR-DGGE技术分析蛋鸡肠道细菌种群结构及多样性[J]. 畜牧兽医学报, 2008 , 39 (7) :955 –961.
( 0) 0)
|
| [2] |
BARNES E M, MEAD G C, BARNUM D A, et al. The intestinal flora of the chicken in the period 2 to 6 weeks of age, with particular reference to the anaerobic bacteria[J].
BARNES E M, MEAD G C, BARNUM D A, et al. The intestinal flora of the chicken in the period 2 to 6 weeks of age, with particular reference to the anaerobic bacteria[J]. British Poultry Science, 1972 , 13 (3) : 311 –326.
DOI: 10.1080/00071667208415953 ( 0) 0)
|
| [3] |
GONG J H, FORSTER R J, YU H, et al. Molecular analysis of bacterial populations in the ileum of broiler chickens and comparison with bacteria in the cecum[J].
GONG J H, FORSTER R J, YU H, et al. Molecular analysis of bacterial populations in the ileum of broiler chickens and comparison with bacteria in the cecum[J]. FEMS Microbiology Ecology, 2002 , 41 (3) : 171 –179.
DOI: 10.1111/fem.2002.41.issue-3 ( 0) 0)
|
| [4] |
GONG J, YU H, LIU T, et al. Effects of zinc bacitracin, bird age and access to range on bacterial microbiota in the ileum and caeca of broiler chickens[J].
GONG J, YU H, LIU T, et al. Effects of zinc bacitracin, bird age and access to range on bacterial microbiota in the ileum and caeca of broiler chickens[J]. Journal of Applied Microbiology, 2008 , 104 (5) : 1372 –1382.
DOI: 10.1111/j.1365-2672.2007.03699.x ( 0) 0)
|
| [5] |
ZHU X Y, ZHONG T Y, PANDYA Y, et al. 16S rRNA-based analysis of microbiota from the cecum of broiler chickens[J].
ZHU X Y, ZHONG T Y, PANDYA Y, et al. 16S rRNA-based analysis of microbiota from the cecum of broiler chickens[J]. Applied and Environmental Microbiology, 2002 , 68 (1) : 124 –137.
DOI: 10.1128/AEM.68.1.124-137.2002 ( 0) 0)
|
| [6] |
QIN N, YANG F L, LI A, et al. Alterations of the human gut microbiome in liver cirrhosis[J].
QIN N, YANG F L, LI A, et al. Alterations of the human gut microbiome in liver cirrhosis[J]. Nature, 2014 , 513 (7516) : 59 –64.
DOI: 10.1038/nature13568 ( 0) 0)
|
| [7] |
王立志, 徐谊英, 蔡永华. 高通量测序分析人工养殖成年林麝粪便古菌菌群多样性[J].
王立志, 徐谊英, 蔡永华. 高通量测序分析人工养殖成年林麝粪便古菌菌群多样性[J]. 动物营养学报, 2016 , 28 (2) :477 –484.
( 0) 0)
|
| [8] |
ZENG B, HAN S S, WANG P, et al. The bacterial communities associated with fecal types and body weight of rex rabbits[J].
ZENG B, HAN S S, WANG P, et al. The bacterial communities associated with fecal types and body weight of rex rabbits[J]. Scientific Reports, 2015 , 5 : 9342 .
DOI: 10.1038/srep09342 ( 0) 0)
|
| [9] |
EDGAR R C, HAAS B J, CLEMENTE J C, et al. UCHⅡME improves sensitivity and speed of chimera detection[J].
EDGAR R C, HAAS B J, CLEMENTE J C, et al. UCHⅡME improves sensitivity and speed of chimera detection[J]. Bioinformatics, 2011 , 27 (16) : 2194 –2200.
DOI: 10.1093/bioinformatics/btr381 ( 0) 0)
|
| [10] |
CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al. QⅡME allows analysis of high-throughput community sequencing data[J].
CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al. QⅡME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010 , 7 (5) : 335 –336.
DOI: 10.1038/nmeth.f.303 ( 0) 0)
|
| [11] |
WERNER J J, KOREN O, HUGENHOLTZ P, et al. Impact of training sets on classification of high-throughput bacterial 16S rRNA gene surveys[J].
WERNER J J, KOREN O, HUGENHOLTZ P, et al. Impact of training sets on classification of high-throughput bacterial 16S rRNA gene surveys[J]. International Society for Microbial Ecology Journal, 2012 , 6 (1) : 94 –103.
( 0) 0)
|
| [12] |
WILLIAMS S T, FOSTER P G, LITTLEWOOD D T J. The complete mitochondrial genome of a turbinid vetigastropod from MiSeq Illumina sequencing of genomic DNA and steps towards a resolved gastropod phylogeny[J].
WILLIAMS S T, FOSTER P G, LITTLEWOOD D T J. The complete mitochondrial genome of a turbinid vetigastropod from MiSeq Illumina sequencing of genomic DNA and steps towards a resolved gastropod phylogeny[J]. Gene, 2013 , 533 (1) : 38 –47.
( 0) 0)
|
| [13] |
SHAUFI M A M, SIEO C C, CHONG C W, et al. Deciphering chicken gut microbial dynamics based on high-throughput 16S rRNA metagenomics analyses[J].
SHAUFI M A M, SIEO C C, CHONG C W, et al. Deciphering chicken gut microbial dynamics based on high-throughput 16S rRNA metagenomics analyses[J]. Gut Pathogens, 2015 , 7 (1) : 1 –12.
DOI: 10.1186/s13099-015-0049-1 ( 0) 0)
|
| [14] |
APAJALAHTI J, KETTUNEN A, GRAHAM H. Characteristics of the gastrointestinal microbial communities, with special reference to chickens[J].
APAJALAHTI J, KETTUNEN A, GRAHAM H. Characteristics of the gastrointestinal microbial communities, with special reference to chickens[J]. Worlds Poultry Science Journal, 2004 , 60 (2) : 223 –232.
DOI: 10.1079/WPS20040017 ( 0) 0)
|
| [15] |
STANLEY D, GEIER M S, HUGHES R J, et al. Highly variable microbiota development in the chicken gastrointestinal tract[J].
STANLEY D, GEIER M S, HUGHES R J, et al. Highly variable microbiota development in the chicken gastrointestinal tract[J]. PLoS One, 2013 , 8 (12) : e84290 .
DOI: 10.1371/journal.pone.0084290 ( 0) 0)
|
| [16] |
RITCHIE L E, STEINER J M, SUCHODOLSKI J S. Assessment of microbial diversity along the feline intestinal tract using 16S rRNA gene analysis[J].
RITCHIE L E, STEINER J M, SUCHODOLSKI J S. Assessment of microbial diversity along the feline intestinal tract using 16S rRNA gene analysis[J]. FEMS Microbiology Ecology, 2008 , 66 (3) : 590 –598.
DOI: 10.1111/fem.2008.66.issue-3 ( 0) 0)
|
| [17] |
SUCHODOLSKI J S, CAMACHO J, STEINER J M. Analysis of bacterial diversity in the canine duodenum, jejunum, ileum, and colon by comparative 16S rRNA gene analysis[J].
SUCHODOLSKI J S, CAMACHO J, STEINER J M. Analysis of bacterial diversity in the canine duodenum, jejunum, ileum, and colon by comparative 16S rRNA gene analysis[J]. FEMS Microbiology Ecology, 2008 , 66 (3) : 567 –578.
( 0) 0)
|
| [18] |
COSTA M C, SILVA G, RAMOS R V, et al. Characterization and comparison of the bacterial microbiota in different gastrointestinal tract compartments in horses[J].
COSTA M C, SILVA G, RAMOS R V, et al. Characterization and comparison of the bacterial microbiota in different gastrointestinal tract compartments in horses[J]. The Veterinary Journal, 2015 , 205 (1) : 74 –80.
DOI: 10.1016/j.tvjl.2015.03.018 ( 0) 0)
|
| [19] |
周小娟, 张日俊, 郭宝林. 不同品种、日龄和饲养方式对肉鸡肠道菌群的影响[J].
周小娟, 张日俊, 郭宝林. 不同品种、日龄和饲养方式对肉鸡肠道菌群的影响[J]. 中国微生态学杂志, 2012 , 24 (7) :577 –587.
( 0) 0)
|
| [20] |
ZHOU M J, ZENG D, NI X Q, et al. Effects of Bacillus licheniformis on the growth performance and expression of lipid metabolism-related genes in broiler chickens challenged with Clostridium perfringens-induced necrotic enteritis[J].
ZHOU M J, ZENG D, NI X Q, et al. Effects of Bacillus licheniformis on the growth performance and expression of lipid metabolism-related genes in broiler chickens challenged with Clostridium perfringens-induced necrotic enteritis[J]. Lipids in Health and Disease, 2016 , 15 : 48 .
DOI: 10.1186/s12944-016-0219-2 ( 0) 0)
|
| [21] |
李可, 罗建杰, 孟昆, 等. 不同益生菌对肉鸡肠道菌群结构的影响[J].
李可, 罗建杰, 孟昆, 等. 不同益生菌对肉鸡肠道菌群结构的影响[J]. 动物营养学报, 2015 , 27 (11) :3516 –3526.
( 0) 0)
|
| [22] |
WITZIG M, CAMARINHA DA SILVA A, GREEN-ENGERT R, et al. Spatial variation of the gut microbiota in broiler chickens as affected by dietary available phosphorus and assessed by T-RFLP analysis and 454 pyrosequencing[J].
WITZIG M, CAMARINHA DA SILVA A, GREEN-ENGERT R, et al. Spatial variation of the gut microbiota in broiler chickens as affected by dietary available phosphorus and assessed by T-RFLP analysis and 454 pyrosequencing[J]. PLoS One, 2015 , 10 (12) : e0143442 .
( 0) 0)
|
| [23] |
BUFFIE C G, JARCHUM I, EQUINDA M. Profound alterations of intestinal microbiota following a single dose of clindamycin results in sustained susceptibility to Clostridium difficile-induced colitis[J].
BUFFIE C G, JARCHUM I, EQUINDA M. Profound alterations of intestinal microbiota following a single dose of clindamycin results in sustained susceptibility to Clostridium difficile-induced colitis[J]. Infection and Immunity, 2012 , 80 (1) : 62 –73.
DOI: 10.1128/IAI.05496-11 ( 0) 0)
|
| [24] |
GULINO L M, OUWERKERK D, KANG A Y H, et al. Shedding light on the microbial community of the macropod foregut using 454-amplicon pyrosequencing[J].
GULINO L M, OUWERKERK D, KANG A Y H, et al. Shedding light on the microbial community of the macropod foregut using 454-amplicon pyrosequencing[J]. PLoS One, 2013 , 8 (4) : e61463 .
DOI: 10.1371/journal.pone.0061463 ( 0) 0)
|
| [25] |
MARTIN R, NAUTA A J, BEN AMOR K, et al. Early life: gut microbiota and immune development in infancy[J].
MARTIN R, NAUTA A J, BEN AMOR K, et al. Early life: gut microbiota and immune development in infancy[J]. Beneficial Microbes, 2010 , 1 (4) : 367 –382.
DOI: 10.3920/BM2010.0027 ( 0) 0)
|




