甲烷是一种与全球气候变化密切相关的温室气体。据统计,畜牧业贡献了全球9%~18%的温室气体排放,其中反刍动物排放占有率为80%,同时反刍动物的甲烷形成过程也导致机体损失2%~12%的净能量[1-2]。因此,反刍动物的甲烷排放越来越受到人们的关注。对反刍动物而言,甲烷主要是由瘤胃甲烷菌利用饲料消化过程中所产生的中间代谢产物,如氢气、甲酸、甲醇及甲胺等,还原二氧化碳而生成的。因此,研究反刍动物瘤胃甲烷菌结构能够为甲烷调控提供依据。梅花鹿(Cervus nippon)是我国的一种珍稀名贵鹿科反刍动物,鹿茸是成年梅花鹿的主要产品,具有很高的药用经济价值。因此,研究梅花鹿瘤胃甲烷菌结构有助于了解甲烷菌在梅花鹿瘤胃生态中的作用。目前,国内外学者基于非培养技术已经研究了多种反刍动物瘤胃甲烷菌结构,结果表明宿主特异性可能是影响瘤胃甲烷菌结构的一个重要因素[3-4]。本实验室前期研究发现普雷沃氏菌属(Prevotella spp.)是梅花鹿瘤胃优势细菌[5],而瘤胃细菌是为甲烷菌生长提供底物的一类重要微生物。因此,梅花鹿瘤胃可能栖息着独特的甲烷菌群落结构。另外,饲粮组成也显著影响着瘤胃甲烷菌组成及丰度。玉米秸秆和玉米青贮是家养梅花鹿常用粗饲料,柞树叶是放牧条件下梅花鹿喜欢采食的一种富含单宁的粗饲料。本实验室前期研究发现富含单宁的植物饲料降低反刍动物甲烷产量并影响其瘤胃甲烷菌结构[6]。这表明采食柞树叶、玉米秸秆和玉米青贮这3种粗饲料的梅花鹿的瘤胃甲烷菌结构可能有所不同。因此,本研究拟对采食上述3种粗饲料的梅花鹿的瘤胃甲烷菌结构进行比较分析,旨在为调控梅花鹿瘤胃甲烷产量提供理论基础。
1 材料与方法 1.1 试验动物及样品采集选取3只2岁龄的装有永久性瘤胃瘘管的成年雄性梅花鹿为研究对象,平均体重120 kg,单只单圈饲养于中国农业科学院特产研究所茸鹿试验基地。采用3×3拉丁方设计,3只试验动物分别饲喂以柞树叶(OL组)、玉米秸秆(CS组)和玉米青贮(CI组)为粗饲料的饲粮,每只梅花鹿每天饲喂相同精饲料2.0 kg,粗饲料在饲粮中约占50%(干物质基础),饲粮组成及营养水平见表 1。试验动物每天定时定量饲喂2次,自由饮水。每只试验动物每种粗饲料预饲1周,正试期为4周。试验期末通过瘤胃瘘管采集瘤胃内容物(约为200 g),置于冰盒中迅速带回实验室,保存于-80 ℃冰箱备用。
|
|
表 1 饲粮组成及营养水平(风干基础) Table 1 Composition and nutrient levels of diets (air-dry basis) |
采用珠磨与QIAGEN试剂盒相结合的方法提取瘤胃内容物总基因组DNA。具体方法如下:称取约0.2 g瘤胃内容物至灭菌后的离心管中,加入约0.7 g直径为0.2 mm的硅珠和1.4 mL ASL溶液;混合样品迅速置于FastPrep -24(MP Biomedicals)以6.5 m/s的速度振荡45 s,之后按照粪便基因组DNA试剂盒说明进行操作。
1.3 PCR扩增与Illumina Miseq测序采用引物A519F(5′-CAGCMGCCGCGGTAA-3′)和A976R(5′-CCGGCGTTGAMTCCAATT-3′)[7]扩增甲烷菌16S rRNA基因V3~V4区。PCR产物纯化后送至上海美吉生物医药科技有限公司进行PCR产物定量、DNA序列修饰、文库构建及Illumina Miseq PE250平台测序。
1.4 数据处理与生物信息学分析对双端测序对原始数据进行质量控制,舍弃低质量序列(50个连续碱基平均质量<25、序列长度<50的序列)。采用软件Flash连接通过质量控制的序列对应的两端序列(错配率为0),获得分析序列。采用QIIME1.7.0软件包分析梅花鹿瘤胃甲烷菌多样性[8]。根据条形码(barcode)信息对序列进行拆分,同时根据以下条件剔除低质量序列:1)序列最短长度为400 bp,最长长度为500 bp;2)50个连续碱基最低质量为25;3)barcode序列最大错误数为0;4)序列中同聚物最大长度为6;5)引物错配率为0。采用Usearch61根据97%序列相似性将所有序列归为操作分类单元(operational taxonomic unit,OTU)[9]。OTU代表序列与Greengenes数据库[10]进行PyNAST比对,采用Chimera Slayer软件去除嵌合体[11]。选取至少出现在5个样品中的OTU序列构建OTU表。选取每个OTU代表序列,利用Blast程序在NCBI中搜索相似性最高序列,对OTU序列进行分类。采用QIIME1.7.0软件包计算菌群丰富度指数(Chao1指数)、覆盖度指数(coverage index)及多样性指数[香农-威纳(Shannon-Wiener)指数和辛普森(Simpson)指数][8]。
以嗜热菌(Aquifex pyrophilus)为外群,选取21种甲烷菌16S rRNA基因序列和所有OTU代表序列,利用MEGA 5.05软件中的ClustalW比对后输出为同一长度序列,利用Kimura-two参数矩阵模型和邻接(neighbor-joining,NJ)法进行系统发育分析,设置Bootstrap值为1 000[12]。FigTree v1.4.0软件显示发育树。
1.5 统计分析基于SigmaPlot 12.0软件采用三因素方差分析对不同组别甲烷菌的相对丰度进行显著性检验,甲烷菌相对丰度和多样性指数等数据结果表示为平均值±标准误,P<0.05为差异显著。
2 结果与分析 2.1 测序结果与多样性指数本试验中9个样本共获得600 352条高质量甲烷菌16S rRNA基因序列,每个样品的平均序列数为66 705。以97%序列相似性为阈值,600 352个序列归为111个OTU。样品覆盖度指数表明试验所得OTU代表了瘤胃中99%的甲烷菌,满足后续分析。3组中丰富度指数(Chao1指数)和多样性指数(Shannon-Wiener指数和Simpson指数)无显著差异(P>0.05),但CS组与CI组Shannon-Wiener指数和Simpson指数较OL组有升高的趋势(表 2)。
|
|
表 2 高通量测序数据概况 Table 2 Summary of high throughput sequencing data |
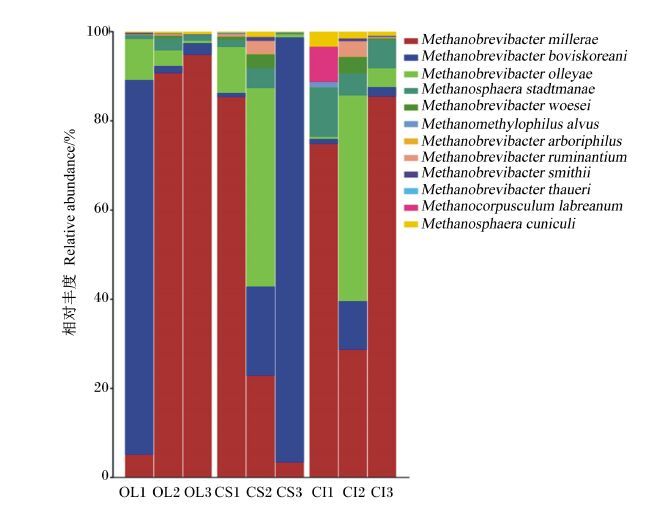
111个OTU代表序列与Genbank数据库序列进行Blast分析,结果如图 1所示。结果表明,这些序列归属为:甲烷短杆菌属(Methanobrevibacter spp.),OL组、CS组与CI组瘤胃中比例分别为(97.80±6.00)%、(97.10±1.50)%、(87.50±5.50)%;甲烷球菌属(Methanosphaera spp.),OL组、CS组与CI组瘤胃中比例分别为(2.09±0.51)%、(2.60±1.34)%、(9.40±2.45)%;甲烷粒菌属(Methanocorpusculum spp.),OL组、CS组与CI组瘤胃中比例分别为(0.01±0.01)%、(0.02±0.00)%、(2.60±2.00)%;Methanomethylophilus spp.,OL组、CS组与CI组瘤胃中比例分别为(0.01%±0.00)%、(0.10%±0.01)%、(0.50±0.30)%。甲烷短杆菌属序列在种水平分类(表 3)发现,OL组、CS组和CI组中分别有(63.50±20.00)%、(37.20±20.00)%和(63.00±10.00)%的OTU与Methanobrevibacter millerae的16S rRNA相似(相似性:97%~99%);OL组、CS组和CI组分别有(29.40±20.00)%、(38.80±20.00)%和(4.70±3.00)%的OTU与Methanobrevibacter boviskoreani的16S rRNA相似(相似性:95%~99%);OL组、CS组和CI组分别有(4.30±2.00)%、(18.40±10.00)%和(16.80±10.00)%的OTU与Methanobrevibacter olleyae的16S rRNA相似(相似性:97%~98%);OL组、CS组和CI组分别有(0.20±0.10)%、(1.30±0.90)%和(1.40±0.90)%的OTU与Methanobrevibacter woesei的16S rRNA相似(相似性:95%~97%);OL组、CS组和CI组分别有(0.20±0.09)%、(1.10±0.80)%和(1.20±1.00)%的OTU与Methanobrevibacter ruminantium的16S rRNA相似(相似性:97%~99%)。甲烷球菌属中,OL组、CS组和CI组分别有(1.70±0.50)%、(2.20±1.00)%和(7.50±1.00)%的OTU与Methanosphaera stadtmanae的16S rRNA相似(相似性:96%~99%);OL组、CS组和CI组分别有(0.39±0.09)%、(0.40±0.30)%和(1.90±0.70)%的OTU与Methanosphaera cuniculi的16S rRNA相似(相似性:95%~96%)。甲烷粒菌属中,OL组、CS组和CI组分别有(0.010±0.005)%、(0.020±0.004)%和(2.600±2.000)%的OTU与Methanocorpusculum labreanum的16S rRNA相似(相似性:98%)。另外,OL组、CS组和CI组中分别有(0.010±0.004)%、(0.100±0.010)%和(0.500±0.300)%的OTU与Candidatus Methanomethylophilus alvus的16S rRNA相似(相似性:93%~98%)。而且,CI组中Methanosphaera stadtmanae相对丰度显著高于OL组与CS组(P<0.05)。
|
OL=柞树叶组,CS=玉米秸秆组,CI=玉米青贮组。 OL=Oak leaf group,CS=corn stover group and CI=corn silage group. 图 1 OL、CS与CI组梅花鹿瘤胃甲烷菌平均相对丰度分布 Figure 1 he relative abundance distribution of methanogen on average in the rumen of sika deer in the OL,CS and CI groups |
|
|
表 3 采食不同粗饲料梅花鹿瘤胃甲烷菌种水平组成 Table 3 Composition of methanogen at species level in the rumen of sika deer fed different forages |
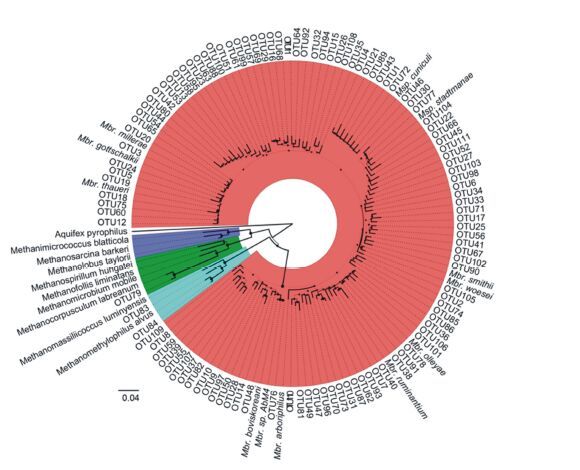
系统发育分析(图 2)表明,111个OTU聚为三大簇。99个OTU与Methanobrevibacter spp.和Methanosphaera spp.聚为一簇;OTU83和OTU84与Methanomethylophilus alvus和Methanomassiliicoccus luminyensis聚为一簇;OTU79与Methanocorpusculum labreanum聚为一簇。
|
图 2 梅花鹿瘤胃甲烷菌16S rRNA基因系统进化分析 Figure 2 Phylogenetic analysis of methanogen 16S rRNA genes from the rumen of sika deer |
瘤胃是自然界中多样且复杂的生态系统,其中栖息有大量的细菌、甲烷菌、真菌、原虫和少量噬菌体等。瘤胃微生物通过复杂的协同作用将饲料中的有机聚合物降解为单体并最终转化为挥发性脂肪酸、二氧化碳和氢气,其中挥发性脂肪酸是宿主的主要能量来源。瘤胃甲烷菌能够利用二氧化碳作为碳源,氢作为主要的电子供体形成发酵副产物——甲烷。甲烷的生成能避免瘤胃氢气压强升高而引起的电子转移反应中微生物酶活性受到抑制,尤其是还原型辅酶Ⅰ脱氢酶(NADH),当NADH积累一定程度会抑制瘤胃发酵的正常进行,因此甲烷的生成能够保证瘤胃发酵系统的有序进行[13]。然而,甲烷的形成不但会导致机体能量的损失,而且会产生温室效应[1-2]。因此,明确瘤胃甲烷菌结构对于理解瘤胃发酵及调控都具有重要意义。本研究首次采用高通技术深入研究了采食3种粗饲料梅花鹿瘤胃甲烷菌的结构,这一研究结果将为调控梅花鹿瘤胃甲烷生成,减少甲烷排放量提供依据。
首先,本研究发现梅花鹿瘤胃优势甲烷菌为Methanobrevibacter spp.,这与国内外在其他草食动物,如委内瑞拉绵羊[14]、非洲野生黑斑羚[15]、北美地区荷斯坦牛[16-17]和中国德昌水牛瘤胃[18]等上的研究结果一致,表明Methanobrevibacter spp.在草食动物前肠甲烷形成中起重要作用。其次,本研究证明Methanobrevibacter millerae是梅花鹿瘤胃优势甲烷菌,这与鹿科动物矮鹿的研究结果[7]相同,但与牛科反刍动物却有一定差异,比如,委内瑞拉绵羊瘤胃主要甲烷菌是Methanobrevibacter gottschalkii[14],北美荷斯坦牛[16-17]与挪威驯鹿瘤胃[19]优势甲烷菌是Methanobrevibacter ruminantium,印度水牛[20]、中国青藏高原奶牛和牦牛瘤胃[21]瘤胃古菌C簇(RCC)最丰富,这些共性与差异性说明宿主特异性可能对瘤胃甲烷菌的种水平有显著影响。已有研究表明甲烷菌的发育型或菌株型在影响瘤胃甲烷生成量方面可能起着更为重要的作用[22],然而,瘤胃甲烷产量又受到细菌型[23]及瘤胃体积大小[24]的影响。因此,这提示我们瘤胃甲烷产量的调控可能需要基于瘤胃微生态和宿主多角度考虑。Methanobrevibacter millerae是一种能够以氢气或甲酸作为电子供体还原二氧化碳生成甲烷的甲烷菌[25],而梅花鹿与矮鹿瘤胃优势细菌Prevotella spp.是一类通过琥珀酸或丙烯酸途径利用氢气生成丙酸的细菌[26]。这说明瘤胃内氢气流转代谢可能是影响Methanobrevibacter millerae丰度的一个重要因素。有关Prevotella spp.与Methanobrevibacter millerae之间的关系值得深入研究。RCC是一类与Methanomassiliicoccus luminyensis和Candidatus Methanomethylophilus alvus相似性低的甲烷菌,它们在瘤胃中的比例大约为15.8%[3],能以甲胺类化合物或甲醇为底物生成甲烷,但不能利用二氧化碳,而且是调控反刍动物甲烷产量的目标甲烷菌[27-28]。RCC在梅花鹿瘤胃的丰度不足1%表明梅花鹿瘤胃甲烷产量可能较低。
其次,本研究发现粗饲料种类显著影响梅花鹿瘤胃甲烷菌结构。OL组Methanosphaera spp.与Methanobrevibacter olleyae相对丰度低于CS组和CI组,这可能与柞树叶中含有单宁(含量:98 mg/kg)有关。研究发现瘤胃某些甲烷菌对单宁比较敏感,而且单宁对产甲烷菌生长所需的酶具有抑制作用。另外,饲粮中的单宁不但可与瘤胃内细菌细胞壁的碳水化合物形成复合物或与细胞结合性胞外酶发生反应抑制蛋白质降解菌的生长,而且能够与饲粮中蛋白质形成复合物从而降低蛋白质的降解[29],这也与我们前期研究发现采食富含单宁柞树叶梅花鹿瘤胃异丁酸和异戊酸含量降低[5]的结果一致。由于Methanosphaera spp.利用甲醇和氢气生成甲烷,不能单独利用二氧化碳或甲酸[30],而Methanobrevibacter olleyae利用氢气或甲酸还原二氧化碳生成甲烷[25],而且不同甲烷菌生长所需的氢气阈值也有所差异[31]。这表明饲粮在瘤胃降解过程中产生的氢气可能是影响甲烷菌组成的一个重要方面。此外,本试验中CI组Methanobrevibacter boviskoreani相对丰度低于CS组。研究发现Methanobrevibacter boviskoreani是一种利用氢气或甲酸产生甲烷的甲烷菌,其适宜生长pH为6.5~7.0[32]。陶莲等[33]发现玉米秸秆经过青贮发酵后乳杆菌目细菌数量及乳酸和乙酸 含量显著增加,而pH显著下降到3.8。因此,CI组较低的pH可能抑制了Methanobrevibacter boviskoreani的生长。
4 结 论本研究发现Methanobrevibacter spp.是梅花鹿瘤胃优势甲烷菌。
| [1] |
GILL M, SMITH P, WILKINSON J M. Mitigating climate change:the role of domestic livestock[J]. Animal, 2010 , 4 (3) : 323 –333.
DOI: 10.1017/S1751731109004662 ( 0) 0)
|
| [2] |
JOHNSON K A, JOHNSON D E. Methane emissions from cattle[J]. Journal of Animal Science, 1995 , 73 (8) : 2483 –2492.
DOI: 10.2527/1995.7382483x ( 0) 0)
|
| [3] |
JANSSEN P H, KIRS M. Structure of the archaeal community of the rumen[J]. Applied and Environmental Microbiology, 2008 , 74 (12) : 3619 –3625.
DOI: 10.1128/AEM.02812-07 ( 0) 0)
|
| [4] |
ST-PIERRE B, WRIGHT A D G. Diversity of gut methanogens in herbivorous animals[J]. Animal, 2013 , 7 (Suppl.1) : 49 –56.
( 0) 0)
|
| [5] |
LI Z P, WRIGHT A D G, LIU H L, et al. Bacterial community composition and fermentation patterns in the rumen of sika deer (Cervus nippon) fed three different diets[J]. Microbial Ecology, 2015 , 69 (2) : 307 –318.
DOI: 10.1007/s00248-014-0497-z ( 0) 0)
|
| [6] |
TAN H Y, SIEO C C, ABDULLAH N, et al. Effects of condensed tannins from Leucaena on methane production,rumen fermentation and populations of methanogens and protozoa in vitro[J]. Animal Feed Science and Technology, 2011 , 169 (3/4) : 185 –193.
( 0) 0)
|
| [7] |
LI Z P, ZHANG Z G, XU C, et al. Bacteria and methanogens differ along the gastrointestinal tract of Chinese roe deer (Capreolus pygargus)[J]. PLoS One, 2014 , 9 (12) : e114513 .
DOI: 10.1371/journal.pone.0114513 ( 0) 0)
|
| [8] |
CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al. QIIME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010 , 7 (5) : 335 –336.
DOI: 10.1038/nmeth.f.303 ( 0) 0)
|
| [9] |
EDGAR R C. Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics, 2010 , 26 (19) : 2460 –2461.
DOI: 10.1093/bioinformatics/btq461 ( 0) 0)
|
| [10] |
DESANTIS T Z, HUGENHOLTZ P, LARSEN N, et al. Greengenes,a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J]. Applied and Environmental Microbiology, 2006 , 72 (7) : 5069 –5072.
DOI: 10.1128/AEM.03006-05 ( 0) 0)
|
| [11] |
HAAS B J, GEVERS D, EARL A M, et al. Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons[J]. Genome Research, 2011 , 21 (3) : 494 –504.
DOI: 10.1101/gr.112730.110 ( 0) 0)
|
| [12] |
TAMURA K, PETERSON D, PETERSON N, et al. MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011 , 28 (10) : 2731 –2739.
DOI: 10.1093/molbev/msr121 ( 0) 0)
|
| [13] |
HUNGATE R E. Hydrogen as an intermediate in the rumen fermentation[J]. Archiv für Mikrobiologie, 1967 , 59 (1/2/3) : 158 –164.
( 0) 0)
|
| [14] |
WRIGHT A D G, MA X L, OBISPO N E. Methanobrevibacter phylotypes are the dominant methanogens in sheep from Venezuela[J]. Microbial Ecology, 2008 , 56 (2) : 390 –394.
DOI: 10.1007/s00248-007-9351-x ( 0) 0)
|
| [15] |
CERSOSIMO L M, LACHANCE H, ST-PIERRE B, et al. Examination of the rumen bacteria and methanogenic archaea of wild impalas (Aepyceros melampus melampus) from Pongola,South Africa[J]. Microbial Ecology, 2015 , 69 (3) : 577 –585.
DOI: 10.1007/s00248-014-0521-3 ( 0) 0)
|
| [16] |
HOOK S E, STEELE M A, NORTHWOOD K S, et al. Impact of high-concentrate feeding and low ruminal pH on methanogens and protozoa in the rumen of dairy cows[J]. Microbial Ecology, 2011 , 62 (1) : 94 –105.
DOI: 10.1007/s00248-011-9881-0 ( 0) 0)
|
| [17] |
KONG Y H, XIA Y, SEVIOUR R, et al. Biodiversity and composition of methanogenic populations in the rumen of cows fed alfalfa hay or triticale straw[J]. FEMS Microbiology Ecology, 2013 , 84 (2) : 302 –315.
DOI: 10.1111/femsec.2013.84.issue-2 ( 0) 0)
|
| [18] |
杨承剑, 韦升菊, 梁辛, 等. 利用16S rRNA基因克隆文库技术分析德昌水牛瘤胃产甲烷菌的多样性[J].
杨承剑, 韦升菊, 梁辛, 等. 利用16S rRNA基因克隆文库技术分析德昌水牛瘤胃产甲烷菌的多样性[J]. 湖南农业大学学报:自然科学版, 2014 , 40 (4) :382 –388.
( 0) 0)
|
| [19] |
SUNDSET M A, EDWARDS J E, CHENG Y F, et al. Rumen microbial diversity in svalbard reindeer,with particular emphasis on methanogenic archaea[J]. FEMS Microbiology Ecology, 2009 , 70 (3) : 553 –562.
DOI: 10.1111/fem.2009.70.issue-3 ( 0) 0)
|
| [20] |
CHAUDHARY P P, SIROHI S K, SAXENA J. Diversity analysis of methanogens in rumen of Bubalus bubalis by 16S riboprinting and sequence analysis[J]. Gene, 2012 , 493 (1) : 13 –17.
DOI: 10.1016/j.gene.2011.11.041 ( 0) 0)
|
| [21] |
HUANG X D, TAN H Y, LONG R J, et al. Comparison of methanogen diversity of yak (Bos grunniens) and cattle (Bos taurus) from the Qinghai-Tibetan plateau,China[J]. BMC Microbiology, 2012 , 12 (1) : 237 .
DOI: 10.1186/1471-2180-12-237 ( 0) 0)
|
| [22] |
ZHOU M, HERNANDEZ-SANABRIA E, GUAN L L. Characterization of variation in rumen methanogenic communities under different dietary and host feed efficiency conditions,as determined by PCR-denaturing gradient gel electrophoresis analysis[J]. Applied and Environmental Microbiology, 2010 , 76 (12) : 3776 –3786.
DOI: 10.1128/AEM.00010-10 ( 0) 0)
|
| [23] |
KITTELMANN S, PINARES-PATIÑO C S, SEEDORF H, et al. Two different bacterial community types are linked with the low-methane emission trait in sheep[J]. PLoS One, 2014 , 9 (7) : e103171 .
DOI: 10.1371/journal.pone.0103171 ( 0) 0)
|
| [24] |
GOOPY J P, DONALDSON A, HEGARTY R, et al. Low-methane yield sheep have smaller rumens and shorter rumen retention time[J]. British Journal of Nutrition, 2014 , 111 (4) : 578 –585.
DOI: 10.1017/S0007114513002936 ( 0) 0)
|
| [25] |
REA S, BOWMAN J P, POPOVSKI S, et al. Methanobrevibacter millerae sp.nov.and Methanobrevibacter olleyae sp.nov.,methanogens from the ovine and bovine rumen that can utilize formate for growth[J]. International Journal of Systematic and Evolutionary Microbiology, 2007 , 57 (3) : 450 –456.
DOI: 10.1099/ijs.0.63984-0 ( 0) 0)
|
| [26] |
PURUSHE J, FOUTS D E, MORRISON M, et al. Comparative genome analysis of Prevotella ruminicola and Prevotella bryantii:insights into their environmental niche[J]. Microbial Ecology, 2010 , 60 (4) : 721 –729.
DOI: 10.1007/s00248-010-9692-8 ( 0) 0)
|
| [27] |
BORREL G, HARRIS H M B, TOTTEY W, et al. Genome sequence of "Candidatus Methanomethylophilus alvus" Mx1201,a methanogenic archaeon from the human gut belonging to a seventh order of methanogens[J]. Journal of Bacteriology, 2012 , 194 (24) : 6944 –6945.
DOI: 10.1128/JB.01867-12 ( 0) 0)
|
| [28] |
POULSEN M, SCHWAB C, JENSEN B B, et al. Methylotrophic methanogenic Thermoplasmata implicated in reduced methane emissions from bovine rumen[J]. Nature Communications, 2013 , 4 : 1428 .
DOI: 10.1038/ncomms2432 ( 0) 0)
|
| [29] |
MCSWEENEY C S, PALMER B, MCNEILL D M, et al. Microbial interactions with tannins:nutritional consequences for ruminants[J]. Animal Feed Science and Technology, 2001 , 91 (1/2) : 83 –93.
( 0) 0)
|
| [30] |
FRICKE W F, SEEDORF H, HENNE A, et al. The genome sequence of Methanosphaera stadtmanae reveals why this human intestinal archaeon is restricted to methanol and H2 for methane formation and ATP synthesis[J]. Journal of Bacteriology, 2006 , 188 (2) : 642 –658.
DOI: 10.1128/JB.188.2.642-658.2006 ( 0) 0)
|
| [31] |
CARLOLINE CHAE-HYUN K.Identification of rumen methanogens,characterization of substrate requirements and measurement of hydrogen thresholds[D].MSc.Thesis.Palmerston North:Massey University,2012.
( 0) 0)
|
| [32] |
LEE J H, KUMAR S, LEE G H, et al. Methanobrevibacter boviskoreani sp.nov.,isolated from the rumen of Korean native cattle[J]. International Journal of Systematic and Evolutionary Microbiology, 2013 , 63 (11) : 4196 –4201.
( 0) 0)
|
| [33] |
陶莲, 刁其玉. 青贮发酵对玉米秸秆品质及菌群构成的影响[J].
陶莲, 刁其玉. 青贮发酵对玉米秸秆品质及菌群构成的影响[J]. 动物营养学报, 2016 , 28 (1) :198 –207.
( 0) 0)
|




