研究瘤胃内微生物对饲料蛋白质的降解和利用机制是调控反刍动物瘤胃氮代谢的基础。阐明蛋白质在瘤胃原虫和细菌中的降解与脱氨的机制是实现瘤胃蛋白质代谢调控的前提。蛋白质是反刍动物饲粮的最主要氮源[1],反刍动物氮利用率低的一个重要原因是饲粮蛋白质在瘤胃内被大量降解的过程中造成显著的氮浪费。瘤胃微生物(主要包括细菌和原虫)分泌的酶把蛋白质水解成多肽、二肽或三肽后继续被水解成氨基酸,最终被发酵成氨氮[2]。瘤胃原虫长期以来都被认为是脱氨基作用的积极参与者和瘤胃氮代谢低效率的主要原因[3-5]。有研究报道,瘤胃纤毛虫合成蛋白质时对氨基酸的需求取决于其摄取的细菌,而不利用氨(NH4+)作为氮源[6-8]。研究表明,具有原虫的瘤胃内氨氮浓度是去原虫的瘤胃内氨氮浓度的2倍[9]。原虫细胞抽提物中氨基酸脱氨基酶的活力至少是细菌的3倍[10]。目前,对于瘤胃原虫产氨效率高于细菌的原因尚不十分清楚。瘤胃微生物与蛋白质分解代谢相关的酶的活力高低是影响反刍家畜饲粮蛋白质降解的重要因素,本试验探讨了瘤胃细菌和原虫细胞中与蛋白质分解代谢相关的蛋白酶(使蛋白质水解为肽或氨基酸)、谷丙转氨酶、谷草转氨酶(催化氨基转移及生成氨基酸)和谷氨酸脱氢酶(glutamate dehydrogenase, GDH)[催化氨基酸脱氨及利用NH4+合成氨基酸]4种酶在瘤胃内的动态变化规律与酶促反应动力学特性,为深入认识绵羊瘤胃细菌、原虫蛋白质分解代谢特征提供相关酶学依据。
1 材料与方法 1.1 试验动物本试验选用平均体重为(32.00±1.36) kg、1岁左右的中国美利奴(新疆型)细毛公羊6只,在试验前安装永久性瘤胃瘘管。饲喂精粗比为30:70的饲粮。每天09:00、21:00各饲喂1次,精料与粗料(玉米秸秆)分2次等量饲喂,将精料与粗料混合后饲喂。自由饮水。饲粮组成及营养水平见表 1。
|
|
表 1 饲粮组成及营养水平(干物质基础) Table 1 Composition and nutrient levels of the diet (DM basis) |
每只羊每周采样1次,共采3次。采样时间点为饲喂前(0 h)和饲喂后1.5、3.0、6.0、9、12.0 h。抽取用20目尼龙网过滤的瘤胃液,每个时间点每只羊抽取120 mL瘤胃液,从中量取100 mL瘤胃液,所有羊的瘤胃液混合在一起,再从中量取480 mL用于分离瘤胃液原虫、细菌,制备无细胞破碎提取物,以测定酶活力。
1.3 瘤胃液细菌、原虫分离和破碎将混合瘤胃液480 mL,150×g离心10 min后弃沉淀,将上清液650×g离心20 min,将细菌和原虫分离,沉淀为原虫组分,上清液为细菌组分。
将沉淀(原虫组分)用4 ℃过夜预冷的磷酸盐缓冲液(25 mL)溶解后,低温高速离心机4 ℃ 650×g离心20 min (重复2次)。将上清液(细菌组分)10 000×g离心20 min,弃上清液,收集所有沉淀,用4 ℃过夜预冷的磷酸盐缓冲液(50 mL)溶解后,低温高速离心机4 ℃ 10 000×g离心20 min (重复2次)。之后,分别收集沉淀,加入预冷的生理盐水溶解,定容至40 mL,混匀后破碎(用超声波破碎机,超声破碎15 min,功率400 W,超声3 s,间隔5 s,工作60次,重复3次)。破碎液经低温高速离心机4 ℃ 10 000×g离心20 min,收集上清液,-20 ℃保存,测定酶活力、谷氨酸脱氢酶体系的米氏常数(Km)值、谷氨酸和氨和蛋白质含量。
1.4 蛋白质含量、酶活力的测定瘤胃细菌、原虫破碎液的蛋白质含量测定参考张龙翔等[11]的方法进行。标准蛋白质为牛血清白蛋白。
瘤胃液细菌和原虫蛋白酶、谷丙转氨酶、谷草转氨酶及谷氨酸脱氢酶活力的测定参考Palmquist等[12]的方法进行。分光光度计使用紫外可见分光光度计(754PC),石英比色皿(光程1.0 cm),水浴锅为SSW型微电脑电热恒温水槽(上海博讯实业有限公司),还原型烟酰胺腺嘌呤二核苷酸(NADH)、烟酰胺腺嘌呤二核苷酸(NAD)为Sigma公司产品,其余均为国产分析纯试剂。
1.5 瘤胃液细菌、原虫谷氨酸脱氢酶Km值的测定谷氨酸脱氢酶催化的脱氨反应为:
谷氨酸+NAD⇌α-酮戊二酸+NADH+氨。
本试验分别测定催化反应中谷氨酸脱氢酶对反应式中5种物质的Km值。
瘤胃液细菌、原虫谷氨酸脱氢酶Km值测定时底物谷氨酸浓度分别为2.5、5.0、10.0、20.0、40.0 mmol/L;NAD浓度分别为0.05、0.10、0.50、1.00、2.00、4.00 mmol/L;NADH浓度分别为0.157、0.313、0.625、0.250、0.500 mmol/L;α-酮戊二酸浓度分别为1.25、2.50、5.00、10.00、20.00 mmol/L;氨浓度分别为10、20、40、80、160 mmol/L。各浓度底物的测定步骤均与测定谷氨酸脱氢酶活力相同。测定出各底物浓度的反应初速度,按Linewaver Burk法作图,将米氏方程两侧取双倒数,得方程式:
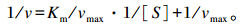
|
式中:v为反应初速度(mol/min);[S]为底物浓度(mol/L);vmax为最大反应速度[mol/min];横轴截距为-1/Km,纵轴截距为1/vmax,用图解法求出vmax和Km值。
1.6 瘤胃液细菌、原虫谷氨酸、氨含量的测定谷氨酸、氨含量采用酶法测定,参考吉尔鲍特[13]的方法进行。测定谷氨酸含量时,谷氨酸标准液的终浓度分别为0、10、25、50、100、200 mmol/L,NAD的终浓度为2 mmol/L,加入谷氨酸脱氢酶100 U,反应总体积为3 mL;混匀后置于37 ℃水浴40 min,取出后于紫外可见分光光度计(754PC)上测定340 nm处的吸光度值,建立标准曲线,调整待测样品的谷氨酸含量在标准浓度范围内进行同样操作,测定。测定氨含量时,氨标准液的终浓度分别为0、1、2、4、7、10 mmol/L,NADH的终浓度为4 mmol/L,加入谷氨酸脱氢酶100 U,反应总体积为3 mL;混匀后置于37 ℃水浴40 min,取出后于紫外可见分光光度计(754PC)上测定340 nm处的吸光度值,建立标准曲线,调整待测样品的氨含量在标准浓度范围内进行同样操作,测定。
1.7 数据处理试验数据以平均值±标准差表示,数据统计分析采用SPSS 16.0软件进行单因素方差分析,平均值的多重比较采用Duncan氏法进行,以独立样本t检验法对细菌和原虫进行比较。
2 结果与分析 2.1 绵羊瘤胃细菌、原虫蛋白质分解代谢主要相关酶活力的动态变化从表 2可见,细菌、原虫破碎液中的蛋白酶、谷丙转氨酶、谷草转氨酶、谷氨酸脱氢酶4种酶的活力随饲喂后时间的延长均呈现“低-高-低”的动态变化规律,且除了细菌谷丙转氨酶活力外均在饲喂后1.5 h达到最大值,在饲喂后12.0 h活力基本恢复到饲喂前0 h的水平。各时间点的原虫破碎液中蛋白酶活力比细菌高出近50%,两者差异极显著(P < 0.01),而原虫破碎液中谷丙转氨酶、谷草转氨酶、谷氨酸脱氢酶3种酶的活力几乎是细菌的2倍,差异极显著(P < 0.01)。
|
|
表 2 绵羊瘤胃细菌、原虫破碎液中蛋白质分解代谢几种相关酶的活力 Table 2 Activities of enzymes related to protein catabolism in rumen bacteria and protozoa crushing liquid of sheep (n=3) |
从表 3可见,细菌和原虫破碎液谷氨酸含量随饲喂时间的延长均呈现先升高后降低的动态变化规律。饲喂后1.5 h,细菌和原虫破碎液谷氨酸含量均达到最大值。饲喂前、后各时间点原虫破碎液谷氨酸含量均极显著高于细菌(P < 0.01)。细菌和原虫破碎液氨含量随饲喂时间的延长均呈现先升高后降低的动态变化规律。饲喂后1.5 h,细菌和原虫破碎液氨含量均达到最大值。在饲喂后1.5 h,原虫破碎液氨含量极显著高于细菌(P < 0.01)。在饲喂后6.0、9.0和12.0 h,原虫破碎液氨含量显著高于细菌(P < 0.05)。
|
|
表 3 绵羊瘤胃细菌、原虫破碎液中谷氨酸、氨含量 Table 3 Contents of glutamate and NH4+ in rumen bacteria and protozoa crushing liquid of sheep (n=3) |
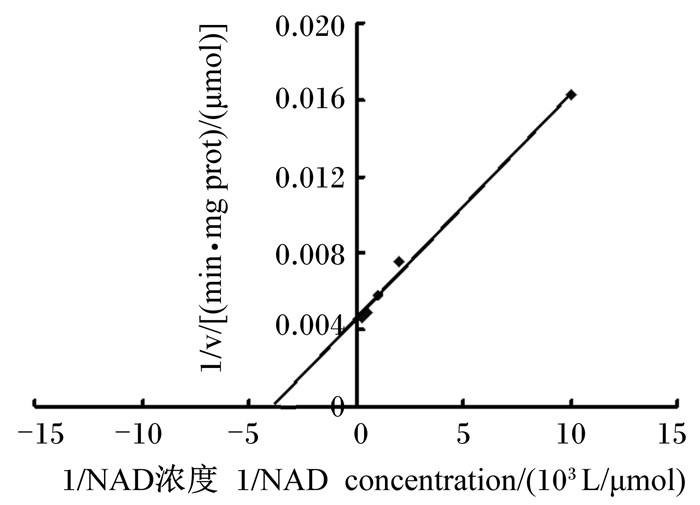
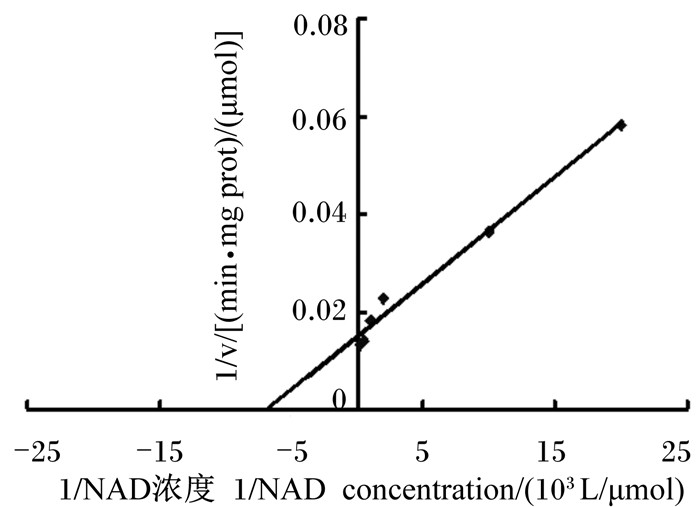
按Linewaver Burk法作图,得到的典型结果如图 1、图 2所示。图解计算出的Km值和vmax结果见表 4。从表可见,谷氨酸脱氢酶对NAD+:细菌Km值为2.60×10-7 mol/L,vmax为2.17×10-4 mol/(L·min);原虫Km值为1.48×10-7 mol/L,vmax为6.70×10-5 mol/(L·min),原虫Km值较小。谷氨酸脱氢酶对谷氨酸:细菌Km值为8.41×10-6 mol/L,vmax为1.35×10-4 mol/(L·min);原虫Km值为4.91×10-6 mol/L,vmax为1.79×10-4 mol/(L·min),原虫Km值较小。谷氨酸脱氢酶对NADH:细菌Km值为3.80×10-8 mol/L,vmax为1.89×10-4 mol/(L·min);原虫Km值为2.70×10-8 mol/L,vmax为2.73×10-4 mol/(L·min),原虫Km值较小。谷氨酸脱氢酶对α-酮戊二酸:细菌Km值为1.16×10-6 mol/L,vmax为4.00×10-4 mol/(L·min);原虫Km值为2.07×10-6 mol/L,vmax为6.67×10-4 mol/(L·min),细菌Km值较小。谷氨酸脱氢酶对氨:细菌Km值为2.97×10-5 mol/L,vmax为3.57×10-4 mol/(L·min);原虫Km值为1.40×10-5 mol/L,vmax为3.33×10-4 mol/(L·min),原虫Km值较小。
|
图 1 瘤胃细菌破碎液谷氨酸脱氢酶体系对NAD的Km值 Figure 1 Km value of GDH to NAD in rumen bacteria crushing liquid |
|
图 2 瘤胃原虫破碎液谷氨酸脱氢酶体系对NAD的Km值 Figure 2 Km value of GDH to NAD in rumen protozoa crushing liquid |
|
|
表 4 绵羊瘤胃液细菌、原虫破碎液谷氨酸脱氢酶的Km值比较 Table 4 Km values of GDH to NAD in rumen bacteria and protozoa crushing liquid |
本试验测定了在饲粮精粗比为30:70时,绵羊瘤胃细菌、原虫破碎液在饲喂前、后各时间点蛋白质分解主要相关酶活力的变化。从酶活力的动态变化来看,瘤胃细菌和原虫蛋白酶、谷丙转氨酶、谷草转氨酶及谷氨酸脱氢酶的活力变化均呈现先升高后降低的动态变化规律,总体上在饲喂后1.5 h上升至最大值,之后缓慢降低。Chen等[14]的研究表明,绵羊瘤胃中的肽浓度随饲喂后时间的延长呈现出先升高后缓慢降低的变化规律,且在1.5 h达到最高。瘤胃蛋白质的快速降解在采食后1.5 h左右达到最活跃的阶段,这可能导致小肽和氨基酸的迅速积累[8]。王梦芝[15]和翟卫爽[16]的研究指出,绵羊瘤胃液氨态氮浓度在不同饲粮间均表现出有相似的变化趋势,采食后1.5~3.0 h较高,之后逐渐降低。氨态氮浓度的变化规律表明,在采食后1.5 h,瘤胃中的脱氨反应达到最活跃的阶段。
瘤胃液中氨态氮浓度受饲粮蛋白质含量的直接影响,也与原虫数量和种类有关。饲粮蛋白质含量高,瘤胃细菌可利用的蛋白质底物含量也增加,蛋白酶、谷丙转氨酶、谷草转氨酶及谷氨酸脱氢酶活力也随之提高,在瘤胃液中氨态氮浓度也就升高。瘤胃原虫细胞内的这4种酶活力都显著高于瘤胃细菌,去原虫能显著降低瘤胃液中氨态氮的浓度[9]。本试验从蛋白酶、谷氨酸脱氢酶、谷丙转氨酶及谷草转氨酶4种酶活力的变化规律为瘤胃液中肽、氨态氮浓度的变化规律提供了酶学理论依据。
3.2 绵羊瘤胃细菌、原虫破碎液谷氨酸脱氢酶反应体系Km值比较有关瘤胃细菌、原虫细胞内谷氨酸脱氢酶反应体系从Km值尚未见有报道。Shino等[17]研究显示,黄色短杆菌(Brevibacterium flavum)谷氨酸脱氢酶对氨的Km值为3.08 mmol/L,对谷氨酸的Km值为100.00 mmol/L,对α-酮戊二酸的Km值为5.72 mmol/L。Kujo等[18]研究显示,厌氧超嗜热菌(Anaerobic hyperthermophiles)谷氨酸脱氢酶对NAD的Km值为0.025 mmol/L,对谷氨酸Km值为0.170 mmol/L,对NADH的Km值为0.005 mmol/L,对α-酮戊二酸的Km值为0.066 mmol/L,对氨的Km值为9.700 mmol/L。Newbold等[19]研究表明,大肠杆菌的谷氨酸脱氢酶对氨和α-酮戊二酸的亲和力较强,Km值分别为2.33和0.71 mmol/L,对谷氨酸的亲和力较弱,Km值为98.00 mmol/L。本试验中得到的瘤胃细菌破碎液中谷氨酸脱氢酶对反应底物的Km值的规律与前人研究的几种细菌的Km值规律相似。综合谷氨酸脱氢酶的活力及Km值的比较来看:1)瘤胃原虫谷氨酸脱氢酶活力高于细菌,且对谷氨酸、NAD+的Km值比细菌低42%~43%,这表明原虫细胞内谷氨酸脱氢酶与谷氨酸、NAD+的亲和力高出细菌的42%~43%,反应催化速度更高,这也是为什么原虫谷氨酸脱氢酶的活力高于细菌,且产氨速度高于细菌的重要原因之一;细菌α-酮戊二酸的Km值低于原虫,表明细菌谷氨酸脱氢酶与α-酮戊二酸的亲和力更高,更有利于逆反应谷氨酸的合成,说明在细菌中逆反应的催化速度高于原虫,这也可能导致细菌产氨效率低于原虫。2)从氨的利用角度看,在细菌细胞中底物ɑ-酮戊二酸的Km值为1.16×10-6 mol/L,低于氨的2.97×10-5 mol/L;原虫细胞内的趋势与细菌相似。因此,在细菌、原虫细胞内ɑ-酮戊二酸的供应充足与否是细菌、原虫利用氨合成重新氨基酸的主要限制步骤,细菌、原虫细胞内的ɑ-酮戊二酸的含量和如何影响氨基酸的重新合成等问题需要进一步研究。
饲粮精粗比为30:70是反刍家畜一般饲养中较为普遍的饲粮结构。饲料精粗比例影响瘤胃内细菌、原虫的增殖和数量。因此,随着精粗比的增加,瘤胃内细菌、原虫数量也会随之发生变化,细胞内的酶活力也将发生变化。从谷氨酸脱氢酶反应体系的Km值来说,特定酶在特定反应条件下是酶的属性,因此,谷氨酸脱氢酶反应体系的Km值并不随底物浓度的变化(即饲粮精粗比例)而改变。
3.3 瘤胃原虫细胞内可能存在利用氨重新合成氨基酸的机制原虫不能利用氨而以吞噬小饲料颗粒和细菌的形式获得氮源,“瘤胃原虫是瘤胃内的净产氨者”,这是长久以来对瘤胃原虫氨代谢的一个基本认定。然而,Williams等[20]研究发现,纤毛虫可以从利用吞噬菌壁中的二氨基庚二酸合成赖氨酸,同时也认为全毛虫可利用氨重新合成氨基酸。本试验测得细菌、原虫破碎液底物氨的Km值分别为2.97×10-5、1.40×10-5 mol/L,这表明原虫的谷氨酸脱氢酶与氨的亲和力更高,更有利于原虫利用氨进行逆反应合成氨基酸,并为后续的谷丙转氨酶、谷草转氨酶提供更多的底物。这提示,瘤胃原虫中不仅存在谷氨酸转氨机制,可能还存在利用氨重新合成氨基酸的机制。
4 结论①总体上,绵羊瘤胃细菌、原虫中蛋白酶、谷氨酸脱氢酶、谷丙转氨酶、谷草转氨酶的活力在饲喂后1.5 h达到峰值,之后逐渐降低。
②绵羊瘤胃原虫中蛋白酶、谷丙转氨酶、谷草转氨酶和谷氨酸脱氢酶的活力均极显著高于细菌,原虫中蛋白质分解代谢更旺盛。
③瘤胃原虫中不仅存在谷氨酸转氨机制,可能还存在利用氨重新合成氨基酸的机制。
| [1] | 冯仰廉. 反刍动物营养学[M]. 北京: 科学出版社, 2004 : 35 . |
| [2] | RUSSELL J B, ONODERA R, HINO T.Ruminal protein fermentation:new perspectives on previous contradictions[M]//TSUDA T, SASAKI Y, KAWASHIMA R.Physiological aspects of digestion and metabolism in ruminants.Tokyo:Academic Press, 1991:681-697. |
| [3] | JOUANY J P, USHIDA K. The role of protozoa in feed digestion[J]. Asian-Australasian Journal of Animal Sciences, 1999 , 12 (1) : 113 –128. DOI: 10.5713/ajas.1999.113 |
| [4] | BOGUHN J, KLUTH H, RODEHUTSCORD M. Effect of total mixed ration composition on amino acid profiles of different fractions of ruminal microbes in vitro[J]. Journal of Dairy Science, 2006 , 89 (5) : 1592 –1603. DOI: 10.3168/jds.S0022-0302(06)72226-3 |
| [5] | LENG R A. Dynamics of protozoa in the rumen of sheep[J]. British Journal of Nutrition, 1982 , 48 (2) : 399 –415. DOI: 10.1079/BJN19820123 |
| [6] | WALLACE R J, ONODERA R, COTTA M A.Metabolism of nitrogen-containing compounds[M]//HOBSON P N, STEWART C S.The rumen microbial ecosystem.Netherlands:Springer, 1997:283-328. |
| [7] | WALLACE R J. Ruminal microbial metabolism of peptides and amino acids:altering ruminal nitrogen metabolism to improve protein utilization[J]. The Journal of Nutrition, 1996 , 126 (4) : 1326S –1334S. |
| [8] | WALLACE R J, BRODERICK G A, BRAMMALL M L. Protein degradation by ruminal microorganisms from sheep fed dietary supplements of urea, casein, or albumin[J]. Applied and Environmental Microbiology, 1987 , 53 (4) : 751 –753. |
| [9] | FIRKINS J L, YU Z, MORRISON M. Ruminal nitrogen metabolism:perspectives for integration of microbiology and nutrition for dairy[J]. Journal of Dairy Science, 2007 , 90 (Suppl.1) : E1 –E16. |
| [10] | HINO T, RUSSELL J B. Effect of reducing-equivalent disposal and NADH/NAD on deamination of amino acids by intact rumen microorganisms and their cell extracts[J]. Applied and Environmental Microbiology, 1985 , 50 (6) : 1368 –1374. |
| [11] | 张龙翔, 张庭芳, 李令媛. 生化实验方法和技术[M]. 2版. 北京: 高等教育出版社, 1997 : 135 -140. |
| [12] | PALMQUIST D L, BALDWIN R L. Enzymatic techniques for the study of pathways of carbohydrate utilization in the rumen[J]. Applied and Environmental Micobiology, 1966 , 14 (1) : 60 –69. |
| [13] | 吉尔鲍特G G.酶法分析手册[M].缪辉南, 陈石根, 译.上海:上海科学技术出版社, 1983:89-96. |
| [14] | CHEN G, RUSSELL J B, SNIFFEN C J. A procedure for measuring peptides in rumen fluid and evidence that peptide uptake can be a rate-limiting step in ruminal protein degradation[J]. Journal of Dairy Science, 1987 , 70 (6) : 1211 –1219. DOI: 10.3168/jds.S0022-0302(87)80133-9 |
| [15] | 王梦芝.山羊瘤胃原虫与细菌吞噬关系和微生物AA变化机制的研究[D].博士学位论文.扬州:扬州大学, 2008:56-59. http://cdmd.cnki.com.cn/Article/CDMD-11117-2008086933.htm |
| [16] | 翟卫爽.不同精粗比日粮时绵羊瘤胃液细菌、原虫内与葡萄糖代谢相关酶活力的比较研究[D].硕士学位论文.乌鲁木齐:新疆农业大学, 2014:34-39. |
| [17] | SHIIO I, OZAKI H. Regulation of nicotinamide adenine dinucleotide phosphate-specific glutamate dehydrogenase from Brevibacterium flavum, a glutamate-producing bacterium[J]. Journal of Biochemistry, 1970 , 68 (5) : 633 –647. |
| [18] | KUJO C, OHSHIMA T. Enzymological characteristics of the hyperthermostable NAD-dependent glutamate dehydrogenase from the archaeon Pyrobaculum islandicum and effects of denaturants and organic solvents[J]. Applied and Environmental Microbiology, 1998 , 64 (6) : 2152 –2157. |
| [19] | NEWBOLD C J, MCEWAN N R, CALZA R E, et al. An NAD+-dependent glutamate dehydrogenase cloned from the ruminal ciliate protozoan, Entodinium caudatum[J]. FEMS Microbiology Letters, 2005 , 247 (2) : 113 –121. DOI: 10.1016/j.femsle.2005.04.034 |
| [20] | WILLIAMS A G, COLEMAN G S. The rumen protozoa[M]. New York: Springer, 1992 : 135 -138. |




