青贮饲料是反刍动物营养和饲料十分重要的来源之一,尤其对冬季较长地区的反刍动物更为重要。而全株玉米青贮是反刍动物最重要的青贮饲料之一,在欧、美等发达地区早已广泛应用[1-2]。随着我国农业结构战略性调整和畜牧业的快速发展,大力推进全株青贮玉米的种植和利用微生物发酵技术青贮饲料,提高青贮饲料品质、增加畜牧生产性能、改善畜牧产品质量是非常重要的[3-4]。因此,在青贮过程中微生物的组成、变化及其对青贮饲料发酵品质的重要影响更是研究者关注的焦点。
多年来,青贮中微生物多样性及群落变化的研究常用的传统培养法,由于培养条件的局限性,传统培养法存在费时、费力、片面的缺陷,无法真正完全解析微生物的组成、丰度及其变化情况,低估了微生物的多样性[5]。近10多年来,随着分子生物学技术的不断发展,基于聚合酶链式反应(PCR)技术出现了许多非培养的方法,如16S rRNA克隆建库、限制性片段长度多态性(RFLP)、变性梯度凝胶电泳PCR(DGGE-PCR)等,这些方法被广泛应用于环境、发酵食品、青贮饲料及其他微生态环境中微生物多样性的研究[6-8]。基于微生物宏基因组学的高通量焦磷酸测序技术具有高通量、快速、省力等优点,已逐渐被广泛应用于土壤、肠道、水体、发酵食品等各微生态环境中微生物多样性的检测和研究[9-11]。然而,高通量焦磷酸测序技术在青贮发酵饲料方面应用的研究方面报道较少。Li等[12]利用高通量测序技术在青贮过程中检测到30多个菌属群落结构的变化情况。刘晶晶[13]应用Miseq高通量测序技术在柳枝稷青贮60 d时检测微生物多样性。陶莲等[14]通过实验室检测手段和Miseq高通量测序技术相结合,分析了青贮饲料品质和青贮前后整个菌落构成及丰度变化的信息,进而为发酵过程的认清微生物组成提供了依据。Bao等[15]利用第3代单分子测序技术(SMRT)检测了苜蓿青贮前后微生物组成的变化及其对苜蓿品质的影响,表明SMRT测序平台可用于评估青贮饲料微生物组成变化和青贮饲料品质。然而,采用高通量测序技术检测和分析全株玉米青贮过程中微生物多样性还未有报道。因此,本试验利用宏基因组学技术,研究和分析全株玉米青贮发酵过程中和发酵后暴露空气后菌群构成及其演替规律,为全面了解全株玉米自然青贮发酵过程中微生物组成及变化、发掘全株玉米附生有益微生物种类、提高青贮饲料营养价值和品质提供理论基础和新方法。
1 材料与方法 1.1 试验材料及样品采集全株玉米来源于内蒙古通辽市查金台第一农场,取自机械化收割后的样品(收割后的全株玉米由机器自动切割成长度为1~2 cm的小段)。青贮试验于2016年9月在内蒙古民族大学动物科学技术学院实验室进行。将样品装入双层聚乙烯袋(45 cm×30 cm)中,排空气、压实、封口,每袋约3.0 kg,压实密度约550 kg/m3,将青贮发酵袋装于塑料贮存箱内室温发酵。分别在青贮第5天(F5组)、青贮第40天(F40组)和青贮40 d开袋暴露空气后第3天(A3组)进行取样,每次取3袋作为重复,共9袋,取样时打开袋,上层弃用,取袋中间位置的样品,一部分装入无菌50 mL冻存离心管中,于-80 ℃超低温冰箱保存,用于微生物多样性检测;另一部分(约60 g)装入封口袋中,保存于-20 ℃冰箱,用于青贮发酵品质和营养成分的测定。
1.2 全株玉米青贮发酵品质和营养成分的测定取全株玉米青贮3个阶段点样品各20 g,加入180 mL蒸馏水,搅拌均匀,用组织捣碎机搅碎1 min,先后用4层纱布和定性滤纸过滤,滤出草渣得到浸出液,将其分为4份,分别采用pH计、苯酚-次氯酸钠比色法、对羟基联苯比色法和中和滴定法测定pH及氨态氮(AN)[14]、乙酸和乳酸[16]含量。参考文献[17]的描述,分别采用烘箱干燥法、凯氏定氮法、范氏洗涤纤维法测定全株玉米青贮的干物质(DM)、总氮(TN)、粗蛋白质(CP)、中性洗涤纤维(NDF)和酸性洗涤纤维(ADF)含量;采用蒽酮分光光度法[18]测定全株玉米青贮的水溶性碳水化合物(WSC)含量。
1.3 青贮饲料微生物宏基因组测序方法 1.3.1 细菌基因组DNA的提取将以上每次取的3个重复样品混合在一起,按照FastDNA® SPIN Kit for Soil (MP Biomedicals,Santa Ana,CA,美国)试剂盒的步骤说明,抽提青贮饲料中的细菌基因组DNA。对抽提到的基因组DNA采用紫外分光光度计进行浓度和纯度检测,并采用0.8%琼脂糖凝胶电泳进行DNA完整性检测。
1.3.2 16S rDNA的扩增使用细菌16S rRNA基因V3~V4区引物338F(ACTCCTACGGGAGGCAGCA)及806R(GGACTACHVGGGTWTCTAAT)[19],对以上获得DNA模板进行序列扩增。
PCR反应体系为20 μL,包括4.0 μL的5×FastPfu Buffer,2.0 μL的dNTPs(dATP、dTTP、dGTP和dCTP各2.5 mmol/L),0.4 μL的FastPfu DNA Polymerase,10 ng模板DNA,0.8 μL正向引物338F(5 μmol/L),0.8 μL的反向引物806R(5 μmol/L),0.2 μL的胎牛血清(BSA)(0.1%),由灭菌ddH2O补至20 μL。95 ℃预变性3 min,95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸45 s,反应循环数为27,最后72 ℃延伸10 min。
1.3.3 测序使用2%琼脂糖凝胶电泳检测PCR产物,如果产物浓度合适,目的条带大小正确,则进行上机测序。测序系统采用Illumina Miseq测序平台(Illumina Corporation,San Diego,美国),测序公司为上海美吉生物医药科技有限责任公司。
1.3.4 生物信息学和数据分析 1.3.4.1 原始双端序列的拼接与质控这一过程主要涉及杂质reads的去除及双端序列的拼接,所用软件为FLASH及Trimmomatic。首先对reads尾部50 bp窗口内平均质量值低于20的窗口碱基进行截除,之后对50 bp以下的reads进行过滤,并去除含N碱基的reads。拼接主要根据测序所得的双端序列之间的重叠关系将成对reads拼接成一条,要求最小重叠数大于10 bp,碱基错配率低于0.2%。最后根据序列两端的barcode及引物区分样本得到有效序列,不允许barcode有错配。
1.3.4.2 序列过滤及操作分类单元(OTU)聚类使用Usearch(vsesion 7.0 http://drive5.com/uparse/)软件平台进行序列过滤及OTU聚类。对1.3.3获得的有效序列进行非重复序列的冗余分析,去除没有重复的单序列,获得优化序列。按照序列97%相似性的原则对优化序列进行OTU聚类,过程中去除嵌合体,生成OTU代表序列,并与优化序列比对产生OTU表格。
1.3.4.3 生物信息学分析物种分类学采用RDP classifier贝叶斯算法(version 2.2,http://sourcef orge.net/projects/rdp-classifier/)及Qiime平台(http://qiime.org/scripts/assign_taxonomy.html)进行分析,设0.7为置信度阈值。将OTU代表序列与Silva数据库(Release128,http://www.arb-silva.de)进行BLAST比对,获得OTU代表序列的分类学信息,分别在各分类学水平上分析各样本的群落组成。
采用mothur软件平台(version v.1.30.1,http://www.mothur.org/wiki/Schloss_SOP#Alpha_diversity)进行稀释曲线及Alpha多样性分析。Venn图、Heatmap和主成分分析(PCA)均采用R语言相关软件包进行分析及作图。
2 结果 2.1 全株玉米青贮期间和暴露空气后发酵品质及营养成分变化由表 1可知,相比青贮第5天,全株玉米青贮第40天和青贮40 d后开袋第3天,氨态氮/总氮、乳酸和乙酸含量均有显著增加(P < 0.05),但pH有相反趋势,出现显著降低(P < 0.05),且青贮第40天和青贮40 d后开袋第3天之间差异不显著(P>0.05)。全株玉米青贮前、青贮第5天、青贮第40天、青贮40 d后开袋第3天营养成分变化结果(表 1)表明,随着青贮时间的延长,全株玉米DM、CP、NDF、ADF和WSC含量均有减少的趋势,开袋后第3天虽略有升高,但与青贮40 d后差异不显著(P>0.05),其中WSC含量下降的最明显,与青贮前相比,青贮第5天、青贮第40天、青贮40 d后开袋第3天比青贮前分别下降了38.60%、68.17%和65.86%,差异均达到显著水平(P < 0.05)。
|
|
表 1 全株玉米青贮期间和暴露空气后的发酵品质及营养成分 Table 1 Fermentation quality and nutritional components of whole-plant maize during ensilage and after air exposure |
通过序列过滤、抽取及双端拼接,3组样本共获得了122 371条有效序列,总碱基数54 758 721个,平均长度447 bp,经过对有效序列的优化处理,得到共110 173条优化序列用于OTU聚类及分类学分析,有效优化序列数目达90.03%(表 2)。
|
|
表 2 不同样本序列信息 Table 2 Sequence information of different samples |
经过序列相似性大于97%水平上的OTU聚类,共得到的OTU的数目为239,单个样本中序列丰度大于1%的OUT的数目却很少,分别是12(F5组)、8(F40组)和10(A3组)。3个样本的覆盖度均达到了0.99以上,稀释曲线及Shannon曲线(图 1)均显示曲线已经趋于平坦,表明样本测序量已经饱和,足够反映样本中绝大部分细菌物种的信息。有效序列及优化序列数随青贮时间延长而升高,同时OTU数目也在增加,说明青贮期间及开袋暴露空气后菌群持续增殖,多样性也在增加。但样本中序列丰度大于1%的OTU的数目不仅减少,还出现青贮后期比青贮前期的低的情况,说明青贮对于主体菌群来说多样性是降低的。
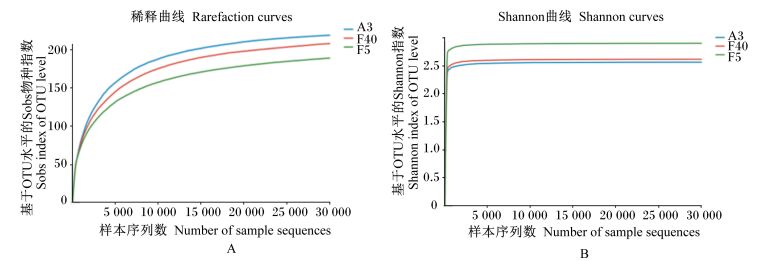
|
图 1 全株玉米青贮和暴露空气样本稀释曲线(A)及Shannon曲线(B) Figure 1 Rarefaction curves (A) and Shannon curves (B) of samples from whole-plant maize during ensilage and after air exposure |
由图 2的主成分分析可知,A3、F40和F5组三者彼此距离互不接近,形成了一个三角形,表明在三者在微生物组成上有很大的差别,三者分别代表了不同的发酵时期,即发酵初期(F5组)、发酵后期(F40组)及开袋期(A3组)。F40组分别与A3及F5组距离较近,说明三者相比,发酵后期分别与发酵初期及开袋期微生物群落构成相似性较大,也表明发酵初期、发酵后期和开袋期三者之间的承继关系。
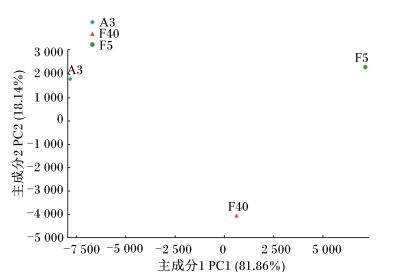
|
图 2 全株玉米青贮期间和暴露空气后微生物群落主成分分析(OUT水平) Figure 2 Principal component analysis of microbial community from whole-plant maize during ensilage and after air exposure (OUT level) |
经过序列相似性大于97%水平上的OTU聚类,共获得了239个OTU,分属163个属,93个科,54个目,26个纲,16个门。所有样本的微生物群落可分为16个门,如图 3所示,丰度大于1%的门有5个,依次为厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、蓝细菌门(Cyanobacteria)和放线菌门(Actinobacteria),它们占了极大的比例。发酵初期(F5组)中主要优势菌群为厚壁菌门和变形菌门,分别占到57.57%和27.54%,其次是蓝细菌门(7.05%)、拟杆菌门(6.49%)和放线菌门(1.08%),它们的总丰度达到99.73%。发酵后期(F40组)中主要有3个门的丰度在1%以上,它们是厚壁菌门、变形菌门和拟杆菌门,分别占到74.65%、20.36%和3.10%,这3个门菌群的总丰度达到98.10%。开袋期(A3组)中主要优势菌群为厚壁菌门和变形菌门,丰度分别为78.82%和17.02%,其次是拟杆菌门和放线菌门,丰度分别是2.17%和1.20%,这4个门菌落的总丰度达到99.21%。
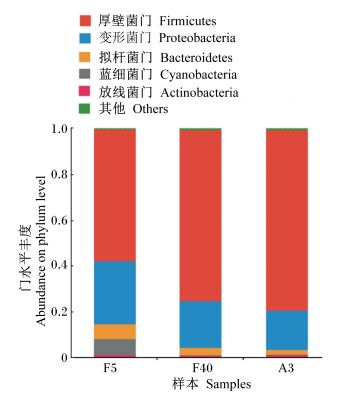
|
图 3 基于门水平的全株玉米青贮期间和暴露空气后微生物群落结构 Figure 3 Microbial community structure of whole-plant maize during ensilage and after air exposure on the phylum level |
比较不同发酵时期的3个样本可知,随着青贮的进行及后续开袋暴露空气,厚壁菌门获得了持续增长,变形菌门和拟杆菌门所占比例持续降低,蓝细菌门只在F5组中出现,放线菌门只在发酵初期和开袋期丰度高于1%。
2.5 基于属水平的微生物群落结构分析图 4显示了在属水平上全株玉米青贮期间及暴露空气后微生物群落结构的变化(丰度>1%),3个样本共鉴定出163个属。
发酵初期(F5组)鉴定出133个属,丰度>1%的菌属有11个(含蓝细菌门),总丰度占到86.33%,其中乳杆菌属(Lactobacillus)占优势地位,丰度达49.78%,随后依次为克雷伯氏菌属(Klebsiella)(9.08%)、蓝细菌门(7.05%)、拉恩氏菌属(Rahnella)(3.45%)、金黄杆菌属(Chryseobacterium)(3.14%)、片球菌属(Pediococcus)(3.18%)、乳球菌属(Lactococcus)(2.67%)、肠杆菌属(Enterobacter)(2.52%)、鞘氨醇单胞菌属(Sphingomonas)(2.34%)、泛菌属(Pantoea)(1.78%)、鞘氨醇杆菌属(Sphingobacterium)(1.33%),可见其他的菌属虽然丰度不大,但具有一定的数量。
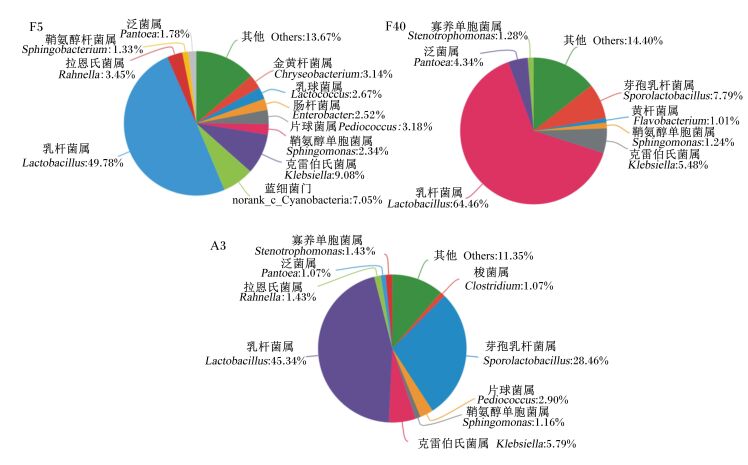
|
图 4 基于属水平的全株玉米青贮期间和暴露空气后微生物群落结构 Figure 4 Microbial community structure of whole-plant maize during ensilage and after air exposure on the genus level |
发酵后期(F40组)鉴定出146个属,丰度>1%的菌属有7个,总丰度占到85.60%,其中乳杆菌属占优势地位,丰度达64.46%,随后依次为芽孢乳杆菌属(Sporolactobacillus)(7.79%)、克雷伯氏菌属(5.48%)、泛菌属(4.34%)、寡养单胞菌属(Stenotrophomonas)(1.28%)、鞘氨醇单胞菌属(1.24%)、黄杆菌属(Flavobacterium)(1.01%)。
开袋期(A3组)鉴定出153个属,丰度>1%的属有9个,总丰度占到88.65%,主要种类有乳杆菌属(45.34%)、芽孢乳杆菌属(28.46%)、克雷伯氏菌属(5.79%)、片球菌属(2.90%)、寡养单胞菌属(1.43%)、拉恩氏菌属(1.43%)、鞘氨醇单胞菌属(1.16%)、泛菌属(1.07%)、梭菌属(Clostridium)(1.07%)。
发酵初期、发酵后期、开袋期乳杆菌属的丰度分别是49.78%、64.46%、45.34%,占绝对优势,在青贮期间丰度呈增长趋势,在青贮40 d达到峰值,在开袋暴露空气后丰度有所下降,而芽胞乳杆菌属,由发酵初期未出现,到发酵后期的7.79%,再到开袋暴露空气3 d后增长到28.46%,说明暴露空气有利于其生长。其他一些菌属则相对稳定而又连续存在,其中克雷伯氏菌属丰度相对较大,发酵初期、发酵后期和开袋期的丰度分别是9.08%、5.48%和5.79%,梭菌属在发酵期间检测不到而在开袋暴露空气3 d后增长到1.07%。此外,丰度相对较小的菌属还包括泛菌属、鞘氨醇单胞菌属等。因此,发酵产生的优势菌群是乳杆菌属,有利于青贮饲料品质的提升,而暴露空气则改变了菌群的菌属结构,滋生了有害菌群。
2.6 微生物群落的Alpha多样性分析表 3为全株玉米青贮期间和暴露空气后的Alpha多样性指数。Alpha多样性用来表示样本内微生物群落的多样性,其中Chao、Ace指数是计算菌群丰度的指数,数值越大,表示菌群丰度越高;而Simpson指数和Shannon指数用来计算菌群多样性的指数,Shannon指数越大,群落多样性越低,Simpson指数越大,群落多样性越高。
|
|
表 3 不同样本的Alpha多样性指数 Table 3 Alpha diversity metrics of different samples |
Alpha多样性分析表明,随着青贮的进行,菌群数量及组成呈现一定的变化。总体来说,3个时期的样本序列数量较大,OTU数目、Chao指数、Ace指数等指标上均处在较高的水平,说明样本菌群数量较大,多样性水平也相对较高。相比发酵前期,发酵后期及开袋期序列数量、OTU数目、Chao指数、Ace指数等指标均有增长,而Shannon值有所降低,说明发酵促进了微生物群落的繁殖生长,并且使很多菌群物种得到了发展,因此微生物多样性得到了提高,菌群丰度也相应提高。
对于短期开袋暴露空气而言,其相对于发酵后期的序列数量、OTU数目、Chao指数、Ace指数等指标的增幅均很小,Shannon指数的降幅也很小,表明短期开袋暴露空气后,在新的气体及温度环境下,菌群生长及物种多样性并没有产生太大变化。
2.7 微生物群落的Venn图和Heatmap分析由Venn图(图 5-A)可见,3个样本的239个OTU中,有173个核心序列,占比为73.49%,说明青贮期间和暴露空气后核心序列占比很大;发酵前期和发酵后期2个样本的223个OTU中,有180个核心序列,占比为80.72%,占比有所增加;发酵后期和开袋期2个样本的238,有198个核心序列,占比为83.19%。F5、F40和A3组的独有物种均很少,分别为1、9和16种。F5组与F40、A3组共有物种较少,其值分别为7和8;而F40组与A3组共有物种较多,为25,表明这2个时期在物种组成上更为接近。总之,Venn图分析显示F40组与A3组在微生物群落组成上更为接近,这说明开袋暴露空气3 d虽然改变了微生物群落组成,但仍然与发酵后期保持了较大的相似性,在一定程度上保证了青贮饲料的发酵品质。
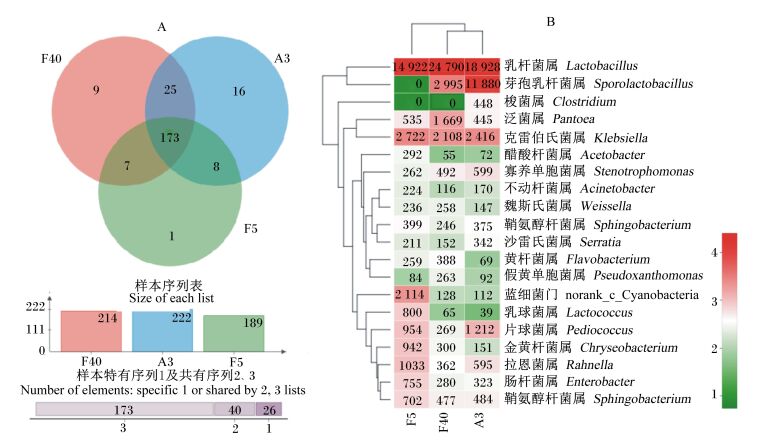
|
图 5 全株玉米青贮期间和暴露空气后微生物群落的Venn图(A)和Heatmap分析(B) Figure 5 Venn map (A) and Heatmap analysis (B) of microbial community of whole-plant maize during ensilage and after air exposure |
微生物群落的Heatmap(图 5-B)显示了在属水平上排名前20的物种丰度,图中不同色块的颜色代表了一个样品中对应一个菌属的丰度,色块内的数值为相应菌属含有的序列数。乳杆菌属在3个青贮时期内均保持了很深的色块深度,即丰度均很大。另外,克雷白氏杆菌属也保持了较好的连续性和较高的丰度,而芽孢乳杆菌属在发酵后期和开袋期色块颜色加深,尤其是在开袋期。梭菌属在开袋期突然出现,丰度也较高。由Heatmap还可以看出,大部分菌属在3个样本中均具有较好的连续性,仅少数菌属出现了中断(色块显示为绿色),表明发酵过程及发酵后开袋暴露空气3 d时乳酸菌等有益菌种类始终保持了优势地位,但也有个别菌属在丰度上随着发酵时间的持续及开袋后培养条件的改变出现了此消彼长变化。
3 讨论全株玉米青贮与玉米秸秆青贮相比,营养价值高、适口性好、消化率高、能值高,可长期保存和四季均衡供应,是解决牛、羊等反刍动物所需青粗饲料的最有效途径。本试验从全株玉米青贮第5天、第40天和青贮40 d后开袋第3天每次取样时观察发现感官品质良好,色泽分别呈黄绿色、黄褐色和黄褐色,无霉变和粘手现象,有酒酸香味和无臭味。从全株玉米青贮的发酵品质来看,青贮发酵40 d后和青贮发酵40 d后短期开袋时氨态氮/总氮均低于9%,pH均低于4.00,乳酸含量高于9%,乙酸含量低于3%,与Zhou等[20]报道在20或25 ℃青贮发酵条件下全株玉米青贮的品质相似。综合考虑上述发酵品质指标,说明全株玉米青贮发酵40 d后具有较好的发酵品质。青贮发酵作用能够使全株玉米中NDF和ADF的含量下降,但也由于乳酸菌占主导的优势菌消耗了大量的可溶性糖类,使得WSC含量显著降低,这在本试验中也得到验证。因此,为进一步降低青贮全株玉米中NDF和ADF的含量,减少WSC消耗量,一些研究采取了在青贮饲料中添加糖类、纤维素酶或青贮添加剂等措施[21-23],以改善青贮饲料的营养价值。
许多欧美国家已将全株玉米青贮作为奶牛和肉牛的全混合日粮配方中不可或缺的一种粗饲料[24],全株玉米青贮发酵过程和发酵完成后微生物群落组成及其丰度变化都很大,影响着青贮饲料的营养价值和品质[20, 25]。Dolci等[26]研究表明,青贮饲料发酵品质的好坏很大程度上取决于发酵初期的优良发酵菌种的数量和该菌种在整体菌群中占的比例。本研究采用MiSeq高通量测序技术对全株玉米青贮期间和暴露空气后微生物群落结构进行分析,发现全株玉米青贮第5天,乳杆菌属迅速发展成为优势菌群,丰度达到49.78%,从而确立菌群发展的走势,其他菌株的繁殖受到抑制,随着发酵时间的延长,乳杆菌属进一步扩大其优势地位,在发酵后期,即青贮第40天时,其丰度达到了64.46%,拥有绝对优势地位。这也为青贮饲料发酵品质的提升提供了菌群保障,乳杆菌属作为一种产乳酸菌(LAB),能够产生乳酸,降低青贮饲料pH,抑制霉菌及其他有害杂菌的繁殖,增加青贮饲料的风味[27]。本研究中,全株玉米青贮发酵期间,乳杆菌属的丰度远大于陶莲等[14]报道的玉米秸秆青贮中乳杆菌属的丰度(9.73%),由于青贮过程中氧气的耗尽,假单胞菌属(Pseudomonas)及霉菌等有害的需氧菌受到抑制,需氧菌属的丰度始终低于1%。在本试验中,除丰度最高的乳杆菌属外,还存在数量较低的其他青贮饲料中常见的典型LAB菌种,如乳球菌属、片球菌属、肠球菌属(Enterococcus)和魏斯氏菌属(Weissella)等,但均未能成为优势菌群。
本研究表明,开袋暴露空气对全株玉米青贮微生物群落组成影响较大,开袋3 d后,乳杆菌属丰度下降严重,由发酵后期的64.46%降为45.34%,这是由于开袋后氧气含量增加,抑制了乳酸菌的繁殖生长,而一些好氧菌或兼性好氧菌得到了发展,如芽孢乳杆菌属,由青贮发酵后期的7.79%上升到了28.46%。有关芽孢乳杆菌属在青贮饲料中的报道的较少。Smoker[28]发现在实验室自然青贮窖条件下芽孢乳杆菌属可利用不同碳水化合物发酵产生乳酸,可作为青贮发酵剂。Kharaziana等[29]研究发现在全株玉米青贮中添加土芽孢乳杆菌(Sporolactobacillus terrae)抑制一些植物致病真菌具有一定的作用。刘磊等[30]在肉仔鸡饲粮中添加100 mg/kg的芽孢乳杆菌能促进肠道发育,提高消化吸收功能,抑制其空肠内的大肠杆菌繁殖。但芽孢乳杆菌作为青贮发酵剂,对青贮饲料微生物菌群、发酵品质和反刍动物健康的作用机制还有待进一步研究。
全株玉米青贮发酵过程对隔绝空气的要求较为严格,如果空气泄露,会导致霉菌滋生,造成青贮饲料的霉变,饲料中养分被降解,产生有害霉菌毒素,危及牲畜安全[31-32]。Vissers等[33]报道,革兰氏阳性芽孢杆菌属(Bacillus)是青贮饲料中主要的腐败菌,主要降解蛋白质和氨基酸,在好氧条件下对青贮变质起重要作用,也是青贮开窖后短期内迅速发展起来的有害微生物,但在本研究中不论是青贮期间还是开袋后第3天均未出现芽孢杆菌属。但值得关注的是有害菌梭菌属,在开袋第3天其丰度达到1.07%,可能对青贮品质产生不利影响。McDonald等[34]在发酵品质较差的青贮饲料中发现有梭菌属,其在厌氧或微需氧条件下能分解糖、有机酸和蛋白质,是青贮有害微生物,这与本研究开袋暴露空气后出现梭菌属的结果一致。
本试验结果表明,克雷伯氏菌属在3个采样期内都保持了较高的丰度,对全株玉米青贮可能具有重要的作用,但目前有关这方面的报道较少,其具体作用机制及产生的作用还需要进一步研究。泛菌属、鞘氨醇单胞菌属等菌属丰度虽低,但在3个采样期中均存在,表明了它们在玉米青贮过程中持续起作用。肠杆菌属只在发酵初期出现,而寡养单胞菌属在发酵后期及开袋期出现,拉恩菌属在发酵前期及开袋期出现,说明青贮发酵及开袋暴露空气影响了某些菌群的发生。陶莲等[14]和Dunière[35]均报道了青贮玉米中寡养单胞菌及鞘氨醇单胞菌属的存在,同本研究结果一致,说明这2种菌属在青贮玉米中是普遍存在的。
4 结论① 全株玉米经过40 d青贮发酵可使pH及NDF、ADF含量显著下降,乳酸含量显著升高,具有良好的发酵品质和营养价值,且发酵40 d后短时间开袋暴露空气对其发酵品质和营养价值无显著影响。
② 主成分分析显示3个发酵时期样本的微生物多样性差异很大,分属3个不同的群体,表明全株玉米青贮在发酵前期、发酵后期及开袋期微生物群落组成变化明显。
③ 在3个发酵时期的样本中,共鉴定出16个门,163个属的细菌。基于门的水平,厚壁菌门和变形菌门始终占优势地位。基于属的水平,发酵前期优势菌群主要是乳杆菌属、克雷伯氏菌属和蓝细菌属,发酵后期和短时间开袋暴露空气后优势菌群均为乳杆菌属、芽孢乳杆菌属和克雷伯氏菌属,但相比发酵前期和发酵后期,短时间开袋暴露空气后出现了梭菌属。
| [1] |
BERTHIAUME R, MANDELL I, FAUCITANO L, et al. Comparison of alternative beef production systems based on forage finishing or grain-forage diets with and without promotants:1.Feedlot performance, carcass quality, and production costs[J]. Journal of Animal Science, 2006, 84(8): 2168-2177. DOI:10.2527/jas.2005-328 |
| [2] |
HATEW B, BANNINK A, VAN LAAR H, et al. Increasing harvest maturity of whole-plant corn silage reduces methane emission of lactating dairy cows[J]. Journal of Dairy Science, 2016, 99(1): 354-368. DOI:10.3168/jds.2015-10047 |
| [3] |
ELLIS J L, BANNINK I K, HINDRICHSEN R D, et al. The effect of lactic acid bacteria included as a probiotic or silage inoculant on in vitro rumen digestibility, total gas and methane production[J]. Animal Feed Science and Technology, 2016, 211: 61-74. DOI:10.1016/j.anifeedsci.2015.10.016 |
| [4] |
CARRILLO J A, HE Y H, LI Y K, et al. Integrated metabolomic and transcriptome analyses reveal finishing forage affects metabolic pathways related to beef quality and animal welfare[J]. Scientific Reports, 2016, 6: 25948. DOI:10.1038/srep25948 |
| [5] |
LIN C J, BOLSEN K K, BRENT B E, et al. Epiphytic lactic acid bacteria succession during the pre-ensiling and ensiling periods of alfalfa and maize[J]. Journal of Applied Microbiology, 2010, 73(5): 375-387. |
| [6] |
MUYZER G, DE WAAL E C, UITTERLINDEN A G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction amplified gene encoding for 16S rRNA[J]. Applied and Environmental Microbiology, 1993, 59(3): 695-700. |
| [7] |
韩吉雨, 侯先志, 杨凯, 等. PCR-DGGE方法分析内蒙古不同地区青贮玉米中乳酸菌群多样性[J]. 动物营养学报, 2009, 21(6): 974-981. |
| [8] |
王红梅, 孙启忠, 屠焰, 等. 呼伦贝尔草原野生牧草青贮中优良乳酸菌的分离及鉴定[J]. 草业学报, 2016, 25(8): 189-196. DOI:10.11686/cyxb2016092 |
| [9] |
CAPORASO J G, LAUBER C L, WALTERS W A, et al. Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms[J]. The ISME Journal, 2012, 6(8): 1621-1624. DOI:10.1038/ismej.2012.8 |
| [10] |
张和平, 于洁. 乳酸菌基因组学研究新进展[J]. 中国食品学报, 2016, 16(2): 1-8. |
| [11] |
JIANG Y, OGUNADE I M, QI S, et al. Effects of the dose and viability of Saccharomyces cerevisiae.1.Diversity of ruminal microbes as analyzed by Illumina MiSeq sequencing and quantitative PCR[J]. Journal of Dairy Science, 2017, 100(1): 325-342. DOI:10.3168/jds.2016-11263 |
| [12] |
LI L H, SUN Y M, YUAN Z H, et al. Effect of microalgae supplementation on the silage quality and anaerobic digestion performance of Manyflower silvergrass[J]. Bioresource Technology, 2015, 189: 334-340. DOI:10.1016/j.biortech.2015.04.029 |
| [13] |
刘晶晶. 生物添加剂对柳枝稷青贮的作用及机理研究[D]. 博士学位论文. 北京: 中国农业大学, 2015. http://cdmd.cnki.com.cn/Article/CDMD-10019-1015564036.htm
|
| [14] |
陶莲, 刁其玉. 青贮发酵对玉米秸秆品质及菌群构成的影响[J]. 动物营养学报, 2016, 28(1): 198-207. |
| [15] |
BAO W C, MI Z H, XU H Y, et al. Assessing quality of Medicago sativa silage by monitoring bacterial composition with single molecule, real-time sequencing technology and various physiological parameters[J]. Scientific Reports, 2016, 6: 28358. DOI:10.1038/srep28358 |
| [16] |
万楚筠, 钮琰星, 黄凤洪, 等. 对羟基联苯比色法测定乳酸显色反应条件的研究[J]. 食品工业科技, 2013, 34(7): 322-324, 353. |
| [17] |
张丽英. 饲料分析及饲料质量检测技术[M]. 2版. 北京: 中国农业大学出版社, 2003, 53-74.
|
| [18] |
OWENS V N, ALBRECHT K A, MUCK R E, et al. Protein degradation and fermentation characteristics of red clover and alfalfa silage harvested with varying levels of total nonstructural carbohydrates[J]. Crop Science, 1999, 39(6): 1873-1880. DOI:10.2135/cropsci1999.3961873x |
| [19] |
DENNIS K L, WANG Y W, BLATNER N R, et al. Adenomatous polyps are driven by microbe-instigated focal inflammation and are controlled by IL-10-producing T cells[J]. Cancer Research, 2013, 73(19): 5905-5913. DOI:10.1158/0008-5472.CAN-13-1511 |
| [20] |
ZHOU Y, DROUIN P, LAFRENIÈRE C. Effect of temperature (5-25℃) on epiphytic lactic acid bacteria populations and fermentation of whole-plant corn silage[J]. Journal of Applied Microbiology, 2016, 121(3): 657-671. DOI:10.1111/jam.2016.121.issue-3 |
| [21] |
兴丽, 韩鲁佳, 刘贤, 等. 乳酸菌和纤维素酶对全株玉米青贮发酵品质和微生物菌落的影响[J]. 中国农业大学学报, 2004, 9(5): 38-41. |
| [22] |
许庆方, 张翔, 崔志文, 等. 不同添加剂对全株玉米青贮品质的影响[J]. 草地学报, 2009, 17(2): 157-161. DOI:10.11733/j.issn.1007-0435.2009.02.005 |
| [23] |
SANTOS A O, ÁVILA C L S, SCHWAN R F. Selection of tropical lactic acid bacteria for enhancing the quality of maize silage[J]. Journal of Dairy Science, 2013, 96(12): 7777-7789. DOI:10.3168/jds.2013-6782 |
| [24] |
MUCK R E. Recent advances in silage microbiology[J]. Agriculture Food Science, 2013, 22(1): 3-15. |
| [25] |
RANJIT N K, TAYLOR C C, KUNG L, Jr. Effect of Lactobacillus buchneri 40788 on the fermentation, aerobic stability and nutritive value of maize silage[J]. Grass Forage Science, 2002, 57(2): 73-81. DOI:10.1046/j.1365-2494.2002.00304.x |
| [26] |
DOLCI P, TABACCO E, COCOLIN L, et al. Microbial dynamics during aerobic exposure of corn silage stored under oxygen barrier or polyethylene films[J]. Applied and Environmental Microbiology, 2011, 77(21): 7499-7507. DOI:10.1128/AEM.05050-11 |
| [27] |
TOHNO M, KOBAYASHI H, NOMURA M, et al. Identification and characterization of lactic acid bacteria isolated from mixed pasture of timothy and orchardgrass, and its badly preserved silage[J]. Journal of Animal Science, 2002, 83(4): 318-330. |
| [28] |
SMOKER A N.Screening of Sporolactobacillus and Bacillus Strains for Use in Silage Inoculation[D].Master Thesis.Corvallis:Oregon State University, 1999.
|
| [29] |
KHARAZIANA Z A, JOUZANIA G S, AGHDAS M, et al. Biocontrol potential of Lactobacillus strains isolated from corn silages against some plant pathogenic fungi[J]. Biological Control, 2017, 110(1): 33-43. |
| [30] |
刘磊, 朱立贤. 芽孢乳杆菌对肉仔鸡生产性能、肠道发育和微生物菌群的影响[J]. 动物营养学报, 2011, 23(12): 2136-2142. DOI:10.3969/j.issn.1006-267x.2011.12.014 |
| [31] |
DRIEHUIS F, ELFERINK S J W H O. The impact of the quality of silage on animal health and food safety:a review[J]. Veterinary Quarterly, 2000, 22(4): 212-216. DOI:10.1080/01652176.2000.9695061 |
| [32] |
WAMBACQ E, VANHOUTTE I, AUDENAERT K, et al. Occurrence, prevention and remediation of toxigenic fungi and mycotoxins in silage:a review[J]. Journal of the Science of Food and Agriculture, 2016, 96(7): 2284-2302. DOI:10.1002/jsfa.2016.96.issue-7 |
| [33] |
VISSERS M M M, TE GIFFEL M C, DRIEHUIS F, et al. Minimizing the level of Bacillus cereus spores in farm tank milk[J]. Journal of Dairy Science, 2007, 90(7): 3286-3293. DOI:10.3168/jds.2006-873 |
| [34] |
MCDONALD P, HENDERSON A R, HERON S J E. The biochemistry of silage[M]. 2nd ed. Marlow: Chalcombe Publications, 1991, 81-151.
|
| [35] |
DUNIÈRE L, SINDOU J, CHAUCHEYRAS-DURAND F, et al. Silage processing and strategies to prevent persistence of undesirable microorganisms[J]. Animal Feed Science and Technology, 2013, 182(1/2/3/4): 1-15. |




