固有免疫(innate immunity)是指机体出生就具有的非特异性天然免疫防御功能,也称为非特异性免疫(nonspecific immunity)。固有免疫是机体免疫系统识别自身和异己物质抵抗微生物感染的第1道防线。宿主机体对侵袭微生物感染做出了广泛的免疫应答反应来阻挡、限制它的感染与复制,甚至从体内清除,而这种应答的前提是固有免疫系统的模式识别受体(pattern recognition receptor,PRRs)对病原相关分子模式(pathogen associated molecule patterns,PAMPs)进行识别,发生受体配体反应,从而诱导产生白细胞介素(interleukin, IL)、肿瘤坏死因子(tumor necrosis factor, TNF)和干扰素(interferon, IFN)等一系列相关的细胞因子来介导机体的抗病原效应。Janeway[1]首先提出了PPRs的模式识别作用,并明确指出了固有免疫的重要生物学意义。PPRs表达于抗原递呈细胞上,特别是树突状细胞(DCs),导致相关效应信号激活适应性免疫反应。目前,一些PRRs的类别已经能够被确定并有相应的特征,包括Toll样受体(Toll-like receptors, TLRs)、核苷酸寡聚结合域样受体(nucleotide-binding oligomerization domain-like receptors, NLRs)、视黄酸诱导基因1样受体(RIG-1-like receptors, RLRs)、C型凝集素受体(C-type lectin receptors, CLRs)和黑素瘤缺乏因子-2样受体(absent in melanoma-2-like receptors, ALRs)。
1 PRRsPPRs识别靶点微生物的结构种类复杂,主要包括多糖、糖脂、脂蛋白、核苷酸和核酸等。PPRs通过特异的配体识别域——富含亮氨酸重复单元、C型凝集素域和多种核酸绑定域,来探测微生物靶点结构(表 1)[2]。除了具有自身的结构和特异性之外,PPRs具有组织特异性表达和定位于不同的细胞区室,如浆膜、核内小体、溶酶体和细胞质[3]。这些模式表达的差异与固有免疫系统和免疫应答2种常规的识别模式有关:细胞内识别由感染细胞胞质感应器介导起作用,并且所有类型的细胞在给定的一类病原体侵染下能够表达相应的感应器,如病毒核酸感受器。细胞外识别不需要感染细胞表达PPRs,而是表达于特定的病原监测细胞表面,如上皮细胞和髓样细胞[4]。
|
|
表 1 模式识别受体与病原相关分子模式 Table 1 PRRs and PAMPs |
开展TLRs研究最初涉及到的分子是白介素1受体(IL-1R)。IL-1是一种多效性的促炎症因子,在20世纪80年代也被报道其参与T细胞的活化、产热原性、促进软骨组织的分解和激活急性期反应[5]。1988年,基因编辑IL-1R克隆,由于细胞质内区域没有可识别基序并没有找到信号转导机制。1991年,发现这种结构域与黑腹果蝇胞浆域蛋白Toll同源[6]。Toll决定着黑腹果蝇胚胎背腹侧轴线的发育。随后研究发现,高度相似的Toll和IL-1R共享核转录因子-κΒ(NF-κΒ)信号所必需的氨基酸序列,并与抗菌肽的生成相关,从而Toll样受体作为固有免疫受体研究揭开序幕[7]。
脂多糖(LPS)作为内毒素的重要组成部分而被广泛研究,这种物质被认为是革兰氏阴性病原体成分并引起细菌性脓毒症。Poltorak等[8]研究克隆了LPSd基因,这项工作也获得了其后的诺贝尔生理学奖,Qureshi等[9]也证实了LPSd实际上就是TLR4。Hoshino等[10]研究发现,TLR4基因被敲除小鼠对LPS没有应答,进一步证实了TLR4是LPS刺激信号受体。随着TLR4参与LPS的识别,一些其他的微生物成分也被用于测试TLRs可能的配体。研究中发现了人的10个TLRs基因和小鼠的12个TLRs基因,并且利用基因敲除小鼠模型确定了多种TLRs所对应识别的特定配体分子[11]。TLR2能够识别细菌的脂肽结构,与TLR1或者TLR6形成异质二聚体分别识别三酰脂肽和二酰脂肽[12]。TLR5能够识别鞭毛虫的鞭毛蛋白[13],进一步的研究表明了TLR5能够调节细菌引起的肠道固有免疫反应和适应性免疫反应[14]。小鼠TLR11被发现能够检测到一种尿路感染菌的成分[15],并且与TLR12共同作用绑定鼠弓形虫鞭毛蛋白[16]。TLR13也被发现能够识别细菌rRNA[17]。TLRs识别配体具有多样性,可以来源于细菌和寄生虫,也可以来源于病毒。TLR3能够识别病毒双链RNA,介导激活NF-κΒ和Ⅰ型IFN信号途径[18]。TLR9被发现能够作为非甲基化的胞嘧啶-磷酸-鸟嘌呤基序(CpGDNA)的受体,后续研究发现血浆树突状细胞TLR9也能够识别孢疹病毒DNA[19]。TLR7能够感受激发抗病毒化学合成咪喹莫特,与TLR8均能识别病毒单链RNA[20]。因此,TLRs受体家族在感知危险信号如微生物侵染或组织损伤,能够启动固有免疫和炎症反应做出相应的抵抗。
Toll/IL-1受体同源区(Toll/IL-1-receptor homologous region,TIR)是TLRs信号传导的起始点,能够绑定结合IL-1R一起活化NF-κΒ信号途径。髓样分化因子(myeloid differentiation factor 88, MyD88) 是最早发现含有TIR结构域的TLRs街头分子,TIR结构域与含有TIR同源域受体相互作用参与MyD88信号传导。此外,MyD88含有一个死亡结构域(death domain, DD),经噬同作用募集含有DD的IL-1相关蛋白激酶(interleukin-1 receptor-associated kinases,IRAKs)。IRAKs与肿瘤坏死因子受体关联因子6(TNF receptor-associated factor, TRAF)6相互作用,与TAB-3、TAB-2和TAK-1结合形成复合物,激活丝裂原活化蛋白激酶激酶(MKK)或κB抑制蛋白激酶(IKK)α/IKKβ/IKKγ,引起丝裂原活化蛋白激酶(MAPK)的c-jun氨基末端激酶(JUN)和p38丝裂原激活蛋白激酶(p38) 途径以及NF-κΒ途径,从而诱导产生促炎症因子,另外在胞内体主要产生干扰素调节因子(interferon-regulatory factors, IRFs),诱导产生Ⅰ型IFN[21]。其次发现的TIR衔接蛋白是MyD88样衔接蛋白(MAL,即TIRAP)。Kawai等[22]利用LPS刺激MyD88基因敲除小鼠发现通过IRF3诱发了Ⅰ型IFN产生,并且延迟了TLR4下游信号NF-κΒ激活,表明非依赖型MyD88信号能够激活TLRs下游信号。然而,研究表明MAL并不能独立完成非依赖型MyD88信号传导,而是作为连接MyD88与TLR4或者TLR2的桥梁存在[23]。Oshiumi等[24]研究表明,TIR衔接蛋白诱导IFNβ(TRIF或TICAM1) 能够引起MyD88基因敲出小鼠产生TLR3通路,也说明了非依赖型MyD88信号通路的存在,以及TRIF作为第3类TIR衔接蛋白分子的作用。另外,研究发现在TLR4途径中存在TRIF与TLR4的连接分子Toll样受体相关分子(TRAM或TICAM2),在膜吞噬体TLR4信号通路中,TRAM连接TRIF与TLR4形成复合体将信号传递至TRAF3或TRAF6前体信号体产生IRF3或NF-κΒ,进而调控转录表达。随着研究的不断深入,目前TLRs也广泛应用于相关疾病治疗中,如TLR7的配体治疗生殖器疣,通过TLR2与TLR4通路疫苗制剂治疗肺结核,TLR7和TLR9抑制剂潜在治疗全身性红斑狼疮以及TLR2抑制抗体有效限制缺血再灌注损伤等。
1.2 参与固有免疫识别的NLRsNLRs家族蛋白是胞浆型PRRs,能够在胞质内探测到PAMPs以及一些内源性分子,在机体固有免疫发挥重要的功能。NLRs结构上由3个结构域组成,N端蛋白相互作用结构域如半胱天冬酶募集结构域(caspase activation and recruitment domain, CARD)、热蛋白结构域(pyrin domain, PYD)或BIR域(baculovirus IAP repeats domain),中间核苷酸寡聚结合域(NOD)核酸绑定和低聚化结构域以及C端富含亮氨酸重复单元结构域(LRR)。NLRs按照N端结构可以分为5个亚科单元,包括NLRA、NLRB、NLRC、NLRP和NLRX。目前报道已经发现人类NLRs基因23个和鼠科NLRs基因34个,但是大多数的NLRs生理学功能有待深入研究[25]。NLRs家族蛋白中最具代表性的是NOD1和NOD2,能够识别肽聚糖(PGN)的相关结构。其中,NOD1存在于革兰氏阴性菌和少数革兰氏阳性菌中,识别PGN的产物内消旋二氨基庚二酸(g-D-glutamyl-meso-diaminopimelic acid, iE-DAP);NOD2存在于所有革兰氏阳性菌和革兰氏阴性菌中,能够识别PGN的组成元件胞壁酰二肽(muramyl dipeptide, MDP)。
NOD1参与胞内多种病原体微生物的识别,如埃希氏菌属大肠杆菌、痢疾福氏杆菌、假单胞杆菌、衣原体类、流感嗜血杆菌和幽门螺杆菌等;NOD2参与肺炎链球杆菌和结核分歧杆菌的识别[26];单核细胞增生利斯特菌能够激活NOD1和NOD2识别。当NLRs的C端LRR识别PAMPs或者危险相关模式分子,通过构象重组引发NOD低聚化,使NLRs暴露N端效应结构域,通过同型相互作用,效应结构域CARD或PYD分别募集其相应效应分子,使分子之间相互接近而使其活化,从而参与激活多种信号通路。例如,NLR家族蛋白NOD1和NOD2与受体相互作用蛋白激酶(receptor interacting protein kinase, RIPK)2相互作用诱导NF-κΒ和MAPK信号。此外,NOD1和NOD2能够通过募集ATG16L1自噬相关蛋白到浆膜控制诱发自噬作用[27]。病毒侵染时,NOD2可以结合线粒体抗病毒信号蛋白(mitochondrial antiviral signalling protein, MAVS)诱导产生Ⅰ型IFNs[28]。NLR家族成员NLRP1、NLRP3和NLRC4可以组装大量蛋白复合物形成炎性小体,激活炎症性细胞凋亡蛋白酶(caspase-1) 产生白介素-1β(IL-1β)和IL-18。如NLRP3通过同型PYD作用募集配体蛋白凋亡相关的斑点样蛋白(apoptosis-associated speck-like protein containing CARD, ASC),ASC通过同型CARD作用绑定促凋亡蛋白酶caspase-1,导致caspase-1的激活并引起促炎症细胞因子IL-1β和IL-18产生,放大抗病毒固有免疫反应。为了维持NLRs家族信号介导的自我平衡,NLRX1和NLRC5蛋白起到了重要的调控作用,研究表明NLRX1和NLRC5可以抑制NF-κΒ和Ⅰ型IFNs介导的信号通路[29]。NLRs家族主要的生物学功能是增强机体免疫系统探测微生物感染的能力,介导产生IL-1β等促炎细胞因子,以此调节免疫应答和炎症反应。
1.3 参与固有免疫识别的RLRs病毒入侵细胞质在产生dsRNA的过程中被机体感应激活抗病毒信号通路。这种模式应答存在于所有免疫和非免疫细胞的细胞质中,除了上述某些TLRs家族蛋白而言,就是RLRs蛋白家族。RLRs具有3个成员存在于细胞质中识别病毒RNA,即视黄酸诱导基因1(retinoic acid inducible gene 1, RIG-1)、蛋白黑色素瘤分化相关分子-5(melanoma differentiation-associated gene-5, MDA5) 及遗传学和生理学实验室蛋白2(laboratory of genetics and physiology-2, LGP2)[30]。RIG-1作为RLRs典型的代表,N端为CARD,可以与其他同型CARD相互作用,中间为结合RNA解旋酶结构域以及C端抑制结构域(RD)绑定RNA。在细胞静息时,RIG-1处于自我抑制状态,当病毒侵染时,RNA绑定引发构象改变激活RIG-1配体解除自我抑制,促进CARD募集作用下游信号分子。MDA5同样包含1个CARD和解旋酶结构域,但是并不确定C端是否有RD功能。LGP2包含解旋酶结构域和RD,但是缺乏CARD。研究表明,LGP2能够负向调控病毒诱导的反应,LGP2通过RD绑定RIG-1干扰其构象缔结从而抑制免疫信号应答[31]。
在细胞质中,RIG-1主要识别多种ssRNA病毒,包括副黏病毒、甲型流感病毒、水疱性口炎病毒以及乙型脑炎病毒;MDA5参与其他RNA病毒的识别,如小核糖核酸病毒脑心肌炎病毒,同样也参与多聚肌苷酸(poly 1:C)的识别。病毒侵袭时,最初RIG-1和MDA5与MAVS相互作用,通过刺激干扰素基因刺激物(STING)和TANK结合激酶1(TBK1) 激活信号级联,导致Ⅰ型IFNs的表达;此外MAVS信号可以通过RIPK1激活活化蛋白转录因子1(AP-1),产生固有免疫应答发挥免疫作用[31]。由于这种对应的识别,可以研究通过小鼠RIG-1或MDA5基因敲除或双敲后特定病毒侵染来确定被识别的病毒RNA对应的配体结构[32]。RIG-1被体外转录的RNA激活,而MDA5通过合成的poly l:C激活,试验证实了RIG-1可以识别ssRNA的5-三磷酸部分。此外,RIG-1能够识别21~27个核苷酸dsRNA,MDA5则可以识别长链dsRNA。尽管LGP2作为负向调控者,但是LGP2基因敲除小鼠在不同类型的RNA病毒侵染时显现出不同的反应水平,poly l:C和水疱性口炎病毒侵染后提高了Ⅰ型IFN水平,脑心肌炎病毒侵染后降低了Ⅰ型IFN水平[33]。
1.4 参与固有免疫的其他受体目前CLRs是研究比较热门的一类PRRs,CLRs蛋白群能够识别许多不同的微生物。所有CLR都含有1个C型凝集素样结构域(CTLD),最初CTLD被认定为是一个双环结构域,能够绑定钙和糖类,后来才发现CTLD连接的蛋白能够绑定其他类型的配体,基于不同的结构CLRs蛋白家族可以被分为17个不同的亚群[34],树突状细胞相关性C-型凝集素(dendritic cell-associated C-type lectin, Dectin)-1和Dectin-2就是CLRs典型的代表。Dectin-1表达于DCs细胞、巨噬细胞、中性粒细胞以及单核细胞,属于跨膜蛋白包含1个细胞外不规则的CTLD结构域和胞内修饰的免疫受体酪氨酸激活基序(immunoreceptor tyrosine-based activation motif,ITAM)。Dectin-1识别的配体大多数是真菌β-1, 3-葡聚糖,一经配体绑定,Dectin-1通过吞噬作用促进配体摄取,并启动调节基因表达和细胞因子产生的信号级联反应,参与真菌侵染防御。此外,Dectin-1还可以识别分泌型免疫球蛋白A、黏蛋白以及其他微生物β-1, 3-葡聚糖[35]。另外Dectin-2连接ITAM轴承分子Fc受体γ(FcRγ)传导信号,含有的胞内部分没有信号传导功能。这也是Dectin-1与Dectin-2信号传导时ITAM用法的区别,下游信号传递基本一致。Dectin-2主要识别真菌细胞壁的α-甘露聚糖[36],以及一些曼氏血吸虫和结核分歧杆菌[35]。
ALRs是一类能够识别细胞内DNA的PRRs,含有1个蛋白相互作用的结构域包含热蛋白和HIN结构域蛋白(pyrin and HIN domain-containing protein, PYHIN)和1个DNA绑定结构域HIN200。机体探测到细胞内DNA侵染时,受体黑素瘤缺乏因子2(AIM2) 与配体ASC相互作用促进炎性小体的形成,启动固有免疫反应分泌促炎性细胞因子IL-1β和IL-18,快速诱发细胞凋亡显现发炎症状[37]。干扰素诱导蛋白16(IFI16) 作为ALRs蛋白家族的另一成员,能够识别单纯孢疹病毒(HSV)、巨细胞病毒、卡波氏肉瘤病毒(KSHV)和人类免疫缺陷病毒(HIV)。IFI16作为细胞质内的感受器,与其他DNA受体一样可以激活STING或者炎性小体。由于病毒DNA的复制循环出现在细胞核,因此受体的识别也会出现在细胞核。如HSV和KSHV的识别发生在细胞核,但是信号最终通过细胞质的炎性小体或STING激活,因此,启动孢疹病毒防御需要配体诱导的IFI16运动至细胞质信号场所[38]。IFI16作为胞质和核内外源DNA识别有着重要的作用。然而,IFI16在细胞核内如何区别自身与外源DNA的感应有待科学进一步探讨。
2 不同PRRs信号途径的相互影响TLRs、NLRs和RLRs在激活固有免疫防御侵染病原体的信号传导过程中存在着紧密的相互作用(图 1)。在产生炎症的过程中,PRRs在不同的物种、组织、细胞和细胞器中对PAMPs的识别有部分重叠和相互补偿发生。革兰氏阴性菌LPS对TLR4的激活至关重要,但是研究也发现胞质caspase-4/5/11能够识别LPS并引发固有免疫信号[39]。RLRs和TLRs途径存在着抑制的现象,经病毒刺激RLRs诱发的IRF3信号通过竞争性占有IL-12b启动子后会抑制经细菌刺激TLRs诱发的IRF5信号,这也在分子机制上解释了机体在病毒感染后的细菌性双重感染的原理[40]。TLR7小分子激动剂通过抑制信号转导和激活转录因子(p-STAT1/2) 和IRF9复合物的磷酸化达到抑制核酸介导的TLR3、TLR9和RIG-1依赖型Ⅰ型IFN信号的目的[41]。此外,RIG-1在炎性小体的形成和IL-1β的分泌过程中起积极的作用,一经RNA病毒刺激,RIG-1与配体ASC相互作用引发caspase-1依赖型炎性小体的活化和IL-1β的成熟[42]。与之相反的是,NLRs在调节RLRs介导的Ⅰ型IFN反应过程中通过NLRC5靶向作用于RIG-1和MDA5而抑制信号分子或者通过NLRX1与MAVS作用破坏RLR-MAVS的连接,NLRC3可以妨碍STING与TBK1的相互作用以及NLRP4引起TBK1的降解[31]。TLRs和NOD蛋白对于机体防御细菌感染非常重要,也存在着相互作用。一方面,NOD1和NOD2激动剂可以与TLR2、TLR3、TLR4和TLR9激动剂协同作用从而促进树突状细胞和嗜碱性粒细胞的成熟;另一方面,NLRs抑制TLRs信号的调控,NLRX1和NLRC3通过干扰TRAF6-NF-κΒ途径,NLRC5破坏IKKα和IKKβ的磷酸化以及NLRP6和NLRP12靶向攻击MAPK和NF-κΒ的活化[43]。这些PRRs的相互协同与拮抗作用使得PRRs信号通路交叉复杂,也使PRRs对PAMPs微生物侵袭做出更精准和迅速的反应。
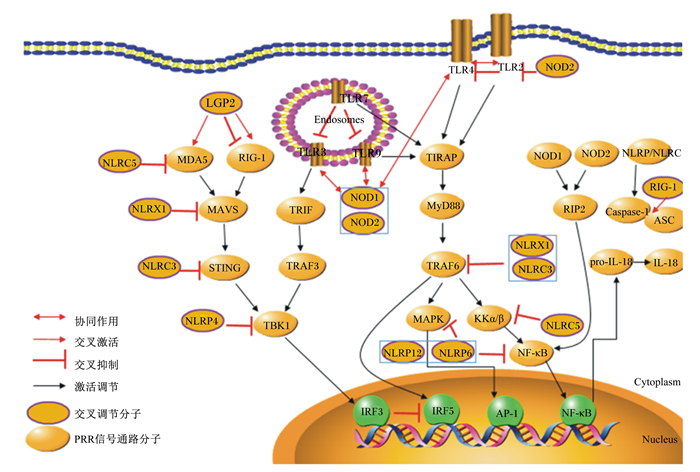
|
MyD88:髓样分化因子88;MAPK:丝裂原活化蛋白激酶;LGP2:遗传学和生理学实验室蛋白2;RIG-1:视黄酸诱导基因;MDA5:蛋白黑色素瘤分化相关分子;MAVS:线粒体抗病毒信号蛋白;STING:干扰素基因刺激物;TBK1:TANK结合激酶1;TRIF:含TIR域配体分子;TRAF3/6:肿瘤坏死因子受体关联因子3/6;IRF:干扰素效应因子;AP-1:活化蛋白转录因子1;TIRAP:含TIR域配体蛋白;IKK:NF-κΒ激酶抑制分子;RIP2:受体相互作用蛋白;ASC:凋亡相关的斑点样蛋白;Caspase-1:半光天冬氨酸特异性蛋白酶1;Cytoplasm:细胞质;Nucleus:细胞核。 图 1 TLRs、RLRs与NLRs信号途径交互作用 Figure 1 Interplay across of TLRs, RLRs and NLRs signaling pathways[43] |
固有免疫系统在不同的细胞类型和不同的亚细胞结构中有多种识别机制。机体出现的每一种PRRs信号通路在消除病原体和维持免疫耐受起着不可或缺的作用,因为PRRs或者其中的信号分子的变异紧密联系着许多炎症性疾病、免疫缺陷和自身免疫疾病。1种PAMP在某些情况下可以引起多种不同的PRRs的识别,协同性的诱发炎症反应并引起适应性免疫反应。因此,如何更好地了解复杂的PRRs之间相互作用以及固有免疫与适应性免疫的联动控制,对有效和定量治疗免疫相关疾病药物的开发显得尤为重要。这些问题也将更好地推动免疫学的发展和理论体系的完善。
| [1] |
JANEWAY C J. Approaching the asymptote? Evolution and revolution in immunology[J]. Cold Spring Harbor Symposia on Quantitative Biology, 1989, 54: 1-13. DOI:10.1101/SQB.1989.054.01.003 |
| [2] |
AKIRA S, UEMATSU S, TAKEUCHI O. Pathogen recognition and innate immunity[J]. Cell, 2006, 124(4): 783-801. DOI:10.1016/j.cell.2006.02.015 |
| [3] |
BARTON G M, KAGAN J C. A cell biological view of Toll-like receptor function:regulation through compartmentalization[J]. Nature Reviews Immunology, 2009, 9(8): 535-542. DOI:10.1038/nri2587 |
| [4] |
IWASAKI A, MEDZHITOV R. Control of adaptive immunity by the innate immune system[J]. Nature Immunology, 2015, 16(4): 343-353. DOI:10.1038/ni.3123 |
| [5] |
DINARELLO C A. Inflammatory cytokines:interleukin-1 and tumor necrosis factor as effector molecules in autoimmune diseases[J]. Current Opinion in Immunology, 1991, 3(6): 941-948. DOI:10.1016/S0952-7915(05)80018-4 |
| [6] |
GAY N J, KEITH F J. Drosophila Toll and IL-1 receptor[J]. Nature, 1991, 351(6325): 355-356. |
| [7] |
HEGUY A, BALDARI C T, MACCHIA G, et al. Amino acids conserved in interleukin-1 receptors (IL-1Rs) and the Drosophila toll protein are essential for IL-1R signal transduction[J]. Journal of Biological Chemistry, 1992, 267(4): 2605-2609. |
| [8] |
POLTORAK A, HE X, SMIRNOVA I, et al. Defective LPS signaling in C3H/HeJ and C57BL/10ScCr mice:mutations in TLR4 gene[J]. Science, 1998, 282(5396): 2085-2088. DOI:10.1126/science.282.5396.2085 |
| [9] |
QURESHI S T, LARIVIERE L, LEVEQUE G, et al. Endotoxin-tolerant mice have mutations in Toll-like receptor 4(TLR4)[J]. Journal of Experimental Medicine, 1999, 189(4): 615-625. DOI:10.1084/jem.189.4.615 |
| [10] |
HOSHINO K, TAKEUCHI O, KAWAI T, et al. Cutting edge:Toll-like receptor 4(TLR4)-deficient mice are hyporesponsive to lipopolysaccharide:evidence for TLR4 as the Lps gene product[J]. Journal of Immunology, 1999, 162(7): 3749-3752. |
| [11] |
KAWAI T, AKIRA S. The roles of TLRs, RLRs and NLRs in pathogen recognition[J]. International Immunology, 2009, 21(4): 17-337. |
| [12] |
JIN M S, KIM S E, HEO J Y, et al. Crystal structure of the TLR1-TLR2 heterodimer induced by binding of a tri-acylated lipopeptide[J]. Cell, 2007, 130(6): 1071-1082. DOI:10.1016/j.cell.2007.09.008 |
| [13] |
HAYASHI F, SMITH K D, OZINSKY A, et al. The innate immune response to bacterial flagellin is mediated by Toll-like receptor 5[J]. Nature, 2001, 410(6832): 1099-1103. DOI:10.1038/35074106 |
| [14] |
UEMATSU S, FUJIMOTO K, JANG M H, et al. Regulation of humoral and cellular gut immunity by lamina propria dendritic cells expressing Toll-like receptor 5[J]. Nature Immunology, 2008, 9(7): 769-776. DOI:10.1038/ni.1622 |
| [15] |
ZHANG D, ZHANG G, HAYDEN M S, et al. A Toll-like receptor that prevents infection by uropathogenic bacteria[J]. Science, 2004, 303(5663): 1522-1526. DOI:10.1126/science.1094351 |
| [16] |
ANDRADE W A, SOUZA M C, RAMOS-MARTINEZ E, et al. Combined action of nucleic acid-sensing Toll-like receptors and TLR11/TLR12 heterodimers imparts resistance to Toxoplasma gondii in mice[J]. Cell Host Microbe, 2013, 13(1): 42-53. DOI:10.1016/j.chom.2012.12.003 |
| [17] |
OLDENBURG M, KRUGER A, FERSTL R, et al. TLR13 recognizes bacterial 23S rRNA devoid of erythromycin resistance-forming modification[J]. Science, 2012, 337(6098): 1111-1115. DOI:10.1126/science.1220363 |
| [18] |
ALEXOPOULOU L, HOLT A C, MEDZHITOV R, et al. Recognition of double-stranded RNA and activation of NF-kappaB by Toll-like receptor 3[J]. Nature, 2001, 413(6857): 732-738. DOI:10.1038/35099560 |
| [19] |
LUND J, SATO A, AKIRA S, et al. Toll-like receptor 9-mediated recognition of Herpes simplex virus-2 by plasmacytoid dendritic cells[J]. Journal of Experimental Medicine, 2003, 198(3): 513-520. DOI:10.1084/jem.20030162 |
| [20] |
VOLLMER J, TLUK S, SCHMITZ C, et al. Immune stimulation mediated by autoantigen binding sites within small nuclear RNAs involves Toll-like receptors 7 and 8[J]. Journal of Experimental Medicine, 2005, 202(11): 1575-1585. DOI:10.1084/jem.20051696 |
| [21] |
FLANNERY S, BOWIE A G. The interleukin-1 receptor-associated kinases:critical regulators of innate immune signalling[J]. Biochemical Pharmacology, 2010, 80(12): 1981-1991. DOI:10.1016/j.bcp.2010.06.020 |
| [22] |
KAWAI T, TAKEUCHI O, FUJITA T, et al. Lipopolysaccharide stimulates the MyD88-independent pathway and results in activation of IFN-regulatory factor 3 and the expression of a subset of lipopolysaccharide-inducible genes[J]. Journal of Immunology, 2001, 167(10): 5887-5894. DOI:10.4049/jimmunol.167.10.5887 |
| [23] |
HORNG T, BARTON G M, FLAVELL R A, et al. The adaptor molecule TIRAP provides signalling specificity for Toll-like receptors[J]. Nature, 2002, 420(6913): 329-333. DOI:10.1038/nature01180 |
| [24] |
OSHIUMI H, SASAI M, SHIDA K, et al. TIR-containing adapter molecule (TICAM)-2, a bridging adapter recruiting to Toll-like receptor 4 TICAM-1 that induces interferon-beta[J]. Journal of Biological Chemistry, 2003, 278(50): 49751-49762. DOI:10.1074/jbc.M305820200 |
| [25] |
TING J P, LOVERING R C, ALNEMRI E S, et al. The NLR gene family:a standard nomenclature[J]. Immunity, 2008, 28(3): 285-287. DOI:10.1016/j.immuni.2008.02.005 |
| [26] |
KANNEGANTI T D, LAMKANFI M, NUNEZ G. Intracellular NOD-like receptors in host defense and disease[J]. Immunity, 2007, 27(4): 549-559. DOI:10.1016/j.immuni.2007.10.002 |
| [27] |
TRAVASSOS L H, CARNEIRO L A, RAMJEET M, et al. Nod1 and Nod2 direct autophagy by recruiting ATG16L1 to the plasma membrane at the site of bacterial entry[J]. Nature Immunology, 2010, 11(1): 55-62. DOI:10.1038/ni.1823 |
| [28] |
SABBAH A, CHANG T H, HARNACK R, et al. Activation of innate immune antiviral responses by Nod2[J]. Nature Immunology, 2009, 10(10): 1073-1080. DOI:10.1038/ni.1782 |
| [29] |
ARNOULT D, SOARES F, TATTOLI I, et al. An N-terminal addressing sequence targets NLRX1 to the mitochondrial matrix[J]. Journal of Cell Science, 2009, 122(17): 3161-3168. DOI:10.1242/jcs.051193 |
| [30] |
BRUBAKER S W, BONHAM K S, ZANONI I, et al. Innate immune pattern recognition:a cell biological perspective[J]. Annual Review of Immunology, 2015, 33: 257-290. DOI:10.1146/annurev-immunol-032414-112240 |
| [31] |
CAO X. Self-regulation and cross-regulation of pattern-recognition receptor signalling in health and disease[J]. Nature Reviews Immunology, 2015, 16(1): 35-50. DOI:10.1038/nri.2015.8 |
| [32] |
KATO H, TAKEUCHI O, SATO S, et al. Differential roles of MDA5 and RIG-Ⅰ helicases in the recognition of RNA viruses[J]. Nature, 2006, 441(7089): 101-105. DOI:10.1038/nature04734 |
| [33] |
VENKATARAMAN T, VALDES M, ELSBY R, et al. Loss of DExD/H box RNA helicase LGP2 manifests disparate antiviral responses[J]. Journal of Immunology, 2007, 178(10): 6444-6455. DOI:10.4049/jimmunol.178.10.6444 |
| [34] |
ZELENSKY A N, GREADY J E. The C-type lectin-like domain superfamily[J]. Federation of European Biochemical Societies, 2005, 272(24): 6179-6217. |
| [35] |
HOVING J C, WILSON G J, BROWN G D. Signalling C-type lectin receptors, microbial recognition and immunity[J]. Cellular Microbiology, 2014, 16(2): 185-194. DOI:10.1111/cmi.12249 |
| [36] |
SAIJO S, IKEDA S, YAMABE K, et al. Dectin-2 recognition of alpha-mannans and induction of Th17 cell differentiation is essential for host defense against Candida albicans[J]. Immunity, 2010, 32(5): 681-691. DOI:10.1016/j.immuni.2010.05.001 |
| [37] |
LAMKANFI M, DIXIT V M. Mechanisms and functions of inflammasomes[J]. Cell, 2014, 157(5): 1013-1022. DOI:10.1016/j.cell.2014.04.007 |
| [38] |
LI T, DINER B A, CHEN J, et al. Acetylation modulates cellular distribution and DNA sensing ability of interferon-inducible protein IFI16[J]. Proceedings of the National Academy of Sciences, 2012, 109(26): 10558-10563. DOI:10.1073/pnas.1203447109 |
| [39] |
SHI J, ZHAO Y, WANG Y, et al. Inflammatory caspases are innate immune receptors for intracellular LPS[J]. Nature, 2014, 514(7521): 187-192. |
| [40] |
NEGISHI H, YANAI H, NAKAJIMA A, et al. Cross-interference of RLR and TLR signaling pathways modulates antibacterial T cell responses[J]. Nature Immunology, 2012, 13(7): 659-666. DOI:10.1038/ni.2307 |
| [41] |
FORSBACH A, MULLER C, MONTINO C, et al. Negative regulation of the type Ⅰ interferon signaling pathway by synthetic Toll-like receptor 7 ligands[J]. Journal of Interferon and Cytokine Research, 2012, 32(6): 254-268. DOI:10.1089/jir.2011.0091 |
| [42] |
HENAO-MEJIA J, ELINAV E, STROWIG T, et al. Inflammasomes:far beyond inflammation[J]. Nature Immunology, 2012, 13(4): 321-324. DOI:10.1038/ni.2257 |
| [43] |
LIU J, CAO X. Cellular and molecular regulation of innate inflammatory responses[J]. Cellular and Molecular Immunology, 2016, 13(6): 711-721. DOI:10.1038/cmi.2016.58 |




