木质纤维素是含有纤维素、半纤维素、木质素等成分的植物生物质的统称[1]。瘤胃是自然界中降解木质纤维素能力最强的生态系统之一,能够在48 h内降解饲粮中60%~65%的木质纤维素,为反刍动物提供营养物质,可见瘤胃内蕴藏着丰富的木质纤维素降解微生物和酶系[2]。瘤胃内微生物种类繁多、数量巨大,共有超过3 000种微生物,既包括细菌、原虫、真菌和古细菌,也有噬菌体和病毒[3]。瘤胃是一座巨大的生物资源库,蕴藏着许多有具有工业应用潜力的木质纤维素降解酶系。Zhou等[4]从牦牛瘤胃中发现了一个双功能酶,同时具有葡萄糖苷酶及木糖苷酶的活性。Huang等[5]从湖羊瘤胃中获得了一种嗜低温的木聚糖酶基因。但瘤胃是一个严格厌氧的环境,绝大多数瘤胃微生物是不可培养的,这就大大限制了人们对瘤胃基因资源的探索研究。随着高通量测序技术的发展,宏基因组测序已成功运用于分析特定环境中的微生物组成、代谢通路研究以及酶蛋白的功能分析等[6]。
目前,已有许多应用高通量测序技术研究反刍动物瘤胃微生物的报道。Hess等[7]在2011年首次对奶牛瘤胃微生物的宏基因组进行了测序,共发现了27 755个木质纤维素降解酶基因,其中糖苷水解酶(glycoside hydrolase,GH)家族GH13(3 442个)和GH3(2 844个)的基因最多,而含有黏连模块和对接模块的基因分别为80和188个,这些基因为发掘新型木质纤维素降解酶提供了可能性。Patel等[8]利用青草、干草饲喂水牛,采样后进行宏基因组高通量测序,结果发现青草组的水牛瘤胃中GH28及果胶裂解酶的基因数量显著多于干草组。Singh等[9]利用高通量测序技术对印度水牛瘤胃微生物宏基因组进行测序,共获得了137 270个重叠群(contigs),预测其中2 614个具有降解木质纤维素的功能。王继文等[10]对山羊瘤胃与粪便微生物基因组进行了比对研究,发现二者存在较大差异,瘤胃微生物主要以拟杆菌门为主,厚壁菌门次之,相对丰度最高的属为普雷沃菌属,而粪便中的微生物以厚壁菌门为主,拟杆菌门次之,相对丰度最高的属为瘤胃球菌科下未分类的属。
海子水牛属沼泽型水牛,分布于江苏北部沿海地区,是我国主要的地方良种之一,它适应当地的自然生态条件,抗逆性突出,耐粗饲,在草质较差的情况下,仍能较好地生长及劳役,可见海子水牛瘤胃内蕴藏着独特的木质纤维素降解微生物和酶系。然而,到目前为止,对于沼泽型水牛瘤胃内的微生物组成及其功能基因尚鲜见报道。本研究借助于高通量测序技术,首次获得海子水牛瘤胃微生物的宏基因组数据,经数据库比对分析后,获得功能注释,结合其他物种肠胃宏基因组信息进行比对研究,旨在系统探讨海子水牛瘤胃内的微生物组成及木质纤维素降解酶系,以期从中发掘新型具有工业应用潜力的木质纤维素降解酶。
1 材料与方法 1.1 样品采集选取3头2.5岁左右的海子水牛,平均体重为493 kg。试验水牛的饲养管理条件一致,每天饲喂2次,自由采食,饲粮组成及营养水平见表 1。饲喂60 d后进行称重并屠宰,屠宰后立刻采集瘤胃液样品,经4层灭菌纱布过滤后,迅速置于液氮中。
|
|
表 1 饲粮组成及营养水平(风干基础) Table 1 Composition and nutrient levels of the diet (air-dry basis) |
在相同的饲粮及生活环境下,牛瘤胃内的微生物群落组成较为相似[7, 11],因此我们随机选取1头海子水牛的瘤胃液进行下一步测序分析。
1.2 宏基因组DNA的提取按照QIAamp DNA micro kit (德国Qiagen)试剂盒说明书的要求,进行瘤胃液宏基因组DNA的分离和纯化。使用1.0%的琼脂糖凝胶电泳和Nano Drop分光光度计,对分离纯化后的DNA进行检测和评价。
1.3 宏基因组测序及分析纯化后的样品送上海欧易生物公司进行高通量测序。首先利用Covaris M220进行宏基因组DNA片段化处理,打断至300 bp左右,末端连接Y字形接头,通过接头PCR将DNA片段连接到芯片上形成“桥”并制成文库,随后通过Illumina HiSeq 2000高通量测序平台进行测序。
获得测序数据后,首先进行宿主基因的过滤和质量剪切等优化处理;其次使用软件SOAP denovo 2.04对测序数据进行拼接组装,组装时选用的kmer参数为65;然后使用软件prodigal 2.6.3对基因组序列进行基因预测;最后进行数据库比对分析。
通过BLAST将预测得到的基因序列与GenBank的非冗余核苷酸序列数据库(Nonredundant database,NR)进行比对(ftp://ftp.ncbi.nih.gov/pub/taxonomy),将evalue≤1e-5的序列作为有意义的序列,并获得物种注释信息;最后将预测基因与COG数据库(ftp://ftp.ncbi.nih.gov/pub/COG/COG/kyva)、GO数据库(http://www.geneontology.org/)和KEGG数据库(http://www.genome.jp/kegg/pathway.html)进行比对,获得基因功能注释。
1.4 碳水化合物基因分析使用CAZy数据库(http://www.cazy.org/)的对应工具hmmscan(http://hmmer.janelia.org/search/hmmscan)将预测基因与CAZy数据库进行比对,获得基因对应的碳水化合物活性酶注释信息,然后使用碳水化合物活性酶对应的基因丰度总和计算该碳水化合物活性酶的丰度。根据蛋白质结构域中氨基酸序列的相似性,可以将不同物种来源的碳水化合物活性酶分成GH、糖基转移酶(glycosyl transferase,GT)、多糖裂合酶(polysaccharide lyase,PL)、碳水化合物酯酶(carbohydrate esterase,CE)、碳水化合物结合模块(carbohydrate-binding module,CBM)、辅助氧化还原酶(auxiliary activity,AA)共6大类蛋白质家族。同时将海子水牛的CAZy分析结果,与奶牛及印度水牛瘤胃、白蚁后肠及鸡盲肠宏基因组中的CAZy分析结果进行比对。
2 结果与分析 2.1 宏基因组测序结果经Illumina Hiseq高通量测序平台测序,获得77 283 638条reads,过滤水牛宿主基因(GCA_000003055.5, Bostaurus Bos_taurus_UMD_3.1.1) 后共获得了10.79 GB的测序数据。使用软件SOAP denovo 2.04对测序数据进行拼接组装,共得到744 712个scaffold,约250 Mbp的宏基因组序列(表 2)。
|
|
表 2 组装结果统计表 Table 2 The result of the assembly |
使用软件prodigal 2.6.3对基因组序列进行基因预测,预测得到氨基酸序列共827 044条,基因平均长度274 bp。
2.2 基因注释结果将827 044条基因序列与NR数据库进行BLAST比对,选取evalue≤1e-5的序列,共498 644条。如图 1所示,与NR数据库中序列的相似度主要分布在50%以上,只有7.8%的序列与NR数据库序列高度相似(相似度>95%),表明此次测序结果中含有许多新基因。
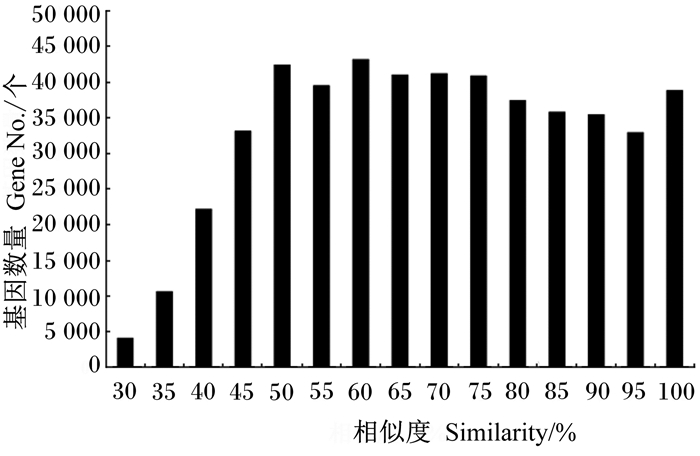
|
图 1 与NR数据库序列的相似度 Figure 1 Similarity with sequences in NR database |
将测序数据与数据库比对,获得分析序列的蛋白质功能注释信息。同时统计基因比对到各个物种的比例,结果显示蛋白质编码基因来源于拟杆菌门(55.3%)的最多,来源于厚壁菌门的比例可达到12.7%, 3.23%及2.71%的基因分别来源于纤维杆菌门及变形菌门(表 3)。
|
|
表 3 最多的15个门的分布 Table 3 Distribution of the top 15 phyla |
此外,将样品中物种水平上的分类层级关系绘制成进化分支图(图 2),从根部开始向外,依次为界—门—纲—目—科—属—种—菌株的分类。共分类出来了3个界:细菌界、古菌界、病毒界,海子水牛瘤胃中的微生物大部分为细菌界。在菌株的分类中,未分类的甲烷短杆菌(Methanobrevibacter unclassified)及猪沙波病毒(Porcine sapelovirus)分别属于古菌界及病毒界。而细菌界中共测得8种主要菌株,分别为栖瘤胃普雷沃氏菌(Prevotella ruminicola)、生黄瘤胃球菌(Ruminococcus flavefaciens)、白色瘤胃球菌(Ruminococcus albus)、未分类的丁酸弧菌(Butyrivibrio unclassified)、链球菌(Streptococcus infantarius)、密螺旋体JC4(Treponema sp. JC4) 及伯氏密螺旋体(Treponema bryantii)、产琥珀酸纤维杆菌(Fibrobacter succinogenes)。
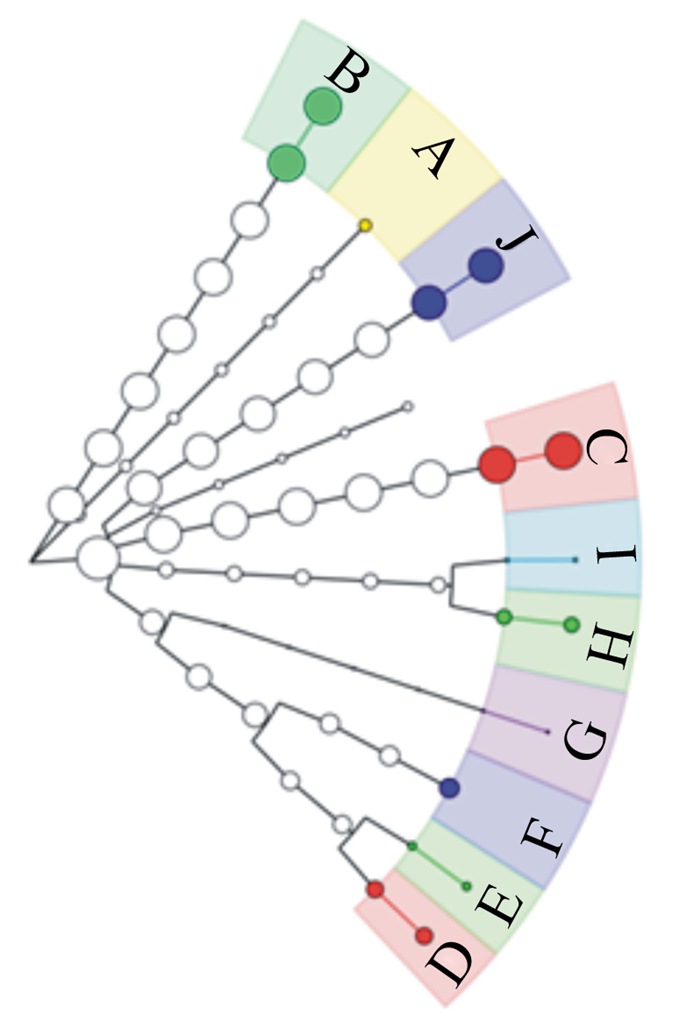
|
A:未分类的甲烷短杆菌Methanobrevibacter unclassified;B:猪萨佩罗病毒Porcine sapelovirus;C:栖瘤胃普雷沃氏菌Prevotella ruminicola;D:生黄瘤胃球菌属Ruminococcus flavefaciens;E:白色瘤胃球菌属Ruminococcus albus;F:未分类的丁酸弧菌属Butyrivibrio unclassified;G:链球菌Streptococcus infantarius;H:梅毒螺旋体菌JC4 Treponema sp. JC4;I:布赖恩蒂密螺旋体Treponema bryantii;J:产琥珀酸纤维杆菌属Fibrobacter succinogenes。 图 2 物种的分类 Figure 2 The classification of species |
对宏基因组中的预测基因进行COG功能分类预测。发现功能集中在碳水化合物转运及代谢的基因是最多的,数量达到37 933个,由此可见该宏基因组内可能含有大量的木质纤维素降解酶基因(图 3)。
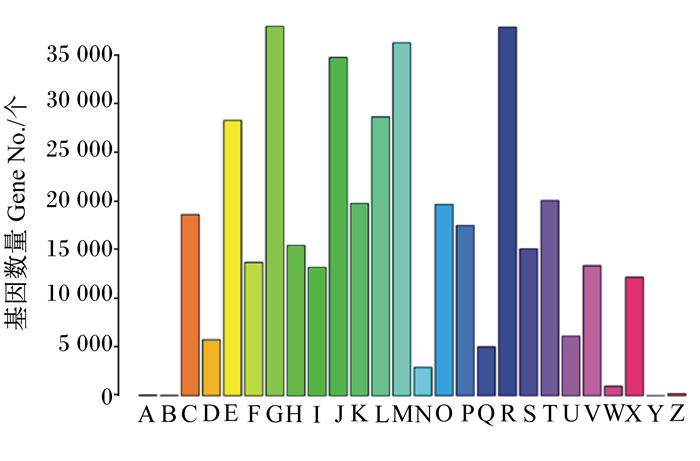
|
A:RNA加工和修改RNA processing and modification;B:染色质结构与动力学Chromatin structure and dynamics;C:能量产生和转换Energy production and conversion;D:细胞周期控制、细胞分裂、染色体分区Cell cycle control, cell division, chromosome partitioning;E:氨基酸运输和代谢Amino acid transport and metabolism;F:核苷酸运输和代谢Nucleotide transport and metabolism;G:碳水化合物的运输和代谢Carbohydrate transport and metabolism;H:辅酶运输和代谢Coenzyme transport and metabolism;I:脂质运输和代谢Lipid transport and metabolism;J:翻译, 核糖体结构和生物转化Translation, ribosomal structure and biogenesis;K:转录Transcription;L:复制、重组和修复Replication, recombinationan drepair;M:细胞壁/膜/包膜生物合成Cell wall/membrane/envelope biogenesis;N:细胞活性Cellmotility;O:蛋白质转译后的修改, 蛋白质转化, 分子伴侣Post translational modification, protein turn over, chaperones;P:无机离子运输和代谢Inorganicion transport and metabolism;Q:次生代谢产物生物合成、运输和分解代谢Secondary metabolites biosynthesis, transport and catabolism;R:通用功能预测General function prediction; S:未知功能Function unknown;T:信号转导机制Signal transduction mechanisms;U:胞内运输、分泌和膜泡运输Intracellular trafficking, secretion, andvesicular transport;V:防御机制Defense mechanisms;W:细胞外结构Extracellular structures;X:移动基因组(噬菌体原、转座子) Mobilome (prophages, transposons);Y:核结构Nuclear structure;Z:细胞骨架Cytoskeleton。 图 3 COG分类 Figure 3 COG classification |
对得到的基因进行GO分类,统计基因在生物过程(biological process,BP)、细胞成分(cellular component,CC)和分子功能(molecular function,MF)3个GO类别中。在BP类中,集中在代谢、细胞外加工的基因最多;在CC类中,集中在细胞、细胞组分的基因最多;在MF类中,集中在结合以及催化功能的基因最多(图 4)。
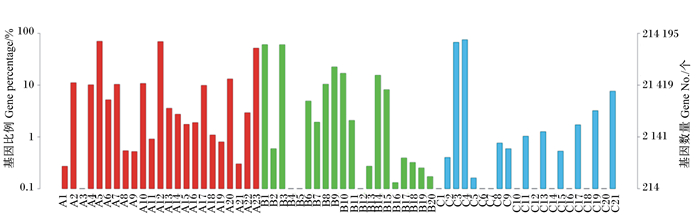
|
A1:生物黏附biological adhesion; A2:生物调节biological regulation; A3:细胞死亡cell killing; A4:组织或生物合成细胞组件cellular component organization or biogenesis; A5:细胞过程cellular process; A6:发育过程developmental process; A7:定位建立establishment of localization; A8:增长growth; A9:免疫系统的过程immune system process; A10:定位localization; A11:移动locomotion; A12:代谢过程metabolic process; A13:多细胞生物的过程multicellular organismal process; A14:多组织过程multi-organism process; A15:生物过程反向调节negative regulation of biological process; A16:生物过程正向调节positive regulation of biological process; A17:生物过程的调节regulation of biological process; A18:繁殖reproduction; A19:生殖过程reproductive process; A20:刺激反应response to stimulus; A21:节律过程rhythmic process; A22:信号signaling; A23:单组织过程single-organism process; B1:细胞cell; B2:细胞连接cell junction; B3:细胞部分cell part; B4:细胞外基质extracellular matrix; B5:细胞外基质部分extracellular matrix part; B6:细胞外区域extracellular region; B7:细胞外区域部分extracellular region part; B8:大分子复合物macromolecular complex; B9:膜membrane; B10:膜部分membrane part; B11:膜封闭腔membrane-enclosed lumen; B12:线粒体相关黏附复合体mitochondrion-associated adherens complex; B13:类核nucleoid; B14:细胞器organelle; B15:细胞器的部分organelle part; B16:共质体symplast; B17:突触synapse; B18:突触部分synapse part; B19病毒粒子virion; B20:病毒粒子部分virion part; C1:D-丙氨酰载体功能D-alanyl carrier activity; C2:抗氧化功能antioxidant activity; C3:结合binding; C4:催化功能catalytic activity; C5:通道调节功能channel regulator activity; C6:化学引诱物功能chemoattractant activity; C7:化学排斥物功能chemorepellent activity; C8:电子载体功能electron carrier activity; C9:酶调节功能enzyme regulator activity; C10:金属伴侣功能metallochaperone activity; C11:分子传感器功能molecular transducer activity; C12:成形素功能morphogen activity; C13:核酸结合转录因子功能nucleic acid binding transcription factor activity; C14:营养储存功能nutrient reservoir activity; C15:蛋白结合转录因子功能protein binding transcription factor activity; C16:蛋白质标记protein tag; C17:受体功能receptor activity; C18:受体调节功能receptor regulator activity; C19:结构分子功能structural molecule activity; C20:翻译调节功能translation regulator activity; C21:运输功能transporter activity。 A代表注释到BP的条目,B代表注释到CC的条目,C代表注释到MF的条目。 A represents the term of BP, B represents the term of CC, and C represents the term of MF. 图 4 最多的20个CO条目分类 Figure 4 Top 20 GO terms classification |
105 560个基因被富集到247条KEGG通路中,表 4为富集最明显的前10条KEGG通路。大部分基因被富集在小分子的代谢中,如氨基酸的生物合成、嘌呤代谢、嘧啶代谢、碳代谢、氨酰tRNA的生物合成等。此外,还有5 236个基因富集在淀粉和蔗糖代谢中。
|
|
表 4 富集最明显的10条KEGG通路 Table 4 Top 10 obviously gathered KEGG pathways |
经hmmscan分析,发现有38 011个基因比对到CAZy数据库中,统计结果如图 5所示,宏基因组中的GH基因数量最多(17 877个),GT(8 637个)、CBM(4 693个)和CE基因(4 214个)次之,PL和AA基因较少,数量分别为1 296和934个。此外还发现了42个黏连蛋白(cohesin)基因、236个对接蛋白(dockerin)基因以及82个S层同源结构域(S-layer homology,SLH)。
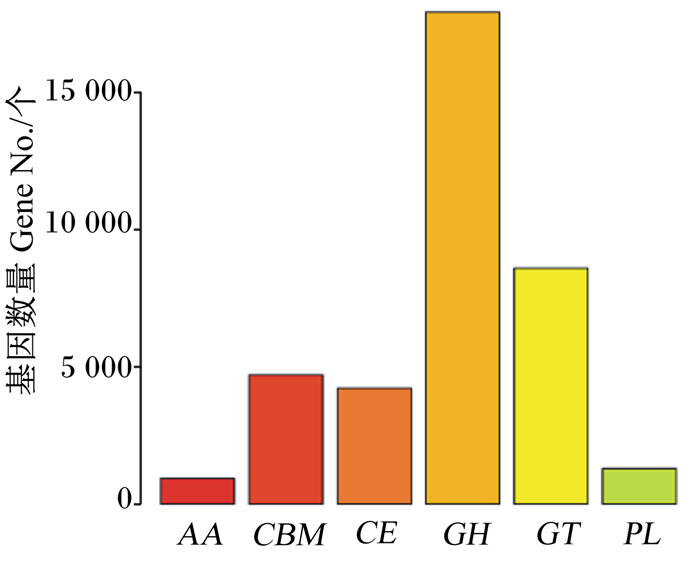
|
AA:辅助氧化还原酶;CBM:碳水化合物结合模块;CE:碳水化合物酯酶;GH:糖苷水解酶;GT:糖基转移酶;PL:多糖裂合酶。 AA: auxiliary activity CBM: carbohydrate binding modules; CE: carbohydrate esterases; GH: glycosyl hydrolases; GT: glycosyl transferases; PL: polysaccharide lyases. 图 5 CAZy数据库比对结果 Figure 5 Distribution of CAZyme encoding genes |
17 877个GH基因分布在不同的GH基因家族,其中GH2、GH43、GH97、GH3家族的基因较多,GH12和GH62家族的基因最少(表 4)。瘤胃中的木质纤维素降解酶可水解植物细胞壁中的纤维素和半纤维素,根据水解底物类型的不同,可将海子水牛瘤胃宏基因组内的GH基因家族分类(表 5),有932个基因编码的蛋白质具备纤维素酶活性,其中GH5及GH9基因家族的数量较多;1 011个基因编码的蛋白质具备半纤维素酶活性,其中GH10基因家族的基因数量最多;分支降解酶(GH67和GH78)的编码基因共有538个;3 266个基因编码的蛋白质具有寡糖降解酶活性,其中GH2基因家族的基因数量最多。
|
|
表 5 海子水牛瘤胃微生物宏基因组中GH基因的分布 Table 5 Distribution of GH genes in Haizi buffalo rumen microorganism metagenome |
|
|
表 6 不同动物的微生物宏基因组中GH基因的分布 Table 6 Distribution of GH genes in microorganism metagenome in different animals |
本试验通过高通量测序技术对海子水牛瘤胃液样本进行组学分析,共获得了77 283 638条reads,并拼接出744 712个Scaffold。将测序数据与COG数据库进行比对发现,功能集中在碳水化合物转运及代谢的基因是最多的,除此之外,富集在氨基酸运输及代谢、细胞壁及细胞膜生物合成基因也较多,这与印度水牛的报道相似[9]。在GO分类分析中发现,集中在结合以及催化功能的基因最多。由此可见,海子水牛瘤胃宏基因组内可能含有大量的木质纤维素降解酶基因。
根据基因注释到物种的比例可知,来源于拟杆菌门的基因较多,可占55.3%。Jami等[13]在奶牛瘤胃中也发现了相同的微生物分布。此外,根据物种分类结果可知,海子水牛瘤胃中的微生物大部分为细菌界,其中的生黄瘤胃球菌、白色瘤胃球菌及产琥珀酸丝状杆菌可产出大量的纤维素酶降解饲粮中的木质纤维素,被认为是瘤胃中降解木质纤维素的主要微生物,而栖瘤胃普雷沃氏菌可降解木聚糖及果胶[14-15]。
纤维素酶、半纤维素酶及果胶酶的协同作用可降解植物的细胞壁,为反刍动物提供营养物质,而这3类酶基因均存在于海子水牛的瘤胃中。GH5及GH9基因家族的含量较高,二者可达到GH基因总量的4.77%。GH10、GH11、GH26和GH28家族的酶具有半纤维素酶活性,其中GH10和GH11家族属于木聚糖酶,它能从木聚糖主链的内部随机切割β-1, 4木糖苷键,将其降解为低聚木糖和少量木糖,而CE1、CE4、CE6及CE7家族的木聚糖酯酶可作用于木聚糖的支链[8, 16]。此外,PL1、PL9及PL10家族的酶具有果胶裂解酶活性,而CE8家族的酶具有果胶甲基酯酶活性,可脱去果胶中的甲酯基团,生成果胶酸[17]。在GH分类中,寡糖降解酶的比例最高,可能是由于经过纤维素酶、半纤维素酶及果胶酶的协同作用后,植物细胞壁被降解为大量的低聚寡糖,这些物质需要被进一步降解后才可被反刍动物吸收利用。而寡糖降解酶中的GH2家族的酶数量最多,主要包括β-D-半乳糖苷酶、β-葡萄糖醛酸酶、β-D-甘露糖苷酶及外切β-氨基葡萄糖苷酶[18]。
此外,本试验还在海子水牛瘤胃内发现了42个黏连蛋白基因、236个对接蛋白基因以及82个S层同源结构域,它们均是构成纤维小体的组分。纤维小体是一种由木质纤维素降解酶依靠脚手架蛋白形成的多酶复合体[19],并借助黏附蛋白固定于细胞表面,可持续、高效地降解木质纤维素。目前已在生黄瘤胃球菌和白色瘤胃球菌中发现了纤维小体[20]。Hess等[7]发现奶牛瘤胃中含有黏连蛋白和对接蛋白的基因分别为80和188个。
将海子水牛的瘤胃宏基因组与奶牛[7]和印度水牛的瘤胃[9]、白蚁后肠[11]及鸡盲肠[12]宏基因组数据中的GH基因进行分类比对,发现在不同的宏基因组中,寡糖降解酶比例均为最高。但与其他4种动物肠胃微生物宏基因组相比,海子水牛中的寡糖降解酶数量最少。海子水牛和奶牛瘤胃宏基因组中的纤维素酶、半纤维素酶和分支降解酶比例接近。印度水牛中的纤维素酶最少,但寡糖降解酶最多。海子水牛瘤胃中纤维素酶及寡糖降解酶的比例与印度水牛瘤胃存在许多不同,这可能与2种水牛饲粮中粗饲料与精饲料的比例有关,印度水牛饲粮中粗饲料与精饲料的比例为75:25[9],而海子水牛的则为85:15,粗饲料含量高可能导致瘤胃内存在高比例的纤维素酶和低比例的寡糖降解酶。但在GH基因家族分布中,2种水牛瘤胃中GH6、GH7、GH12、GH48及GH62家族基因数量均较少[8]。白蚁后肠微生物宏基因组中的纤维素酶和半纤维素酶基因最多,其中GH5(5.71%)和GH10(5.39%)家族的基因比例最高,主要是由于木材中纤维素及半纤维素的比例高于牧草。鸡盲肠中的纤维素酶及半纤维素酶的比例最低,但寡糖降解酶比例较高。由此可见宿主基因型及饲料的不同可影响肠胃GH基因的组成。
4 结论① 通过高通量测序发现海子水牛瘤胃中含有多种木质纤维素降解微生物,如生黄瘤胃球菌、白色瘤胃球菌、产琥珀酸丝状杆菌及栖瘤胃普雷沃氏菌。可见,海子水牛瘤胃内含有丰富的木质纤维素降解微生物。
② 水牛瘤胃中还含有大量的糖苷水解酶基因,其中GH2、GH43、GH97、GH3家族的基因较多,且编码蛋白质具有寡糖降解酶活性的基因数量最多。可见,海子水牛瘤胃内含有丰富的木质纤维素降解酶系。
| [1] |
邓玉营, 黄振兴, 阮文权, 等. 木质纤维素沼气体系中共培养菌群形成及适应性变化研究进展[J]. 应用与环境生物学报, 2016, 22(5): 944-958. |
| [2] |
WANG G R, DUAN Y L. Studies on lignocellulose degradation by rumen microorganism[J]. Advanced Materials Research, 2014, 853: 253-259. |
| [3] |
KIM M, MORRISON M, YU Z T. Status of the phylogenetic diversity census of ruminal microbiomes[J]. FEMS Microbiology Ecology, 2011, 76(1): 49-63. DOI:10.1111/fem.2011.76.issue-1 |
| [4] |
ZHOU J, BAO L, CHANG L, et al. Beta-xylosidase activity of a GH3 glucosidase/xylosidase from yak rumen metagenome promotes the enzymatic degradation of hemicellulosic xylans[J]. Letters in Applied Microbiology, 2012, 54(2): 79-87. DOI:10.1111/lam.2012.54.issue-2 |
| [5] |
HUANG H Q, WANG G Z, ZHAO Y Y, et al. Direct and efficient cloning of full-length genes from environmental DNA by RT-qPCR and modified TAIL-PCR[J]. Applied Microbiology and Biotechnology, 2010, 87(3): 1141-1149. DOI:10.1007/s00253-010-2613-4 |
| [6] |
王禄禄, 王立志, 周美丽. 宏基因组学技术在反刍动物瘤胃微生态系统上的应用研究进展[J]. 中国微生态学杂志, 2017, 29(2): 223-228. |
| [7] |
HESS M, SCZYRBA A, EGAN R, et al. Metagenomic discovery of biomass-degrading genes and genomes from cow rumen[J]. Science, 2011, 331(6016): 463-467. DOI:10.1126/science.1200387 |
| [8] |
PATEL D D, PATEL A K, PARMAR N R, et al. Microbial and carbohydrate active enzyme profile of buffalo rumen metagenome and their alteration in response to variation in the diet[J]. Gene, 2014, 545(1): 88-94. DOI:10.1016/j.gene.2014.05.003 |
| [9] |
SINGH K M, REDDY B, PATEL D, et al. High potential source for biomass degradation enzyme discovery and environmental aspects revealed through metagenomics of Indian buffalo rumen[J]. BioMed Research International, 2014, 2014: 267189. |
| [10] |
王继文, 王立志, 闫天海, 等. 山羊瘤胃与粪便微生物多样性[J]. 动物营养学报, 2015, 27(8): 2559-2571. |
| [11] |
WARNECKE F, LUGINBVHL P, IVANOVA N, et al. Metagenomic and functional analysis of hindgut microbiota of a wood-feeding higher termite[J]. Nature, 2007, 450(7169): 560-565. DOI:10.1038/nature06269 |
| [12] |
QU A, BRULC J M, WILSON M K, et al. Comparative metagenomics reveals host specific metavirulomes and horizontal gene transfer elements in the chicken cecum microbiome[J]. PLoS One, 2008, 3(8): e2945. DOI:10.1371/journal.pone.0002945 |
| [13] |
JAMI E, MIZRAHI I. Composition and similarity of bovine rumen microbiota across individual animals[J]. PLoS One, 2012, 7(3): e33306. DOI:10.1371/journal.pone.0033306 |
| [14] |
FLINT H J, BAYER E A. Plant cell wall breakdown by anaerobic microorganisms from the mammalian digestive tract[J]. Annals of the New York Academy of Sciences, 2008, 1125(1): 280-288. DOI:10.1196/annals.1419.022 |
| [15] |
FLINT H J, BAYER E A, RINCON M T, et al. Polysaccharide utilization by gut bacteria:potential for new insights from genomic analysis[J]. Nature Reviews Microbiology, 2008, 6(2): 121-131. DOI:10.1038/nrmicro1817 |
| [16] |
ZHANG H M, LI J F, WANG J Q, et al. Determinants for the improved thermostability of a mesophilic family 11 xylanase predicted by computational methods[J]. Biotechnology for Biofuels, 2014, 7(1): 3. DOI:10.1186/1754-6834-7-3 |
| [17] |
YADAV S, YADAV P K, YADAV D, et al. Pectin lyase:a review[J]. Process Biochemistry, 2009, 44(1): 1-10. DOI:10.1016/j.procbio.2008.09.012 |
| [18] |
NASCIMENTO A S, MUNIZ J R C, APARÍCIO R, et al. Insights into the structure and function of fungal β-mannosidases from glycoside hydrolase family 2 based on multiple crystal structures of the Trichoderma harzianum enzyme[J]. FEBS Journal, 2014, 281(18): 4165-4178. DOI:10.1111/febs.2014.281.issue-18 |
| [19] |
HYEON J E, JEON S D, HAN S O. Cellulosome-based, Clostridium-derived multi-functional enzyme complexes for advanced biotechnology tool development:advances and applications[J]. Biotechnology Advances, 2013, 31(6): 936-944. DOI:10.1016/j.biotechadv.2013.03.009 |
| [20] |
DASSA B, BOROVOK I, RUIMY-ISRAELI V, et al. Rumen cellulosomics:divergent fiber-degrading strategies revealed by comparative genome-wide analysis of six Ruminococcal strains[J]. PLoS One, 2014, 9(7): e99221. DOI:10.1371/journal.pone.0099221 |




