瘤胃功能的发育完善对于幼龄反刍动物至关重要,瘤胃生酮作用是促进瘤胃发育成熟的关键性因素。过氧化物酶体增殖物激活受体(PPARs)是配体激活的核转录因子超家族成员,在单胃动物上研究表明,其主要参与线粒体内脂肪酸的氧化和能量代谢[1-2]。近年来仅有少数研究报道了PPARs在生酮过程中的作用[3-4],发现了PPARs的部分靶基因,例如3-羟基3-甲基戊二酰辅酶A合成酶2(3-hydroxy-3-methylglutaryl coenzyme A synthase 2, HMGCS2)和胆固醇酰基转移酶1(cholesterol acyltransferase 1, ACAT1)2个基因。目前仅发现了与生酮作用相关的以及受到PPARs调控的部分酶,在小鼠上的研究表明丁酸可以激活PPARs[5],但在反刍动物瘤胃中丁酸是否可以激活PPARs尚不得知,推测瘤胃中的丁酸很可能作为配体激活PPARs进而调控下游靶基因的表达。在单胃动物上仅有少数研究报道了PPARs基因表达的表观遗传学调控机制,例如,小鼠的PPAR-α启动子区域的低甲基化造成了PPAR-α及其靶基因mRNA的表达[6]。丁酸可引起表观遗传学上的改变,因此作者推测瘤胃中的丁酸可以引起PPARs表观遗传上的改变,进而影响PPARs的表达。尽管如此,已有研究对于影响PPARs表达的表观遗传学机制的了解仍然十分有限。本文针对PPARs调控幼龄反刍动物瘤胃生酮的研究进展进行综述,以期为进一步探索PPARs调控瘤胃生酮作用的分子机制提供部分线索,对于更深层次地理解瘤胃发育过程及建立最佳的反刍动物营养供给策略具有重要意义。
1 瘤胃生酮作用的发育特点瘤胃功能的发育是保障反刍动物成年后健康生长的关键[7]。前人的研究表明,羔羊的瘤胃发育分为3个阶段:非反刍阶段(1~3周龄)、过渡阶段(4~8周龄)以及反刍阶段(9周龄以后)。反刍动物刚出生时,主要依靠消化母乳生存,其瘤胃并不具有成年反刍动物的功能。此时,葡萄糖是其主要的能量来源[1]。在随后不断与外界环境接触的过程中,逐渐转变为依靠消化植物性饲料生存。在消化方式转变的过程中,瘤胃也不断发生变化,瘤胃内壁出现高度的角质化,瘤胃上皮细胞能够吸收挥发性脂肪酸(volatile fatty acid, VFA),进行代谢为机体提供能量,以确保瘤胃上皮能够吸收足够的营养物质来满足机体代谢的需要。羔羊从2日龄生长到6月龄对葡萄糖的利用约降低90%,期间瘤胃上皮细胞角质化程度不断增加,上皮细胞代谢的VFA也不断增加,丁酸和乳酸的利用率逐渐升高,丁酸转化为酮体的效率也不断增加。瘤胃中的生酮作用主要指的是丁酸生成乙酰辅酶A,进而转化为酮体乙酰乙酸和β-羟丁酸(BHBA)的过程。3-羟基-3-甲基-戊二酸单酰辅酶A(3-hydroxy-3-methyl-glutaric acid monoacyl-CoA, HMG-CoA)是生酮作用的限速酶,存在2种形式,包括3-羟基3-甲基戊二酰辅酶A合成酶(3-hydroxy-3-methylglutaryl coenzyme A synthase, HMGCS)1和HMGCS2。由于瘤胃中生酮作用主要是线粒体生酮,起主要作用的是HMGCS2。近年来,研究发现,HMGCS2和HMGCS1参与了瘤胃的生酮作用[4]。HMGCS2的表达受到PPARs的调控[3]。因此,PPARs在瘤胃生酮作用中发挥重要调控作用。
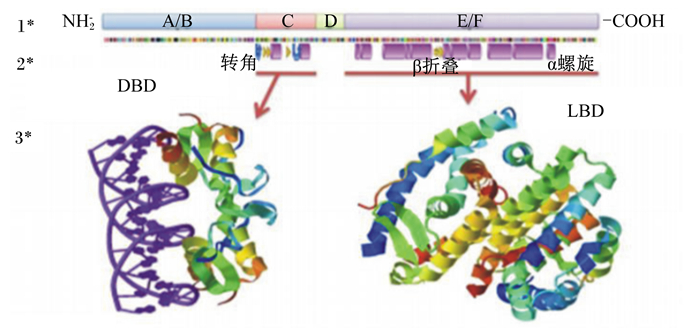
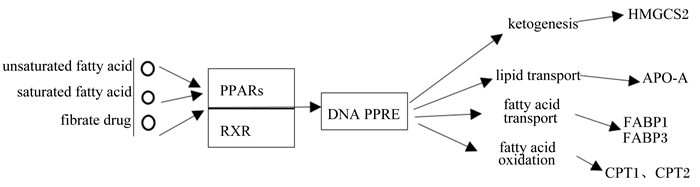
2 PPARs的结构及功能PPARs是一类由配体激活的核转录因子,其包括PPAR-α、PPAR-β以及PPAR-γ 3个成员,属于受体超家族成员[8]。PPARs包含4个功能结构域,人们为了便于研究,将其分为6个区域,用字母A~F表示。A、B 2个结构域存在丝氨酸残基,这些残基能够被丝裂原活化蛋白激酶(mitogen-activated protein kinase, MAPK)磷酸化,影响到PPARs的活性。C结构域为DNA结合结构域(DNA-binding domain, DBD),PPARs通过此结构域与反应元件结合调节基因转录活性。D区域为转录活性调节结构域,此区域与核内辅因子结合调节PPARs的活性。E、F结构域为配体结合结构域(图 1)[9]。PPARs先与配体结合,再与视黄酸类受体(retinoid X receptor, RXR)结合形成异二聚体导致共价键性质的改变,之后招募多种协同刺激因子,与其靶基因上游的启动子过氧化物增殖体反应元件(peroxisome proliferators reactive element, PPRE)识别并结合后发挥调控作用(图 2)[10-11],PPRE是一个六核苷酸的重复(AGGTCA)。
|
DBD:DNA结合结构域DNA-binding domain;LBD:配体结合域ligand binding domain。 图 1 PPARs的一级结构、二级结构和三级结构 Figure 1 PPARs primary, secondary and tertiary structures[9] |
|
PPARs:过氧化物酶体增殖物激活受体peroxisome proliferator-activated receptors;RXR:视黄酸类受体retinoid X receptor;DNA PPRE:DNA过氧化物增殖体反应元件DNA peroxisome proliferators reactive element;HMGCS2:3-羟基3-甲基戊二酰辅酶A合成酶2 3-hydroxy-3-methylglutaryl coenzyme A synthase 2;APO-A:载脂蛋白-A apolipoprotein-A;FABP1:脂肪酸结合蛋白1 fatty acid binding protein 1;FABP3:脂肪酸结合蛋白3 fatty acid binding protein 3;CPT1:肉毒碱棕榈酰基转移酶1 carnitine palmitoyltransferase 1;CPT2:肉毒碱棕榈酰基转移酶2 carnitine palmitoyltransferase 2;unsaturated fatty acid:不饱和脂肪酸;saturated fatty acid:饱和脂肪酸;fibrate drug:贝特类药物;ketogenesis:生酮作用;lipid transport:脂质运输;fatty acid transport:脂肪酸转运;fatty acid oxidation:脂肪酸氧化。 图 2 PPARs信号途径示意图 Figure 2 The sketch map of signal transduction pathways of PPARs[10-11] |
目前,国内外许多学者对PPARs在人类和单胃动物中的功能做了大量研究。PPAR-α、PPAR-β以及PPAR-γ在动物机体组织中的分布存在差异,其生理功能也有很大不同。PPAR-α是最早在哺乳动物上研究的PPARs亚型,在肝脏、心脏和肾脏高度表达,主要参与线粒体内脂肪酸的转运与氧化、能量代谢和氧化应激[12]。PPAR-β在骨骼细胞中高度表达,主要参与脂肪酸的代谢。PPAR-β与PPAR-α具有相似的作用,也参与调控瘤胃上皮的增殖分化,且PPAR-β在瘤胃的表达量要显著高于PPAR-α[13],提示PPAR-β在调控瘤胃上皮细胞增殖中可能发挥更重要的作用,但还需进一步研究证实。在人类癌症的研究中,PPAR-γ在肾癌细胞中表达量显著升高,而正常肾癌细胞中表达量正常[14],提示PPAR-γ在调控细胞增殖过程中可能发挥调控作用。PPAR-γ在反刍动物的瘤胃中同样检测到表达且表达量较PPAR-α高[15]。但PPAR-γ在反刍动物瘤胃中是否发挥调控上皮细胞增殖的作用还有待考究。近年来,PPARs在反刍动物上的作用受到许多专家学者的重视,尤其是PPARs在脂代谢中的作用。脂肪酸及脂肪酸衍生物都可以作为PPARs的配体,激活PPARs。PPARs在脂质代谢和碳水化合物代谢中承担至关重要的角色[16],与特异性配体结合后可以促进脂肪细胞的增殖[17]。PPAR-α在反刍动物上研究相对较少,PPAR-α在奶牛的肾脏和肝脏大量表达,而瘤胃中表达量较少[15]。早在16年前,在山羊上的研究发现,PPAR-α参与了肝脏中的脂肪酸氧化[18]。随后,Schlegel等[19]进一步研究发现PPAR-α通过调控肉毒碱棕榈酰基转移酶1A(carnitine palmitoyltransferase 1A, CPT1A)的表达调节脂肪酸代谢。相比于PPAR-α,虽有研究表明PPAR-β也参与脂肪酸的氧化[20],但还有待于考证。PPAR-γ在反刍动物上研究最多,其在脂肪组织中高度表达,它与脂肪的生成[21]和长链脂肪酸的氧化有关[22]。
3 PPARs调控瘤胃生酮作用的分子机制反刍动物在瘤胃发育的过程中,酮体生成量不断增加,PPARs被配体激活后调控下游靶基因的表达,促进瘤胃生酮和瘤胃上皮细胞的增殖与分化。近年来,随着基因表达谱分析的广泛应用,许多研究发现,伴随瘤胃乳头的生长加快,出现了许多差异表达基因。其中,HMGCS2、ACAT1和脂肪酸结合蛋白3(fatty acid binding protein 3, FABP3)等基因的启动子区域都包含1个PPRE的反应元件,这些基因的表达受到PPAR-α的调控[22]。因此,PPAR-α在瘤胃发育的过程中,具有至关重要的调控作用。Kinoshita等[5]研究发现,VFA可以影响瘤胃的生酮作用以及生酮基因的表达,因此Penner等[23]提出一个假设,在瘤胃从利用葡萄糖转变为利用丁酸生酮的过程中,VFA影响PPAR-α表达进而调控生酮作用。Connor等[24]在探索瘤胃发育的分子机制时,发现PPAR-α与瘤胃上皮细胞的增殖与分化有关。此外,PPAR-α的另一个靶基因——成纤维生长因子21(fibroblast growth factor 21, FGF21)在能量代谢中起重要作用。在小鼠上的研究发现,用PPAR-α的激动剂处理,小鼠的FGF21表达量升高,而FGF21能够使肝脏中CPT1A和HMGCS2的表达量升高,促进生酮作用的发生[25]。但在幼龄反刍动物瘤胃中PPAR-α是否能够诱导FGF21的表达,FGF21是否促进瘤胃中HMGCS2的表达进而促进瘤胃生酮还不曾得知。
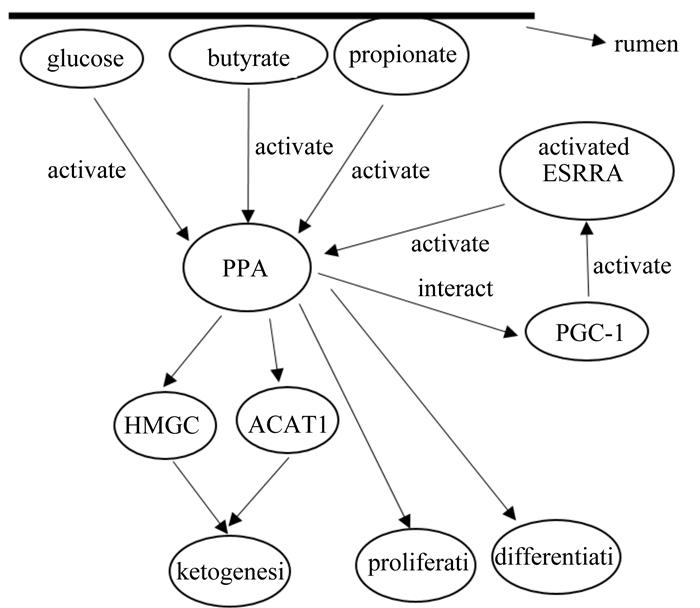
对于单胃动物,人工合成的WY-14643是PPAR-α的特异性配体[26],L165041是PPAR-β的特异性配体[27],TZD是PPAR-γ的特异性配体[28],这些配体都能与PPARs特异性结合,激活PPARs,发挥基因调控作用。而对于反刍动物来说,其体内本身可能存在PPARs的特异性配体。瘤胃内的葡萄糖以及发酵产生的长链脂肪酸(long-chain fatty acid, LCFA)、丙酸、丁酸都有可能作为PPARs的特异性配体激活PPARs,调控靶基因的表达,促进瘤胃生酮作用和瘤胃乳头的发育(图 3)[15, 29-33],但仍待研究证实。在反刍动物体内LCFA是否为真正意义上的PPARs配体还尚待考究,因为在小鼠上研究表明,肝脏中LCFA通过激活其他转录因子来发挥转录调控作用,在肝脏中除PPARs外,肝细胞核因子-4(hepatic nuclear factor 4, HNF4)、肝孤儿受体(liver X receptor, LXR)都可以结合LCFA[31]。此外,研究发现,雌激素相关受体(estrogen-related receptors, ESRRA)可以直接激活PPAR-α的基因表达[32]。Connor等[24]在犊牛上的研究表明,激活的ESRRA进一步激活PPAR-α来调节瘤胃生酮作用。PPAR-α和PPAR-γ为过氧化物酶体增殖物激活受体-γ共激活因子-1(peroxisome proliferator-activated receptor-gamma coactivator-1, PGC-1)的调节因子,而PGC-1是ESRRA的调节因子。因此,PPAR-α和PPAR-γ通过与PGC-1的互作来激活ESRRA(图 3)[33]。阐明PPAR、ESRRA与PGC-1之间的相互关系对于理解PPAR与瘤胃生酮作用来说非常重要。
|
rumen:瘤胃;activated ESRRA:活化的雌激素相关受体activated estrogen-related receptors;PGC-1:过氧化物酶体增殖物激活受体-γ共激活因子-1 peroxisome proliferator-activated receptor-gamma coactivator-1;HMGCS2:3-羟基3-甲基戊二酰辅酶A合成酶2 3-hydroxy-3-methylglutaryl coenzyme A synthase 2;ACAT1:胆固醇酰基转移1 cholesterol acyltransferase 1;glucose:葡萄糖;butyrate:丁酸;propionate:丙酸:ketogenesis:生酮作用;proliferation:增殖;differentiation:分化;activate:活化;interact:互作。 图 3 PPARs调控瘤胃生酮与发育 Figure 3 The regulation of ketogenosis and rumen development by PPARs[15, 29-33] |
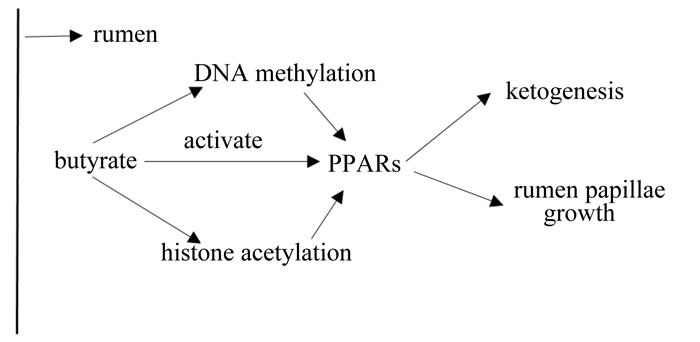
表观遗传学是指除DNA序列以外的变化(环境因素)引起的表型及基因表达改变的分子机制,包括DNA的甲基化、组蛋白修饰、非编码RNA和染色质重塑等现象[34]。在小鼠上的研究发现,丁酸可以激活PPAR-γ[5],且PPAR-α会通过DNA高甲基化或低甲基化被静默或被激活[6]。小鼠饲喂果糖后发现PPAR-α和CPT1A基因启动子区域的高甲基化,导致了PPAR-α和CPT1A的表达量降低,而饲喂甜菜碱显著降低小鼠PPAR-α启动子区甲基化水平,上调了PPAR-α及其靶基因的表达量。在大鼠上同样发现PPAR-α启动子区较低的DNA甲基化,上调PPAR-α的表达量,增加肝脏内脂肪酸的β氧化[35]。在人类[36]以及小鼠[37]的研究都表明,与脂代谢有关的基因表达会受到DNA甲基化的影响。在奶牛饲粮中补充蛋氨酸导致了DNA整体低甲基化和PPAR-α的特异区域的高甲基化,PPAR-α的表达上调[38]。因此,对幼龄反刍动物进行营养调控,与脂代谢和生酮作用相关的基因也会受到DNA甲基化的影响。DNA甲基化的高低影响到PPARs的激活进而影响靶基因的表达,且丁酸在幼龄反刍动物瘤胃发育过程中发挥重要作用。鉴于此,我们提出假设,丁酸调控瘤胃PPARs DNA甲基化,甲基化的改变改变了PPARs及其靶基因的表达,进而改变了瘤胃的生酮作用(图 4)[35-37]。组蛋白修饰引起的染色质重塑在真核生物基因的表达调控中发挥重要作用。组蛋白乙酰化由组蛋白乙酰化酶(histone acetylases, HATs)和组蛋白去乙酰化酶(histone deacetylase, HDAC)催化完成[39]。组蛋白的乙酰化能够激活基因的转录,去乙酰化能够抑制基因的转录。研究表明,丁酸能够使组蛋白高度乙酰化,具有HDAC抑制剂的作用[40]。因此,反刍动物瘤胃内发酵产生的丁酸很可能作为HDAC抑制剂影响PPARs的转录,进而影响到瘤胃的生酮作用和发育(图 4),但有待考证。目前,针对瘤胃发育过程中存在的表观遗传学机制研究甚少,对于调控瘤胃内PPARs表达的表观遗传学机制尚未有研究报道。
|
rumen:瘤胃;PPARs:过氧化物酶体增殖物激活受体peroxisome proliferator-activated receptors;butyrate:丁酸;DNA methylation:DNA甲基化;histone acetylation:组蛋白乙酰化;ketogenesis:生酮作用;rumen papillae growth:瘤胃乳头生长;activate:活化。 图 4 PPARs基因表达的表观遗传学调控机制 Figure 4 Epigenetic regulation of PPARs gene expression[35-37] |
生酮作用是瘤胃成熟的标志,PPARs是调控生酮作用和瘤胃发育的重要转录调节因子。目前仅发现了与生酮作用相关的以及受到PPARs调控的部分酶,在小鼠上研究发现丁酸可以激活PPARs,但丁酸在反刍动物瘤胃内是否激活PPARs尚未报道。PPARs调控瘤胃生酮作用的途径以及激活PPARs的因素尚未清楚,其可能的分子机制是:瘤胃中的葡萄糖以及发酵产生的VFA(尤其是丙酸和丁酸)作为配体激活PPARs,进而调控靶基因的表达。VFA(尤其是丁酸)影响瘤胃DNA的甲基化水平,调控PPARs及相关靶基因的表达,丁酸作为HDAC抑制剂影响PPARs的转录,进而影响到瘤胃的生酮作用和发育。但PPARs调控瘤胃生酮作用的途径以及激活PPARs的因素尚未清楚。PPARs的3个亚型在反刍动物瘤胃中的相对表达量仍需要进一步研究证实,3个亚型之间功能的相对重要性非常值得探索。
| [1] |
胡越, 许军, 刘燕华, 等. PPARα激动剂降血脂作用的研究进展[J]. 中国药科大学学报, 2016, 47(1): 118-124. DOI:10.11665/j.issn.1000-5048.20160118 |
| [2] |
金鹏, 陈保林, 杨永曜. PPARα/PGC-1α信号通路对阿霉素损伤h9c2心肌细胞能量代谢的调节[J]. 临床心血管病杂志, 2016, 32(4): 388-391. |
| [3] |
NAEEM A. Effect of age and dietary plane of nutrition on transcript profiles of ruminal tissue of Holstein bull calves[D]. Ph. D. Thesis. Urbana: University of Illinois at Urbana-Champaign, 2012.
|
| [4] |
WANG W M, CHONG L, LI F D, et al. Effects of early feeding on the host rumen transcriptome and bacterial diversity in lambs[J]. Scientific Reports, 2016, 6: 32479. DOI:10.1038/srep32479 |
| [5] |
KINOSHITA M, SUZUKI Y, SAITO Y. Butyrate reduces colonic paracellular permeability by enhancing PPARγ activation[J]. Biochemical and Biophysical Research Communications, 2002, 293(2): 827-831. DOI:10.1016/S0006-291X(02)00294-2 |
| [6] |
WANG L J, CHEN L, TAN Y Z, et al. Betaine supplement alleviates hepatic triglyceride accumulation of apolipoprotein E deficient mice via reducing methylation of peroxisomal proliferator-activated receptor alpha promoter[J]. Lipids in Health and Disease, 2013, 12(1): 34. DOI:10.1186/1476-511X-12-34 |
| [7] |
JIAO J Z, LI X P, BEAUCHEMIN K A, et al. Rumen development process in goats as affected by supplemental feeding v.Grazing:age-related anatomic development, functional achievement and microbial colonisation[J]. British Journal of Nutrition, 2015, 113(6): 888-900. DOI:10.1017/S0007114514004413 |
| [8] |
GEORGIADI A, KERSTEN S. Mechanisms of gene regulation by fatty acids[J]. Advances in Nutrition, 2012, 3(2): 127-134. DOI:10.3945/an.111.001602 |
| [9] |
林圣宸. 核受体PPARs家族与新型激动剂的结构与功能研究[D]. 博士学位论文. 厦门: 厦门大学, 2013.
|
| [10] |
WAKU T, SHIRAKI T, OYAMA T, et al. Structural insight into PPARgamma activation through covalent modification with endogenous fatty acids[J]. Journal of Molecular Biology, 2009, 385(1): 188-199. DOI:10.1016/j.jmb.2008.10.039 |
| [11] |
FERRÉ P. The biology of peroxisome proliferator-activated receptors:Relationship with lipid metabolism and insulin sensitivity[J]. Diabetes, 2004, 53(1S): 43-50. |
| [12] |
杨亚维, 袁杰. PPARα抗氧化应激作用的研究进展[J]. 东南大学学报(医学版), 2016, 35(1): 147-150. |
| [13] |
NAEEM A, DRACKLEY J K, STAMEY J, et al. Role of metabolic and cellular proliferation genes in ruminal development in response to enhanced plane of nutrition in neonatal Holstein calves[J]. Journal of Dairy Science, 2012, 95(4): 1807-1820. DOI:10.3168/jds.2011-4709 |
| [14] |
COLLET N, THÉOLEYRE S, RAGEUL J, et al. PPARγ is functionally expressed in clear cell renal cell carcinoma[J]. International Journal of Oncology, 2011, 38(3): 851-857. |
| [15] |
BIONAZ M, CHEN S W, KHAN M J, et al. Functional role of PPARs in ruminants:potential targets for fine-tuning metabolism during growth and lactation[J]. PPAR Research, 2013, 2013: 684159. |
| [16] |
PYPER S R, NAVIN V, YU S T, et al. PPARα:energy combustion, hypolipidemia, inflammation and cancer[J]. Nuclear Receptor Signaling, 2010, 8: e002. |
| [17] |
王丽, 那威, 王宇祥, 等. 鸡PPARγ基因的表达特性及其对脂肪细胞增殖分化的影响[J]. 遗传, 2012, 34(4): 454-464. |
| [18] |
CAPPON G D, LIU R C M, FRAME S R, et al. Effects of the rat hepatic peroxisome proliferator, wyeth 14, 643, on the lactating goat[J]. Drug and Chemical Toxicology, 2002, 25(3): 255-266. DOI:10.1081/DCT-120005888 |
| [19] |
SCHLEGEL G, KELLER J, HIRCHE F, et al. Expression of genes involved in hepatic carnitine synthesis and uptake in dairy cows in the transition period and at different stages of lactation[J]. BMC Veterinary Research, 2012, 8: 28. DOI:10.1186/1746-6148-8-28 |
| [20] |
BRENNAN K M, MICHAL J J, RAMSEY J J, et al. Body weight loss in beef cows:Ⅰ.The effect of increased β-oxidation on messenger ribonucleic acid levels of uncoupling proteins two and three and peroxisome proliferator-activated receptor in skeletal muscle[J]. Journal of Animal Science, 2009, 87(9): 2860-2866. DOI:10.2527/jas.2008-1302 |
| [21] |
GARCÍA-ROJAS P, ANTARAMIAN A, GONZÁLEZ-DÁVALOS L, et al. Induction of peroxisomal proliferator-activated receptor gamma and peroxisomal proliferator-activated receptor gamma coactivator 1 by unsaturated fatty acids, retinoic acid, and carotenoids in preadipocytes obtained from bovine white adipose tissue[J]. Journal of Animal Science, 2010, 88(5): 1801-1808. DOI:10.2527/jas.2009-2579 |
| [22] |
SHARMA S, SUN X T, RAFIKOV R, et al. PPAR-gamma regulates carnitine homeostasis and mitochondrial function in a lamb model of increased pulmonary blood flow[J]. PLoS One, 2012, 7(9): e41555. DOI:10.1371/journal.pone.0041555 |
| [23] |
PENNER G B, STEELE M A, ASCHENBACH J R, et al. Ruminant Nutrition Symposium:Molecular adaptation of ruminal epithelia to highly fermentable diets[J]. Journal of Animal Science, 2011, 89(4): 1108-1119. DOI:10.2527/jas.2010-3378 |
| [24] |
CONNOR E E, BALDWIN R L, WALKER M P, et al. Transcriptional regulators transforming growth factor-β1 and estrogen-related receptor-α identified as putative mediators of calf rumen epithelial tissue development and function during weaning[J]. Journal of Dairy Science, 2014, 97(7): 4193-4207. DOI:10.3168/jds.2013-7471 |
| [25] |
LIN Z F, PAN X B, WU F, et al. Fibroblast growth factor 21 prevents atherosclerosis by suppression of hepatic sterol regulatory element-binding protein-2 and induction of adiponectin in mice[J]. Circulation, 2015, 131(21): 1861-1871. DOI:10.1161/CIRCULATIONAHA.115.015308 |
| [26] |
OKINE B N, SPICER C, MILLNS P, et al. Systemic administration of WY-14643, a selective synthetic agonist of peroxisome proliferator activator receptor-alpha, alters spinal neuronal firing in a rodent model of neuropathic pain[J]. Scandinavian Journal of Pain, 2015, 9: 42-48. |
| [27] |
沈桂佳, 安鹏, 聂玉强, 等. PPAR-β/δ及其激动剂l165041对小鼠肝脏成瘤的影响[J]. 胃肠病学和肝病学杂志, 2015, 24(11): 1350-1353. |
| [28] |
HARMS M, SEALE P. Brown and beige fat:development, function and therapeutic potential[J]. Nature Medicine, 2013, 19(10): 1252-1263. DOI:10.1038/nm.3361 |
| [29] |
RUBY M A, GOLDENSON B, ORASANU G, et al. VLDL hydrolysis by LPL activates PPAR-α through generation of unbound fatty acids[J]. Journal of Lipid Research, 2010, 51(8): 2275-2281. DOI:10.1194/jlr.M005561 |
| [30] |
DENGLER F, RACKWITZ R, BENESCH F, et al. Both butyrate incubation and hypoxia upregulate genes involved in the ruminal transport of SCFA and their metabolites[J]. Journal of Animal Physiology and Animal Nutrition, 2015, 99(2): 379-390. DOI:10.1111/jpn.2015.99.issue-2 |
| [31] |
KHAN S A, HEUVEL J P V. Reviews:current topicsrole of nuclear receptors in the regulation of gene expression by dietary fatty acids (review)[J]. Journal of Nutritional Biochemistry, 2003, 14(10): 554-567. DOI:10.1016/S0955-2863(03)00098-6 |
| [32] |
HUSS J M, TORRA I P, STAELS B, et al. Estrogen-related receptor alpha directs peroxisome proliferator-activated receptor alpha signaling in the transcriptional control of energy metabolism in cardiac and skeletal muscle[J]. Molecular & Cellular Biology, 2004, 24(20): 9079-9091. |
| [33] |
SHAO D, LIU Y, LIU X J, et al. PGC-1β-Regulated mitochondrial biogenesis and function in myotubes is mediated by NRF-1 and ERRα[J]. Mitochondrion, 2010, 10(5): 516-527. DOI:10.1016/j.mito.2010.05.012 |
| [34] |
丁勇, 许超, 吴季辉, 等. 表观遗传学研究进展[J]. 中国科学(生命科学), 2017, 47(1): 3-15. |
| [35] |
唐丹, 周永健, 李瑜元, 等. 姜黄素对脂肪变性肝细胞过氧化物酶体增殖物活化受体-α的去甲基化作用[J]. 广东医学, 2014, 35(2): 175-179. |
| [36] |
KIRCHNER H, SINHA I, GAO H, et al. Altered DNA methylation of glycolytic and lipogenic genes in liver from obese and type 2 diabetic patients[J]. Molecular Metabolism, 2016, 5(3): 171-183. DOI:10.1016/j.molmet.2015.12.004 |
| [37] |
OHASHI K, MUNETSUNA E, YAMADA H, et al. High fructose consumption induces DNA methylation at PPARα and CPT1A promoter regions in the rat liver[J]. Biochemical & Biophysical Research Communications, 2015, 468(1/2): 185-189. |
| [38] |
OSORIO J S, JACOMETO C B, ZHOU Z, et al. Hepatic global DNA and peroxisome proliferator-activated receptor alpha promoter methylation are altered in peripartal dairy cows fed rumen-protected methionine[J]. Journal of Dairy Science, 2016, 99(1): 234-244. DOI:10.3168/jds.2015-10157 |
| [39] |
王洁, 张心怡, 郝彩丽, 等. 组蛋白去乙酰化酶抑制剂的研究进展[J]. 现代生物医学进展, 2014, 14(14): 2783-2785. |
| [40] |
MYZAK M C, DASHWOOD R H. Histone deacetylases as targets for dietary cancer preventive agents:Lessons learned with butyrate, diallyl disulfide, and sulforaphane[J]. Current Drug Targets, 2006, 7(4): 443-452. DOI:10.2174/138945006776359467 |




