在反刍动物饲粮中,纤维成分占有很大的比重。它在促进瘤胃蠕动,维持瘤胃正常酸碱环境等方面有着不可替代的作用。以往虽然许多学者都曾研究过纤维的生理功能,但是对纤维的定义并不科学也不统一。现在人们普遍认为,相比其他纤维指标而言,中性洗涤纤维(NDF)是表示动物饲粮纤维水平最好的指标,因为它几乎涵盖了组成纤维的所有成分[1]。NDF不仅具有维护动物胃肠道健康的作用,其降解产物还为动物的生长发育提供了大量能量。但反刍动物本身并不具有消化NDF的能力,其对纤维的利用主要依赖于瘤胃内寄生的微生物。瘤胃微生物的结构与组成决定了宿主对NDF的消化利用程度,而宿主进食的NDF也是瘤胃微生物赖于生长繁殖的重要底物。以往虽然已有饲粮纤维水平对瘤胃微生物影响的研究,但这些研究主要采用的是传统的培养技术和变性梯度电泳等指纹图谱技术[2-6]。培养技术只能分析能在体外培养基中生长的微生物,但瘤胃中99%左右的微生物到目前为止还不能进行体外培养。而指纹图谱技术不仅费时费力,且分辨率低,仅能检测出样品中10%左右的优势微生物。这些技术都严重低估了瘤胃微生物的多样性,因此饲粮NDF水平影响瘤胃微生物结构与组成的规律还不尽为人所知。针对这些问题,本研究拟采用最新的Illumina HiSeq 250PE高通量测序技术,全面揭示饲粮NDF水平对山羊瘤胃细菌结构与组成的影响,研究结果能提高人们对瘤胃微生物适应营养物质水平变化规律的认识,以及营养物质水平对瘤胃微生物多样性的影响等知识的理解,还可为今后通过调控瘤胃微生物促进瘤胃纤维的降解提供参考。
1 材料与方法 1.1 试验动物与饲养管理试验动物为6只健康雄性努比亚黑山羊,平均月龄为8月龄,平均体重为(28.33±3.77) kg。参照我国《肉羊饲养标准》(NY/T 816—2004),以每天每头增重0.1 kg为标准配制各组饲粮。采用3×3拉丁方试验,依据饲粮NDF水平分为低(35.01%,LN组)、中(40.10%,MN组)和高NDF水平组(45.16%,HN组), 每组2只。饲粮组成及营养水平见表 1。饲粮中的干物质(dry matter,DM)、粗蛋白质(crude protein,CP)、钙(calcium,Ca)、磷(phosphorus,P)等常规营养成分参照Chemists[7]的方法进行测定,NDF和酸性洗涤纤维(acid detergent fiber,ADF)含量参照Van Soest等[8]的方法进行测定。试验羊单栏饲养,每天分别于09:00及17:00分2次饲喂,自由饮水。
|
|
表 1 饲粮组成及营养水平(干物质基础) Table 1 Composition and nutrient levels of diets (DM basis) |
分3期进行饲养试验,每期试验20 d,其中预试期14 d,正试期6 d。参照文献[9]中的方法,每期饲养试验结束后于次日晨饲后2 h,将10 mm直径塑料管连接到真空泵,用开口器打开羊的口腔,将塑料管从羊口腔缓慢插入至瘤胃,抽取瘤胃内容物约50 mL,用便携式酸度计进行瘤胃液pH测定后,立即装入充满氮气的样品袋中,置于冰上。反复拍打样品袋以确保固相微生物充分进入液相,然后用4层纱布过滤得到瘤胃液,迅速投入液氮罐中,立即带回实验室转移至-80 ℃超低温冰箱保存备用。
1.3 瘤胃发酵参数检测瘤胃液氨态氮(NH3-N)浓度的测定:参照Broderick等[10]描述的方法进行测定。首先将采集的瘤胃内容物进行预处理;然后制作NH3-N标准曲线,建立线性拟合回归方程;接着把预处理后的待测离心瘤胃液充分混匀,再吸取80 μL加入到预先标好的测试管中,各管按照补加试剂顺序依次加入40 μL甲醇、2.5 mL苯酚、2.0 mL碱性次氯酸钠溶液,在37 ℃水浴10 min,取出后室温下放置10 min,于650 nm处采用酶标仪(美国Molecular Devices公司SpectramaxM2)测定吸光度值;最后将样品液的吸光度值代入线性拟合回归方程,计算出瘤胃液中NH3-N浓度。
瘤胃液挥发性脂肪酸(VFA)浓度的测定:参照Li等[11]描述的方法采用气相色谱分析仪(CP-3800,美国Varian公司)进行测定。首先将采集的瘤胃内容物进行预处理;然后向标准中间液中分别加入0.2 mL偏磷酸溶液,40 μL巴豆酸溶液,混匀,4 ℃放置30 min, 12 000×g离心10 min,接着取上清液0.1 mL加入0.9 mL甲醇,混匀后经0.22 μm有机滤膜过滤。用气相色谱分析仪对滤液中乙酸、丙酸、丁酸的含量进行测定。
1.4 总DNA的提取和高通量测序取200 μL瘤胃液样品,用天根生化科技(北京)有限公司生产的粪便基因组DNA提取试剂盒提取瘤胃细菌总DNA。采用细菌通用引物对(515F:GTGCCAGCMGCCGCGGTAA;806R:GGACTACHVGGGTWTCTAAT)[12],以提取的总DNA为模板针对细菌16S rRNA V4区域进行PCR扩增。采用如下50 μL反应体系:dNTP Mixture (10 mmol/L) 1 μL,上游、下游引物(10 μmol/L)各1.25 μL,总DNA(50 ng/μL) 1 μL,Taq DNA Polymerase(5 U/μL, 含Mg2+) 0.25 μL,10×Taq Buffer 5 μL,加双蒸水至50 μL。PCR反应参数如下:95 ℃预变性2 min;随后变性循环30次(95 ℃,30 s;55 ℃,30 s;72 ℃,30 s);72 ℃延伸5 min,10 ℃冷却。PCR产物待检测合格后送北京诺禾致源生物科技有限公司,采用Illumina HiSeq 250 PE平台进行高通量测序。
1.5 生物信息学分析参照Wright等[13]的方法,利用QIIME 1.8.0软件对测序原始数据进行初次质控,过滤掉低质量序列,去除Barcode和引物序列,然后参照Yáñez-Ruiz等[14]的方法,在Mothur软件中进行拼接;再在QIIME 1.8.0软件中使用Uparse模块将拼接的序列按97%的相似性聚类为运算分类单位(operational taxonomic unit, OTU),并挑选每个OTU中相对丰度最高的序列作为代表序列。将代表序列与RDP数据库(Release 11.1,http://rdp.cme.msu.edu/)比对并构建OTU表,用RDP Classifier将OTU代表性序列在微生物各分类水平进行物种注释,并绘制在门水平物种组成的柱状图。基于去除嵌合体和Singletons处理之后的OTU table、rep_set.tree文件及其抽样最大深度,计算α-多样性指数(Chao1、Shannon、observed species)并绘制各样品的OTU稀释曲线。对样品共享属进行分析,用R软件根据共享属的组成及其在各样品中所占的比例,绘制样品间共享属的聚类热图。
1.6 数据统计分析试验结果用以下一般线性模型表示:
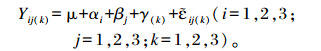
|
式中:Y为某指标的观测值; μ为试验全部观测值总体平均值;α、β、γ分别为本研究中试验期、羊组别和饲粮NDF水平; 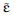
用SPSS 21.0软件中的ANOVA模块用以NDF水平为主要影响因素,且与其他2个影响因素无互作效应的方法对组间进行差异显著性检验,并用Duncan氏法进行多重比较。试验结果以平均值±标准差表示。以P<0.05表示差异显著,P<0.01为差异极显著。
2 结果与分析 2.1 饲粮NDF水平对山羊瘤胃发酵参数的影响从表 2可以看出,山羊瘤胃液pH在3组间差异不显著(P>0.05),但随着饲粮NDF水平的增加有升高趋势;HN组瘤胃液NH3-N浓度极显著低于LN组和MN组(P<0.01),LN组和MN组间的差异不显著(P>0.05);各组间瘤胃液乙酸、丙酸、丁酸及总挥发性脂肪酸(TVFA)浓度差异均不显著(P>0.05),乙酸/丙酸有随着饲粮NDF水平增加而升高的趋势,且LN组和HN组间的差异达到了显著水平(P<0.05)。
|
|
表 2 饲粮NDF水平对山羊瘤胃发酵参数的影响 Table 2 Effects of dietary NDF level on rumen fermentation parameters in goats |
本次试验共得到了1 125 746条有效序列(clean data),平均每个样品含(62 541±9 024)条。将有效序列进行聚类,共得到17 198个OTU。3组间共享OTU数为1 012个,LN组与MN组间共享OTU数为1 197个,LN组与HN组间共享OTU数为1 083个,MN组与HN组间共享OTU数为1 083个(图 1)。
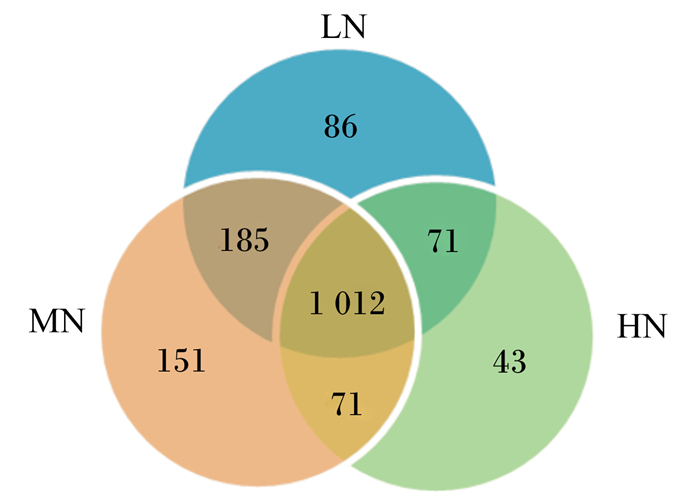
|
图 1 OTU维恩图 Figure 1 Venn diagram of OTU |
各样品的稀释曲线见图 2。由图可见,在本试验的测序深度下(reads=30 154),各样品稀释曲线最终均趋于平缓可以,说明本试验的测序深度覆盖各样品的绝大多数微生物。
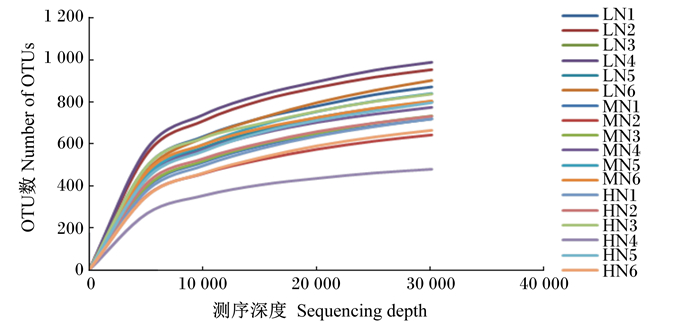
|
图例由组别和样品编号构成。 Legends consisted of group name and sample No. 图 2 样品的稀释曲线 Figure 2 Rarefaction curves of samples |
在取样深度为30 154时对山羊瘤胃细菌的α-多样性指数进行组间差异分析,结果见表 3。Chao1指数和Shannon指数在组间差异均不显著(P>0.05),LN组observed species指数显著高于其他2组(P<0.05),其他2组间则无显著差异(P>0.05)。
|
|
表 3 在取样深度为30 154时各组α多样性指数的对比 Table 3 Comparison of α diversity indexes among groups calculated at a depth of 30 154 |
将本试验所得有效序列在不同分类水平上进行物种注释,结果共得到23个门,44个纲,71个目,121个科,225个属。由图 3可以看出,在门水平上,3组间物种相对丰度差异均不显著(P>0.05)。拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)在3组中均为优势菌门,其次依次为黏胶球形菌门(Lentisphaerae)、软壁菌门(Tenericutes)。
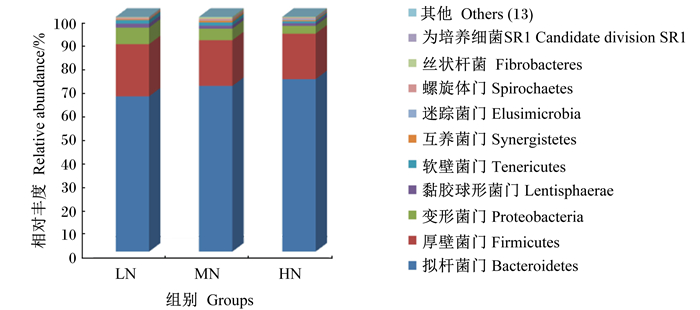
|
图 3 瘤胃细菌在门水平上的组成(相对丰度前10) Figure 3 Composition of rumen bacteria at phylum level (top 10 of relative abundance) |
在属水平上,3组的前2个优势属均依次为普雷沃氏菌属1(Prevotella 1)和理研菌属RC9(Rikenellaceae RC9 gut group)。将225个属中相对丰度低于1%的属聚为其他(215个)后,各样本在属水平上的物种组成见图 4。表 4列出了在属水平上组间相对丰度有显著差异的细菌。普雷沃氏菌科UCG-001(Prevotellaceae UCG-001)、普雷沃氏菌科UCG-003(Prevotellaceae UCG-003)和瘤胃球菌科UCG-014(Ruminococcaceae UCG-014)的相对丰度呈现随饲粮NDF水平增加而升高的变化规律,且HN组显著高于其他2组(P<0.05),其他2组间无显著差异(P>0.05);瘤胃球菌科NK4A214(Ruminococcaceae NK4A214 group)、瘤胃球菌科UCG-005(Ruminococcaceae UCG-005)的相对丰度呈现随饲粮NDF水平增加而升高,且HN组显著高于LN组(P<0.05);SP3-e08和Lachnoclostridium 10的相对丰度呈现随NDF水平增加而降低的变化规律,且HN组显著低于其他2组(P<0.05),其他2组无显著差异(P>0.05);LN组中解琥珀酸菌属(Succiniclasticum)的相对丰度显著高于其他2组(P<0.05),其他2组无显著差异(P>0.05);MN组中赖氨酸芽孢杆菌属(Lysinibacillus)、芽孢杆菌属(Bacillus)和叶杆菌属(Phyllobacterium)的相对丰度显著高于其他2组(P<0.05),食物谷菌属(Victivallis)的相对丰度极显著高于其他2组(P<0.01),其他2组间无显著差异(P>0.05);LN组[Eubacterium] ruminantium group的相对丰度显著高于HN组(P<0.05),但与MN组比较无显著差异(P>0.05)。
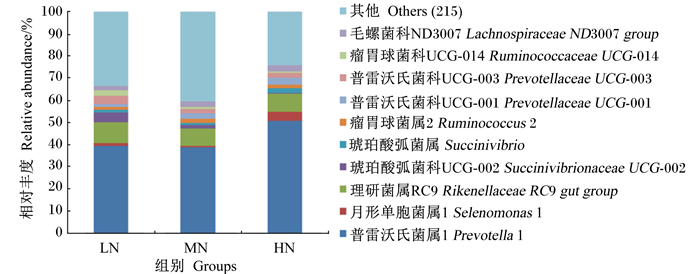
|
图 4 瘤胃细菌在属水平上的组成 Figure 4 Composition of rumen bacteria at genus level |
|
|
表 4 各组相对丰度存在显著差异的属 Table 4 Genus with significantly difference among groups |
经统计,所有样品间共有35个共享属。其中,相对含量在1%以上的主要共享菌属由高到低依次为Prevotella 1[(37.29 ±7.27)%]、Rikenellaceae RC9 gut group[(7.30±0.54)%]、Prevotellaceae UCG-003[(2.31±0.88)%]、毛螺菌科ND3007(Lachnospiraceae ND3007 group)[(2.19±0.52)%]、Prevotellaceae UCG-001[(2.04±0.93)%]、琥珀酸弧菌科UCG-002(Succinivibrionaceae UCG-002)[(1.90±1.75)%]、月形单胞菌属1(Selenomonas 1)[(1.79±4.24)%]、瘤胃球菌属2(Ruminococcus 2)[(1.35±1.77)%]、琥珀酸弧菌属(Succinivibrio)[(1.20±0.58)%]、Ruminococcaceae UCG-014[(1.13±0.81)%]。这些菌属的比例占总菌属的56.50%。共享菌群在属水平上的聚类热图见图 5。
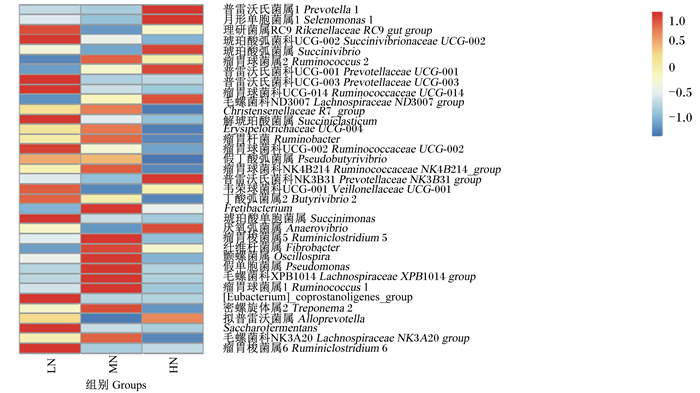
|
图 5 共享菌群在属水平上的聚类热图 Figure 5 Clustering heatmap of bacteria community at genus level |
由于NDF具有调控和维持反刍动物瘤胃正常发酵的作用,所以不同饲粮NDF水平会影响瘤胃的发酵模式。那仁巴图等[15]采用完全随机试验设计在6个饲粮NDF水平(49%、52%、55%、59%、62%和65%)下研究了内蒙古白绒山羊羯羊的瘤胃发酵的变化。结果发现,饲粮NDF水平对瘤胃液pH、NH3-N浓度均有显著影响,但对瘤胃液微生物蛋白(MCP)和乙酸、丙酸和丁酸浓度的影响不显著。王海荣等[16]也分别在3个不同饲粮NDF水平(42.71%、54.59%、64.38%)下研究了苏尼特绵羊瘤胃内环境的变化,结果发现随着NDF水平的增高,绵羊瘤胃液NH3-N浓度会显著降低,瘤胃液乙酸/丙酸显著升高,与本试验结果相一致。
本次研究发现Ruminococcaceae NK4A214 group、Ruminococcaceae UCG-005和Ruminococcaceae UCG-014的相对丰度随饲粮NDF水平增加而升高的变化规律。这些细菌都属于瘤胃球菌科(Ruminococcaceae),以往的研究表明瘤胃菌科微生物与纤维降解密切相关,在瘤胃内可产生纤维酶降解纤维二糖等纤维类物质,是一类典型的纤维降解菌[17-18]。Patra等[19]的研究表明,瘤胃菌科的数量减少会导致纤维消化率降低。Zhao等[20]的研究也证实,瘤胃菌科的微生物与饲粮NDF的消化率显著相关。本研究的结果表明,饲粮NDF水平的增加,山羊瘤胃菌科微生物的增殖加强,这可能是一种底物诱导效应。这也从证实了瘤胃菌科与瘤胃纤维降解的密切关系。
本试验中,Prevotellaceae UCG-001和Prevotellaceae UCG-003的相对丰度表现出随饲粮NDF水平增加而升高的变化规律,说明饲粮NDF水平的提高也能促进这2种细菌的生长。导致这种现象的原因可能与这些细菌的特性有关。这2种细菌属于普雷沃氏菌属(Prevotella),以往体外培养的试验表明,它们具有消化蛋白质和氨基酸的活性,没有直接降解纤维的能力,但与纤维降解菌共培养时却能间接地促进纤维的降解[21-23]。Zhao等[20]在犊牛上的试验表明,饲粮纤维消化率的高低影响着这2种细菌的相对丰度,再结合本研究的结果可以推测,这2种细菌可能是重要的纤维降解协作菌,它们也可从纤维降解过程中获取养分,从而导致本研究中HN组这2种细菌的相对丰度显著高于其他2组。
本次研究还发现,解琥珀酸菌属的相对丰度在组间存在着显著差异。有研究报道,解琥珀酸菌属能发酵降解纤维或纤维二糖产生琥珀酸、乙酸和二氧化碳等,也是一种典型的纤维降解菌[25]。国内外许多学者的研究也发现,解琥珀酸菌属和纤维降解有密切的关系[25-26]。而本课题组以往的研究结果也表明,纤维消化率高的山羊解琥珀酸菌属的相对丰度显著高于纤维消化率低的山羊。根据该菌的特性从理论上分析,当饲粮纤维含量增加时该菌的相对丰度应升高。但本研究中,LN组的相对丰度却显著高于其他2组,造成这种现象的具体原因还不清楚,有可能与LN组饲粮中淀粉含量较高有关。淀粉是解琥珀酸菌属最主要的底物,与纤维相比,淀粉更能刺激解琥珀酸菌属的生长。
4 结论① 饲粮NDF水平在35.01%~45.16%变化时,随NDF水平增加,瘤胃液NH3-N浓度降低, 瘤胃液乙酸/丙酸升高。
② 山羊瘤胃的优势细菌是拟杆菌门、厚壁菌门和变形菌门。
③ 山羊瘤胃中共有13个菌属的相对丰度受饲粮NDF水平的显著影响,其中Prevotellaceae UCG-001、Prevotellaceae UCG-003、和Ruminococcaceae UCG-014等菌属的相对丰度呈现随NDF水平增加而升高的变化规律。
| [1] |
ANNISON G. The role of wheat non-starch polysaccharides in broiler nutrition[J]. Australian Journal of Agricultural Research, 1993, 44(3): 405-422. |
| [2] |
周祥. 日粮不同NDF水平对杂交水牛瘤胃细菌群落结构与瘤胃主要功能细菌的影响[D]. 硕士学业论文. 武汉: 华中农业大学, 2015. http://cdmd.cnki.com.cn/Article/CDMD-10504-1015380622.htm
|
| [3] |
JAMI E, MIZRAHI I. Composition and similarity of bovine rumen microbiota across individual animals[J]. PLoS One, 2012, 7(3): e33306. DOI:10.1371/journal.pone.0033306 |
| [4] |
PITTA D W, KUMAR S, VEICCHARELLI B, et al. Bacterial diversity associated with feeding dry forage at different dietary concentrations in the rumen contents of Mehshana buffalo (Bubalus bubalis) using 16S pyrotags[J]. Anaerobe, 2014, 25: 31-41. DOI:10.1016/j.anaerobe.2013.11.008 |
| [5] |
曹晨霞, 韩琬, 张和平. 第三代测序技术在微生物研究中的应用[J]. 微生物学通报, 2016, 43(10): 2269-2276. |
| [6] |
YANG L Y, CHEN J, CHENG X L, et al. Phylogenetic analysis of 16S rRNA gene sequences reveals rumen bacterial diversity in Yaks (Bos grunniens)[J]. Molecular Biology Reports, 2010, 37(1): 553-562. DOI:10.1007/s11033-009-9794-x |
| [7] |
AOAC. Official methods of analysis[S]. 15th ed. AOAC International, 1990.
|
| [8] |
VAN SOEST P J, ROBERTSON J B, LEWIS B A. Methods for dietary fiber, neutral detergent fiber, and nonstarch polysaccharides in relation to animal nutrition[J]. Journal of Dairy Science, 1991, 74(10): 3583-3597. DOI:10.3168/jds.S0022-0302(91)78551-2 |
| [9] |
WANG L Z, WANG Z S, XUE B, et al. Comparison of rumen archaeal diversity in adult and elderly yaks (Bos grunniens) using 16S rRNA gene high-throughput sequencing[J]. Journal of Integrative Agriculture, 2017, 16(5): 1130-1137. DOI:10.1016/S2095-3119(16)61454-5 |
| [10] |
BRODERICK G A, KANG J H. Automated simultaneous determination of ammonia and total amino acids in ruminal fluid and in vitro media[J]. Journal of Dairy Science, 1980, 63(1): 64-75. DOI:10.3168/jds.S0022-0302(80)82888-8 |
| [11] |
LI Y L, MENG Q X. Effect of different types of fibre supplemented with sunflower oil on ruminal fermentation and production of conjugated linoleic acids in vitro[J]. Archives of Animal Nutrition, 2006, 60(5): 402-411. DOI:10.1080/17450390600884401 |
| [12] |
CAPORASO J G, LAUBER C L, WALTERS W A, et al. Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(Suppl.1): 4516-4522. |
| [13] |
WRIGHT A D G, WILLIAMS A J, WINDER B, et al. Molecular diversity of rumen Methanogens from sheep in western Australia[J]. Applied and Environmental Microbiology, 2004, 70(3): 1263-1270. DOI:10.1128/AEM.70.3.1263-1270.2004 |
| [14] |
YÁÑEZ-RUIZ D R, MACÍAS B, PINLOCHE E, et al. The persistence of bacterial and Methanogenic archaeal communities residing in the rumen of young lambs[J]. FEMS Microbiology Ecology, 2010, 72(2): 272-278. DOI:10.1111/fem.2010.72.issue-2 |
| [15] |
那仁巴图, 贾志海, 卢德勋, 等. 不同纤维水平口粮对内蒙古白绒山羊瘤胃发酵的影响[J]. 中国畜牧杂志, 2008, 44(3): 27-30. |
| [16] |
王海荣, 侯先志, 王贞贞, 等. 不同纤维水平日粮对绵羊瘤胃内环境的影响[J]. 内蒙古农业大学学报(自然科学版), 2008, 29(3): 9-14. |
| [17] |
WOOD T M. Cellulase of Ruminococcus albus[J]. Methods in Enzymology, 1988, 160: 216-221. DOI:10.1016/0076-6879(88)60123-6 |
| [18] |
EZAKI T. Ruminococcus[M]. New York: John Wiley & Sons, Ltd, 2015.
|
| [19] |
PATRA A K, YU Z T. Essential oils affect populations of some rumen bacteria in vitro as revealed by microarray (rumen bact array) analysis[J]. Frontiers in Microbiology, 2015, 6: 297. |
| [20] |
ZHAO X H, CHEN Z D, ZHOU S, et al. Effects of daidzein on performance, serum metabolites, nutrient digestibility, and fecal bacterial community in bull calves[J]. Animal Feed Science and Technology, 2017, 225: 87-96. DOI:10.1016/j.anifeedsci.2017.01.014 |
| [21] |
SCHWINGEL W R, BATES D B. Use of sodium dodecyl sulfate polyacrylamide gel electrophoresis to measure degradation of soluble soybean proteins by Prevotella ruminicola GA33 or mixed ruminal microbes in vitro[J]. Journal of Animal Science, 1996, 74(2): 475-482. DOI:10.2527/1996.742475x |
| [22] |
BEKELE A Z, KOIKE S, KOBAYASHI Y. Genetic diversity and diet specificity of ruminal Prevotella revealed by 16S rRNA gene-based analysis[J]. FEMS Microbiology Letters, 2010, 305(1): 49-57. DOI:10.1111/fml.2010.305.issue-1 |
| [23] |
ROSENBERG E. The family Prevotellaceae[M]//ROSENBERG E, DELONG E F, LORY S, et al. The Prokaryotes. Berlin: Springer, 2014.
|
| [24] |
RAINEY F A. Succiniclasticum[M]//ONYENWOKE R U, WIEGEL J. Bergey's manual of systematics of archaea and bacteria. New York: John Wiley & Sons, Ltd., 2015.
|
| [25] |
AN D D, DONG X Z, DONG Z Y. Prokaryote diversity in the rumen of yak (Bos grunniens) and Jinnan cattle (Bos taurus) estimated by 16s rDNA homology analyses[J]. Anaerobe, 2005, 11(4): 207-215. DOI:10.1016/j.anaerobe.2005.02.001 |
| [26] |
刘利, 唐纪良, 冯家勋. 广西水牛瘤胃中的细菌多样性[J]. 微生物学报, 2009, 49(2): 251-256. |




