功能性寡糖具有调控动物胃肠道微生物生长和改变微生物区系组成的作用,甘露寡糖是功能性寡糖的一种。研究发现甘露寡糖能够改善单胃动物肠道菌群结构,促进有益菌群增殖[1-2],增强免疫力[3],提高动物生产性能[4]。在犊牛饲粮中添加的甘露寡糖在通过小肠时,与侵入病原菌的碳水化合物基团结合,使病原菌既不能繁殖,又不能附着在犊牛的肠壁,被安全排出体外,而对乳酸菌等有益菌没有负面影响[5]。近几年研究人员在成年反刍动物上也进行了甘露寡糖应用研究,发现其在促进瘤胃发酵、瘤胃菌群结构等方面也具有积极作用。Heinrichs等[6]报道,甘露寡糖能够优化牛的胃肠道微生态环境,促进双歧杆菌等有益菌的增殖,抑制大肠杆菌等致病菌的增殖。刘立恒等[7]研究报道,饲粮添加甘露寡糖与其他寡糖组合后,牛瘤胃液细菌的PCR-变性梯度凝胶电泳(PCR-DGGE)图谱条带数明显增加,经测序,其中2个是产琥珀酸丝状杆菌属。甘露寡糖作为主要功能性寡糖在单胃动物上研究较多,目前在反刍动物上的应用研究也备受关注,但甘露寡糖如何改变瘤胃菌群结构目前报道不一。为此,本研究基于16S rDNA高通量测序分析技术,探讨添加甘露寡糖对奶牛瘤胃菌群结构与多样性的影响,为其在奶牛中的应用研究提供理论依据。
1 材料与方法 1.1 试验材料甘露寡糖购买于河南三化生物科技公司,纯度为99%。
1.2 试验动物与饲粮选择4头泌乳天数为20 d、日产奶量30 kg左右、体重为550 kg左右的经产(二胎)中国荷斯坦泌乳奶牛作为试验动物。根据NRC(2001)奶牛饲养标准配制饲粮,精粗比为40 : 60(DM基础),基础饲粮组成及营养水平见表 1。试验动物采用全混合日粮(TMR)饲喂方式,日投料3次,自由采食。全天自由饮水。
|
|
表 1 基础饲粮组成及营养水平(干物质基础) Table 1 Composition and nutrient levels of the basal diet (DM basis) |
将试验牛随机分为2组,对照组饲喂基础饲粮,试验组饲喂基础饲粮,同时添加60 g/头甘露寡糖(参照刘立恒等[7]),通过口腔灌注添加。采用2×2交叉试验设计,每期21 d,其中预试期14 d,采样期7 d。
1.4 样品采集与处理 1.4.1 瘤胃液采集通过牛口腔导管采集瘤胃液,准备牛鼻夹把牛头固定,在口腔中放入硬管(硬塑胶,内径要超过取样软管),插入深度不要超过咽部,外面露出20 cm左右,工作人员能够用手把其固定,然后把软管(2.5 m左右)通过口腔硬管内径逐步送入瘤胃,将牛头压低,通过牛的咀嚼过程使瘤胃液自然流出。
1.4.2 瘤胃液的处理将采集的瘤胃液转移至实验室内,用4层纱布过滤,将过滤后的瘤胃液等量混匀。pH直接用比色计测定;取20 mL瘤胃液于50 mL离心管中,然后将部分转移至含有3 mL 25%偏磷酸和0.6% 2-乙基丁酸的10 mL离心管,于-20 ℃的冰箱内保存,用于挥发性脂肪酸浓度的测定。挥发性脂肪酸浓度采用气相色谱法[9](Agilent-6890N气相色谱仪)测定;另取50 mL瘤胃液于离心管中,放置于-80 ℃冰箱冻存,用于菌群结构分析,共有8个样品,其中添加甘露寡糖的试验组4个样品(MT-1、MT-2、MT-3、MT-4),没有添加甘露寡糖的对照组4个样品(CK-1、CK-2、CK-3、CK-4)。
1.5 DNA样品提取、扩增 1.5.1 DNA提取DNA采用试剂盒提取,试剂盒采购于南京建成生物工程研究所,提取步骤参照试剂盒中的说明书操作。
1.5.2 PCR扩增PCR扩增前用琼脂糖凝胶电泳检测DNA样品的纯度和浓度,于离心管中取适量的样品进行稀释。将稀释后的基因组DNA作为模板。瘤胃液DNA扩增目的片段为V3+V4区,大概长度468 bp,V3+V4区引物为341F-806R[10],引物序列为341F:5′-CCTAYGGGRBGCASCAG-3′;806R:5′-GGACTACNNGGGTATCTAAT-3′。
1.6 PCR产物的混样和纯化PCR产物使用胶浓度为2%的琼脂糖凝胶进行电泳检测;依照PCR产物浓度的检测结果进行等浓度混样,充分混匀后使用胶浓度为2%的琼脂糖凝胶电泳检测PCR产物,使用南京建成生物工程研究所提供的回收试剂盒回收产物。
1.7 Miseq测序将样品送至广州基迪奥生物科技有限公司进行Miseq测序,测序采用Miseq 2500 PE 250平台。
1.8 数据统计分析数据采用Excel 2007进行初步处理,采用SAS 8.2统计软件中的两阶段交叉设计资料的方差分析进行数据处理。P<0.05为差异显著,P<0.01为差异极显著。
2 结果与分析 2.1 甘露寡糖对奶牛瘤胃发酵参数的影响由表 2可以看出,试验组奶牛瘤胃液pH与对照组相比提高了2.46%(P>0.05)。与对照组相比,添加甘露寡糖极显著提高了瘤胃液乙酸的浓度(P < 0.01),乙酸/丙酸提高了8.47%(P>0.05)。
|
|
表 2 甘露寡糖对奶牛瘤胃发酵参数的影响 Table 2 Effects of mannan oligosaccharides on fermentation parameters in the rumen of dairy cows |
对照组和试验组每组取4个样,共取8个瘤胃液样品。对8个瘤胃液样品总DNA进行质量检测,检测结果表明,浓度及纯度均符合Illumina Miseq平台测序要求,可以进行后续试验。对不同样品基因组DNA进行16S rDNA基因V3+V4区扩增,扩增的条带大小正确,浓度合适且条带单一,如图 1所示,适合后续测序试验。
|
9、10、17、18为对照组样品,1、2、25、26为试验组样品。 9, 10, 17 and 18 were samples of control group, 1, 2, 25 and 26 were samples of experimental group. 图 1 样本16Sr DNA基因V3+V4区域扩增结果 Figure 1 Amplification results of V3+V4 region of 16S rDNA gene of samples |
经Illumina Miseq测序结束后,除去低质量序列,通过序列拼接,应用Mothur,根据97%的序列相似度,对序列进行OTU划分。根据OTU聚类分析结果,分析不同样品的聚类信息,依照其共有、特有的OTU信息绘制韦恩图(Venn graph),由图 2可知,添加甘露寡糖后奶牛瘤胃液菌群多样性降低。
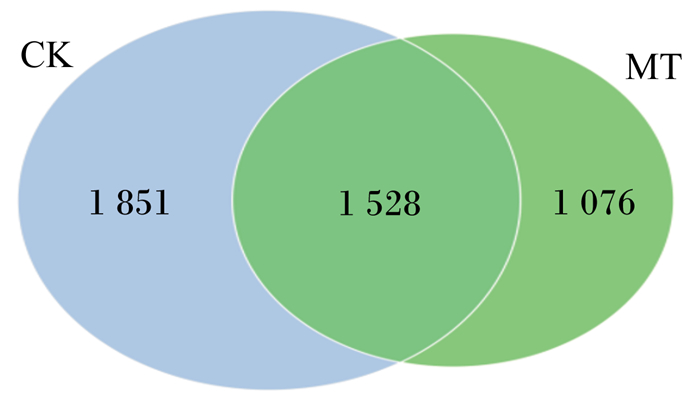
|
CK:对照组;MT:试验组。 CK: control group; MT: experimental group. 图 2 奶牛瘤胃液菌群韦恩图 Figure 2 Venn graph of microflora in rumen fluid of dairy cows |
为了检测测序得到的数据能否科学充分反映试验组与对照组瘤胃液菌群的分布情况,本试验根据获得的OTU数据,以随机抽取的序列数与OTU数量来构建曲线,做出每个样品的稀释曲线,可用来说明样本测序数据量是否足以反映物种多样性。从图 3、图 4中可以看出,测序深度较小时,OTU数量变化剧烈,OTU数量随着测序深度的增加而大幅增加,当测序深度达到5 000 reads时,稀释曲线仍有上升趋势,表明瘤胃内仍有新的细菌尚未被发现。当测序深度达5 000 reads时,Shannon曲线已达到饱和状态,说明测序趋向饱和,即当前的测序量足够进行样品菌群多样性分析。
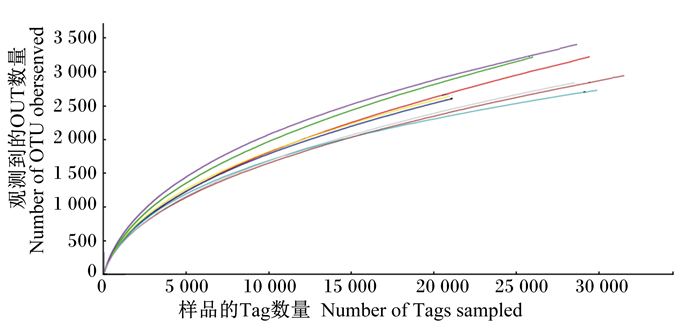
|
图 3 奶牛瘤胃细菌OTU稀释曲线 Figure 3 OTU dilution curve of bacteria in the rumen of dairy cows |
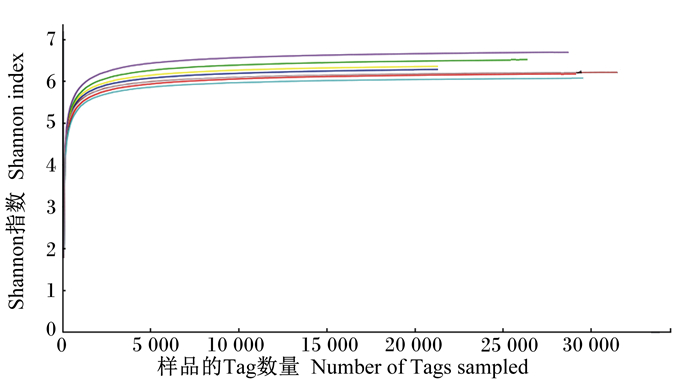
|
图 4 奶牛瘤胃细菌OTU Shannon稀释曲线 Figure 4 OTU Shannon dilution curve of bacteria in the rumen of dairy cows |
Alpha多样性是对单个样品中物种多样性的分析,包括Chao1指数、ACE指数、Shannon指数以及Simpson指数等[11]。Chao1指数和ACE指数是根据所测得的Tags数量和OTU的数量以及相对比例来预测样品中微生物的种类(OTU的数量),是基于已知结果所得相对值。Shannon指数是一个综合反映OTU丰度和OTU均匀度2方面因素的一个多样性指数,Shannon指数越大,越接近于0,则表示该样品中的物种越丰富。
由表 3可知,试验组的ACE指数和Chao1指数均低于对照组,说明添加甘露寡糖使得奶牛瘤胃中细菌丰富度指数降低;Shannon指数小于对照组,Simpson指数大于对照组,说明添加甘露寡糖使得奶牛瘤胃中细菌多样性降低,并且覆盖率均大于93%,说明样品采集足以反映瘤胃菌群情况。
|
|
表 3 样品多样性指数 Table 3 Diversity indexes of samples |
由图 5、表 4可知,奶牛瘤胃液中总共涉及29个门,包括拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、变形细菌门(Proteobacteria)、螺旋菌门(Spirochaetes)、蓝藻门(Cyanobacteria)、疣微菌门(Verrucomicrobi)、丝状杆菌门(Fibrobacteres)、软壁菌门(Tenericutes)、浮霉菌门(Planctomycetes)、放线菌门(Actinobacteria)、迷踪菌门(Elusimicrobia)、互养菌门(Synergistetes)、广古菌门(Euryarchaeota)、梭杆菌门(Fusobacteria)、绿弯菌门(Chloroflexi)、酸杆菌门(Acidobacteria)、装甲菌门(Armatimonadetes)等。其中拟杆菌门、厚壁菌门和变形细菌门占到总菌群比例的90%。与对照组相比,添加甘露寡糖极显著降低了奶牛瘤胃液中蓝藻门的相对丰度(P < 0.01),极显著提高了装甲菌门相对丰度(P<0.01);添加甘露寡糖可以降低瘤胃液中螺旋菌门、丝状杆菌门、梭杆菌门、酸杆菌门的相对丰度,但差异均不显著(P>0.05)。
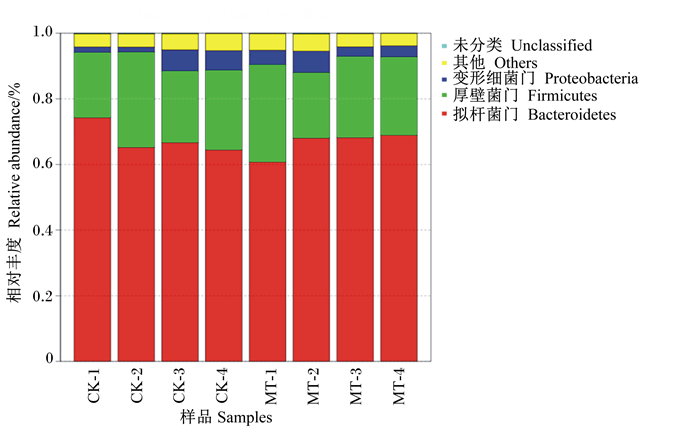
|
图 5 门水平上瘤胃菌群分布图 Figure 5 Distribution diagram of rumen microflora at phylum level |
|
|
表 4 甘露寡糖对奶牛瘤胃菌群在门水平相对丰度的影响 Table 4 Effects of mannan oligosaccharides on relative abundance in rumen microflora of dairy cows at phylum level |
进一步细化分类水平到属,本试验奶牛瘤胃液菌群总共涉及219个属种。由图 6和表 5可知,表中共显示丰度相对较高的25个属种占各样品总菌属91%以上,其中13个菌属的相对丰度较高,分别为普雷沃式菌属(Prevotella)、NA、S24-7_NA、未注释琥珀酸弧菌属(Succinivibrionaceae_NA)、未注释毛螺菌属(Lachnospiraceae_NA)、未注释瘤胃球菌属(Ruminococcaceae_NA)、未注释普雷沃氏菌属(Prevotellaceae_NA)、解琥珀酸弧菌属(Succiniclasticum)、瘤胃球菌属(Ruminococcu)、丁酸弧菌属(Butyrivibrio)、梅毒螺旋属(Treponema)、Shuttleworthia、粪球菌属(Coprococcus)。试验组锥形杆菌属(Pyramidobacter)的相对丰度极显著低于对照组(P < 0.01),梭菌属(Clostridium)的相对丰度显著低于对照组(P < 0.05)。试验组与纤维素降解有关的瘤胃球菌属、假丁酸弧菌属(Pseudobutyrivibrio)的相对丰度,较对照组分别增加了24.34%、12.83%(P>0.05)。试验组与半纤维素分解有关的厌氧螺菌属(Anaerobiospirillum)的相对丰度显著高于对照组(P < 0.05)。试验组未注释毛螺菌属相对丰度较对照组增加了8.16%(P>0.05)。试验组与淀粉降解有关的普雷沃氏菌属的相对丰度较对照组降低了4.66%(P> 0.05)。试验组利用乳酸为主的未注释韦荣氏菌属(Veillonellaceae_NA)的相对丰度较对照组提高了143.11%(P>0.05)。试验组氧化乳酸的脱硫弧菌属(Desulfovibrio)的相对丰度较对照组提高了4.88%(P>0.05)。
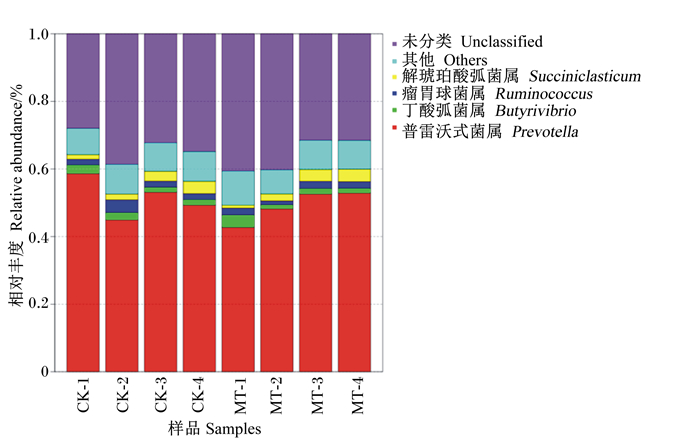
|
图 6 属水平上瘤胃菌群分布图 Figure 6 Distribution diagram of rumen microflora at genus level |
|
|
表 5 甘露寡糖对奶牛瘤胃菌群在属水平相对丰度的影响 Table 5 Effects of mannan oligosaccharides on relative abundance in rumen microflora of dairy cows at genus level |
利用LEFse软件对差异组间进行分析,LEFse先对所有组样品间进行kruskal-Wallis秩和检验(一种多样本比较时常用的检验方法),将筛选出的差异再通过wilcoxon秩和检验(一种两样本成组比较常用的检验方法)进行两两组间比较,最后筛选出的差异使用线性判断分析(LDA)得出的结果进行排序得到图 7-A。图 7-A展示了不同组中相对丰度差异显著的物种(LDA score大于预设值的显著差异物种),柱状图的长度代表差异物种的影响大小(即为LDA score)。随后通过将差异映射到已知层级结构的分类树上的方式得到进化分支图(图 7-B)。在进化分支图中,由内至外辐射的圆圈代表了由门至属(或种)的分类级别。在不同分类级别上的每一个小圆圈代表该水平下的一个分类,小圆圈直径大小与相对丰度大小呈正比。着色原则:无显著差异的物种统一着色为黄色。由图 7-A和图 7-B可知,试验组装甲菌门(Armatimonadetes)、RB046、SJA_176、金氏菌属(Kingella)和无胆甾原体目(Acholeplasmatales)的相对丰度较对照组显著增加;锥形杆菌属、蓝藻菌属(Cyanobacteria)、4C0d_2、YS2和瘤菌属(Ruminicola)的相对丰度较对照组显著降低。
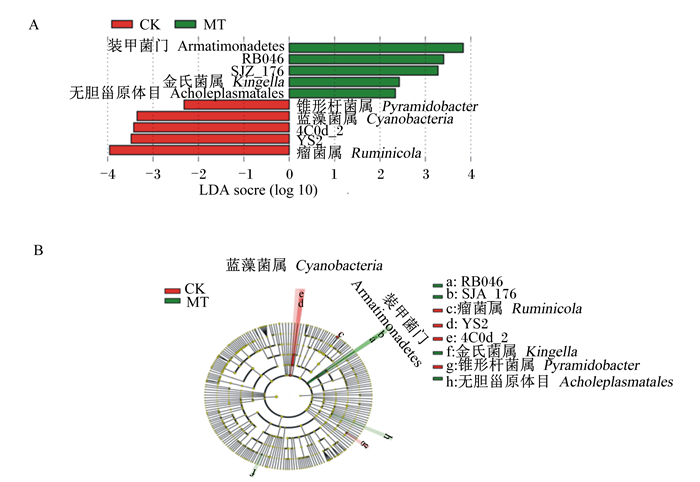
|
A :LDA值分布柱状图;B:进化分支图。CK:对照组;MT:试验组。 A: LDA value distribution histogram; B: cladogram. CK: control group; MT: experimental group. 图 7 LEFse差异分析图 Figure 7 LEFse difference analysis diagram |
瘤胃中的微生物是反刍动物不可缺少的消化菌群,瘤胃为微生物提供相对稳定的生存环境,瘤胃微生物主要包括细菌、原虫和真菌。其中细菌含量极其丰富,且种类繁多,大量的研究表明拟杆菌门和厚壁菌门是哺乳动物胃肠道的优势菌群[12-15]。本试验研究发现,添加甘露寡糖后奶牛瘤胃中菌群多样性降低。闵力[16]研究发现,添加功能性寡糖组合可以增加瘤胃液微生物种类,而且促进部分瘤胃液细菌的生长繁殖,使其成为优势菌群。本研究发现,添加甘露寡糖极显著降低了奶牛瘤胃液蓝藻门的相对丰度,极显著提高装甲菌门的相对丰度。Derakhshani[17]报道奶牛产后瘤胃装甲菌门丰度降低,其原因有可能是基于奶牛产后饲粮变化、能量负平衡等生理应激,产后奶牛饲粮精粗比增加,纤维比例降低,装甲菌门有可能以纤维素和半纤维素为碳源。但Lee[18]研究发现,非纤维碳水化合物能够显著促进装甲菌门细菌的生长。本试验结果表明,添加甘露寡糖可以促进装甲菌门菌群的增殖,甘露寡糖为β-葡聚糖,主要化学结构为β-1, 3葡聚糖和β-1, 6葡聚糖,完全不同于淀粉的主要化学结构, 也不同于纤维素的β-1, 4葡聚糖的化学结构。
粗饲料是反刍动物主要的饲料来源,粗饲料中纤维物质在瘤胃微生物与瘤胃的共同作用下快速降解, 转化为营养物质以满足反刍动物的能量需求[19]。细菌和真菌在分解利用纤维素过程中起着主要作用,细菌、原虫和真菌通过酶的催化使纤维素和半纤维素分解为瘤胃能够吸收的小分子物质[20],其中纤维素分解菌对纤维素的降解作用尤为重要。本试验研究发现,添加甘露寡糖使得与纤维素降解有关的瘤胃球菌属和假丁酸弧菌属的相对丰度分别增加24.34%、12.83%。通过瘤胃发酵参数可以看出,试验组奶牛瘤胃pH与对照组相比提高了2.46%, 为瘤胃纤维降解菌的增殖提供了适宜的pH。刘力恒等[7]添加功能性寡糖组合后,牛瘤胃液中产琥珀酸丝状杆菌属相对丰度增加,产琥珀酸丝状杆菌是瘤胃主要纤维降解菌。本研究发现,添加甘露寡糖显著提高了奶牛瘤胃中与半纤维素分解有关的厌氧螺菌属的相对丰度,试验组未注释毛螺菌属相对丰度较对照组增加了8.16%。闵力[16]研究报道,在添加甘露寡糖等功能性寡糖的组合后,锦江黄牛瘤胃液细菌PCR-DDGE产生了12个特异性条带,经测序分析,其中2株为产琥珀酸丝状杆菌,提高了瘤胃对纤维的降解率,提高粗饲料降解率。
奶牛瘤胃中广泛存在着降解和利用淀粉的普雷沃氏菌属[21]。本试验中,添加甘露寡糖后, 试验组奶牛瘤胃中与淀粉降解有关的普雷沃氏菌属相对丰度较对照组降低了4.66%。由本试验结果可以看出,普雷沃氏菌属相对丰度达到了49%以上,是瘤胃主要的细菌菌属。闵力[16]研究报道,在锦江黄牛的饲粮中添加功能性寡糖组合后瘤胃中增加了1株栖瘤胃普雷沃氏菌属,但并未见瘤胃普雷沃氏菌属丰度变化的报道。添加甘露寡糖之后,奶牛瘤胃梭菌属相对丰度极显著降低,通过本试验可以看出其相对丰度仅占0.5%左右,说明梭菌属并不是瘤胃主要菌属。虽然不是瘤胃中的主要细菌,但其在瘤胃中的种类多,有纤维降解梭菌(Clostridium fibrinolysis)、产气荚膜梭菌(Clostridium perfringens)和丁酸梭菌(Clostridium butyrate),还有菌株可降解蛋白质[22],而且从饲喂高含量淀粉的反刍动物瘤胃中可以分离得到丁酸梭菌[23],丁酸梭菌水解淀粉但不水解纤维素,水解淀粉和糖类的最终代谢产物为丁酸、乙酸和乳酸。与对照组相比,添加甘露寡糖极显著提高了瘤胃液乙酸的浓度,乙酸/丙酸提高了8.47%。同时本研究发现,与对照组相比,添加甘露寡糖后瘤胃中以利用乳酸为主的未注释韦荣氏菌属相对丰度提高了143.11%,氧化乳酸的脱硫弧菌属相对丰度提高了4.88%。与对照组相比,添加甘露寡糖极显著提高了瘤胃乙酸的浓度,但添加甘露寡糖后瘤胃pH比对照组提高了2.46%。与乙酸、丁酸等挥发性有机酸比较,乳酸的酸性更强,对瘤胃pH的贡献更大,甘露寡糖对于瘤胃pH的调控有可能是以利用淀粉为主的菌属与以利用乳酸为主的未注释韦荣氏菌属综合作用的结果,但具体机理还需进一步研究。
4 结论① 本试验条件下,添加甘露寡糖降低了奶牛瘤胃菌群的多样性,门水平上,蓝藻门相对丰度极显著降低,装甲菌门相对丰度极显著提高; 属水平上,与半纤维素分解有关的厌氧螺菌属的相对丰度显著提高,与淀粉降解有关的梭菌属相对丰度显著降低。
② 添加甘露寡糖可以增加与纤维降解有关的奶牛瘤胃主要菌属瘤胃球菌属、假丁酸弧菌属以及毛螺菌属的相对丰度,降低了与淀粉降解有关的普雷沃菌属相对丰度,提高了利用乳酸为主的未注释韦荣氏菌属以及氧化乳酸的脱硫弧菌属的相对丰度,但差异均不显著。
③ 添加甘露寡糖显著增加了奶牛瘤胃液乙酸浓度,提高了瘤胃液pH,但具体机理有待进一步研究。
| [1] |
GIANNENAS I, DOUKAS D, KARAMOUTSIOS A, et al. Effects of Enterococcus faecium, mannan oligosaccharide, benzoic acid and their mixture on growth performance, intestinal microbiota, intestinal morphology and blood lymphocyte subpopulations of fattening pigs[J]. Animal Feed Science and Technology, 2016, 220: 159-167. DOI:10.1016/j.anifeedsci.2016.08.003 |
| [2] |
唐志如, 孙志洪, 印遇龙, 等. 环保型饲料添加剂-寡糖的作用机理[J]. 中国畜牧兽医, 2003, 30(4): 17-19. DOI:10.3969/j.issn.1671-7236.2003.04.004 |
| [3] |
陈小兵, 丁宏标, 乔宇. 甘露寡糖的益生作用、免疫机制与应用技术[J]. 中国畜牧兽医, 2005, 32(8): 6-8. DOI:10.3969/j.issn.1671-7236.2005.08.002 |
| [4] |
PICCOLO G, CENTODUCATI G, MARONO S, et al. Effects of the partial substitution of fish meal by soy bean meal with or without mannanoligosaccharide and fructooligosaccharide on the growth and feed utilization of sharpsnout seabream, Diplodus puntazzo (Cetti, 1777):preliminary results[J]. Italian Journal of Animal Science, 2016, 10(3): e37. |
| [5] |
祁茹, 肖宇, 褚永康, 等. 外源寡糖对反刍动物胃肠道微生物区系的影响[J]. 饲料博览, 2011(12): 32-34. DOI:10.3969/j.issn.1001-0084.2011.12.013 |
| [6] |
HEINRICHS A J, JONES C M, HEINRICHS B S. Effects of mannan oligosaccharide or antibiotics in neonatal diets on health and growth of dairy calves[J]. Journal of Dairy Science, 2003, 86(12): 4064-4069. DOI:10.3168/jds.S0022-0302(03)74018-1 |
| [7] |
刘立恒, 闵力, 瞿明仁, 等. 甘露寡糖、果寡糖和大豆寡糖组合对锦江黄牛瘤胃液细菌多样性的影响[J]. 动物营养学报, 2012, 24(08): 1583-1588. DOI:10.3969/j.issn.1006-267x.2012.08.025 |
| [8] |
冯仰廉, 周建民, 张晓明, 等.我国奶牛饲料产奶净能值测算方法的研究[C]//第三届全国反刍动物营养学术研讨会论文集.北京: 中国畜牧兽医学会, 2011: 6.
|
| [9] |
徐晓锋.缩合单宁与水解单宁对奶牛日粮氮利用影响及作用机制的研究[D].博士学位论文.北京: 中国农业大学, 2013.
|
| [10] |
YU Y, LEE C, KIM J, et al. Group-specific primer and probe sets to detect methanogenic communities using quantitative real-time polymerase chain reaction[J]. Biotechnology & Bioengineering, 2010, 89(6): 670-679. |
| [11] |
SAR C, SANTOSO B, MWENYA B, et al. Manipulation of rumen methanogenesis by the combination of nitrate with β, 1-4 galacto-oligosaccharides or nisin in sheep[J]. Animal Feed Science and Technology, 2004, 115(1/2): 129-142. |
| [12] |
LEY R E, LOZUPONE C A, HAMADY M, et al. Worlds within worlds:evolution of the vertebrate gut microbiota[J]. Nature Reviews Microbiology, 2008, 6(10): 776-788. DOI:10.1038/nrmicro1978 |
| [13] |
OLIVEIRA M L S, ARÊAS A P M, CAMPOS I B, et al. Induction of systemic and mucosal immune response and decrease in Streptococcus pneumoniae colonization by nasal inoculation of mice with recombinant lactic acid bacteria expressing pneumococcal surface antigen A[J]. Microbes and Infection, 2006, 8(4): 1016-1024. DOI:10.1016/j.micinf.2005.10.020 |
| [14] |
QIN J J, LI R Q, RAES J, et al. A human gut microbial gene catalogue established by metagenomic sequencing[J]. Nature, 2010, 464(7285): 59-65. DOI:10.1038/nature08821 |
| [15] |
SINGH K M, AHIR V B, TRIPATHI A K, et al. Metagenomic analysis of Surti buffalo (Bubalus bubalis) rumen:a preliminary study[J]. Molecular Biology Reports, 2012, 39(4): 4841-4848. |
| [16] |
闵力.功能性寡糖组合对锦江黄牛瘤胃微生物区系的影响[D].硕士学位论文.南昌: 江西农业大学, 2012.
|
| [17] |
DERAKHSHANI H.The microbiome composition of the rumen is altered during the peripartal period in dairy cattle[C]//2014 ADSA-ASAS-CSAS Joint annual meeting.Kansas City: ASAS, 2014.
|
| [18] |
LEE C Y K.The first insights into the phylogeny, genomics, and ecology of the novel bacterial phylum armatimonadetes[D].Ph.D.Thesis.New Zealand: University of Waikato, Hamilton, 2015.
|
| [19] |
ASCHENBACH J R, PENNER G B, STUMPFF F, et al. Ruminant nutrition symposium:role of fermentation acid absorption in the regulation of ruminal pH[J]. Journal of Animal Science, 2011, 89(4): 1092-1107. DOI:10.2527/jas.2010-3301 |
| [20] |
ZEBELI Q, ASCHENBACH J R, TAFAJ M, et al. Invited review:role of physically effective fiber and estimation of dietary fiber adequacy in high-producing dairy cattle[J]. Journal of Dairy Science, 2012, 95(3): 1041-1056. DOI:10.3168/jds.2011-4421 |
| [21] |
PURUSHE J, FOUTS D E, MORRISON M, et al. Comparative genome analysis of Prevotella ruminicola and Prevotella bryantii:insights into their environmental niche[J]. Microbial Ecology, 2010, 60(4): 721-729. DOI:10.1007/s00248-010-9692-8 |
| [22] |
裴彩霞. 反刍动物瘤胃微生物及其利用[M]. 北京: 中国科学技术出版社, 2013: 9-10.
|
| [23] |
冯仰廉. 反刍动物营养学[M]. 北京: 科学出版社, 2006: 4-5.
|




