瘤胃是一个复杂的生态系统,内含多种多样的古细菌、真菌及原虫等,它们在维持瘤胃内环境稳定和影响奶牛健康方面起关键作用。这些瘤胃微生物具有高度特异性,它们消化代谢产生的能量可以被奶牛利用,对营养物质的消化起着关键的作用,还可能会提高宿主对入侵的病原体的抵抗力[1]。细菌是瘤胃中数量最多的一种微生物,其中分解粗饲料中的纤维物质主要是靠纤维素分解菌,如丝杆菌(Fibrobacter)、瘤胃球菌(Ruminococcus)、丁酸弧菌(Butyrivibrio)等。产琥珀酸丝状杆菌(Fibrobacter succinogenes)具有很强的分解纤维素能力,其发酵产物有乙酸和琥珀酸,这种菌对抗生素也有很强的耐受力。另外,拟杆菌门(Bacteroidetes)中的普雷沃氏菌属(Prevotella)是瘤胃优势菌属,它们可以分解蛋白质、淀粉、多糖,但不能分解纤维素。总之,多种微生物相互作用、相互制约,共同维持瘤胃内环境稳定,对于促进奶牛消化代谢、维持奶牛健康、提高生产性能具有重要意义。因此,对瘤胃微生物区系变化的研究是十分必要的。
产气法是应用较广、操作较简单、重复性较高的体外发酵方法之一,常用来评价饲料消化率、评估饲料添加剂效果等。大多数体外发酵试验研究底物精粗比和添加剂等对瘤胃微生物区系的影响[2-6],但是在正常饲粮作为发酵底物时,用体外发酵法研究瘤胃微生物区系随时间变化的报道较少。因此,了解产气法系统带来的瘤胃微生物区系变化,完善现有条件下的科研工作具有现实意义。
16S rRNA是细菌上编码rRNA相对应的DNA序列,存在于所有细菌的基因组中,具有高度的保守性和特异性。随着PCR技术的发展,16S rRNA测序已经成为分析微生物区系组成、研究生态系统多样性的重要方法,其准确性、全面性、精确性远优于其他传统的技术[7]。本试验旨在使用16S rRNA测序技术,分析奶牛体外发酵瘤胃微生物区系随时间推移的变化规律,为探究瘤胃微生物繁殖规律及完善体外发酵分析方法提供支持。
1 材料与方法 1.1 体外发酵产气系统建立发酵底物为江苏省南京西岗奶牛场提供的全混合日粮,精粗比为50 : 50,经烘干粉碎后,过20目筛,待用,饲粮组成及营养水平见表 1。
|
|
表 1 饲粮组成及营养水平(干物质基础) Table 1 Composition and nutrient levels of the diet (DM basis) |
发酵液采用新鲜瘤胃液与人工唾液混合而成。新鲜瘤胃液选自3头泌乳中期的成年健康荷斯坦奶牛。具体方法为:在晨饲2 h后,通过瘤胃瘘管接取足量的瘤胃液,4层纱布过滤,灌入充满CO2并经过预热达39 ℃的保温瓶,灌满后把瓶口立即盖紧,送回实验室,再经4层纱布过滤,磁力搅拌器混匀,待用。整个过程持续通入CO2维持厌氧环境。人工唾液参照Theodorou等[8]方法配制,人工唾液组成见表 2。每升人工唾液配制过程为:在558.9 mL蒸馏水中加入0.1 mL溶液A、200 mL溶液B、200 mL溶液C及1 mL溶液D,通入CO2饱和后放置于39 ℃并恒温水浴5~6 h,加入40 mL溶液E,混匀后通入CO2至饱和并加热至39 ℃,继续通CO2 0.5~1.0 h,使人工唾液变为无色,待用。将备好的瘤胃液与人工唾液在厌氧条件下充分混合(体积比1 : 9),pH调至6.7,39 ℃恒温,作为发酵液。
|
|
表 2 人工唾液组成 Table 2 Composition of artificial saliva |
本试验使用160 mL无菌玻璃瓶为发酵瓶,每个发酵瓶中称取1 g发酵底物,持续通入30 s CO2后,使用全自动分装仪进行快速分装,每瓶100 mL发酵液,然后迅速加盖异丁基塑胶塞,并以铝盖密封,摇匀后置于39 ℃的培养箱摇床中恒温培养48 h。
1.2 试验设计试验采用单因素试验设计。分别于培养0、6、12、24、48 h后取出培养瓶,放入冰水浴中使发酵停止,并分别吸取2 mL发酵液置于冻存管,密封后液氮保存。本试验每个时间点设3个重复,共15个样品。同时,另取3个培养瓶,只加入人工唾液进行培养,用于设备参数校正。
1.3 累积产气量的测定培养期间分别在6、12、24、48 h测定并记录体外发酵产气量,根据朱伟云等[9]的方法,使用气压转换器(IGER,UK)定时测定厌氧瘤胃微生物发酵产气量,根据各产气量和气压进行校正,除去空白发酵瓶产气量,计算出累积产气量。
1.4 16S rRNA测序及数据分析首先对样品中的DNA进行提取、PCR扩增和纯化,然后构建Miseq文库并进行测序。接着根据index和barcode序列区分各样本数据,且对数据进行处理,统计有效数据和确定优质序列长度分布。利用QIIME软件,使用RDP分类器[9]以Silva、RDP、Greengene细菌数据库[10]为参比,对序列进行系统分类,分类基于Bergey’s taxonomy,共分为6级,依次为界(kingdom)、门(phylum)、纲(class)、目(order)、科(family)、属(genus),默认阈值为80%。统计每个样品在门和属水平上群落组成,绘制系统分类柱状图,并采用gplots软件绘制瘤胃微生物群落热图[11]。最后,利用USEARCH软件[12],并基于操作分类单位(OTU)[13]进行物种多样性分析。
1.5 数据统计采用SPSS 17.0软件ANOVA过程进行单因素方差分析,试验数据用平均值±标准差表示,P < 0.05为差异显著。
2 结果 2.1 测序质量与合理性对不同样本DNA进行16S rRNA V3~V4区扩增,扩增的条带大小正确,浓度合适且条带单一,说明纯化后基因组DNA中无抑制PCR反应的杂质,可基于16S rRNA基因序列分析微生物多样性。经MiSeq高通量测序平台,共得到748 172条raw_reads序列(每个样品平均49 878条);经去除序列两端的低质量区域、其中的接头序列、含有模糊碱基N的序列程序处理后,对序列进行组装拼接,得到744 200条clean-reads序列(每个样品平均为49 613条),优质序列比率为99.47%,去除clean-reads序列中引物错配过多的序列(≥4 bp), 去除引物序列以及嵌合体后,15个样品共获得341 727条优化序列,每个样品平均22 782条,其中最多为31 305条,最少为14 195条,序列平均长度456 bp。
样品稀释曲线是采取随机抽样的方法获取数据,抽到的序列数与其所代表的OTU数目构建的曲线。当样品稀释曲线趋于平缓时,说明测序数量合理,更多的测序数据对于发现新的OTU的边际效应很小;反之,说明继续测序还可能产生较多新的OTU。本试验15个样品稀释曲线(图 1-A)表明并非所有样品都进入了平台期(即随着抽取数据的增加仍可能检测到新的OTU),但所有样品的覆盖度曲线(图 1-B)都已达到饱和状态,表明本研究样品的测序量及测序深度合理,更深的测序对瘤胃液中检出新的OTU贡献很小。
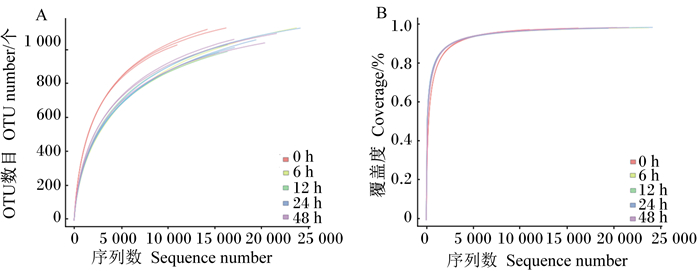
|
A:稀释曲线;B:覆盖度曲线。 A: rarefaction curve; B: coverage curve. 图 1 样品稀释曲线和覆盖度曲线 Figure 1 Rarefaction curve and coverage curve of samples |
从图 2-A可以看出,厚壁菌门(Firmicutes)的相对丰度随着时间变化呈先上升后下降的趋势。然而,拟杆菌门、纤维杆菌门(Fibrobactere)的相对丰度总体上呈现下降的趋势。螺旋体门(Spirochaetae)、黏胶球形菌门(Lentisphaerae)和浮霉菌门(Planctomycetes)的相对丰度呈现先下降后上升的趋势。柔膜菌门(Tenericutes)的相对丰度呈现上升的趋势。
由表 3可知,经过48 h的发酵,纤维杆菌门的相对丰度显著低于其他时间点(P<0.05),其相对丰度为0.22%;拟杆菌门的相对丰度也显著低于其他时间点(P<0.05),其相对丰度为30.69%;黏胶球形菌门在24 h时显著低于其他时间点(P<0.05),其相对丰度为3.41%。浮霉菌门的相对丰度在6、12、24 h时显著低于其他时间点(P<0.05),24 h最低,其相对丰度为0.59%;厚壁菌门的相对丰度在6 h时显著高于其他时间点(P<0.05),其相对丰度为36.69%;变形菌门的相对丰度在12、24、48 h时显著高于其他时间点(P<0.05),24 h最高,其相对丰度为18.23%;螺旋体门的相对丰度在48 h时显著高于其他时间点(P<0.05),其相对丰度为7.05%;柔膜菌门的相对丰度在48 h时显著高于其他时间点(P<0.05),其相对丰度为5.58%。
|
|
表 3 瘤胃微生物在体外发酵条件下细菌组成及其相对丰度(门水平) Table 3 Composition and relative abundance of rumen microbes under in vitro fermentation conditions (phylum level) |
从属水平细菌区系的物种相对丰度分析,相对丰度在0.01%以上的属有72个,不能归类到目前已知菌属的细菌相对丰度高达42.26%。,从图 2-B可以看出,普沃氏菌属(Pevotella)、链球菌属(Streptococcus)和琥珀酸弧菌属(Succinivibrio)的比例随着时间的变化呈现先上升后下降的趋势,丁酸弧菌属(Butyrivibrio)、密螺旋体菌属(Treponema)呈先下降后上升的趋势,丝杆菌属(Fibrobacter)和解琥珀酸菌属(Succiniclasticum)呈下降的趋势。
由表 4可知,随着时间的变化,普沃氏菌属的相对丰度在12 h时显著高于其他时间点(P < 0.05),其相对丰度为31.19%;链球菌属的相对丰度在6 h时显著高于其他时间点(P < 0.05),其相对丰度为23.03%;琥珀酸弧菌属的相对丰度在12 h时显著高于除24 h外的其他时间点(P < 0.05),其相对丰度为5.94%;RC9_gut_group的相对丰度在24 h时显著低于其他时间点(P < 0.05),其相对丰度为1.73%;丁酸弧菌属的相对丰度在12 h时显著低于其他时间点(P < 0.05),其相对丰度为0.26%;密螺旋体菌属的相对丰度在12 h时显著低于其他时间点(P < 0.05),其相对丰度为0.24%;丝杆菌属的相对丰度在48 h时显著低于其他时间点(P < 0.05),其相对丰度为0.22%;解琥珀酸菌属的相对丰度在24、48 h时显著低于其他时间点(P < 0.05),其相对丰度分别为2.22%和2.42%。
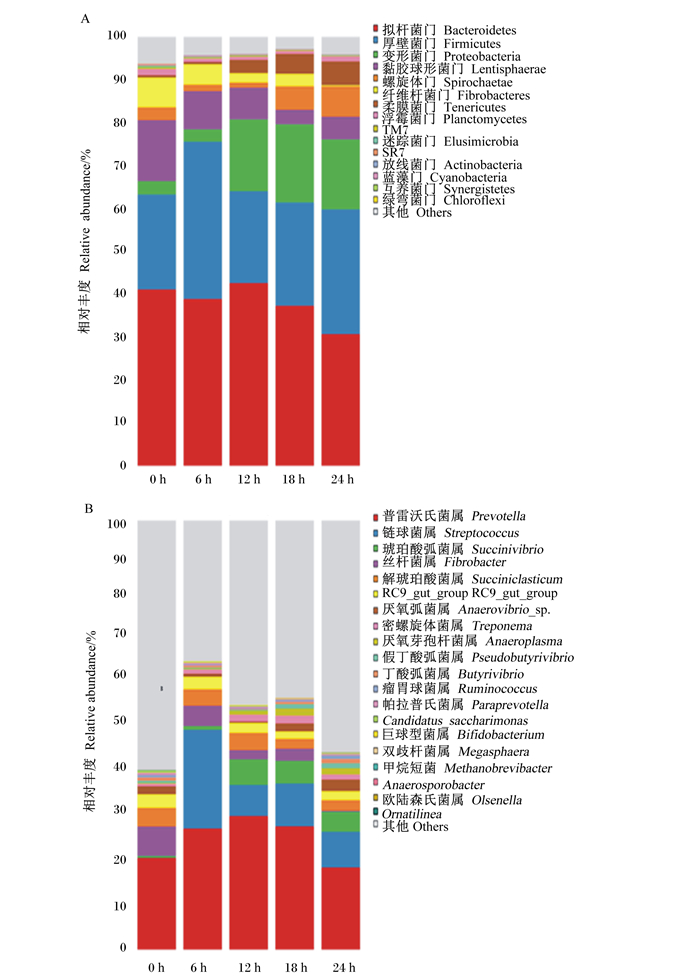
|
A:门水平;B:属水平。 A: phylum level; B: genus level. 图 2 在体外发酵条件下瘤胃细菌组成随时间变化分类柱状图 Figure 2 Classification histogram of rumen microbial composition with time under in vitro fermentation conditions |
|
|
表 4 瘤胃微生物在体外发酵条件下细菌组成及其相对丰度(属水平) Table 4 Composition and relative abundance of rumen microbes under in vitro fermentation conditions (genera level) |
热图是使用渐变的色带直观地展现数据的疏密程度或频率高低,可以简单地聚合大量数据。瘤胃内18种门水平的细菌进行双向聚类分析,结果显示(图 3-A),可从横向将15个样品分成2个大簇——拟杆菌门、厚壁菌门,这2个菌门聚成一类,其余16个菌门聚成一类(又可聚成2小簇);从纵向上将15个样品分成2个大簇,左边的第1大簇包含5个样品(0、6 h),右边的第2大簇包含10个样品(12、24、48 h)。为了进一步了解瘤胃微生物细菌相对丰度随时间的变化规律,在属的水平,随着时间的变化,瘤胃细菌组成存在明显差异(图 3-B),瘤胃内59种菌属进行双向聚类分析结果显示,可从横向将15个样品分成3个大簇,普雷沃氏菌属聚成一类,链球菌属聚成一类,其他菌属又聚成一类;从纵向上将15个样品分成2个大簇,左边的第1大簇包含3个样品(6 h),右边的第2大簇包含12个样品(又可聚成2个小簇)。

|
A:门水平;B:属水平。 A: phylum level; B: genus level. 图 3 瘤胃微生物在体外发酵条件下细菌的双向聚类分析 Figure 3 Two-way cluster analysis of rumen microbes under in vitro fermentation conditions |
由表 5可知,随时间变化,Ace指数、Chao1指数差异不显著(P>0.05),但是Shannon指数与Simpson指数差异显著(P < 0.05)。各样本稀释曲线均达到平台期,说明测序量及测序深度合理,各个样品的覆盖率随着测序的深度均达到了平台期,覆盖率都在97%以上,说明测序数据足够大,可以反映样品中绝大多数微生物的信息。
|
|
表 5 测序结果及α多样性分析 Table 5 Sequencing result and α diversity analysis |
由表 6可知,随时间变化,pH在24 h时显著低于其他时间点(P < 0.05)。随时间变化,累积产气量逐渐增加,在48 h时显著高于其他时间点(P < 0.05)。
|
|
表 6 pH和累积产气量随时间的变化 Table 6 Change of pH and accumulative gas production with timebg |
在细菌群落中,厚壁菌门、拟杆菌门和变形菌门是瘤胃细菌的3大优势菌群[14],其中拟杆菌门的相对比例在本试验中是最高的,这与之前给美国阿拉斯加州和挪威的驼鹿、梅花鹿、水牛以及斯瓦尔巴特群岛的驯鹿饲喂冬季饲粮和给大额牛饲喂树叶和稻草的混合饲粮的研究结果相一致[15-16]。Qin等[17]和Tun等[18]也证实无论是人类还是猫科胃肠道菌群中,拟杆菌门和厚壁菌门均占有较高的丰度。Tajima等[19]、Singh等[20]和Peng等[21]都指出荷斯坦牛和水牛瘤胃内的细菌都以拟杆菌门为主,其余菌门的相对丰度比较少,比如酸杆菌门、蓝藻门等。有研究表明,酸杆菌门、蓝藻门的细菌更适合在江海湖泊等环境中生存[22],它们在瘤胃严格厌氧的环境中无法进行生长与繁殖。拟杆菌门是瘤胃微生物中非纤维性碳水化合物的主要降解者,含有与降解非纤维类多糖有关的基因[23],而纤维分解菌在发酵中后期起主导作用。因此,拟杆菌门的相对丰度在12 h之后减少。奶牛瘤胃内不会产生纤维降解酶分解摄入的纤维物质,而是依靠体内的细菌、真菌等微生物对其进行降解消化[24]。其中纤维杆菌门消化和分解纤维物质起着重要的作用。Liu等[25]的研究表明,纤维杆菌门的纤维杆菌属从0~16和48 h的相对丰度显著增加,在降解植物纤维性物质方面发挥关键的作用。而本试验纤维杆菌门在12~48 h降解纤维时显著减少,这与发酵方法不同有关。本试验是采用的体外发酵法,随着时间的变化,具有分解纤维功能的细菌之间会相互竞争,纤维素、半纤维素物质会不断减少,这将会导致纤维杆菌门相对丰度显著减少。而Liu等[25]采用尼龙袋法,在每个时间点都会供应相同的饲粮,这样可能会缓解纤维分解菌之间的竞争。同时在本试验中主要消化纤维的瘤胃球菌属、丁酸弧菌属的相对丰度在12 h之后会显著增加,这有可能与纤维杆菌门形成竞争关系。也有研究结果表明,链球属中的牛粪球菌属(Coccus mucosa)会造成瘤胃内不耐酸细菌生长受到抑制,比如纤维分解菌,这可能与链球菌属之间存在竞争关系[26]。在本试验中,链球菌属的相对丰度随着时间变化显著增加,而纤维分解菌的相对丰度显著减少,这都有可能引起本试验纤维杆菌门随时间变化显著减少。有研究表明,变形菌门的大部分菌属不仅可以和其他菌属竞争生存,而且在低碳源浓度下也可以适度生长[27]。随着长时间的发酵,淀粉、蛋白质等易降解营养物质基本被微生物降解,变形菌门的相对丰度增加可能与在营养浓度低的环境下与纤维分解菌争夺资源、适度生长有关。有研究表明,厚壁菌门中梭菌纲占很大比例,链球菌属和解琥珀酸菌属是厚壁菌门的主要优势菌属[28],这与本试验结果相一致。厚壁菌门含有大量分解纤维的菌属[如瘤胃球菌属、丁酸弧菌属、假丁酸弧菌属(Pseudobutyrivibrio)][29],这些菌属的相对丰度在12 h之后都增加。因此,厚壁菌门的相对丰度在12 h之后增加。
在属水平上,大量的研究表明,普雷沃氏菌属是广泛存在瘤胃中且数量最多的一类菌,普雷沃氏菌属不仅是活性高的蛋白质降解菌,而且还能利用淀粉以及果胶。由于遗传相关或者高的遗传变异使得这种菌属的成员在瘤胃内占据不同的生态位[30-32]。这与本试验研究结果相一致。有研究表明,普雷沃氏菌属属于耗氢菌,它可以通过琥珀酸和丙烯酸途径发酵糖类物质和乳酸, 产生丙酸[33-34],丙酸途径可能对瘤胃发酵是相对重要的,相应地可能会导致甲烷产量下降,因为琥珀酸-丙酸途径可以与产甲烷菌争夺氢。在本试验中,普雷沃氏菌属的相对丰度随时间变化减少,而甲烷短杆菌的相对丰度随时间增加,普雷沃氏菌属和产甲烷菌属的关系值得进一步研究。普雷沃氏菌属基本是分解蛋白质、淀粉的菌属。淀粉和蛋白质被不断地代谢分解,被普雷沃氏菌属所利用,迅速增殖,随着发酵时间的增加,淀粉、蛋白质等营养物质的不断减少,在12 h之后,一些活性高的纤维分解菌属主要起主导作用。虽然Li等[15]认为普雷沃氏菌属在降解纤维方面发挥潜在的作用,但它不是主要的纤维素分解菌,普雷沃氏菌属生长繁殖可能会受到其他活性高的纤维分解菌的抑制,这将可能造成普雷沃氏菌属的相对丰度随时间变化呈现先增加后减少的趋势。解琥珀酸菌属是Van Gylswyk于1995年从牛瘤胃中分离,其功能可使琥珀酸转化成丙酸,可能是瘤胃微生物区系的正常菌[35],它可以分解淀粉与纤维素。在本试验中解琥珀酸菌属的相对丰度随时间变化而减少,这可能与其他淀粉分解菌和纤维素分解菌存在竞争,营养物质不能充分地供给自身有关。琥珀酸弧菌属最早发现于牛瘤胃中,其主要发酵糊精,终产物主要是乙酸和琥珀酸,它具有淀粉分解活性。在本试验中,在12 h之前,琥珀酸弧菌属的相对丰度增加,在这个阶段,它们主要发酵饲粮中的淀粉,在12 h之后,瘤胃中的纤维分解菌起主要作用,所以它们的相对丰度在这个阶段会下降。链球菌属中的牛链球菌能降解淀粉,但不能降解纤维素,它的作用跟琥珀酸弧菌属类似,但是它的相对丰度在6 h之前增加,原因可能是它产生的淀粉分解酶活性会比琥珀酸弧菌属强。密螺旋体菌属、丁酸弧菌属、瘤胃球菌属是瘤胃内主要的纤维分解菌,它们的相对丰度都在12 h之后升高,可能它们在这个阶段主要消化纤维性物质为主。
α多样性反映了单个样品内部物种的多样性,包括Chao1指数、Shannon指数、Simpson指数、Ace指数。其中Chao1指数、Ace指数反映样品中群落的丰富度,Shannon指数、Simpson指数反映物种的多样性。Chao1指数、Ace指数和Shannon指数值越大,Simpson指数值越小,说明样品中的物种越丰富。各物种指数有一定的变化范围,说明随着时间的变化,细菌组成和多样性存在差异。Chao1指数、Ace指数无显著差异,说明随着时间的变化,细菌总数变化不显著,但是Shannon指数以及Simpson指数有显著差异,说明随着时间的变化,细菌的多样性存在显著差异,与0 h相比,Shannon指数在48 h时显著降低,Simpson指数在48 h时显著增加,说明细菌多样性显著降低,可能与发酵底物消耗以及人工唾液的营养物质减少有关。
产气量是一个综合反映饲粮可发酵程度的重要指标,饲粮中的可发酵有机物含量高,微生物的活性就越高,产气量就越大,反之,微生物的活性较弱,产气量就较少[36]。本试验中随着时间的变化,累计产气量显著增加,微生物都保持较高的活性。pH是影响瘤胃发酵的重要因素。当pH长时间过低时,瘤胃内纤维素降解菌降解纤维素的能力下降[37]。pH在6~7时可以保证瘤胃正常发酵。在本试验中,pH在6.26~6.70,均在适宜的范围。
4 结论奶牛瘤胃体外发酵过程中,瘤胃细菌主要的门以及属的相对丰度以及多样性均随时间推移发生改变,且因瘤胃细菌功能差异在发酵过程的变化更为显著。因此,在使用体外发酵法进行研究时,应充分考虑瘤胃细菌的功能差异与底物以及人工唾液的营养物质减少引起的发酵环境变化的关系。
| [1] |
MUYZER G, DE WAAL E C, UITTERLINDEN A G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J]. Applied and Environmental Microbiology, 1993, 59(3): 695-700. |
| [2] |
GETACHEW G, BLVMMEL M, MAKKAR H P S, et al. In vitro gas measuring techniques for assessment of nutritional quality of feeds:a review[J]. Animal Feed Science and Technology, 1998, 72(3/4): 261-281. |
| [3] |
朱智, 朱伟云. 不同精粗比底物时添加大蒜油对体外瘤胃微生物发酵的影响[J]. 南京农业大学学报, 2012, 35(2): 125-130. |
| [4] |
陈亚迎, 刘壮, 吕朋安, 等. 溶菌酶对瘤胃体外发酵、甲烷生成及微生物菌群结构的影响[J]. 微生物学报, 2017, 57(5): 758-768. |
| [5] |
HUMER E, ADITYA S, KALTENEGGER A, et al. Graded substitution of grains with bakery by-products modulates ruminal fermentation, nutrient degradation, and microbial community composition in vitro[J]. Journal of Dairy Science, 2018, 101(4): 3085-3098. DOI:10.3168/jds.2017-14051 |
| [6] |
JOHNSON R R, DEHORITY B A, MCCLURE K E, et al. A comparison of in vitro fermentation and chemical solubility methods in estimating forage nutritive value[J]. Journal of Animal Science, 1964, 23(4): 1124-1128. DOI:10.2527/jas1964.2341124x |
| [7] |
CUNHA I S, BARRETO C C, COSTA O Y A, et al. Bacteria and archaea community structure in the rumen microbiome of goats (Capra hircus) from the semiarid region of Brazil[J]. Anaerobe, 2011, 17(3): 118-124. DOI:10.1016/j.anaerobe.2011.04.018 |
| [8] |
THEODOROU M K, WILLIAMS B A, DHANOA M S, et al. A simple gas production method using a pressure transducer to determine the fermentation kinetics of ruminant feeds[J]. Animal Feed Science and Technology, 1994, 48(3/4): 185-197. |
| [9] |
朱伟云, 毛胜勇, 王全军, 等. 厌氧真菌体外发酵筛选技术的研究[J]. 南京农业大学学报, 2001, 24(3): 44-48. |
| [10] |
WANG Q, GARRITY G M, TIEDJE J M, et al. Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied and Environmental Microbiology, 2007, 73(16): 5261-5267. DOI:10.1128/AEM.00062-07 |
| [11] |
PRUESSE E, QUAST C, KNITTEL K, et al. SILVA:a comprehensive online resource for quality checked and aligned ribosomal RNA sequence data compatible with ARB[J]. Nucleic Acids Research, 2007, 35(21): 7188-7196. DOI:10.1093/nar/gkm864 |
| [12] |
GENTLEMAN R C, CAREY V J, BATES D M, et al. Bioconductor:open software development for computational biology and bioinformatics[J]. Genome Biology, 2004, 5(10): R80. DOI:10.1186/gb-2004-5-10-r80 |
| [13] |
EDGAR R C. UPARSE:highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [14] |
BHATT V D, DANDE S S, PATIL N V, et al. Molecular analysis of the bacterial microbiome in the forestomach fluid from the dromedary camel (Camelus dromedarius)[J]. Molecular Biology Reports, 2013, 40(4): 3363-3371. DOI:10.1007/s11033-012-2411-4 |
| [15] |
LI Z P, LIU H L, LI G Y, et al. Molecular diversity of rumen bacterial communities from tannin-rich and fiber-rich forage fed domestic Sika deer (Cervus nippon) in China[J]. BMC Microbiology, 2013, 13: 151. DOI:10.1186/1471-2180-13-151 |
| [16] |
PITTA D W, KUMAR S, VEICCHARELLI B, et al. Bacterial diversity associated with feeding dry forage at different dietary concentrations in the rumen contents of Mehshana buffalo (Bubalus bubalis) using 16S pyrotags[J]. Anaerobe, 2014, 25: 31-41. DOI:10.1016/j.anaerobe.2013.11.008 |
| [17] |
QIN J J, LI R Q, RAES J, et al. A human gut microbial gene catalogue established by metagenomic sequencing[J]. Nature, 2010, 464(7285): 59-65. |
| [18] |
TUN H M, BRAR M S, KHIN N, et al. Gene-centric metagenomics analysis of feline intestinal microbiome using 454 junior pyrosequencing[J]. Journal of Microbiological Methods, 2012, 88(3): 369-376. DOI:10.1016/j.mimet.2012.01.001 |
| [19] |
TAJIMA K, ARAI S, OGATA K, et al. Rumen bacterial community transition during adaptation to high-grain diet[J]. Anaerobe, 2000, 6(5): 273-284. |
| [20] |
SINGH K M, AHIR V B, TRIPATHI A K, et al. Metagenomic analysis of Surti buffalo (Bubalus bubalis) rumen:a preliminary study[J]. Molecular Biology Reports, 2012, 39(4): 4841-4848. DOI:10.1007/s11033-011-1278-0 |
| [21] |
PENG S, YINJ G, LIU X L, et al. First insights into the microbial diversity in the omasum and reticulum of bovine using Illumina sequencing[J]. Journal of Applied Genetics, 2015, 56(3): 393-401. DOI:10.1007/s13353-014-0258-1 |
| [22] |
QUAISER A, OCHSENREITER T, LANZ C, et al. Acidobacteria form a coherent but highly diverse group within the bacterial domain:evidence from environmental genomics[J]. Molecular Microbiology, 2003, 50(2): 563-575. DOI:10.1046/j.1365-2958.2003.03707.x |
| [23] |
RUSSELL J B, DIEZ-GONZALEZ F. The effects of fermentation acids on bacterial growth[J]. Advances in Microbial Physiology, 1998, 39: 205-234. |
| [24] |
WRIGHT A D G, KLIEVE A V. Does the complexity of the rumen microbial ecology preclude methane mitigation?[J]. Animal Feed Science and Technology, 2011, 166-167: 248-253. DOI:10.1016/j.anifeedsci.2011.04.015 |
| [25] |
LIU J H, ZHANG M L, XUE C X, et al. Characterization and comparison of the temporal dynamics of ruminal bacterial microbiota colonizing rice straw and alfalfa hay within ruminants[J]. Journal of Dairy Science, 2016, 99(12): 9668-9681. DOI:10.3168/jds.2016-11398 |
| [26] |
NOCEK J E. Bovine acidosis:implications on laminitis[J]. Journal of Dairy Science, 1997, 80(5): 1005-1028. DOI:10.3168/jds.S0022-0302(97)76026-0 |
| [27] |
BULYGINA E S, GALCHENKO V F, GOVORUKHINA N I, et al. Taxonomic studies on methylotrophic bacteria by 5S ribosomal RNA sequencing[J]. Journal of General Microbiology, 1990, 136(3): 441-446. |
| [28] |
KIM M, MORRISON M, YU Z T. Status of the phylogenetic diversity census of ruminal microbiomes[J]. FEMS Microbiology Ecology, 2011, 76(1): 49-63. DOI:10.1111/fem.2011.76.issue-1 |
| [29] |
EVANS N J, BROWN J M, MURRAY R D, et al. Characterization of novel bovine gastrointestinal tract Treponema isolates and comparison with bovine digital dermatitis treponemes[J]. Applied and Environmental Microbiology, 2011, 77(1): 138-147. DOI:10.1128/AEM.00993-10 |
| [30] |
RAMŠAK A, PETERKA M, TAJIMA K, et al. Unravelling the genetic diversity of ruminal bacteria belonging to the CFB phylum[J]. FEMS Microbiology Ecology, 2000, 33(1): 69-79. DOI:10.1111/fem.2000.33.issue-1 |
| [31] |
PURUSHE J, FOUTS D E, MORRISON M, et al. Comparative genome analysis of Prevotella ruminicola, and Prevotella bryantii:insights into their environmental niche[J]. Microbial Ecology, 2010, 60(4): 721-729. |
| [32] |
AVGUŠTIN G, WALLACE R J, FLINT H J. Phenotypic diversity among ruminal isolates of Prevotella ruminicola:proposal of Prevotella brevis sp.nov.Prevotella bryantii sp.nov.and Prevotella albensis sp.nov.and redefinition of Prevotella ruminicola[J]. International Journal of Systematic Bacteriology, 1997, 47(2): 284-288. DOI:10.1099/00207713-47-2-284 |
| [33] |
FLINT H J, BAYER E A, RINCON M T, et al. Polysaccharide utilization by gut bacteria:potential for new insights from genomic analysis[J]. Nature Reviews Microbiology, 2008, 6(2): 121-131. |
| [34] |
NEWBOLD C J, LÓPEZ S, NELSON N, et al. Propionate precursors and other metabolic intermediates as possible alternative electron acceptors to methanogenesis in ruminal fermentation in vitro[J]. British Journal of Nutrition, 2005, 94(1): 27-35. DOI:10.1079/BJN20051445 |
| [35] |
冯仰廉. 反刍动物营养学[M]. 北京: 中国农业出版社, 2004: 1-12.
|
| [36] |
雷冬至, 金曙光, 乌仁塔娜. 用体外产气法评价不同粗饲料与相同精料间的组合效应[J]. 饲料工业, 2009, 30(3): 30-33. |
| [37] |
ZHOU X, ZEITZ J O, MEILE L, et al. Influence of pH and the degree of protonation on the inhibitory effect of fatty acids in the ruminal methanogen Methanobrevibacter ruminantium strain M1[J]. Journal of Applied Microbiology, 2016, 119(6): 1482-1493. |




