研究表明,采用理想蛋白质模型,在现行推荐的生长育肥猪饲养标准下使饲粮蛋白质水平降低2%~4%,通过添加合成氨基酸来满足动物对氨基酸的需要,并不影响动物生长性能和健康状况[1-2]。按照这种方式配制的低蛋白质水平饲粮可以节约蛋白质类饲料成本,并可降低动物粪尿中氮的排泄,在养猪生产实践中应用可提高饲料的利用率,并减少排泄物对环境造成的污染。猪肠道中栖居着数量庞大、种类多样的微生物,其与宿主之间的物质交换信息传递,对动物养分的消化吸收、机体免疫功能与健康的维护起着重要的作用[3-4]。研究表明,不同的氮水平和氮模式,对肠道微生物的结构和组成存在巨大差异,进而影响宿主对营养物质的吸收和利用[5]。目前低蛋白质水平饲粮在生长育肥猪上的研究大部分针对其生长性能、肉品质及血液生化指标等,但有关低蛋白质水平饲粮对猪肠道菌群影响的报道还较为鲜见。本试验以商业生产条件下生长育肥猪为研究对象,将饲粮蛋白质水平在《猪饲养标准》(NY/T 65—2004)推荐的标准上降低约2%,同时补充必需氨基酸,研究饲粮蛋白质水平的降低对生长育肥猪生长性能和肠道微生物菌群的影响,为进一步完善猪氨基酸理想模型以及低蛋白质水平饲粮的合理配制提供理论依据。
1 材料与方法 1.1 试验动物和试验设计试验采用单因子随机试验设计,选用遗传背景相同、初始体重为(45.50±3.64) kg的“长×大”二元杂交生长育肥猪140头,按照体重相近,性别比例一致的原则随机分为2个组,每组5个重复,每个重复14头猪。对照组饲喂蛋白质水平为15.05%的正常蛋白质水平饲粮,试验组饲喂蛋白质水平为12.97%的低蛋白质水平饲粮,低蛋白质水平饲粮添加相应的合成氨基酸,使2组饲粮中氨基酸的标准回肠消化率(standardized ileal digestibility, SID)水平保持相同。生长育肥猪饲粮配制参考我国《猪饲养标准》(NY/T 65—2004)推荐的生长育肥猪营养需要量标准。试验饲粮组成及营养水平见表 1。
|
|
表 1 试验饲粮组成及营养水平(干物质基础) Table 1 Composition and nutrient levels of experimental diets (DM basis) |
本试验在北京市房山区喜庆民丰养殖场进行。预试期为5 d,正试期为30 d。试验猪按重复分圈饲养,自由采食,自由饮水。试验期间按照该猪场常规程序进行清洁和消毒,保持环境卫生良好。试验设专门人员负责管理,严格按照试验设计进行试验。试验过程中,每天观察并记录试验猪的健康状况,记录好腹泻、发病和死亡情况。
1.3 生长性能测定分别于试验初期和末期进行个体称重,试验全程记录采食量。生长性能评定指标包初重、末重、平均日增重(ADG)、平均日采食量(ADFI)和料重比(F/G)。
1.4 样品的采集在试验结束前1天,每重复随机选择6头猪采集新鲜粪便样品,每3头等量混合在一起,立刻装入无菌离心管内,置于干冰中,带回实验室保存于-80 ℃冰箱中,用于提取细菌总DNA。
1.5 样品的DNA提取及测序粪便样品中的总DNA采用E.Z.N.A. Stool DNA提取试剂盒(Omega Bio-Tek公司,美国),按照说明书上的具体步骤进行提取。提取后的总DNA纯化后,用琼脂糖凝胶电泳检测浓度和纯度。选择16S rRNA的V3~V4保守区域,采用细菌通用引物338F (5′-ACTCCTACGGGAGGCAGCA-3′)和806R(5′-GGACTACHVGGGTWTCT-AAT-3′)对粪便DNA进行扩增。扩增体系和程序如表 2所示。PCR产物经2%琼脂糖凝胶电泳检测,使用AxyPrep DNA凝胶回收试剂盒(Axygen公司,美国)切胶回收PCR产物。扩增子采用双尾法在Illumina MiSeq平台上进行测序(上海美吉生物医药科技有限公司,上海)。PCR反应程序:95 ℃ 3 min,95 ℃ 30 s,55 ℃ 30 s,72 ℃ 45 s,27个循环;72 ℃ 10 min。
|
|
表 2 PCR扩增体系 Table 2 PCR reaction system |
生长性能数据结果采用SSPS 17.0软件包中的one-way ANOVA程序进行单因素方差分析,若组间差异显著,则采用Duncan氏法进行多重比较,以P < 0.05为差异显著性标准。
粪便菌群测序得到的Reads首先根据Overlap关系进行拼接,之后采用Trimmomatic和Qiime v.1.5.0进行去杂和过滤,采用Uchime algorithm v.4.2.40去嵌合体。优化后的高质量序列依据Silva 16S序列数据库进行操作分类单元(OTU)聚类(相似水平为97%),之后采用RDP classifier对菌种进行分类,并在各分类水平统计每个样本的菌群组成。采用Qiime软件进行Alpha多样性分析,包括多样性指数Shannon指数和Simpson指数,丰富度指数ACE指数和Chao1指数。采用基于Bray-Curtis距离的PCoA对粪便样品微生物组成的组间距离进行分析。采用LEfSe算法对各分类水平物种差异菌种进行分析。
2 结果与分析 2.1 饲粮蛋白质水平对生长育肥猪生长性能的影响由表 3可知,与对照组相比,试验组生长育肥猪ADG、ADFI和F/G等生长性能指标差异均不显著(P>0.05)。但试验组生长育肥猪ADG略高于对照组,ADFI和F/G略低于对照组。上述结果说明在本次试验条件下,饲粮在正常蛋白质水平上降低约2%,同时添加合成氨基酸,不会对生长育肥猪生长性能产生影响。
|
|
表 3 饲粮蛋白质水平对生长育肥猪生长性能的影响 Table 3 Effects of dietary protein level on growth performance of growing-finishing pigs |
本试验采用高通量测序的方法对对照组和试验组生长育肥猪粪便中的微生物群落进行了测序分析。本次试验产生的20个样品共测到有效序列1 230 704条,序列平均长度为436.20 bp。序列长度绝大部分分布于421~460 bp之间,占所有序列条数的99.94%。对获得的20个有效样本进行OTU聚类,样本平均聚类形成OTU的数量为1 098。采用随机抽样的方法,模拟输入序列数目与OTU个数产出间关系,绘制稀疏曲线。由图 1所示,本次测序所有样品的稀释性曲线均趋向平坦,说明测序数据量合理,可以反映样本中绝大多数微生物的信息。
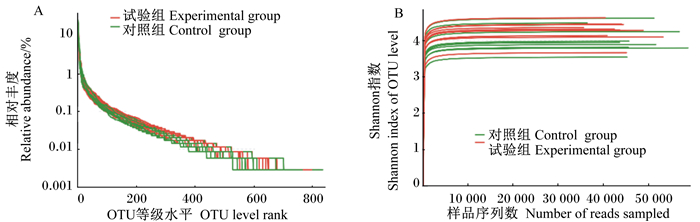
|
图A、图B分别为OTU丰度等级曲线和稀释性曲线。 Fig.A and Fig.B indicated the OUT rank-abundance curve and dilution curve, respectively. 图 1 样品丰度等级曲线和稀释性曲线 Figure 1 OUT rank-abundance curve and dilution curve of samples |
从表 4可知,虽然各多样性和丰度度指数在2组间差异均未达到显著水平(P>0.05),但低蛋白质水平饲粮组Shannon指数、ACE指数和Chao1指数均高于正常蛋白质水平饲粮组,且Simpson指数低于正常蛋白质水平饲粮组。这说明饲喂低蛋白质水平饲粮有利于提高育肥猪粪便中细菌的多样性和丰富度。
|
|
表 4 饲粮蛋白质水平对生长育肥猪肠道菌群多样性的影响 Table 4 Effects of dietary protein level on intestine microbiota diversity of growing-finishing pigs |
从图 2-A可知,在门分类水平上,试验组和对照组试验猪粪便中优势菌群主要由厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes)两大类组成,其中厚壁菌门约占近80%,拟杆菌门约占近15%。其次为螺旋菌门(Spirochaetae)、软壁菌门(Tenericutes)、蓝藻菌门(Cyanobacteria)等,它们的总占比平均为5.69%。试验组和对照组厚壁菌门和拟杆菌门占比非常接近(分别为80.07% vs.79.64%和14.10% vs. 14.82%),组间差异较小,其他门水平细菌组间略有差异。
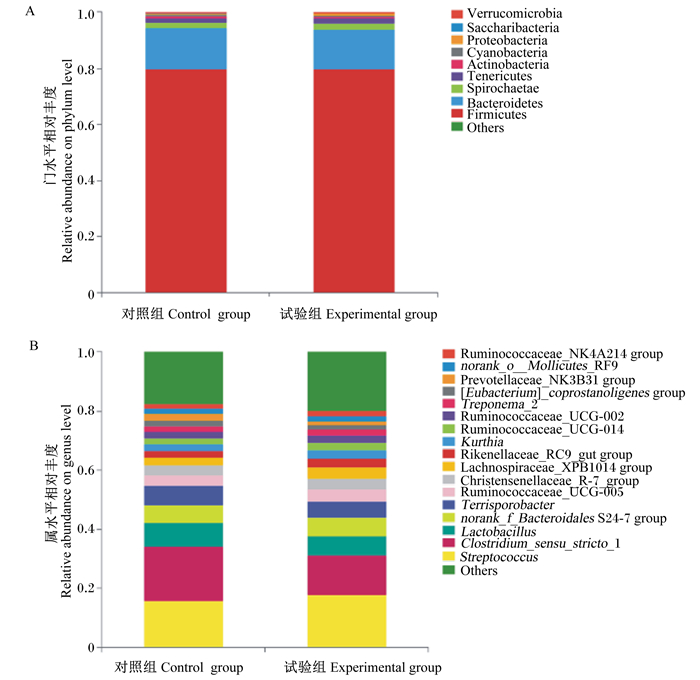
|
图A代表门水平。Fig.A represented the phylum level. Firmicutes:厚壁菌门;Bacteroidetes:拟杆菌门;Spirochaetae:螺旋菌门;Tenericutes:软壁菌门;Actinobacteria:放线菌门;Cyanobacteria:蓝藻菌门;Proteobacteria:变形菌门;Verrucomicrobia:疣微菌门;Others:其他。 图B代表属水平。Fig.B represented the genus level. Streptococcus:链球菌属;Clostridium_sensu_stricto_1:狭义梭菌属1;Lactobacillus:乳杆菌属;norank_f_Bacteroidales S24-7 group:norank f拟杆菌目S24-7群;Ruminococcaceae_UCG-005:疣微菌科UCG-005;Lachnospiraceae_XPB1014 group:毛螺菌科XPB1014群;Rikenellaceae_RC9_gut group:理研菌科RC9肠道群;Kurthia:库特氏菌属;Ruminococcaceae_UCG-014:疣微菌科UCG-0014;Ruminococcaceae_UCG-002:疣微菌科UCG-002;Treponema_2:密螺旋体属2;_coprostanoligenes group:真杆菌属coprostanoligenes群;Prevotellaceae_NK3B31 group:普雷沃氏菌科NK3B31群;norank_o__Mollicutes_RF9:norank o柔膜菌纲RF9;Ruminococcaceae_NK4A214 group:疣微菌科NK4A214群;Others:其他。 图 2 粪便中的优势菌群分布 Figure 2 Distribution of fecal dominant microbiota |
从图 2-B可知,在属水平分类水平上,2组在菌群组成上有较大差异。属水平共计分类得出205个菌属,其中相对丰度度大于1%的菌属有36个,总相对丰度约占82.98%。试验组相对丰度较高的菌属分别为链球菌属(Streptococcus,17.95%)、狭义梭菌属1(Cloustridium sensu stricto 1,13.22%)、乳杆菌属(Lactobacillus,6.52%)和norank_f拟杆菌目S24-7(norank_f Bacteroidales S24-7,6.24%),而对照组相对丰度较高的则为Cloustridium sensu stricto 1(18.38%)、Streptococcus(15.90%)、Lactobacillus(7.81%)和Terris-porobacter(6.50%)。此外,试验组中和对照组中均有几个独有菌属,但相对丰度都较低。
2.2.4 物种差异显著性分析基于LEfSe的多级物种差异判别分析表明(图 3),在门水平和纲水平上试验组和对照组没有相对丰度显著差异细菌,而其他分类水平上2组间均有相对丰度差异细菌。如在属水平上,试验组毛螺菌属(Lachnospira)、Pseudobutyrivibrio、Butyrivibrio和颤杆菌科(Oscillibacter)等的相对丰度均显著高于对照组(P<0.05),而对照组Paraeggerthella和狭义梭菌属6(Clostridium sensu stricto 6)等属的相对丰度显著高于试验组(P<0.05)。
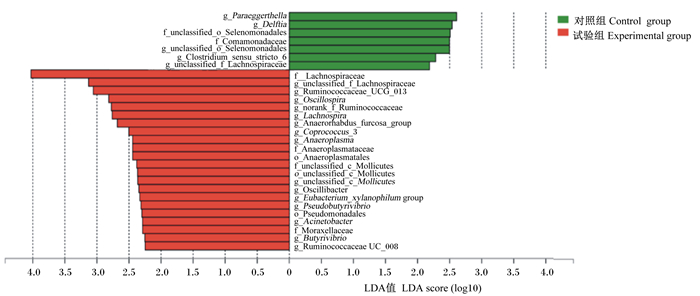
|
对照组control group:g_Delftia:代尔夫特菌属;f_Comamonadaceae:丛毛单胞菌科;g_Clostridium_sensu_stricto_6:狭义梭菌属6。 试验组experimental group:f__Lachnospiraceae:毛螺菌科;g_unclassified_f_Lachnospiraceae:未分类毛螺菌科;g_Ruminococcaceae_UCG_013:疣微菌科UCG 013;g_Oscillospira:颤螺旋菌属;g_norank_f_Ruminococcaceae:norank f疣微菌科;g_Lachnospira:毛螺菌属;g_Anaerorhabdus_furcosa_group:棍状厌氧菌furcosa群;g_Coprococcus_3:粪球菌属3;g_Anaeroplasma:厌氧原体属;f_Anaeroplasmataceae:厌氧原体科;o_Anaeroplasmatales:厌氧原体目;f_unclassified_c_Mollicutes:未分类c柔膜菌纲;o_unclassified_c_Mollicutes:未分类c柔膜菌纲(目水平);g_unclassified_c_Mollicutes:未分类c柔膜菌纲(属水平);g_Oscillibacter:颤杆菌科;g_Eubacterium_xylanophilum group:真杆菌属xylanophilum群;g_Pseudobutyrivibrio:假丁酸弧菌属;o_Pseudomonadales:假单胞菌目;g_Acinetobacter:不动杆菌属;f_Moraxellaceae:莫拉氏菌科;g_Butyrivibrio:丁酸弧菌属;g_Ruminococcaceae UC_008:疣微菌科UC 008。 图 3 LEfSe多级物种差异判别分析 Figure 3 Discriminant analysis of multistage species difference in LEfSe |
图 4为2组生长育肥猪粪便微生物群落组成的PCoA聚类分析图及偏最小二乘法判别式分析。由图可以看出,试验组的菌群能够较好地聚积在一起,并且能与对照组区分开来,说明试验组相比于对照组有着不同的肠道微生物区系。
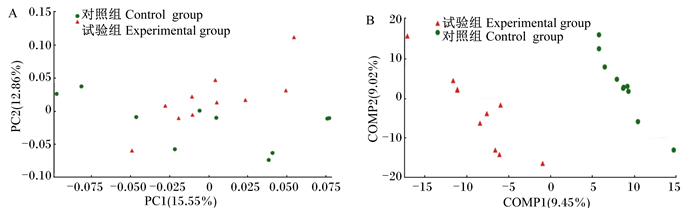
|
图A、图B分别为PCoA和PLS-DA聚类分析图。 Fig.A and Fig.B were the PCoA and PLS-DA cluster analysis diagrams, respectively. 图 4 粪便菌群聚类分析图 Figure 4 Fecal microbiota cluster analysis diagram |
已有较多研究结果表明,饲粮蛋白质水平降低2%~3%,并补充必需氨基酸满足猪的营养需要或符合理想氨基酸比例,对生长肥育猪的生长性能没有显著影响[2, 6-7]。而饲粮蛋白质水平降低3%或3%以上时,对生长肥育猪生长性能的影响则报导不一,生长育肥猪生长性能随饲粮蛋白质水平降低程度、必需氨基酸补充的种类和水平的不同而表现不同[8-10]。当饲粮蛋白质水平降到一定程度时,无论怎样补充和平衡饲粮氨基酸,均无法避免对生长性能造成的影响。此外,随着合成氨基酸技术的日益成熟,低蛋白质水平饲粮配制的成本不会明显升高,反而会因为豆粕类蛋白质饲料投入的减少,使其整体饲料成本有所降低。本研究结果表明,生长育肥阶段饲粮蛋白质水平降低约2%对猪生长性能没有显著影响。试验组平均日增重和平均日采食量略高于对照组,料重比低于对照组,差异不显著。按照当时的饲料原料价格核算,每头猪实际耗料成本有所降低,加之生长性能的提高使其生产效益有所提升。该试验结果与前人研究结果一致,再次证明,饲粮蛋白质水平降低约2%,补充适当的必需氨基酸,并不影响生长育肥猪生长性能,且能提高其生产效益。
3.2 低蛋白质水平饲粮对生长育肥猪肠道菌群多样性和丰度的影响动物肠道菌群的数量和结构对宿主的消化和健康起到很重要的作用[4, 11]。Zhang等[12]研究表明,饲粮因素引起的肠道微生物变化占到所有因素的57%,说明饲粮组分会明显影响肠道微生物数量和组成。Shannon指数和Simpson指数通常反映肠道菌群的多样性,而肠道微生物多样性通常与微生物区系的稳定性和抵抗致病菌感染的能力呈正相关[13]。本研究中,低蛋白质水平饲粮增加了生长育肥猪粪便菌群的多样性,说明低蛋白质水平饲粮有益于生长育肥猪肠道健康。此外,本研究中发现低蛋白质水平饲粮组生长育肥猪粪便的丰富度也高于正常蛋白质水平饲粮组,这和曹克飞[14]研究结果一致,其研究结果表明低蛋白质水平饲粮可以提高生长育肥猪肠道菌群丰富度和多样性。Fan等[15]研究表明,当饲粮蛋白质水平从16%降到13%时,回肠菌群丰度和多样性提高,有益菌乳杆菌属比例提高,结肠有益菌巨型球菌属比例也明显提高,但当饲粮蛋白质水平降到10%时,生长育肥猪回肠、结肠菌群多样性下降,且猪的生长性能降低。这说明饲粮蛋白质水平一定程度的降低对肠道微生物多样性有促进调节作用,而饲粮蛋白质水平过低反而降低肠道微生物多样性,影响生长性能。
3.3 低蛋白质水平饲粮对生长育肥猪肠道菌群组成的影响肠道菌群在门水平上主要由Firmicutes(35%~80%)和Bacteroidetes(17%~60%)构成[16]。本试验中,Firmicutes和Bacteroidetes是各组中猪肠道微生物相对丰度最高的菌群,且组间差异不显著,表明降低蛋白质水平在门水平上对其主要菌群结构无明显影响。前期研究[17]和前人的研究[8]均表明,适度降低饲粮蛋白质水平并补充相应合成氨基酸可以提高育肥猪氮的表观生物学价值,有利于育肥猪氮代谢平衡。而这种氮的利用率的提高一定程度上与低蛋白质水平饲粮引起的肠道菌群变化相关。在属水平上Streptococcus是2组中的优势菌属,饲喂低蛋白质水平饲粮的生长育肥猪粪便中Streptococcus的相对丰度有所增加,该属细菌主要利用氨基酸合成菌体蛋白质[18],Streptococcus的增加可能是由于低蛋白质水平饲粮中补充了限制性氨基酸,使得可供Streptococcus代谢的氨基酸增加所致,菌体蛋白合成的增加会影响氮代谢和利用效率。Clostridium sensu stricto 1也是优势菌属之一,该菌属丰度在低蛋白质水平饲粮生长育肥猪粪便中有明显降低。Fan等[15]研究也表明,随着饲粮蛋白质水平的降低,猪回肠中Clostridium sensu strict 1的丰度显著降低,分析原因认为是饲粮蛋白质水平的降低导致发酵底物氮源的缺乏而引起的。低蛋白质水平饲粮粪便中Lachnospiraceae[19]、Ruminococcaceae[19]、Butyrivibrio[20]和Pseudobutyrivibrio[20]菌属的相对丰度显著增加,这几个菌属的细菌可以利用饲粮中的碳水化合物和纤维代谢产生丁酸类物质,而丁酸对肠上皮细胞的能量代谢和免疫应答都有调节作用[21]。此外,丁酸还能够刺激肠道细胞产生抗菌肽类物质,协助抵御外来致病菌的侵袭,有助于抑制肠道炎症的发生和保护肠道健康[22]。此类细菌种属的增加可能是由于低蛋白质水平饲粮中碳水化合物及淀粉的比例显著增加,为这类细菌提供了足够的代谢底物。Paraeggerthella在正常蛋白质水平饲粮组中的相对丰度显著高于低蛋白质水平饲粮组,该菌属已被证明能够引起免疫功能不全人群的侵入性感染,易通过受损的肠上皮引起菌血症等疾病[23]。正常饲粮蛋白质组中数量较高的Delftia也是一个典型的机会致病菌,说明适当降低饲粮蛋白质水平引起的菌群改变可能有助于降低有害菌感染的机率。而Wellock等[24]和Bhandari等[25]研究也发现,降低断奶仔猪饲粮蛋白质水平可使仔猪肠道和粪便中大肠杆菌等致病菌的数量明显降低。低蛋白质水平饲粮大肠杆菌数量降低的原因被认为可能是供其增殖的底物减少造成的。此外,本研究中低蛋白质水平饲粮组Oscillibacter相对丰度显著高于对照组,Oscillibacter是一种主要产生戊酸的厌氧菌[26],过量戊酸对肠黏膜有刺激作用,可能会对肠道产生一定的调节和影响。曹克飞[14]研究表明,育肥猪饲喂用棉籽粕和玉米胚芽粕50%等氮替代豆粕的低蛋白质水平饲粮,可显著增高直肠粪便中Oscillibacter的相对丰度。本试验中生长育肥猪饲粮蛋白质水平的降低还显著影响了一些未分类菌群,这些未分类菌属的功能特性目前尚不清楚,因此,低蛋白质水平饲粮条件下这类细菌丰度增加或减少的原因还有待进一步阐明。
3.4 低蛋白质水平饲粮对生长育肥猪肠道菌群区系的影响由组间及物种差异性分析得出2组生长育肥猪肠道微生物群落被明显地分为2类,且低蛋白质水平饲粮组生长育肥猪肠道菌群能够较好地聚在一起,这说明低蛋白质水平饲粮能够明显地改善生长育肥猪肠道微生物区系。从以上的肠道菌群组成及物种差异显著性分析可以看出,饲喂低蛋白质水平饲粮的菌群中蛋白质和氨基酸降解菌以及丁酸产生菌等有益菌的数量增多,有害菌属的数量降低,表明饲粮蛋白质水平的适当降低能够使生长育肥猪肠道菌群向着更加有益健康和平衡的方向发展,对生长育肥猪的健康生长有促进作用。罗振等[27]研究结果也表明,低蛋白质水平饲粮组和正常蛋白质水平饲粮组肠道微生物存在明显差异,低蛋白质水平饲粮组微生物能较好的与正常蛋白质水平饲粮组分开,说明饲粮蛋白质水平的变化调节了肠道微生物区系。
4 结论在本次试验条件下,生长育肥猪饲粮蛋白质在正常水平基础上降低约2%并补充相应的合成氨基酸,不会影响生长育肥猪生长性能及健康状况,同时能够改善生长育肥猪肠道微生物的组成和区系,使其向着有利于生长育肥猪健康的方向发展。
| [1] |
KERR B J, SOUTHERN L L, BIDNER T D, et al. Influence of dietary protein level, amino acid supplementation, and dietary energy levels on growing-finishing pig performance and carcass composition[J]. Journal of Animal Science, 2003, 81(12): 3075-3087. DOI:10.2527/2003.81123075x |
| [2] |
KERR B J, MCKEITH F K, EASTER R A. Effect on performance and carcass characteristics of nursery to finisher pigs fed reduced crude protein, amino acid-supplemented diets[J]. Journal of Animal Science, 1995, 73(2): 433-40. DOI:10.2527/1995.732433x |
| [3] |
ISAACSON R, KIM H B. The intestinal microbiome of the pig[J]. Animal Health Research Reviews, 2012, 13(1): 100-109. DOI:10.1017/S1466252312000084 |
| [4] |
YANG Y X, DAI Z L, ZHU W Y. Important impacts of intestinal bacteria on utilization of dietary amino acids in pigs[J]. Amino Acids, 2014. DOI:10.1007/s00726-014-1807-y |
| [5] |
范沛昕.低蛋白饲粮对断奶仔猪和育肥猪肠道微生物区系的影响[D].硕士学位论文.北京: 中国农业大学, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10019-1016085516.htm
|
| [6] |
ZERVAS S, ZIJLSTRA R T. Effects of dietary protein and fermentable fiber on nitrogen excretion patterns and plasma urea in grower pigs[J]. Journal of Animal Science, 2002, 80(12): 3247-3256. DOI:10.2527/2002.80123247x |
| [7] |
PRANDINI A, SIGOLO S, MORLACCHINI M, et al. Microencapsulated lysine and low-protein diets:effects on performance, carcass characteristics and nitrogen excretion in heavy growing-finishing pigs[J]. Journal of Animal Science, 2013, 91(9): 4226-4234. DOI:10.2527/jas.2013-6412 |
| [8] |
邓敦.低蛋白饲粮补充必需氨基酸对猪营养生理效应的研究[D].博士学位论文.长沙: 中国科学院亚热带农业生态研究所, 2007.
|
| [9] |
TUITOEK K, YOUNG L G, DE LANGE C F M, et al. The effect of reducing excess dietary amino acids on growing-finishing pig performance:an elevation of the ideal protein concept[J]. Journal of Animal Science, 1997, 75(6): 1575-1583. DOI:10.2527/1997.7561575x |
| [10] |
FIGUEROA J L, LEWIS A J, MILLER P S, et al. Nitrogen metabolism and growth performance of gilts fed standard corn-soybean meal diets or low-crude protein, amino acid-supplemented diets[J]. Journal of Animal Science, 2002, 80(11): 2911-2919. DOI:10.2527/2002.80112911x |
| [11] |
KIM H B, ISAACSON R E. The pig gut microbial diversity:understanding the pig gut microbial ecology through the nest generation high throughput sequencing[J]. Veterinary Microbiology, 2015, 177(3/4): 242-251. |
| [12] |
ZHANG C, ZHANG M, WANG S, et al. Interactions between gut microbiota, host genetics and diet relevant to development of metabolic syndromes in mice[J]. The ISME Journal, 2009, 4(2): 232-241. |
| [13] |
KONSTANTINOV S R, FAVIER C F, ZHU W Y, et al. Microbial diversity studies of the porcine gastrointestinal ecosystem during weaning transition[J]. Animal Research, 2004, 53(4): 317-324. DOI:10.1051/animres:2004019 |
| [14] |
曹克飞.不同氮源对猪肠道微生物多样性及主要产蛋白酶菌株的影响[D].硕士学位论文.长春: 吉林农业大学, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10193-1016732406.htm
|
| [15] |
FAN P X, LIU P, SONG P X, et al. Moderate dietary protein restriction alters the composition of gut microbiota and improves ileal barrier function in adult pig model[J]. Scientific Reports, 2017, 7: 43412. DOI:10.1038/srep43412 |
| [16] |
SHOAIE S, KARLSSON F, MARDINOGLU A, et al. Understanding the interactions between bacteria in the human gut through metabolic modeling[J]. Scientific Reports, 2013, 3: 2532. DOI:10.1038/srep02532 |
| [17] |
曾燕霞, 王晶, 季海峰, 等. 低蛋白质饲粮对育肥猪生产性能、氮代谢及血液生化指标的影响[J]. 家畜生态学报, 2017, 38(6): 30-36. DOI:10.3969/j.issn.1673-1182.2017.06.007 |
| [18] |
DAI Z L, LI X L, XI P B, et al. Metabolism of select amino acids in bacteria from the pig small intestine[J]. Amino Acids, 2012, 42(5): 1597-1608. DOI:10.1007/s00726-011-0846-x |
| [19] |
BARRIOS C, BEAUMONT M, PALLISTER T, et al. Gut-microbiota-metabolite axis in early renal function decline[J]. PLoS One, 2015, 10(8): e0134311. DOI:10.1371/journal.pone.0134311 |
| [20] |
DENG Y F, LIU Y Y, ZHANG Y T, et al. Efficacy and role of inulin in mitigation of enteric sulfur-containing odor in pigs[J]. Journal of the Science of Food and Agriculture, 2017, 97(8): 2382-2391. DOI:10.1002/jsfa.2017.97.issue-8 |
| [21] |
DONOHOE D R, GARGE N, ZHANG X X, et al. The microbiome and butyrate regulate energy metabolism and autophagy in the mammalian colon[J]. Cell Metabolism, 2011, 13(5): 517-526. DOI:10.1016/j.cmet.2011.02.018 |
| [22] |
DOU X J, HAN J L, SONG W T, et al. Sodium butyrate improves porcine host defense peptide expression and relieves the inflammatory response upon toll-like receptor 2 activation and histone deacetylase inhibition in porcine kidney cells[J]. Oncotarget, 2017, 8(16): 26532-26551. |
| [23] |
LEE M R, HUANG Y T, LIAO C H, et al. Clinical and Microbiological characteristics of bacteremia caused by Eggerthella, Paraeggerthella, and Eubacterium species at a university hospital in Taiwan from 2001 to 2010[J]. Journal of Clinical Microbiology, 2012, 50(6): 2053-2055. DOI:10.1128/JCM.00548-12 |
| [24] |
WELLOCK I J, FORTOMARIS P D, HOUDIJK J G M, et al. The effect of dietary protein supply on the performance and risk of post-weaning enteric disorders in newly weaned pigs[J]. Animal Science, 2006, 82(3): 327-335. |
| [25] |
BHANDARI S K, OPAPEJU F O, KRAUSE D O, et al. Dietary protein level and probiotic supplementation effects on piglet response to Escherichia coli K88 challenge:performance and gut microbial population[J]. Livestock Science, 2010, 133(1/2/3): 185-188. |
| [26] |
ⅡNO T, MORI K, TANAKA K, 等. Oscillibacter valericigenes gen.nov., sp.nov., a valerate-producing anaerobic bacterium isolated from the alimentary canal of a Japanese corbicula clam[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(8): 1840-1845. |
| [27] |
罗振, 成艳芬, 朱伟云. 低蛋白饲粮对育肥猪盲肠代谢产物及菌群的影响[J]. 畜牧与兽医, 2015, 47(10): 5-9. |




