咖啡貂(Mustela iutreola)作为一种严格的食肉目鼬科动物,在野生条件下主要以小型啮齿动物、蛙类、鱼类等为食。在人工饲养条件下以畜、禽类副产品、海产品以及副产品等动物性饲料原料为主。在长期进化过程中,水貂形成了其独特的消化方式,肠道较短且直,结构较简单,排空速度快。家养成年短毛黑水貂肠道微生物主要以厚壁菌门、拟杆菌门、梭杆菌门、放线菌门和变形菌门为主,其中厚壁菌门约占细菌总数的60%[1]。正常的肠道菌群对宿主健康具有重要影响,在物质代谢、营养吸收、免疫调节和保护屏障等方面发挥重要作用[2],而饲粮改变会影响动物肠道微生物种类及数量[3]。咖啡貂作为我国近年来饲养的主要毛皮动物品种之一,对其肠道菌群的研究较少。本试验以舍饲的咖啡貂(丹麦引种咖啡貂第2代成年貂)为研究对象,饲喂营养水平基本一致但组成不同的饲粮,利用高通量测序技术对水貂后肠细菌中的16S rDNA序列进行定性分析,研究饲喂不同饲粮组成的咖啡貂肠道菌群多样性特征以及异同,为研究饲粮因素对肠道菌群组成与宿主特异性关系提供基础数据。
1 材料与方法 1.1 试验动物与饲粮管理从辽宁大连咖啡貂饲养场选取平均体重为(1.39±0.36) kg、(1.00±0.15)岁、健康的雄性水貂20只,随机分为2组,分别饲喂以海杂鱼、鸡骨架等为主的鲜饲料和50%鲜饲料+50%市售商品饲粮(加水)(由北京丰硕饲料有限公司提供,商品饲粮:水=1.7 : 1.0)的混合饲粮,每组10只,每只动物单笼饲养。试验期为30 d。试验饲粮组成及营养水平见表 1。早晚定时饲喂,自由饮水,试验期30 d,所有动物饲养管理均相同。
|
|
表 1 试验饲粮组成及营养水平(干物质基础) Table 1 Composition and nutrient levels of experimental diets (DM basis) |
水貂经一氧化碳窒息法处死后,立即取肠道后段(约30 cm)内容物,放入已灭菌的无菌塑料袋中。做好标记后,立即放入低温冰盒中,运回试验室,马上进行肠道细菌基因组DNA的提取。试验期间,分别于正式试验前、试验中、试验结束前1天采集鲜饲料和混合饲粮样品,根据文献[4]的方法进行饲料营养成分分析。
1.3 肠道菌群细菌组DNA提取及扩增利用FastDNA SPIN Kit for feces试剂盒(MP Pro,美国),严格按照操作说明书进行。将提取的DNA取2 μL利用电泳仪(120 V,30 min)检测,选取具有单一条带的DNA作为模板,扩增其16S rRNA V4~V5区,扩增引物采用515F、907R[5]。PCR采用20 μL扩增体系:4 μL 5×Fast Pfu Buffer,2 μL 2.5 mmol/L dNTPs,0.4 μL上游引物,0.4 μL下游引物,0.4 μL Fast Pfu Polymerase,10 ng Template DNA,加灭菌水补至20 μL。扩增条件:95 ℃预变性2 min;25个循环,95 ℃ 30 s,55 ℃ 30 s,72 ℃ 45 s;72 ℃延伸10 min。2%琼脂糖凝胶电泳检测PCR产物,采用QIA quick GelExtraction Kit(Qiagen,德国)回收纯化目的条带胶,将纯化产物送至上海美吉生物有限公司,测序平台为Illumina MiSeq。
1.4 生物信息学及统计学分析对有效序列进行去杂和甄别。设定具有97%以上相似性的序列为1个分类操作单元(OTU),特定的1个OTU代表某一特定细菌类群。通过QIIME计算菌群相对丰度和α多样性。本次分析选取评估指数包括:Ace指数、Chao1指数、Simpson指数、Shannon指数。Ace指数和Chao1指数用来计算菌群相对丰度,Simpson指数和Shannon指数用来计算菌群多样性。通过Shannon指数评估每个样品微生物群落的生态多样性。在门和属2个分类水平上统计样品的物种相对丰度和多样性。对所取得OTU数据进行主坐标分析(PCoA),根据β多样性距离矩阵进行层次聚类分析[6],结合不同细菌群落柱状图,使用非加权组平均法(UPGMA)构建树状结构,分析不同组别以及个体间微生物群落菌群多样性的分类学差异。利用SPSS 19.0单因素方差分析,将不同种类的主要细菌,在门、属水平进行差异显著性检验,P<0.05表示差异显著。
2 结果与分析 2.1 多样性指数所有样品均得到覆盖深度极深的测序数据(表 2)。20个样品,共获得761 866条高质量细菌序列用于分析。序列平均长度为357.24 bp。通过归类分析,共获得6 288个OTUs,所有OTUs通过与SILVA (SSU123)16S rRNA数据库进行BLAST比较后确定其微生物归类。本研究所获OTUs归类于25个菌门,425个属。表 2展示了OTUs在97%[与SILVA (SSU123)16S rRNA数据库对比]的置信度区间内的α多样性,2个组别的覆盖度基本均为100%。同时我们也分析了Ace指数、Chao1指数、Shannon指数、Simpson指数,样本的多样性分析(α多样性)可以反映微生物群落的相对丰度和多样性,从本研究数据可知,鲜饲料组与混合饲粮组相比,水貂肠内微生物多样性差异不明显。
|
|
表 2 鲜饲料组及混合饲粮组肠道菌群多样性指数(97%置信度区间) Table 2 Intestinal flora diversity index in fresh feed group and mixed diet group (97% confidence interval) |
在门分类水平进行远端肠道菌群结构分析,这些菌群在分类学上归属于6个门(图 1),分别为厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、未分类细菌门(Bacteria_Unclassified)、蓝菌门(Cyanobacteria)与拟杆菌门(Bacteroidetes)。其中厚壁菌门为水貂肠内相对丰度最高的细菌,在混合饲粮组和鲜饲料组分别为84.08%和83.50%,其次分别为变形菌门(9.37%和7.22%)、放线菌门(3.85%和5.72%)、未分类细菌门(1.69%和2.18%)、蓝细菌门(0.66%和0.85%)以及拟杆菌门(0.11%和0.32%)。这6个门占所有门类的99%以上。2组间微生物组成有差异,混合饲粮组第5只(H5)水貂肠道厚壁菌门的相对丰度(98.33%)高于其他动物,但差异不显著(P>0.05)。鲜饲料组及混合饲粮组水貂肠内微生物组成及数量变化趋势相似,所有细菌相对丰度未见显著差异(P>0.05)。2组间不同菌门统计学分析,未见显著差异(P>0.05)(图 2)。
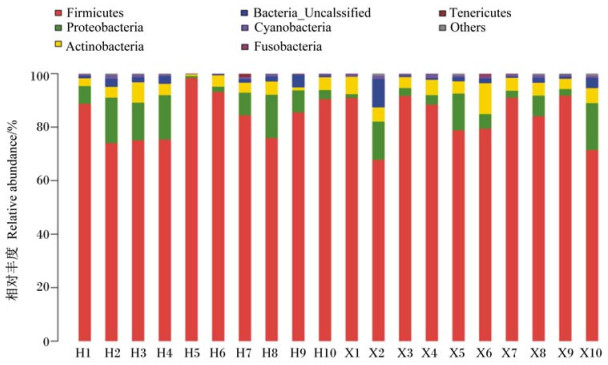
|
Firmicutes:厚壁菌门; Proteobacteria:变形菌门; Actinobacteria:放线菌门; Bacteria_Uncalssified:未分类细菌门;Cyanobacteria:蓝菌门;Fusobacteria:梭杆菌门; Tenericutes:柔膜菌门;Others:其他。 H1~H10代表混合饲粮组(H)水貂,X1~X10代表鲜饲料组(X)水貂。不同颜色柱形图代表不同的细菌门。图 3、图 5、图 6同。 H1 to H10 represent mink in mixed diet group, X1 to X10 represent mink in fresh feed group. Different color bars signal different phylum of bacteria. The same as Fig. 3, Fig. 5 and Fig. 6. 图 1 水貂肠道菌群门分类水平相对丰度图 Fig. 1 Relative abundance figure of bacterial community at phylum level in mink gut |
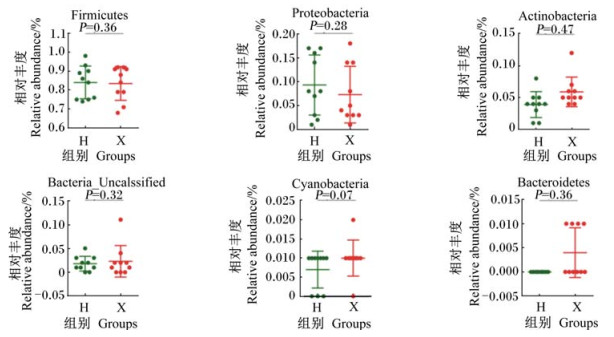
|
Firmicutes:厚壁菌门; Proteobacteria:变形菌门; Actinobacteria:放线菌门; Bacteria_Uncalssified:未分类细菌门;Cyanobacteria:蓝菌门;Bacteroidetes:拟杆菌门。 红色点代表鲜饲料组(X)水貂,绿色点代表混合饲粮组(H)水貂。图 4同。The red dot labeled for mink in fresh feed group (X), and green dot labeled for mink in mixed diet group (H). The same as Fig. 4. 图 2 门分类水平下不同菌群相对丰度差异分析 Fig. 2 Variation analysis of different bacteria relative abundance at phylum level |
在属分类水平上水貂肠道内细菌,初步归类为66个细菌属,约占所有细菌总数的95%以上,其中还含有少量其他细菌。本研究选择相对丰度高于0.1%的细菌属,绘制柱状图,其余归类于其他菌类(图 3),并对相对丰度最高的6个细菌属进行了差异显著性分析(图 4)。除混合饲粮组第5只动物(H5)外,乳酸杆菌属在其他9只动物中所占比例最高,相对丰度为21.4%~66.7%,平均相对丰度32.9%,鲜饲料组乳酸杆菌属相对丰度13.9%~37.2%,平均相对丰度22.2%,2组间差异不显著(P=0.41)。其次为魏斯氏菌属,混合饲粮组和鲜饲料组平均相对丰度分别为23.8%和32.2%,2组间差异不显著(P=0.48)。混合饲粮组与鲜饲料组芽孢杆菌属相对丰度分别为12.2%和14.0%。混合饲粮组与鲜饲料组链球菌属平均相对丰度分别为2.2%和1.5%。以上细菌占水貂肠道细菌总数的70%以上,其他细菌在数量上2组间差异均不显著(P>0.05)。但是尤其需要指出的是混合饲粮组第5只动物(H5)肠道内细菌组成结构与其他动物均不同,其中罗尔斯通菌属相对丰度为61.3%,曲霉菌属相对丰度为11.3%,其他细菌相对丰度为16.7%,乳酸杆菌相对丰度仅为3.1%。
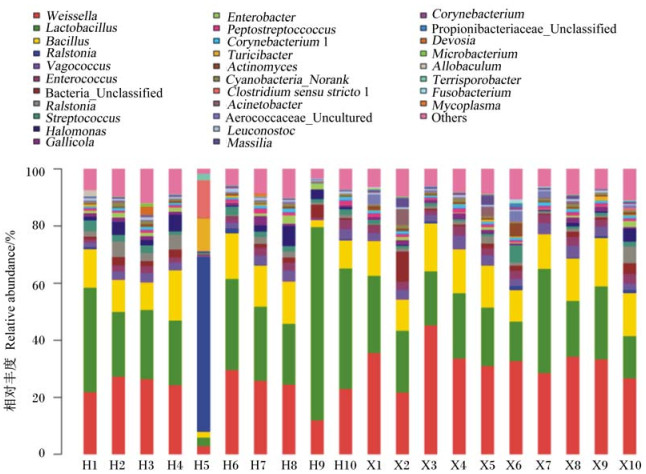
|
Weissella:魏斯氏菌属;Lactobacillus:乳杆菌属;Bacillus:芽孢杆菌属;Vagococcus:漫游球菌属;Enterococcus:肠球菌属;Bacteria_Unclassified:未分类细菌属;Ralstonia:罗尔斯通菌属;Streptococcus:链球菌属;Halomonas:盐单胞菌属;Enterobacter:肠杆菌属;Corynebacterium 1:棒状杆菌属1;Turicibacter:曲霉菌属;Actinomyces:放线菌属;Cyanobacteria_Norank:未分类细菌属;Clostridium sensu stricto 1:狭长梭菌属;Acinetobacter:不动杆菌属;Aerococcaceae_Uncultured:不可培养气球菌科;Leuconostoc:明串珠菌属;Massilia Corynebacterium:棒状杆菌属;Propionibacteriaceae_Unclassified:未分类丙酸杆菌科;Devosia:德沃斯氏菌属;Microbacterium:微杆菌属;Allobaculum Terrisporobacter:变形杆菌;Fusobacterium:梭杆菌属;Mycoplasma:支原体属;Others:其他。 图 3 水貂肠道菌群属分类水平相对丰度图 Fig. 3 Relative abundance figure of bacterial community at genus level in mink gut |
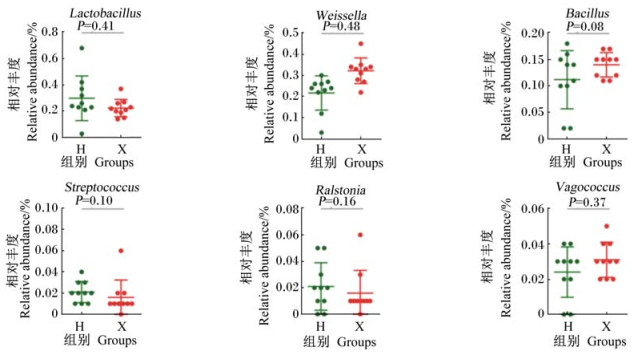
|
Lactobacillus:乳杆菌属;Weissella:魏斯氏菌属;Bacillus:芽孢杆菌属;Streptococcus:链球菌属;Ralstonia:罗尔斯通菌属;Vagococcus:漫游球菌属。 图 4 属分类水平下不同菌群相对丰度差异分析 Fig. 4 Variation analysis of different bacteria relative abundance at genus level |
通过上述水平的分析初步表明,饲喂不同饲粮组成的水貂肠道微生物群落结构虽然存在一定区别,但差异并不明显。为了进一步分析2组间肠道微生物群落结构的差别以及产生这些差别的原因,我们继续利用所有样品的OTUs信息构建了未加权的Unifrace距离矩阵,并通过样本聚类树与柱状图结合分析。基于Unifrace矩阵进行了PCoA分析。由图 5可知,前2个主成分分别解释了总变量的81.16%和7.43%,除混合饲粮组第5和第9只动物(H5、H9)在主成分1(PC1)轴的方向上距离较远,其他动物距离均较近。由图 6可知,除混合饲粮第5和第9只动物(H5、H9)外,其他组别动物均处于相近的分支,进一步说明其他动物肠道微生物群落结构相似。综合图 5和图 6可知,PCoA分析中,PC1在很大程度上代表了2组动物肠道微生物组成发生偏移的饲粮因素。由此说明混合饲粮组动物肠道菌群结构个体差异较大,而鲜饲料组肠道菌群结构个体差异小。
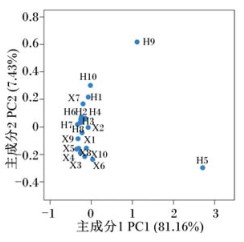
|
横、纵坐标轴的刻度只代表相对距离,并无实际意义。 The scale of horizontal and vertical axes only represent relative distance, and has no practical meaning. 图 5 咖啡貂肠道微生物结构的PCoA分析 Fig. 5 PCoA analysis of intestinal microbial structure of minks |
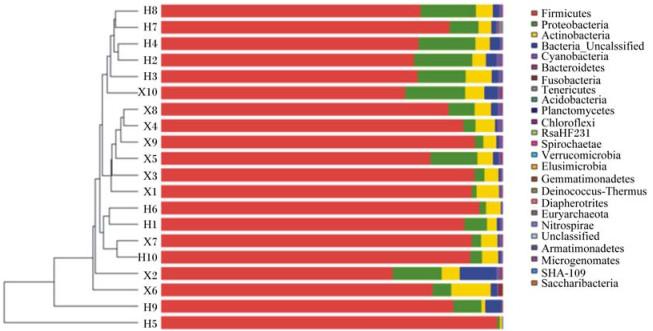
|
Firmicutes:厚壁菌门;Proteobacteria:变形菌门;Actinobacteria:放线菌门;Bacteria_Uncalssified:未分类细菌门;Cyanobacteria:蓝菌门;Bacteroidetes:拟杆菌门;Fusobacteria:梭杆菌门;Tenericutes:柔膜菌门;Acidobacteria:嗜酸菌门;Planctomycetes:浮霉菌门;Chloroflexi:绿弯菌门;Spirochaetae:螺旋体;Verrucomicrobia:疣微菌门;Elusimicrobia:迷踪菌门;Gemmatimonadetes:芽单胞菌门;Deinococcus-Thermus:奇异球菌门;Euryarchaeota:广域古细菌;Nitrospirae:硝化螺旋菌门;Unclassified:未分类;Armatimonadetes:装甲菌门;Microgenomates:微基因菌门;Saccharibacteria:糖菌门。 左侧是样本间基于群落组成的层次聚类分析(bray-curtis算法[7]),右侧是样本的群落结构柱状图。左侧是相似度树状图,样本之间的差异越小,样本便会处在相近的同一分支上;右侧柱状图,展示样本中微生物的群落结构, 不同颜色代表不同微生物。 The left shows the hierarchical clustering (bray-curtis algorithm[7]) based on the community composition of samples, and the right shows a histogram of the community structure of samples. In the similarity tree (left), samples with a smaller difference appear on the same branches close to each other. In the histogram (right), the microbiota community structure of the samples is showed, and different colors represent different microbiota. 图 6 聚类树与柱状图组合分析图 Fig. 6 Mcrobial community barplot with cluster tree |
水貂肠道较短,整个肠道为身体的3.5~4.0倍,食物排空速度快。由于较快的排空率可能会影响肠道微生物的群落和增殖,导致肠道菌群不能充分利用通过的营养物质,缺乏足够的代谢时间,进而难以为肠道厌氧菌提供适合的营养环境[8]。为了揭示一定时间内,基本相同营养水平的不同饲粮组成如何影响水貂肠道菌群组成,我们设计了本试验。
研究结果表明,饲喂鲜饲料和混合饲粮的水貂肠道细菌组成相似,但在数量方面存在一些差异,这也进一步验证了饲粮组成会影响动物后肠微生物的组成,但核心菌群保持相对稳定[9]。肠道中丰富的微生物已被证明在生物环境中起着重要作用,对宿主具有举足轻重的影响[9]。虽然本研究中个别动物的微生物群落组成有明显的差异,但根据报道在短期禁食状态下,水貂肠道黏膜伴生菌群基本不受环境的影响[3],对于产生这种结果的原因,我们分析可能是由于遗传因素或个体差异所导致,因为除环境影响以外,遗传因素在肠道菌群组成中发挥了重要作用[10]。为了应对复杂的饲粮环境,具有相对简单的消化系统的肉食动物肠道微生态区系具有较强的稳定性,才得以维护动物肠道正常的健康状态,但对于这一点仍需要进一步深入研究。
在本研究中,2组咖啡貂肠道内厚壁菌门平均相对丰度约为84%,远远高于其他菌门,这也进一步证实,厚壁菌门细菌广泛存在于哺乳动物肠道内[11],在短毛黑水貂、印度豹、狼、豺等肉食性动物粪便中厚壁菌门数量最多[12-14]。厚壁菌门是革兰氏阳性菌中数量最大、种类最丰富的细菌群类,同时也是研究最多的细菌种类[15],包含动物肠道中的正常菌群,如乳酸菌、肠球菌等[11, 15-16]。它们参与宿主的代谢过程,对食物的消化发挥重要作用[17]。在咖啡貂肠道内数量最多的乳酸杆菌属、魏斯氏菌属、芽孢杆菌属和消化链球菌属均属于厚壁菌门,在以往的报道中,这4类细菌主要参与肠道中的蛋白质、脂肪等物质的降解[18],这可能与动物的肉食性生物学习性有关。
变形菌门为咖啡貂肠道内第2大细菌门类,其中混合饲粮组和鲜饲料组所占的相对比例分别为9.37%和7.22%。β变形菌门中的罗尔斯通菌属所占比例最高达1.5%~2.2%。据报道,中国沿海地区水产养殖区水域中变形菌门为主要优势菌[19],在养殖区入水口和养殖区底泥中相对丰度分别为83.11%和53.39%[20]。因为本研究水貂饲粮中约含有29%的海杂鱼,因此变形菌门相对丰度高,可能与饲料中海杂鱼含量较高有关,但对于这一点仍需进一步研究验证。这与Fan等[1]所报道肠道细菌相对丰度有些差异,在他的研究中水貂肠道第2大丰富菌门为拟杆菌门(16.20%),其次分别为梭杆菌门(11.54%)和变形菌门(3.55%),其中变形菌门为第4大菌门,我们分析这可能与品种不同、饲养地区不同并且饲粮组成也具有较大差别有关。这可能也进一步说明遗传因素、饲粮组成等对动物肠道菌群结构和组成具有重要影响。
本研究中放线菌属相对丰度较高(混合饲粮组和鲜料组,分别为3.85%和5.72%),放线菌属广泛存在于动物肠道中[18, 21],其生长环境不同,习性和功能也存在差异,目前已获得的放线菌属多因其较强的抗菌素分泌能力而被分离[22]。有研究表明,东北虎肠道内也含有丰富的放线菌属群(15%)[23],这可能表明食性相近的动物,肠道菌群组成也较相近。但对于其具体的功能以及在食物消化中发挥的作用,也许可以通过进行分离培养,才能进一步验证。
拟杆菌门是单胃杂食和反刍动物肠道内的主要菌群[24],是饲粮多糖的重要降解菌,可将复杂的结构性碳水化合物降解成短链脂肪酸,为动物提供能量[25]。较高的厚壁菌门与拟杆菌门的比例可能更易促进宿主吸收储存能量[26]。本研究中拟杆菌门相对丰度远低于东北虎[22]、豺[14]等肉食性动物,甚至也远远低于短毛黑水貂。我们推测产生这种结果的原因可能与咖啡貂饲粮组成以及宿主特性有关,本研究所选择水貂为丹麦引种水貂,饲粮组成与国内水貂具有一定差异,主要以海杂鱼等动物性原料为主,其中植物性成分含量较少(<14%饲粮)。同时遗传因素也会对肠道菌群具有较大影响,具有宿主特异性的肠道微生态系统一旦建立,通常将伴随其一生[14],对于这一点仍需进一步研究验证。
在本研究中,咖啡貂肠内含有一定比例的蓝细菌门,蓝细菌通常普遍生长在淡水、海水和土壤中,在我国近海地区海水中存在一定含量的蓝细菌门细菌[19],因此在辽宁地区水貂肠道中检测到的蓝细菌可能与饲粮组成中含有较高比例海杂鱼有关。此外本研究中还存在一定数量的未分类细菌门,通过分子生物学技术的不断进步,会在分类学上逐步破译这些神秘的细菌。在不同动物消化道内某些细菌的相对含量随动物个体不同而有一些变化,很多低丰度菌门也可能存在特定的功能,但是咖啡貂肠道菌群的组成及功能特点仍需进行大量研究分析验证。
饮食结构会影响肠道菌群数量。高脂肪、高蛋白质饲粮使大鼠粪便拟杆菌属数量增加,而双歧杆菌属等数量下降,高碳水化合物饲粮会增加肠道拟杆菌门普雷沃氏菌属数量[27],本研究中咖啡貂肠内普雷沃氏菌属相对丰度与双歧杆菌属相对丰度均较低,可能也与饲粮中脂肪、蛋白质含量较高而植物性成分含量较低有关。此外本研究结果表明,咖啡貂肠道内存在0.1%~0.2%的Allobaculum,作为支原体科细菌,可能对动物的健康具有潜在的不利影响。
4 结论本研究表明鲜饲料或者混合饲粮对成年咖啡貂肠道菌群结构未产生较大影响,但不同分类细菌在相对丰度方面仍存在一些差异,混合饲粮组咖啡貂肠道菌群个体差异较大。在饲养的咖啡貂肠道内存在复杂的微生物区系,其中厚壁菌门、变形菌门、放线菌门为咖啡貂肠道最优势的细菌种类。
| [1] |
FAN Z Y, LIU H L, WANG F, et al. Determination of bacterial community composition in the distal gut of mink (Mustela vison) by high-throughput sequencing[J]. Microbiology China, 2016, 43(1): 123-130. |
| [2] |
NAGATA T, YAMADA H, KIRⅡKE N. Treatment of conditions preceding eating disorders, with social anxiety disorders as an example[J]. Seishin Shinkeigaku Zasshi, 2007, 109(12): 1129-1134. |
| [3] |
XU Z J, KNIGHT R. Dietary effects on human gut microbiome diversity[J]. British Journal of Nutrition, 2015, 113: S1-S5. DOI:10.1017/S0007114515001087 |
| [4] |
杨胜. 饲料分析及饲料质量检测技术[M]. 北京: 北京农业大学出版社, 1993.
|
| [5] |
SUN D L, JIANG X, WU Q L, et al. Intragenomic heterogeneity of 16S rRNA genes causes overestimation of prokaryotic diversity[J]. Applied and Environmental Microbiology, 2013, 79(19): 5962-5969. DOI:10.1128/AEM.01282-13 |
| [6] |
JIANG X T, PENG X, DENG G H, et al. Illumina sequencing of 16S rRNA tag revealed spatial variations of bacterial communities in a mangrove wetland[J]. Microbial Ecology, 2013, 66(1): 96-104. DOI:10.1007/s00248-013-0238-8 |
| [7] |
SRINIVASAN S, HOFFMAN N G, MORGAN M T, et al. Bacterial communities in women with bacterial vaginosis:high resolution phylogenetic analyses reveal relationships of microbiota to clinical criteria[J]. PLoS One, 2012, 7(6): e37818. DOI:10.1371/journal.pone.0037818 |
| [8] |
WILLIAMS C, ELNIF J, BUDDINGTON R K. The gastrointestinal bacteria of mink (Mustela vison L.):influence of age and diet[J]. Acta Veterinaria Scandinavica, 1998, 39(4): 473-482. |
| [9] |
BENJAMINO J, LINCOLN S, SRIVASTAVA R, et al. Low-abundant bacteria drive compositional changes in the gut microbiota after dietary alteration[J]. Microbiome, 2018, 6: 86. DOI:10.1186/s40168-018-0469-5 |
| [10] |
MURPHY K, O'SHEA C A, RYAN C A, et al. The gut microbiota composition in dichorionic triplet sets suggests a role for host genetic factors[J]. PLoS One, 2015, 10(4): e0122561. DOI:10.1371/journal.pone.0122561 |
| [11] |
ZHANG M, HANG X M, TAN J, et al. The host genotype and environment affect strain types of Bifidobacterium longum subsp. Longum inhabiting the intestinal tracts of twins[J]. Applied and Environmental Microbiology, 20156, 81(14): 4774-4781. |
| [12] |
BECKER A A M J, HESTA M, HOLLANTS J, et al. Phylogenetic analysis of faecal microbiota from captive cheetahs reveals underrepresentation of Bacteroidetes and Bifidobacteriaceae[J]. BMC Microbiology, 2014, 14: 13. DOI:10.1186/1471-2180-14-13 |
| [13] |
ZHANG H H, CHEN L. Phylogenetic analysis of 16S rRNA gene sequences reveals distal gut bacterial diversity in wild wolves (Canis lupus)[J]. Molecular Biology Reports, 2010, 37(8): 4013-4022. DOI:10.1007/s11033-010-0060-z |
| [14] |
WU X Y, ZHANG H H, CHEN J, et al. Comparison of the fecal microbiota of dholes high-throughput Illumina sequencing of the V3-V4 region of the 16S rRNA gene[J]. Applied Microbiology and Biotechnology, 2016, 100(8): 3577-3586. DOI:10.1007/s00253-015-7257-y |
| [15] |
ARSÈNE-PLOETZE F, NICOLOFF H, BRINGEL F. Lactobacillus plantarum ccl gene is non-essential, arginine-repressed and codes for a conserved protein in Firmicutes[J]. Archives of Microbiology, 2005, 183(5): 307-316. DOI:10.1007/s00203-005-0774-9 |
| [16] |
BERNINI L J, SIMÃO A N C, ALFIERI D F, et al. Beneficial effects of Bifidobacterium lactis on lipid profile and cytokines in patients with metabolic syndrome:a randomized trial.Effects of probiotics on metabolic syndrome[J]. Nutrition, 2016, 32(6): 716-719. DOI:10.1016/j.nut.2015.11.001 |
| [17] |
HUANG S W, ZHANG H Y. The impact of environmental heterogeneity and life stage on the hindgut microbiota of Holotrichia parallela larvae (Coleoptera:Scarabaeidae)[J]. PLoS One, 2013, 8(2): e57169. DOI:10.1371/journal.pone.0057169 |
| [18] |
LEY R E, HAMADY M, LOZUPONE C, et al. Evolution of mammals and their gut microbes[J]. Science, 2008, 320(5883): 1647-1651. DOI:10.1126/science.1155725 |
| [19] |
LIU M, DONG Y, ZHANG W C, et al. Diversity of bacterial community during spring phytoplankton blooms in the central Yellow Sea[J]. Canadian Journal of Microbiology, 2013, 59(5): 324-332. DOI:10.1139/cjm-2012-0735 |
| [20] |
赵晓伟, 丁君, 窦妍, 等. 基于MiSeq测序技术分析红鳍东方鲀养殖环境菌群多样性[J]. 生态学杂志, 2015, 34(10): 2965-2970. |
| [21] |
ZHU L, WU Q, DAI J, et al. Evidence of cellulose metabolism by the giant panda gut microbiome[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(43): 17714-17719. DOI:10.1073/pnas.1017956108 |
| [22] |
MATSUI T, TANAKA J, NAMIHIRA T, et al. Antibiotics production by an actinomycete isolated from the termite gut[J]. Journal of Basic Microbiology, 2012, 52(6): 731-735. DOI:10.1002/jobm.v52.6 |
| [23] |
CAO Y R, JIANG Y, JIN R X, et al. Enteractinococcus coprophilus gen. nov., sp.nov., of the family Micrococcaceae, isolated from Panthera tigris amoyensis faeces, and transfer of Yaniella fodinae Dhanjal et al.2011 to the genus Enteractinococcus as Enteractinococcus fodinae comb. nov[J]. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(Pt 11): 2710-2716. DOI:10.1099/ijs.0.034249-0 |
| [24] |
DICK L K, BERNHARD A E, BRODEUR T J, et al. Host distributions of uncultivated fecal Bacteroidales bacteria reveal genetic markers for fecal source identification[J]. Applied and Environmental Microbiology, 2005, 71(6): 3184-3191. DOI:10.1128/AEM.71.6.3184-3191.2005 |
| [25] |
BECKER A A M J, HESTA M, HOLLANTS J, et al. Phylogenetic analysis of faecal microbiota from captive cheetahs reveals underrepresentation of Bacteroidetes and Bifidobacteriaceae[J]. BMC Microbiology, 2014, 14: 43. DOI:10.1186/1471-2180-14-43 |
| [26] |
TURNBAUGH P J, LEY R E, MAHOWALD M A, et al. An obesity-associated gut microbiome with increased capacity for energy harvest[J]. Nature, 2006, 444(7122): 1027-1031. DOI:10.1038/nature05414 |
| [27] |
QIAO Y, SUN J, DING Y Y, et al. Alterations of the gut microbiota in high-fat diet mice is strongly linked to oxidative stress[J]. Applied Microbiology and Biotechnology, 2013, 97(4): 1689-1697. DOI:10.1007/s00253-012-4323-6 |




