动物肠道不仅是营养物质消化吸收的器官,还是机体最大的免疫器官,肠道微生物主要分布在肠腔内流动食糜中,且黏附于肠道黏膜及相关的黏膜层,发挥营养物质代谢调控、肠道上皮细胞修复、肠道黏膜免疫激活、宿主行为调控、抵抗病原微生物等重要作用[1-2]。肠道微生物与宿主高度互惠共生,被认为是一个混合器官,称作共生功能体。
人粪样中有3.3百万个非冗余微生物基因来自1 150多个物种,超过人体蛋白质复制基因约150倍[3],其中厚壁门菌与拟杆菌门最丰富,其次是变形菌门[4]。肠道微生物区系的平衡是宿主保持良好健康和发挥正常生产性能所必需的条件[5-6]。因此,阐明肠道微生物及其代谢产物的作用功能将有助于保障宿主的健康。
1 动物肠道微生物的分布通过对猪粪便微生物测序获得了770万个非冗余基因,719种微生物信息,并且猪的性别、年龄和基因型均会影响肠道微生物群的组成与分布[7]。畜禽肠道微生物的分布呈空间多样化,由于小肠和大肠内氧气和营养物质的差异,其肠道微生物组成显著不同。鸡小肠主要分布乳酸杆菌、肠球菌和各种梭菌[8],盲肠要分布厚壁菌门、变形菌与放线菌[9];猪盲肠黏膜微生物种类与肠腔微生物也有差异[10]。胃肠道微生物的密度从近端到远端逐步增加(胃内容物10个/g、十二指肠103个/g、空肠104个/g、回肠107个/g、结肠1012个/g),且沿组织-腔轴呈递增趋势[11]。但胃肠道微生物丰富度和多样性受胃肠道pH、胆汁酸浓度、食糜停留时间、黏液性质和宿主防御等因素的影响[12]。微生物在肠道的高密度分布对免疫系统是一个巨大的负荷,肠道免疫系统通过减少肠道微生物与上皮细胞表面的直接接触面积,以及环绕微生物的肠部组织网限制微生物在全身免疫区域的运动,进而控制和减少潜在病理隐患[13]。因此,微生物的丰富度和多样性变化时刻影响和调节肠道内环境的稳定。
仔猪断奶期受饲料、群体与环境改变的应激,应激诱导肠道微生物菌群的组成与分布发生变化,可导致仔猪出现厌食与肠道感染,直接影响其生长性能与健康[14]。研究显示,健康30日龄断奶仔猪肠道内的优势菌群是双歧杆菌、乳酸杆菌和梭菌[15]。断奶后藏猪肠道内大肠杆菌、乳酸杆菌和梭状杆菌数量显著多于长白猪[16]。断奶和经产荣昌猪肠道微生物定植类群高于长白、杜洛克猪[17]。通过构建猪肠道微生物的系统网络,发现猪与人的微生物网络极其相似,主要为普雷沃氏菌和瘤胃球菌2个肠型,肠型与60日龄猪体重和平均日增重显著相关,与猪的生长表型密切相关[18]。拟杆菌属可以分解碳水化合物,此肠型的人相对不易发胖;普氏菌属可以分解引发肠道疼痛的肠黏液;而某些瘤胃球菌属有助于细胞对糖分的吸收,导致体重增加[19]。因此,肠型不仅可以评价猪生长性能,更可以评估人体的膳食偏好与健康状况。
研究还发现,肠道微生物群数量越多、分配的均匀度越高,菌群之间的相互依赖和制约能力就越强,当机体遭受应激刺激或者外界环境波动较大时菌群能更好地保持动态平衡,从而增加机体应对不良因素的能力[20]。例如,肠道微生物嗜黏蛋白-艾克曼菌(Akkermansia muciniphila,A. muciniphila)有助于调节宿主肥胖,减少肥胖鼠的肠道A. muciniphila的丰度,并且采用普通的填喂法再引入该细菌,能够提高上皮细胞屏障功能和减少饮食诱导的肥胖[21]。而以粪球菌替代抗生素,猪肠道菌群丰度和多样性会降低,粪球菌和抗生素均显著降低纤维菌门的丰度,二者对猪肠道菌群造成的变化相似[22]。因此,肠道微生物的多样性及其组成与分布均与畜禽生长性能以及人体健康密切相关,值得我们研究与关注。
2 肠道微生物代谢产物动物肠道微生物可帮助宿主消化复杂的食物,并合成基本代谢产物,例如维生素K和B族维生素[28]。哺乳动物血液中10%的代谢产物来自肠道微生物[23]。肠道微生物也通过直接参与饮食成分消化分解产生代谢产物、利用生化修饰宿主消化产物合成代谢产物和重新合成的代谢产物等途径参与宿主生理、免疫的发展与调控[24]。
肠道微生物能够通过生化修饰参与营养物质的分解与加工促进宿主营养代谢。肠道中含丰富的乙酸、丙酸和丁酸等短链脂肪酸(SCFAs),其主要来自大肠植物发酵宿主酶不能消化的多糖和纤维素[25],其中肠道内拟杆菌门主要生产乙酸与丙酸,厚壁菌门能够低效的利用多糖产生丁酸,促进SCFAs的产生与利用[26]。肠道微生物还参与胆汁酸盐的代谢,在小肠中部分原代胆汁酸可以被局部微生物吸收和转化,其大部分被结肠微生物转化为次级胆汁酸被宿主有效地吸收和利用,有利于宿主肠道营养物质的代谢与吸收[27]。
肠道微生物也可自身合成代谢产物,维持宿主的免疫与健康。研究表明,肠道微生物能自身合成ATP,无菌小鼠的肠道ATP浓度比对照组低[28]。而普通肠道细菌脆弱拟杆菌可以生产两性离子多糖A(PSA),参与免疫细胞的活化[29]。肠道微生物通过产生芳香烃受体(AhR)的配体恢复AhR信号通路对代谢综合征的有益作用[30]。肠道微生物衍生的代谢产物(如丁酸盐)通过胰腺内先天性淋巴细胞(ILCs)控制白细胞介素(IL)-22分泌[31]。因此,肠道微生物的代谢产物均可直接或间接参与肠道免疫,促进宿主营养吸收和肠道健康。
3 肠道微生物与机体免疫 3.1 肠道微生物对免疫细胞的影响肠道微生物群的改变及其代谢产物均可激活免疫细胞和诱导其分泌免疫因子,进而对机体肠道免疫进行调控。研究发现,某些菌株使免疫球蛋白(Ig)A+B细胞的数量显著增加,其中革兰氏阳性菌可以增加分泌IL-10的细胞数量(图 1-A),革兰氏阴性菌可以增加分泌IL-12的细胞数量(图 1-B),相反,IL-10分泌量不足的小鼠肠道微生物群的组成与多样性降低;结肠炎引起肠黏膜免疫受损的小鼠肠道菌群的大肠杆菌、放线菌、变形菌等微生物的数量提高,产气芽孢梭菌等微生物的数量降低[32](图 1-C)。调节性T细胞(Tregs)及其亚群在免疫调控中发挥重要作用,无菌饲养的动物的CD4+ T淋巴细胞发育欠佳,而脆弱类杆菌可以通过释放PSA弥补这种缺陷,并且无菌饲养的动物的CD4+ T淋巴细胞比普通饲养的同龄动物的要少,而脆弱类杆菌的转入可以促进这类免疫细胞的发育[33](图 1-D)。因此,肠道微生物参与免疫细胞的发育与成熟,促进宿主免疫系统的完善。
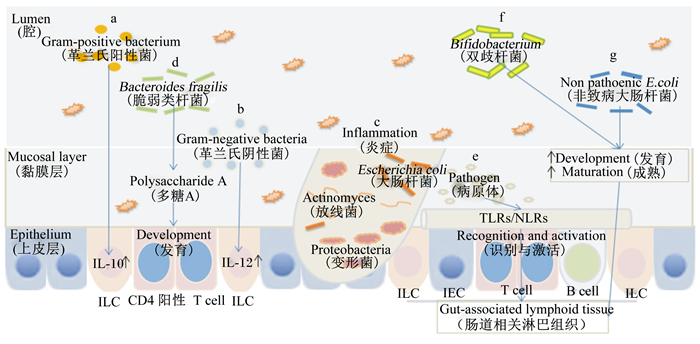
|
a:革兰氏阳性菌激活白细胞介素-10的表达Gram positive bacteria activate the expression of interleukin-10;b:革兰氏阴性菌激活白细胞介素-12的表达Gram negative bacteria activate the expression of interleukin-12;c:肠道炎症的发生The occurrence of intestinal inflammation;d:脆弱类杆菌产生多糖A,诱导CD4+T淋巴细胞的发育Production of polysaccharide A by Bacteroidesfragilis induces the development of CD4+T lymphocytes;e:肠上皮表面的Toll样受体与NOD样受体识别病原体并激活肠道相关淋巴组织Identification of pathogens and activation of gut associated lymphoid tissue by TLRs and NLRs on the intestinal epithelium surface;f:双歧杆菌促进肠道相关淋巴组织的发育与成熟Bifidobacterium promote the development and maturation of gut associated lymphoid tissue;g:非致病性的埃希氏大肠杆菌刺激肠道相关淋巴组织的发育与成熟Non pathogenic Escherichia coli stimulates the development and maturation of gut associated lymphoid tissue; TLRs:Toll样受体Toll-like receptor;NLRs:NOD样受体NOD-like receptors;ILC:先天性淋巴细胞Innate lymphoid cells;IEC:肠上皮细胞Intestinal epithelial cells;T cell:T细胞;B cell:B细胞。 图 1 肠道微生物对肠道免疫细胞与淋巴组织的影响 Fig. 1 Effects of intestinal microbial on intestinal immune cells and lymphoid tissue[32-36] |
肠上皮屏障由肠道上皮细胞、上皮内淋巴细胞等组成,在其表面具有Toll样受体(TLRs)和NOD样受体(NLRs)上的病原体或病原体相关分子模式,肠道免疫系统每天都面临环境及食物中的大量抗原,如Peyer氏斑(次级淋巴组织)和淋巴滤泡(三级淋巴组织)能够响应抗原刺激释放细胞因子或IgA[34](图 1-E)。研究表明,婴儿早期粪便中双歧杆菌的数量和黏膜分泌型免疫球蛋白A(SIgA)浓度显著相关,表明双歧杆菌的种类及数量可以促进黏膜SIgA分泌系统发育及成熟[35](图 1-F);此外,给予新生仔猪口服非致病性的埃希氏大肠杆菌,结果表明非致病菌摄入初期能够引起肠道黏膜以及宿主体液免疫反应,之后肠道特异性免疫应答受到抑制,进而出现口服耐受[36](图 1-G)。因此,肠道微生物能够促进宿主肠道免疫组织的发育,维持宿主的生命健康。
4 肠道微生物代谢产物对肠道免疫的影响 4.1 SCFAs对免疫的影响研究表明,在饮食过程中适量摄入含纤维食物,不仅有助于食物的充分消化,更有助于预防炎症,保障胃肠道内环境的稳定与健康[37]。SCFAs影响肠道免疫系统的主要表现为抗炎特性。在结肠炎的小鼠模型中,SCFAs被证明通过游离脂肪酸受体2(FFAR2)和羟基羧酸受体2(HCAR2)激活NOD样受体家族蛋白3(NLRP3)炎症小体以减轻疾病严重性,诱导上皮修复细胞因子IL-18的分泌[38-39]。可见,SCFAs可以促进肠道上皮屏障的完整性。在细胞水平,SCFAs通过核Ⅰ类组蛋白脱乙酰酶(HDAC)抑制调节基因表达调节B细胞的能量代谢以促进抗体的产生[40]。同时SCFAs也可促进效应细胞和Tregs的产生[41](图 2-a)。因此肠道微生物通过纤维分解产生的SCFAs有助于维持肠道免疫系统的稳态,促进宿主的肠道健康。
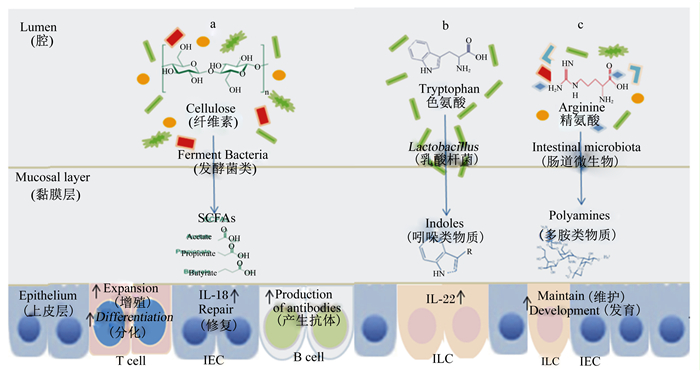
|
a:发酵菌类通过粗纤维发酵产生短链脂肪酸,调控肠上皮细胞与免疫细胞的作用Fermentation bacteria produce SCFAs through cellulose fermentation, and regulate the function of intestinal epithelial cells and immune cells;b:乳杆菌分解色氨酸产生吲哚类物质,激活白细胞介素-22的表达Lactobacillus decomposes tryptophan to produce indole substance, and activates the expression of interleukin-22;c:肠道微生物分解精氨酸产生多胺类物质,促进肠黏膜与免疫细胞的发育与维持Intestinal microbes decompose arginine to produce polyamines, which promote the development and maintenance of intestinal mucosa and immune cells;ILC:先天性淋巴细胞Innate lymphoid cells;IEC:肠上皮细胞Intestinal epithelial cells;SCFAs:短链脂肪酸Short chain fatty acids;IL-18:白细胞介素-18 Interleukin-18;IL-22:白细胞介素-22 Interleukin-22;T cell:T细胞;B cell:B细胞。 图 2 肠道微生物代谢产物对肠道免疫的影响 Fig. 2 Effects of intestinal microbial metabolites on intestinal immune[41-47] |
肠道微生物群如乳杆菌可将色氨酸分解代谢为芳香烃受体(AhR)的配体吲哚-3-醛[42]。AhR由ILC表达,并且其激活IL-22的表达,IL-22是调节肠黏膜稳态的关键细胞因子,并且对白色念珠菌具有抗性[43](图 2-b)。此外,多胺如高反应性多胺腐胺(二胺,N2)、亚精胺(N3)和精胺(N4)都是由肠道微生物产生和分泌的精氨酸衍生物[44]。多胺口服给药增强了肠黏膜和常驻免疫细胞的发育和维持[45],且在局部和全身炎症的小鼠模型中表现出保护性抗炎作用[46-47],但具体作用机制尚不清楚(图 2-c)。因此,肠道微生物代谢产物能参与并维持肠道免疫系统的健康。
4.3 肠道微生物自身合成代谢产物对肠道免疫的影响肠道微生物也可自身合成代谢产物,如ATP与PSA。在生物化学水平上,细胞外ATP是配体门控离子通道受体(P2X)的唯一激动剂,ATP与P2X7的结合导致钠离子(Na+)和钙离子(Ca2+)的流入和钾离子(K+)流出,并引起LPS刺激的巨噬细胞中的NLRP3炎性体的激活,导致分泌IL-18,促进上皮屏障的维持和功能的发挥[48]。PSA能激活在树突状细胞(DC)表面Toll样受体2(TLR2)的表达,进而促进T细胞产生IL-10[49],并且PSA与在FoxP3+Treg上表达的TLR2的直接结合,可进一步增强IL-10的产生[50]。PSA通过诱导IL-10的产生进而抑制黏膜效应T细胞,特别是TH17细胞的活性,从而对小鼠结肠炎具有保护作用[51]。因此,肠道微生物自身代谢产物在维持宿主肠道免疫与健康中发挥重要作用。
5 小结与展望肠道微生物与机体免疫之间的相互作用机制十分复杂,至今尚未完全阐明,深入了解两者之间的关系将有助于我们认识“肠道微生物-畜禽健康”的相互调控机理。我国地方畜禽遗传资源丰富,普遍具有耐粗饲、适应性好、抗逆性强的优点,进一步开展肠道微生物及其代谢产物与地方畜禽耐粗饲、适应性和抗逆性的调控机制研究,将有助于利用我国地方丰富畜禽遗传资源,开发新型饲用微生态物制剂,解决目前畜禽养殖过程中存在的发病率高、适应性不好等问题,促进畜禽健康养殖可持续发展。另外,相较于小鼠等模式动物而言,猪更适合作为人类疾病的模型动物,解析人类疾病特别是肠道疾病与肠道微生物的相互调控关系及其作用机制,从而研发新药或开发新的治疗手段,为人类健康做贡献。
| [1] |
BLASER M J. The microbiome revolution[J]. The Journal of Clinical Investigation, 2014, 124(10): 4162-4165. DOI:10.1172/JCI78366 |
| [2] |
HOOPER L V. Epithelial cell contributions to intestinal immunity[J]. Advances in Immunology, 2015, 126: 129-172. DOI:10.1016/bs.ai.2014.11.003 |
| [3] |
QIN J J, LI R Q, RAES J, et al. A human gut microbial gene catalogue established by metagenomic sequencing[J]. Nature, 2010, 464(7285): 59-65. DOI:10.1038/nature08821 |
| [4] |
The Human Microbiome Project Consortium. Structure, function and diversity of the healthy human microbiome[J]. Nature, 2012, 486(7402): 207-214. DOI:10.1038/nature11234 |
| [5] |
GAO W, MENG Q X. Numeration of normal bacterial populations in gastrointestinal tract of finishing swine[J]. Journal of China Agricultural University, 2000, 5(5): 88-93. |
| [6] |
BRODIN P, JOJIC V, GAO T X, et al. Variation in the human immune system is largely driven by non-heritable influences[J]. Cell, 2016, 160(1/2): 37-47. |
| [7] |
XIAO L, ESTELLÉ J, KⅡLERICH P, et al. A reference gene catalogue of the pig gut microbiome[J]. Nature Microbiology, 2016, 1: 16161. DOI:10.1038/nmicrobiol.2016.161 |
| [8] |
STANLEY D, HUGHES R J, MOORE R J. Microbiota of the chicken gastrointestinal tract:influence on health, productivity and disease[J]. Applied Microbiology and Biotechnology, 2014, 98(10): 4301-4310. DOI:10.1007/s00253-014-5646-2 |
| [9] |
OAKLEY B B, LILLEHOJ H S, KOGUT M H, et al. The chicken gastrointestinal microbiome[J]. FEMS Microbiology Letters, 2014, 360(2): 100-112. DOI:10.1111/fml.2014.360.issue-2 |
| [10] |
KELLY J, DALY K, MORAN A W, et al. Composition and diversity of mucosa-associated microbiota along the entire length of the pig gastrointestinal tract; dietary influences[J]. Environmental Microbiology, 2017, 19(4): 1425-1438. DOI:10.1111/1462-2920.13619 |
| [11] |
SEKIROV I, RUSSELL S L, ANTUNES L C M, et al. Gut microbiota in health and disease[J]. Physiological Reviews, 2010, 90(3): 859-904. DOI:10.1152/physrev.00045.2009 |
| [12] |
BUFFIE C G, PAMER E G. Microbiota-mediated colonization resistance against intestinal pathogens[J]. Nature Reviews Immunology, 2013, 13(11): 790-801. DOI:10.1038/nri3535 |
| [13] |
MILLER C P, BOHNHOFF M, RIFKIND D. The effect of an antibiotic on the susceptibility of the mouse's intestinal tract to Salmonella infection[J]. Transactions of the American Clinical and Climatological Association, 1957, 68: 51-58. |
| [14] |
GRESSE R, CHAUCHEYRAS-DURAND F, FLEURY M A, et al. Gut microbiota dysbiosis in postweaning piglets:understanding the keys to health[J]. Trends in Microbiology, 2017, 25(10): 851-873. DOI:10.1016/j.tim.2017.05.004 |
| [15] |
孙笑非, 孙冬岩, 王文娟, 等. 猪肠道微生物定植规律和生理功能[J]. 饲料研究, 2011(9): 70-72. |
| [16] |
李江凌, 陈晓晖, 刘锐, 等. 藏猪、长白猪肠道微生物菌群的定量分析及比较研究[J]. 中国猪业, 2016, 11(3): 61-64. DOI:10.3969/j.issn.1673-4645.2016.03.023 |
| [17] |
杨柳, 张邑帆, 郑华, 等. 荣昌、长白、杜洛克猪肠道微生物ERIC-PCR-DGGE指纹图谱比较分析[J]. 家畜生态学报, 2011, 32(5): 21-25. DOI:10.3969/j.issn.1673-1182.2011.05.005 |
| [18] |
RAMAYO-CALDAS Y, MACH N, LEPAGE P, et al. Phylogenetic network analysis applied to pig gut microbiota identifies an ecosystem structure linked with growth traits[J]. The ISME Journal, 2016, 10(12): 2973-2977. DOI:10.1038/ismej.2016.77 |
| [19] |
JONES N. Gut study divides people into three types[J]. Nature, 2011. DOI:10.1038/news.2011.249 |
| [20] |
HOOPER L V, MACPHERSON A J. Immune adaptations that maintain homeostasis with the intestinal microbiota[J]. Nature Reviews Immunology, 2010, 10(3): 159-169. DOI:10.1038/nri2710 |
| [21] |
EVERARD A, BELZER C, GEURTS L, et al. Cross-talk between Akkermansia Muciniphila and intestinal epithelium controls diet-induced obesity[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(22): 9066-9071. DOI:10.1073/pnas.1219451110 |
| [22] |
LI P H, NIU Q, WEI Q T, et al. Microbial shifts in the porcine distal gut in response to diets supplemented with Enterococcus Faecalis as alternatives to antibiotics[J]. Scientific Reports, 2017, 7: 41395. DOI:10.1038/srep41395 |
| [23] |
CUMMINGS J H, MACFARLANE G T. Role of intestinal bacteria in nutrient metabolism[J]. Clinical Nutrition, 1997, 16(1): 3-11. DOI:10.1016/S0261-5614(97)80252-X |
| [24] |
WIKOFF W R, ANFORA A T, LIU J, et al. Metabolomics analysis reveals large effects of gut microflora on mammalian blood metabolites[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(10): 3698-3703. DOI:10.1073/pnas.0812874106 |
| [25] |
POSTLER T S, GHOSH S. Understanding the holobiont:how microbial metabolites affect human health and shape the immune system[J]. Cell Metabolism, 2017, 26(1): 110-130. DOI:10.1016/j.cmet.2017.05.008 |
| [26] |
MORRISON D J, PRESTON T. Formation of short chain fatty acids by the gut microbiota and their impact on human metabolism[J]. Gut Microbes, 2016, 7(3): 189-200. DOI:10.1080/19490976.2015.1134082 |
| [27] |
LEVY M, THAISS C A, ELINAV E. Metabolites:messengers between the microbiota and the immune system[J]. Genes & Development, 2016, 30(14): 1589-1597. |
| [28] |
MARTÍNEZ-AUGUSTIN O, DE MEDINA F S. Intestinal bile acid physiology and pathophysiology[J]. World Journal of Gastroenterology, 2008, 14(37): 5630-5640. DOI:10.3748/wjg.14.5630 |
| [29] |
HIRONAKA I, IWASE T, SUGIMOTO S, et al. Glucose triggers ATP secretion from bacteria in a growth-phase-dependent manner[J]. Applied and Environmental Microbiology, 2013, 79(7): 2328-2335. DOI:10.1128/AEM.03871-12 |
| [30] |
NATIVIDAD J M, AGUS A, PLANCHAIS J, et al. Impaired aryl hydrocarbon receptor ligand production by the gut microbiota is a key factor in metabolic syndrome[J]. Cell Metabolism, 2018. DOI:10.1016/j.cmet.2018.07.001 |
| [31] |
MIANI M, LE NAOUR J, WAECKEL-ENÉE E, et al. Gut microbiota-stimulated innate lymphoid cells support β-defensin 14 expression in pancreatic endocrine cells, preventing autoimmune diabetes[J]. Cell Metabolism, 2018. DOI:10.1016/j.cmet.2018.06.012 |
| [32] |
SHARMA S, ERICKSON K M, TROUTMAN J M. Complete tetrasaccharide repeat unit biosynthesis of the immunomodulatory Bacteroides fragilis capsular polysaccharide A[J]. ACS Chemical Biology, 2017, 12(1): 92-101. DOI:10.1021/acschembio.6b00931 |
| [33] |
SEKELJA M, RUD I, KNUTSEN S H, et al. Abrupt temporal fluctuations in the chicken fecal microbiota are explained by its gastrointestinal origin[J]. Applied and Environmental Microbiology, 2012, 78(8): 2941-2948. DOI:10.1128/AEM.05391-11 |
| [34] |
BREGENHOLT S. Cells and cytokines in the pathogenesis of inflammatory bowel disease:new insights from mouse T cell transfer models[J]. Experimental and Clinical Immunogenetics, 2000, 17(3): 115-129. DOI:10.1159/000019131 |
| [35] |
MAGRONE T, JIRILLO E. The interplay between the gut immune system and microbiota in health and disease:nutraceutical intervention for restoring intestinal homeostasis[J]. Current Pharmaceutical Design, 2013, 19(7): 1329-1342. |
| [36] |
POZO-RUBIO T, DE PALMA G, MUJICO J R, et al. Influence of early environmental factors on lymphocyte subsets and gut microbiota in infants at risk of celiac disease; the PROFICEL study[J]. Nutricion Hospitalaria, 2013, 28(2): 464-473. |
| [37] |
CUKROWSKI I, CUKROWSKA E. Direct zinc determination in commercial sulphuric acid by dpasv[J]. Talanta, 1993, 40(2): 205-211. DOI:10.1016/0039-9140(93)80323-J |
| [38] |
DESAI M S, SEEKATZ A M, KOROPATKIN N M, et al. A dietary fiber-deprived gut microbiota degrades the colonic mucus barrier and enhances pathogen susceptibility[J]. Cell, 2016, 167(5): 1339-1353. DOI:10.1016/j.cell.2016.10.043 |
| [39] |
MACIA L, TAN J, VIEIRA A T, et al. Metabolite-sensing receptors GPR43 and GPR109A facilitate dietary fibre-induced gut homeostasis through regulation of the inflammasome[J]. Nature Communications, 2015, 6: 6734. DOI:10.1038/ncomms7734 |
| [40] |
SINGH N, GURAV A, SIVAPRAKASAM S, et al. Activation of Gpr109a, receptor for niacin and the commensal metabolite butyrate, suppresses colonic inflammation and carcinogenesis[J]. Immunity, 2014, 40(1): 128-139. DOI:10.1016/j.immuni.2013.12.007 |
| [41] |
KIM M, QIE Y Q, PARK J, et al. Gut microbial metabolites fuel host antibody responses[J]. Cell Host & Microbe, 2016, 20(2): 202-214. |
| [42] |
PARK J, KIM M, KANG S G, et al. Short-chain fatty acids induce both effector and regulatory T cells by suppression of histone deacetylases and regulation of the mTOR-S6K pathway[J]. Mucosal Immunology, 2015, 8(1): 80-93. DOI:10.1038/mi.2014.44 |
| [43] |
ZELANTE T, IANNITTI R G, CUNHA C, et al. Tryptophan catabolites from microbiota engage aryl hydrocarbon receptor and balance mucosal reactivity via interleukin-22[J]. Immunity, 2013, 39(2): 372-385. DOI:10.1016/j.immuni.2013.08.003 |
| [44] |
LAMAS B, RICHARD M L, LEDUCQ V, et al. CARD9 impacts colitis by altering gut microbiota metabolism of tryptophan into aryl hydrocarbon receptor ligands[J]. Nature Medicine, 2016, 22(6): 598-605. DOI:10.1038/nm.4102 |
| [45] |
MICHAEL A J. Biosynthesis of polyamines and polyamine-containing molecules[J]. Biochemical Journal, 2016, 473(15): 2315-2319. DOI:10.1042/BCJ20160185 |
| [46] |
PÉREZ-CANO F J, GONZÁLEZ-CASTRO A, CASTELLOTE C, et al. Influence of breast milk polyamines on suckling rat immune system maturation[J]. Developmental & Comparative Immunology, 2010, 34(2): 210-218. |
| [47] |
TER STEEGE J C A, FORGET P P, BUURMAN W A. Oral spermine administration inhibits nitric oxide-mediated intestinal damage and levels of systemic inflammatory mediators in a mouse endotoxin model[J]. Shock, 1999, 11(2): 115-119. DOI:10.1097/00024382-199902000-00008 |
| [48] |
SUTTERWALA F S, HAASKEN S, CASSEL S L. Mechanism of NLRP3 inflammasome activation[J]. Annals of the New York Academy of Sciences, 2014, 1319: 82-95. DOI:10.1111/nyas.2014.1319.issue-1 |
| [49] |
YARON J R, GANGARAJU S, RAO M Y, et al. K+ regulates Ca2+ to drive inflammasome signaling:dynamic visualization of ion flux in live cells[J]. Cell Death & Disease, 2015, 6(10): e1954. |
| [50] |
TELESFORD K M, YAN W, OCHOA-REPARAZ J, et al. A commensal symbiotic factor derived from Bacteroides fragilis promotes human CD39+Foxp3+ T cells and Treg function[J]. Gut Microbes, 2015, 6(4): 234-242. DOI:10.1080/19490976.2015.1056973 |
| [51] |
ROUND J L, LEE S M, LI J, et al. The Toll-like receptor 2 pathway establishes colonization by a commensal of the human microbiota[J]. Science, 2011, 332(6032): 974-977. DOI:10.1126/science.1206095 |




