豆粕作为大豆加工的副产品,其粗蛋白质含量为40%~50%[1],已广泛用于畜禽的饲料。然而豆粕中所含的多种抗营养因子如抗原蛋白不利于断奶仔猪的消化吸收[2],易导致腹泻[3-4],阻碍了仔猪的生长,降低其成活率。利用微生物发酵豆粕可以有效地降低豆粕中抗营养因子的含量[5-6],提高养分利用率[7],提高仔猪的生长性能[8]。单达聪等[9]研究结果表明,发酵豆粕替代普通豆粕用于断奶仔猪可使日增重提高9.9%~58.0%,料重比降低8.3%~16.1%, 腹泻率降低13.3%~77.9%。发酵豆粕还可使仔猪的粗蛋白质表观消化率提高2.8%~20.8%[7, 10-11]。此外,发酵所用的益生菌还能增加肠道微生物多样性,降低病原菌数量,减少肠道疾病发生[12-13]。
研究表明,每克肠道内容物中微生物分属30多个属,500多个种[14],总数高达1013~1014个[15]。猪具有发达的大肠,但肠道微生物易受年龄、饲养环境、抗生素的使用和食物类型及形态等[16]的影响。前人在研究肠道微生物时多用粪便中的微生物来代替,原因是粪便微生物组成与大肠内微生物组成相似度可达到75%[17]。采用平皿培养粪便微生物只能对可分离培养的细菌进行计数,而对粪便微生物的16S rRNA进行高通量测序能高度精准地对所有微生物进行分类鉴定,从而反映肠道微生物的真实组成和比例[18]。此外,由于发酵豆粕时所用菌种、原料、发酵工艺及饲喂仔猪的日龄等的不同,其在断奶仔猪饲粮中的添加量未达到一致。因此,本研究通过对饲喂不同含量发酵豆粕饲粮的断奶仔猪的粪便进行Illumina MiSeq高通量测序,来探讨发酵豆粕对断奶仔猪肠道微生物组成及代谢功能的影响,为研究发酵豆粕在仔猪饲粮中的合理使用以及肠道菌群功能提供理论基础。
1 材料与方法 1.1 试验材料发酵豆粕的制备采用以乳酸菌为主要发酵菌,酿酒酵母菌、芽孢杆菌为辅助菌种的复合发酵菌,按照5%接种量进行密闭式发酵。
1.2 试验设计选用36日龄的“杜×长×大”三元杂交健康断奶仔猪80头,随机分为4组,分别为A、B、C、D组,每组2个重复,每个重复10头。D组为对照组,饲喂基础饲粮;A、B、C组为试验组,分别在基础饲粮中添加5%、10%、15%的发酵豆粕。饲粮为玉米-豆粕型饲粮,其组成及营养水平见表 1,参照NY/T 65—2004《猪饲养标准》配制。
|
|
表 1 饲粮组成及营养水平(风干基础) Table 1 Composition and nutrient levels of diets (DM basis) |
所有仔猪断奶后进入保育舍并由专人管理。仔猪饲养在保育床上,自由采食和饮水,每日清扫粪便,其他饲养管理按猪场常规程序进行。预试期为3 d,正试期为28 d。
1.4 仔猪粪便样品采集在试验期第28天,每个重复随机采集2头仔猪的新鲜无污染粪便,装入2 mL冻存管,并标记好猪的编号,迅速投入液氮中保存。送回实验室后,将粪便样品存放于-80 ℃冰箱保存以待测序。
1.5 高通量测序分析本试验由上海派森诺生物科技股份有限公司的Illumina MiSeq平台对粪便中的微生物菌群进行16S rRNA高通量测序。
1.6 数据计算和统计方法利用FLASH软件对原始测序数据进行拼接,QIIME软件进行过滤和去除嵌合体后得到每个样本的有效数据。随后调用UCLUST序列比对工具,按照97%的序列相似度进行操作分类单元(OTUs)划分和分类鉴定。用QIIME软件进行样品的多样性分析。Metastats分析使用Mouthur软件对门和属水平的各个OTU在组间的绝对丰度差异进行两两比较检验,得到P值,然后利用Benjamini and Hochberg False Discovery Rate方法对P值进行修正。菌群代谢功能采用PICRUSt软件,将测序数据与代谢功能已知的Greengenes数据库相比较,实现对细菌代谢功能的预测分析。
2 结果与分析 2.1 16S rRNA基因测序结果及菌群多样性 2.1.1 粪便样品16S rRNA基因测序结果采用Illumina MiSeq测序平台对4组粪便样本进行测序,得到的原始数据经过质控初筛、拼接、疑问序列剔除等过滤后得到755 484条高质量有效序列,平均每个样本reads数为47 218,reads的平均长度为397 bp,最小为181 bp,最大为476 bp。
2.1.2 OTUs分析和物种注释按照97%的序列相似度作为OTUs的划分阈值,将4组样品的有效序列数进行聚类,共得到3 581个OTUs。4组OUTs数目分别为2 623、2 465、2 335、2 218个。各组特有的OUTs数目分别为195、208、102、101个。4组共有1 178个OUTs。
选取每个OTUs中丰度最高的序列作为该类OTUs的代表序列,与Greengenes数据库进行比对,将OUTs注释到门、纲、目、科、属和种水平上的柱状图(图 1)。所有样品共注释了12个门、21个纲、33个目、64个科、111个属和137个种。
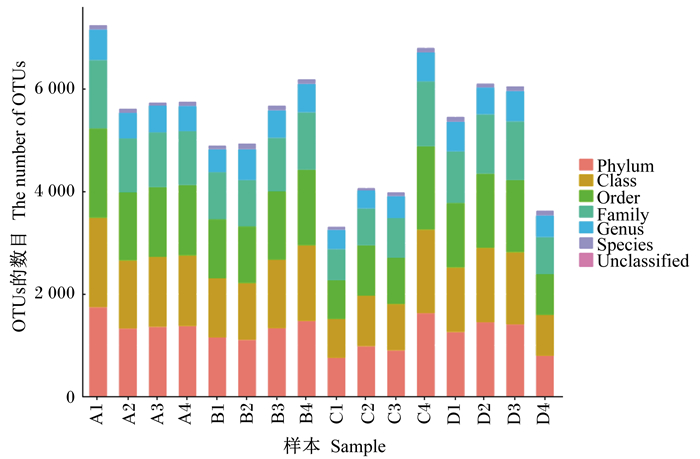
|
Phylunm:门;Class:纲;Order:目;Family:科;Genus:属;Species:种;Unclassified:未分类。 A1~A4、B1~B4、C1~C4、D1~D4分别为A、B、C、D组的样本。图 3、图 4同。A1 to A4, B1 to B4, C1 to C4, D1 to D4 were the samples of groups A, B, C, D, respectively. The same as Fig. 3 and Fig. 4. 图 1 样品在各分类水平上的OUTs数目 Fig. 1 Number of OUTs at each classification level of all samples |
α多样性用于反映样品内微生物群落的丰富度和多样性。目前,可以用稀释曲线、物种累计曲线、丰度等级曲线来评判每个样本的测序深度是否足以反映所包含的微生物多样性。4组样本的稀释曲线(图 2)能够达到较一致的平缓趋势,表明样本的多样性已接近饱和,继续增加测序深度已无法检测到大量尚未发现的新OTUs。因此,该测序结果可以反映粪便中微生物大部分的真实信息。
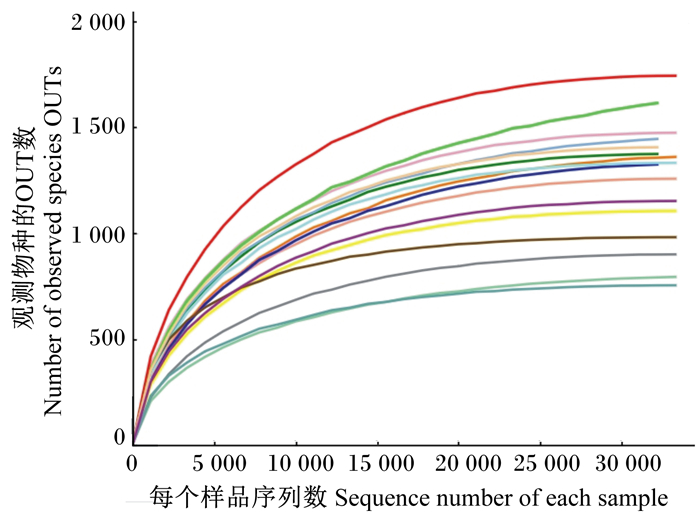
|
图 2 所有样本的稀释曲线 Fig. 2 Dilution curves of all samples |
4组间的菌群多样性指数见表 2。其中,ACE和Chao1指数反映了粪便中微生物群落丰富度。Chao1指数倾向于预测低丰度的物种。ACE指数默认将序列量10以下的OUTs都用来估计群落中的物种数。Simpson和Shannon指数均能反映生物群落的多样性,但是前者侧重于均匀度,后者综合考虑了群落的丰富度和均匀度。B组的Shannon指数高于其他3组,说明该组样本的丰富度和均匀度最好,即多样性最高;但是,其估测的OTUs数量不如A组,说明A组包含的低丰度物种数较多。4组样品的多样性指数间无显著性差异(P>0.05)。
|
|
表 2 菌群多样性指数 Table 2 Microbial diversity indexes |
β多样性分析主要是比较不同样本间微生物的相似性,常用基于UniFrac距离的非度量多维尺度(NMDS)分析描述样本的分布特征,其结果见图 3。对于结果复杂的数据,该分析能使计算结果更稳定。图中2点之间的距离越近,表明2个样本之间的微生物群落结构相似度越高,差异越小。4组样品能够很明显的区分开,A、B、D组内样本间聚集度较高,差异性较小,较稳定。C组样本间分布零散,差异较大。与B组相比,A和D组间的样本距离较近,2组仔猪粪便微生物群落结构相似度较其他组高。
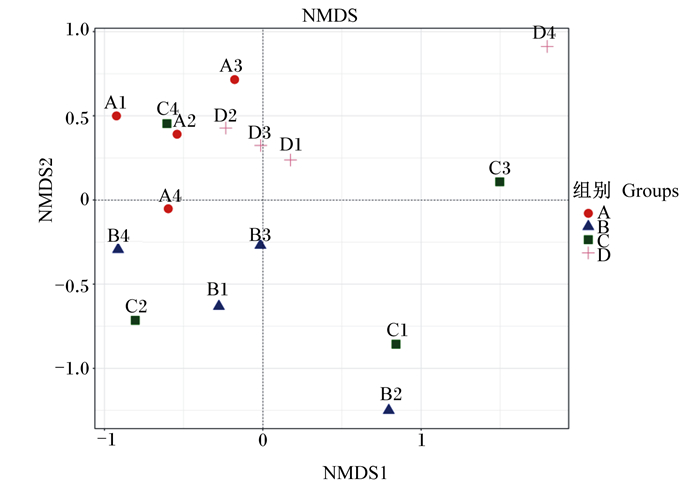
|
图 3 UniFrac非度量多维尺度分析图 Fig. 3 Analysis figure of UniFrac NMDS |
偏最小二乘判别(PLS-DA)分析通过寻找物种丰度矩阵和样本分布或分组信息的最大协方差,从而对样本进行排序来研究组间差异性。由图 4可知,B组与A、D组的样本距离均较远,且各自组内样本的距离较近,说明A、B、D组的分组效果较好。而C组样本与其他3组样本间有重合,尤其是C组包含了B组的4个样本,说明2组肠道中的菌群差异较小。
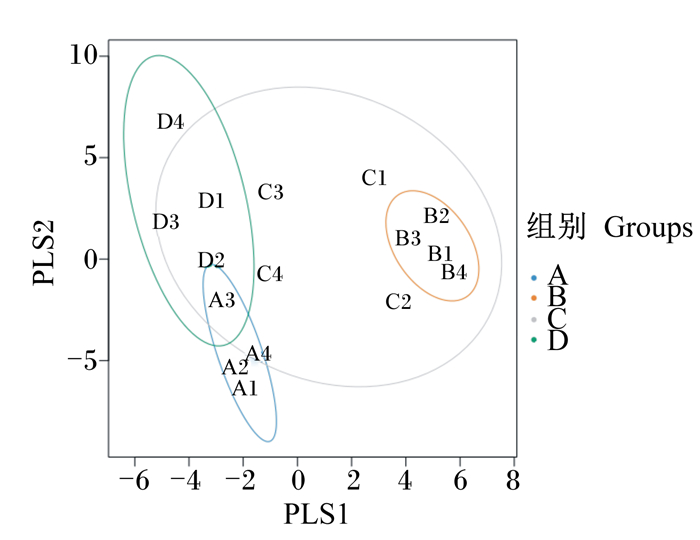
|
图 4 偏最小二乘判别分析图 Fig. 4 Analysis figure of PLS-DA |
4组样本共检测出12个菌门。图 5中不同颜色代表不同菌群,其柱越高在样品中所占的比例越大,相对丰度也越高,科、属水平与门水平与之相同。由图 5可知,仔猪粪便中排名前10的优势菌门分别为厚壁菌门(Firmicutes,81.7%)、变形菌门(Proteobacteria,6.1%)、无壁菌门(Tenericutes,4.6%)、蓝菌门(Cyanobacteria,2.8%)、放线菌门(Actinobacteria,2.4%)、拟杆菌门(Bacteroidetes,1.8%)、螺旋体菌门(Spirochaetes)、TM7菌门、衣原体(Chlamydiae)、脱铁杆菌门(Deferribacteres),上述菌门之和占总菌门的99.0%以上。
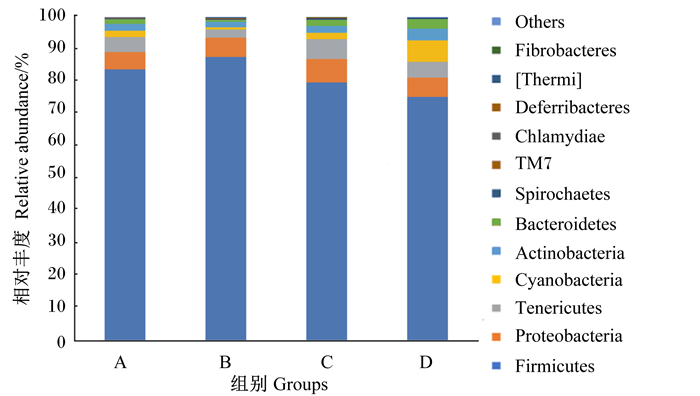
|
Others:其他;Fibrobacteres:纤维杆菌门;[Thermi]:栖热菌门;Deferribacteres:脱铁杆菌门;Chlamydiae:衣原体;TM7:TM7菌门;Spirochaetes:螺旋体门;Bacteroidetes:拟杆菌门;Actinobacteria:放线菌门;Cyanobacteria:蓝菌门;Tenericutes:无壁菌类;Proteobacteria:变形菌门;Firmicutes:厚壁菌门。 图 5 门水平上粪便微生物相对丰度 Fig. 5 Relative abundance of faecal microbiota at phylum level |
组间存在显著差异的菌门为厚壁菌门、蓝菌门、放线菌门、拟杆菌门(表 3)。B组的厚壁菌门相对丰度显著高于D组(P < 0.05),而放线菌门、蓝菌门和拟杆菌门相对丰度极显著低于D组(P < 0.01)。
|
|
表 3 组间差异显著菌门的相对丰度 Table 3 Relative abundance of bacterial phylum with significant difference among groups |
由图 6可知,仔猪粪便中排名前10的优势菌科分别为乳酸菌科(Lactobacillaceae,20.4%)、梭菌科(Clostridiaceae,17.3%)、瘤胃菌科(Ruminococcaceae,12.8%)、韦荣球菌科(Erysipelotrichaceae,9.6%)、毛螺菌科(Lachnospiraceae,7.9%)、假单胞菌科(Pseudomonadaceae,3.4%)、红蝽菌科(Coriobacteriaceae,2.4%)、莫拉式菌科(Moraxellaceae,2.3%)、链球菌科(Streptococcaceae,2.3%)、消化链球菌科(Peptostreptococcaceae,2.2%),以上菌科之和所占总菌科的比例为80.6%。
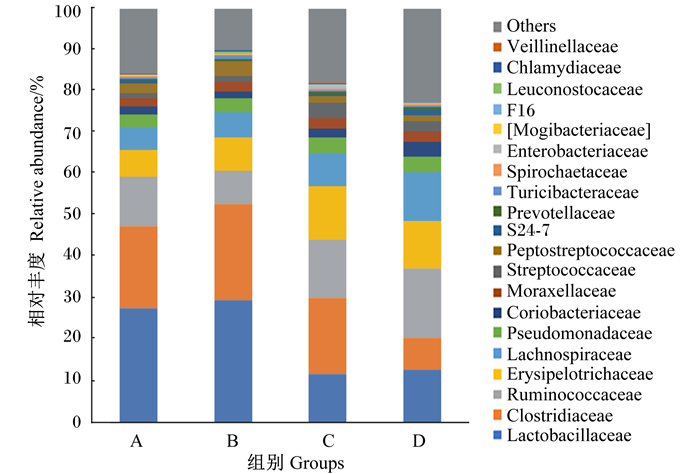
|
Others:其他;Veillonellaceae:韦荣氏菌科;Chlamydiaceae:衣原体科;Leuconostocaceae:明串珠菌科;F16:F16菌科;[Mogibacteriaceae]:艰难杆菌科;Enterobacteriaceae:肠杆菌科;Spirochaetaceae:螺旋体科;Turicibacteraceae:苏黎世杆菌科;Prevotellaceae:普雷沃氏菌科;S24-7:S24-7菌科;Peptostreptococcaceae:消化链球菌科;Streptococcaceae:链球菌科;Moraxellaceae:莫拉氏菌科;Coriobacteriaceae:红蝽菌科;Pseudomonadaceae:假单胞菌科;Lachnospiraceae:毛螺菌科;Erysipelotrichaceae:韦荣球菌科;Ruminococcaceae:瘤胃菌科;Clostridiaceae:梭菌科; Clostridiaceae:梭菌科;Lactobacillaceae:乳杆菌科。 图 6 科水平上粪便微生物相对丰度 Fig. 6 Relative abundance of faecal microbiota at family level |
由图 7可知,粪便中前10位优势菌属分别为乳酸菌属(Lactobacillus,20.4%)、假单胞菌属(Pseudomonas,3.4%)、链型杆菌属(Catenibacterium,3.3%)、布劳特氏菌属(Blautia,2.8%)、链球菌属(Streptococcus,2.2%)、优杆菌属(Eubacterium,2.2%)、Bulleidia(1.9%)、柯林斯菌属(Collinsella,1.4%)、瘤胃球菌属(Ruminococcus,1.1%)、德里奥菌属(Dorea,1.0%),以上菌属之和所占总菌属的比例为39.7%。
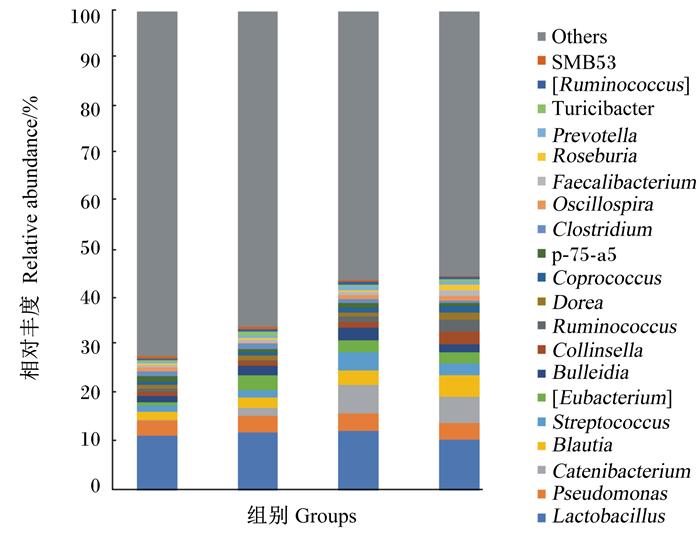
|
Others:其他;SMB53:SMB53菌属;[Ruminococcus]:瘤胃球菌属;Prevotella:普雷沃菌属;Roseburia:螺斯氏菌属;Faecalibacterium:粪杆菌属;Oscillospira:颤螺菌属;Clostridium:梭菌属;p-75-a5:p-75-a5菌属;Coprococcus:粪球菌属;Dorea:德里奥菌属;Ruminococcus:瘤胃球菌属;Collinsella:柯林斯菌属;Bulleidia:布雷德菌属;[Eubacterium]:优杆菌属;Streptococcus:链球菌属;Blautia:布劳特氏菌属;Catenibacterium:链型杆菌属;Pseudomonas:假单胞菌属;Lactobacillus:乳酸菌属。 图 7 属水平上粪便微生物相对丰度 Fig. 7 Relative abundance of faecal microbiota at genus level |
4组间差异显著的菌属共有16种,属于优势菌属的有6种。除柯林斯菌属于放线菌门外,其余5种菌均属于厚壁菌门(表 4)。其中,除瘤胃菌属外,B组的其他5种菌属的相对丰度均高于A组。除优杆菌属外,B组的其他5种菌属的相对丰度均低于C组。除乳酸菌属和优杆菌属外,B组的其他4种菌的相对丰度均低于D组。B组的乳酸菌属、链型杆菌属、优杆菌属的相对丰度显著高于A组(P < 0.05),布劳特氏菌属、柯林斯菌属的相对丰度高于A组,但差异不显著(P>0.05),瘤胃球菌属的相对丰度显著低于A组(P < 0.05),其余菌属间差异不显著(P>0.05)。
|
|
表 4 组间差异显著菌属的相对丰度 Table 4 Relative abundance of bacterial genus with significant difference among groups |
将16S rRNA测序获得的序列进行基因功能预测,然后“映射”到KEGG数据库中,实现对菌群代谢功能的预测。共检测到139个OUTs参与了代谢通路,4组粪便菌群共同参与的代谢通路的基因为4 991个,占到分别各组总基因数的94.5%以上,说明4组菌群所参与的代谢功能基本相似。由图 8可知,菌群主要参与了碳水化合物代谢、氨基酸代谢、能量代谢、核酸代谢、辅酶和维生素代谢,占比分别为10.3%、9.5%、5.6%、4.2%和4.0%。除了核酸代谢外,其余4种代谢通路中菌群的相对丰度依次为B组>C组>A组>D组。核酸代谢的相对丰度依次为A组>B组>C组>D组,但各组的相对丰度差别很小。
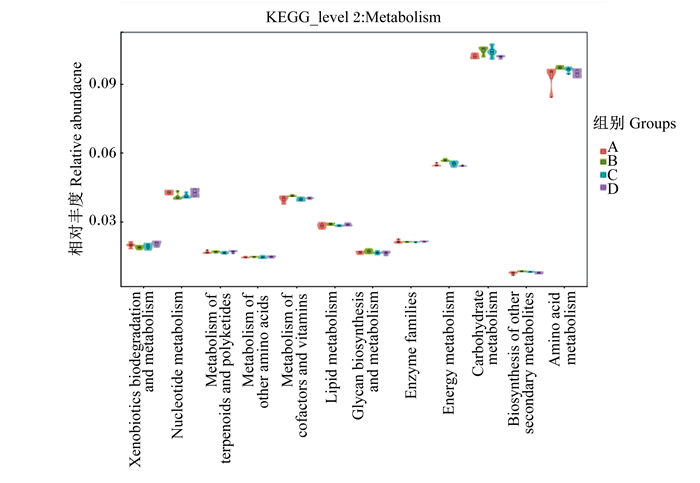
|
Xenobiotics biodegradation and metabolism:外源物的生物降解和代谢;Nucleotide metabolism:核苷酸代谢;Metabolism of terpenoids and polyketides:萜类化合物和聚酮类化合物的代谢;Metabolism of other amino acids:其他氨基酸的代谢;Metabolism of cofactors and vitamins:辅酶和维生素的代谢;Lipid metabolism:脂质代谢;Glycan biosynthesis and metabolism:多糖的生物合成和代谢;Enzyme families:酶家族;Energy metabolism:能量代谢;Carbohydrate metabolism:碳水化合物代谢;Biosynthesis of other secondary metabolites:其他次级代谢物的生物合成;Amino acid metabolism:氨基酸代谢。 图 8 粪便菌群代谢通路分布图 Fig. 8 Distribution figure of metabolic pathways of faecal microbiota |
高通量测序技术具有诸多优点,不仅能准确分析肠道微生物菌群结构和多样性,还能进一步预测肠道微生物的基因功能和代谢途径[19]。Pajarillo等[20]对42日龄的仔猪粪便微生物进行454焦磷酸测序,产生了75 605条reads,鉴定出711个OTUs。杨巧丽[21]采用Illumina HiSeq测序平台对正常仔猪和腹泻仔猪的粪便分别进行测序,平均每个样本reads数为54 748,reads平均长度为253 bp;共获得1 342个OTUs;注释了20个门,34个纲,62个目,81个科,151个属。
4组样品间的α多样性指数差异不显著,说明发酵豆粕含量的变化未对仔猪肠道菌群的多样性产生显著影响。但是,B组多样性最高,说明在断奶仔猪饲粮中添加10%发酵豆粕可以提高仔猪的肠道微生物的多样性,这与张煜[22]的研究结果一致。当发酵豆粕添加量增加到15%时,该组的Simpson指数与B组相同,而Shannon指数低于B组。原因可能是随着发酵豆粕添加量增加,菌群中各成员间的差异性减小,导致群落的丰富度和稀有OUTs的数量的降低。β多样性分析表明,C组内样品聚集度不高且包含了B组的样本,说明当发酵豆粕添加量为10%时,肠道中的菌群已经趋于稳定。
3.2 不同分类水平仔猪粪便菌群组成分析研究表明,不同品种、年龄、环境和饲粮的猪肠道中的微生物组成基本相似,只是相对含量有变化,主要由4个门(厚壁菌门、拟杆菌门、变形菌门、放线菌门)组成[20, 23],且随着仔猪日龄的增大,厚壁菌门和无壁菌门相对含量上升,拟杆菌门相对含量下降[24]。王一冰等[25]研究表明厚壁菌门是仔猪断奶前后的优势菌门,本研究结果与之一致。本研究中, 随着发酵豆粕添加量的增高,以B组为拐点,厚壁菌门相对丰度呈先高后低的趋势,无壁菌门、蓝菌门、放线菌门、拟杆菌门相对丰度呈先低后高趋势。B组厚壁菌的相对丰度最高,可能是因为豆粕经过发酵后产生了大量有机酸,而厚壁菌门细菌在代谢碳水化合物中发挥着重要作用。在科水平上排名前3位的菌科为乳杆菌科、梭菌科、瘤胃菌科,所占50.5%,都属于厚壁菌门。
乳酸菌属是仔猪断奶后的主要优势菌属[26-27],本研究与此相符。提高发酵豆粕含量会增加肠道中乳酸菌属的相对丰度,但B和C组间差异不显著。因此,考虑到乳酸菌属所占比例较大和经济效益,认为发酵豆粕在断奶仔猪饲粮中的添加量为10%比较合适。Yuan等[28]采用平皿培养粪便细菌,结果表明在仔猪饲粮中添加10%发酵豆粕能提高乳酸菌数量,降低大肠杆菌数量。变形菌门在临床研究中对肠道疾病诊断意义重大,该菌门细菌数量庞大、种类多样,多是致病菌,与炎症有关。在科水平上出现的假单胞杆菌、莫拉式菌都属于该菌门。本研究发现随着发酵豆粕添加量的增加,粪便中变形菌门相对丰度略有增加,但组间差异不显著。蓝菌门和放线菌门在1周龄健康与腹泻仔猪粪便菌群中存在显著差异[21],也与仔猪回肠黏膜菌群失调有关[29]。本研究中B组的蓝菌门的相对丰度显著低于其他3组,放线菌门相对丰度显著低于C、D组。而B组的乳酸菌属相对丰度也较高,有可能是因为10%的发酵豆粕提高了肠道中的乳酸菌的数量,该菌可能通过竞争营养和定植位点及产生抑菌物质抑制了致病菌的生长。
3.3 菌群结构和功能的预测对物质代谢和健康的影响研究表明,肠道微生物在宿主的生理功能[30]、营养吸收和能量代谢[31-32]、免疫[33]和机体防御[34]等过程扮演着重要的角色,对于宿主的健康影响巨大。肠道中厚壁菌通过代谢碳水化合物产生短链脂肪酸,与能量获得密切相关[35]。拟杆菌能够促进多糖的降解,利用单糖和低聚糖帮助仔猪进行糖类代谢[20, 36],还是动物胃肠道中主要的食物蛋白质水解菌[37]。乳酸菌能分解肠后端的碳水化合物、蛋白质, 合成维生素,显著抑制病原菌的生长繁殖[38],提高食物的消化率和生物价,促进消化吸收。梭菌属是主要的产丁酸菌,能降解食物中的寡糖和调控肠道微生物区系平衡[39]。瘤胃球菌在成年动物肠道内主要分解纤维素,发酵纤维素和纤维二糖等碳水化合物产生丙酸盐等[40]。
吴先华[41]在保育仔猪饲粮中添加5%、10%、15%的湿态发酵豆粕和10%风干发酵豆粕,与对照组相比,10%湿态和风干发酵豆粕组使仔猪日增重分别显著提高了13.3%和20.7%,粗蛋白质表观消化率分别显著提高了7.8%和5.5%;10%湿态发酵豆粕组腹泻率降低了84.5%;但添加量超过10%时,生长性能及腹泻率几乎不变。章世元[7]等在26日龄的断奶仔猪日龄中添加17.5%和35.0%的发酵豆粕,与对照组相比,日增重分别显著提高了13.90%和50.55%,料重比分别降低了19.47%和4.21%,粗蛋白质消化率分别显著提高了16.97%和20.39%,粗脂肪消化率分别显著提高了22.31%和27.87%。俞晓辉[42]给26~70日龄阶段的断奶仔猪分3个阶段添加10%、8%、6%发酵豆粕,与对照组相比,仔猪日增重显著提高了6.38%,料重比和腹泻率分别显著降低了7.40%和65.37%。这与豆粕经发酵后,减少了抗营养因子的含量,增加了小肽、维生素A、维生素B、多种有机酸等营养物质的含量,增强肠道对干物质、粗蛋白质、总能和磷全肠道表观消化率[43]相关。
本研究中随着发酵豆粕添加量的增加,除核酸代谢外,其他4种代谢通路中菌群的相对丰度均表现为先升后降的趋势,但B、C组间菌群相对丰度差别不大。原因可能是当发酵豆粕添加量从0上升到10%时,菌群的多样性增加导致参加相应代谢通路的菌群数量也随之增加,进而提高了营养物质的消化率。
4 结论对添加发酵豆粕的断奶仔猪粪便进行高通量测序,结果表明饲粮中添加10%发酵豆粕能增加肠道菌群多样性,提高仔猪肠道中厚壁菌门和乳酸菌属的相对丰度,促进糖类、氨基酸、能量、核酸和维生素的代谢,进而促进营养物质的利用率和保持肠道的健康。因此,建议在36日龄后的断奶仔猪饲粮中添加10%的发酵豆粕。
| [1] |
陈斌.微生物发酵对豆粕中抗营养因子及营养价值的影响[D].硕士学位论文.杭州: 浙江大学, 2005. http://d.wanfangdata.com.cn/Thesis_Y745685.aspx
|
| [2] |
SOTAK-PEPER K M, GONZALEZ-VEGA J C, STEIN H H. Concentrations of digestible, metabolizable, and net energy in soybean meal produced in different areas of the United States and fed to pigs[J]. Journal of Animal Science, 2015, 93(12): 5694-5701. DOI:10.2527/jas.2015-9281 |
| [3] |
王格, 董胜奇, 张涛, 等. 基于高通量测序分析豆粕发酵过程中细菌群落结构及多样性[J]. 华中农业大学学报, 2018, 37(5): 89-94. |
| [4] |
SOLOMON S G, OKOMODA V T, OGUCHE O. Nutritional value of raw Canavalia ensiformis and its utilization as partial replacement for soybean meal in the diet of Clarias gariepinus (Burchell, 1822) fingerlings[J]. Food Science & Nutrition, 2018, 6(1): 207-213. |
| [5] |
徐倩.外加酶提高发酵豆粕水解度的研究[D].硕士学位论文.武汉: 华中农业大学, 2009. http://cdmd.cnki.com.cn/Article/CDMD-10504-2010010619.htm
|
| [6] |
NG'ONG'OLA-MANANI T A, ØSTLIE H M, MWANGWELA A M, et al. Metabolite changes during natural and lactic acid bacteria fermentations in pastes of soybeans and soybean-maize blends[J]. Food Science & Nutrition, 2014, 2(6): 768-785. |
| [7] |
章世元, 全丽萍, 徐健超, 等. 发酵豆粕对断奶仔猪生长性能、养分消化率和胃肠道发育的影响[J]. 中国饲料, 2008(16): 8-11. DOI:10.3969/j.issn.1004-3314.2008.16.004 |
| [8] |
徐述亮, 刘晓东, 王丹丹, 等. 不同梯度发酵豆粕对仔猪生长性能和经济效益的影响[J]. 饲料工业, 2013, 34(24): 48-51. |
| [9] |
单达聪, 刘苹苹, 王四新, 等. 发酵豆粕对仔猪生产性能及血液指标的研究[J]. 饲料研究, 2011(6): 1-4. |
| [10] |
王园.豆粕固态发酵条件及其对断奶仔猪饲用效果的研究[D].博士学位论文.北京: 中国农业大学, 2014: 5. http://cdmd.cnki.com.cn/Article/CDMD-10019-1014225949.htm
|
| [11] |
苏恺, 蔡仁贤, 熊罗英, 等. 发酵豆粕的营养价值及其在断奶仔猪日粮中的应用[J]. 西北农业学报, 2014, 23(4): 117-123. |
| [12] |
VAN WINSEN R L, KEUZENKAMP D, URLINGS B A P, et al. Effect of fermented feed on shedding of Enterobacteriaceae by fattening pigs[J]. Veterinary Microbiology, 2002, 87(3): 267-276. DOI:10.1016/S0378-1135(02)00066-4 |
| [13] |
胡远亮.利用分子生物技术研究益生菌对断奶仔猪生长及粪便菌群的影响[D].博士学位论文.武汉: 华中农业大学, 2014: 25-87. http://cdmd.cnki.com.cn/Article/CDMD-10504-1014213592.htm
|
| [14] |
孙笑非, 孙冬岩, 王文娟, 等. 猪肠道微生物定植规律和生理功能[J]. 饲料研究, 2011(9): 70-72. |
| [15] |
POWER S E, O'TOOLE P W, STANTON C, et al. Intestinal microbiota, diet and health[J]. British Journal of Nutrition, 2014, 111(3): 387-402. DOI:10.1017/S0007114513002560 |
| [16] |
刘虹, 王琪, 刘作华, 等. 猪肠道微生物区系的形成及营养调控[J]. 动物营养学报, 2018, 30(7): 2480-2487. DOI:10.3969/j.issn.1006-267x.2018.07.007 |
| [17] |
LIU H Y, IVARSSON E, DICKSVED J, et al. Inclusion of chicory (Cichorium intybus L.) in pigs' diets affects the intestinal microenvironment and the gut microbiota[J]. Applied and Environmental Microbiology, 2012, 78(12): 4102-4109. DOI:10.1128/AEM.07702-11 |
| [18] |
李东萍, 郭明璋, 许文涛. 16S rRNA测序技术在肠道微生物中的应用研究进展[J]. 生物技术通报, 2015, 31(2): 71-77. |
| [19] |
叶雷, 闫亚丽, 陈庆森, 等. 高通量测序技术在肠道微生物宏基因组学研究中的应用[J]. 中国食品学报, 2016, 16(7): 216-223. |
| [20] |
PAJARILLO E A B, CHAE J P, BALOLONG M P, et al. Assessment of fecal bacterial diversity among healthy piglets during the weaning transition[J]. The Journal of General and Applied Microbiology, 2014, 60(4): 140-146. DOI:10.2323/jgam.60.140 |
| [21] |
杨巧丽.腹泻仔猪粪便微生物结构和功能及SLA-DQA基因与粪便菌群的相关性[D].博士学位论文.兰州: 甘肃农业大学, 2017: 6.
|
| [22] |
张煜.新型枯草芽孢杆菌的研发及其发酵饲料在仔猪上的应用研究[D].博士学位论文.杭州: 浙江大学, 2017: 12.
|
| [23] |
PANDA S, EL KHADER I, CASELLAS F, et al. Short-term effect of antibiotics on human gut microbiota[J]. PLoS One, 2014, 9(4): e95476. DOI:10.1371/journal.pone.0095476 |
| [24] |
KIM H B, BOREWICZ K, WHITE B A, et al. Longitudinal investigation of the age-related bacterial diversity in the feces of commercial pigs[J]. Veterinary Microbiology, 2011, 153(1/2): 124-133. |
| [25] |
王一冰, 张小平, 黄怡, 等. 运用454焦磷酸测序技术对断奶前后仔猪肠道菌群的分析[J]. 动物营养学报, 2013, 25(10): 2440-2446. DOI:10.3969/j.issn.1006-267x.2013.10.028 |
| [26] |
SLIFIERZ M J, FRIENDSHIP R M, WEESE J S. Longitudinal study of the early-life fecal and nasal microbiotas of the domestic pig[J]. BMC Microbiology, 2015, 15(1): 184. |
| [27] |
EVANS N J, BROWN J M, MURRAY R D, et al. Characterization of novel bovine gastrointestinal tract Treponema isolates and comparison with bovine digital dermatitis treponemes[J]. Applied and Environmental Microbiology, 2011, 77(1): 138-147. DOI:10.1128/AEM.00993-10 |
| [28] |
YUAN L, CHANG J, YIN Q Q, 等.发酵豆粕改善仔猪生长性能、营养物质消化率和肠道微生物菌群[J].王志林译.广东饲料, 2017, 26(1): 51.
|
| [29] |
AZCARATE-PERIL M A, FOSTER D M, CADENAS M B, et al. Acute necrotizing enterocolitis of preterm piglets is characterized by dysbiosis of ileal mucosa-associated bacteria[J]. Gut Microbes, 2011, 2(4): 234-243. DOI:10.4161/gmic.2.4.16332 |
| [30] |
O'HARA A M, SHANAHAN F. The gut flora as a forgotten organ[J]. EMBO Reports, 2006, 7(7): 688-693. DOI:10.1038/sj.embor.7400731 |
| [31] |
SONNENBURG J L, XU J, LEIP D D, et al. Glycan foraging in vivo by an intestine-adapted bacterial symbiont[J]. Science, 2005, 307(5717): 1955-1959. DOI:10.1126/science.1109051 |
| [32] |
YATSUNENKO T, REY F E, MANARY M J, et al. Human gut microbiome viewed across age and geography[J]. Nature, 2012, 486(7402): 222-227. DOI:10.1038/nature11053 |
| [33] |
CHOW J, LEE S M, SHEN Y, et al. Host-bacterial symbiosis in health and disease[J]. Advances in Immunology, 2010, 107: 243-274. DOI:10.1016/B978-0-12-381300-8.00008-3 |
| [34] |
O'TOOLE P W, CLAESSON M J. Gut microbiota:changes throughout the lifespan from infancy to elderly[J]. International Dairy Journal, 2010, 20(4): 281-291. DOI:10.1016/j.idairyj.2009.11.010 |
| [35] |
WALKER A W, INCE J, DUNCAN S H, et al. Dominant and diet-responsive groups of bacteria within the human colonic microbiota[J]. The ISME Journal, 2011, 5(2): 220-230. DOI:10.1038/ismej.2010.118 |
| [36] |
EL KAOUTARI A, ARMOUGOM F, GORDON J I, et al. The abundance and variety of carbohydrate-active enzymes in the human gut microbiota[J]. Nature Reviews Microbiology, 2013, 11(7): 497-504. DOI:10.1038/nrmicro3050 |
| [37] |
MACFARLANE G T, CUMMINGS J H, ALLISON C. Protein degradation by human intestinal bacteria[J]. Microbiology, 1986, 132(6): 1647-1656. DOI:10.1099/00221287-132-6-1647 |
| [38] |
黄沧海.仔猪复合益生乳酸杆菌制剂及其作用机理的研究[D].博士学位论文.北京: 中国农业大学, 2003. http://cdmd.cnki.com.cn/Article/CDMD-10019-2006023039.htm
|
| [39] |
LOUIS P, FLINT H J. Diversity, metabolism and microbial ecology of butyrate-producing bacteria from the human large intestine[J]. FEMS Microbiology Letters, 2010, 294(1): 1-8. |
| [40] |
FLINT H J, BAYER E A, RINCON M T, et al. Polysaccharide utilization by gut bacteria:potential for new insights from genomic analysis[J]. Nature Reviews Microbiology, 2008, 6(2): 121-131. DOI:10.1038/nrmicro1817 |
| [41] |
吴先华.发酵豆粕在断奶仔猪及生长育肥猪日粮中的应用研究[D].硕士学位论文.南宁: 广西大学, 2014: 6. http://cdmd.cnki.com.cn/Article/CDMD-10593-1014378992.htm
|
| [42] |
俞晓辉.发酵豆粕对断奶仔猪生长性能和肠道微生物的影响[D].硕士学位论文.南京: 南京农业大学, 2008: 6. http://cdmd.cnki.com.cn/Article/CDMD-10307-2009077101.htm
|
| [43] |
KIM Y G, LOHAKARE J D, YUN J H, et al. Effect of feeding levels of microbial fermented soy protein on the growth performance, nutrient digestibility and intestinal morphology in weaned piglets[J]. Asian-Australasian Journal of Animal Sciences, 2007, 20(3): 399-404. DOI:10.5713/ajas.2007.399 |




