现阶段我国人畜争粮问题严重,为了保证畜牧业的可持续发展,开发利用秸秆饲料是一条有效途径。苜蓿草粉是用于伊拉兔饲养的最好粗饲料,但价格较高,花生作为我国北方地区的主要农作物,其副产物花生秧营养丰富,含蛋白质、脂肪、各种矿物质及维生素,且适口性好,其产量相当可观。选择花生秧作为兔用粗饲料可以减少苜蓿草粉用量,在保持兔生长性能基本不变的同时可以降低饲料造价。陆小虹等[1]用40%的花生秧干粉代替等量苜蓿草粉饲喂伊拉兔,结果发现用花生秧粉取代苜蓿草粉对伊拉兔的生长性能没有显著影响,并可降低伊拉兔的单位饲养成本。马佳等[2]在基础饲粮中添加20%花生秧饲喂肉兔,结果表明肉兔对花生秧中粗蛋白质的消化率较高,而对粗纤维的消化率偏低。田小蜜等[3]研究认为,与不添加花生秧粉的饲粮相比,饲粮中添加30%花生秧粉可显著提高繁殖獭兔的产仔数、窝均活仔数以及仔兔断奶体重。李海利[4]研究发现,75日龄健康白色獭兔使用15%的花生秧替代基础饲粮的饲喂效果最好,营养物质的表观消化率相对较高,高或低水平的花生秧添加饲粮都会影响獭兔的消化率。
在养兔业中,肠道疾病是造成兔死亡的主要难题,肠道环境的健康十分重要。肠道菌群的结构正常及稳定,是保持肠道健康的重要因素,菌群紊乱会导致伊拉兔生产能力低下,甚至爆发疾病[5]。健康动物肠道菌群的稳定是保护动物免受病原微生物及毒素侵害的重要保障[6]。伊拉兔的肠道中95%以上都是厌氧菌,需氧菌和兼性厌氧菌只占肠道菌群的5%以下。
本试验旨在研究饲粮中添加不同比例的花生秧对伊拉兔肠道菌群的影响,为花生秧在兔饲粮中添加及推广提供理论依据。
1 材料与方法 1.1 试验材料及试验动物试验所用花生秧来自钟祥市祥楚农业科技有限公司。试验动物为(35±2)日龄、健康状况良好、体重[(1.02±0.15) kg]相近的120只生长期伊拉兔(公母各占1/2),由沈阳富宁兔业有限公司提供。
1.2 试验饲粮参照李福昌[7]推荐的肉兔不同生理阶段饲养标准,设计花生秧添加量分别为0、10.0%、12.5%、15.0%、17.5%和20.0%的6种试验饲粮,其组成及营养水平见表 1。试验饲粮以颗粒料形式饲喂,颗粒料的制作在沈阳立得鑫农牧有限公司进行。
|
|
表 1 试验饲粮组成及营养水平(风干基础) Table 1 Composition and nutrient levels of experimental diets (air-dry basis) |
本试验于2018年5月至2018年7月在沈阳富宁兔业有限公司进行。
1.4 试验设计和饲养管理采用单因素随机化试验设计,将120只生长期伊拉兔随机分为6组,每组4个重复,每个重复5只伊拉兔。6组试验兔分别用花生秧添加量为0(Ⅰ组,作为对照组)、10.0%(Ⅱ组)、12.5%(Ⅲ组)、15.0%(Ⅳ组)、17.5%(Ⅴ组)和20.0%(Ⅵ组)的颗粒料进行饲喂。各组试验兔的饲养管理条件保持一致,免疫程序按兔场常规程序进行,预试期7 d,正试期42 d。试验兔按照单笼饲养的方式分别饲养于兔笼内,每天固定时间喂料2次,给予充足的饮水,自由采食,当料槽内剩料较多时,适当减少饲喂量。
1.5 盲肠微生物的收集及编号试验结束时,从各组的每个重复随机选取1只伊拉兔进行屠宰,剖开腹腔,分离盲肠,用细线结扎,剪下后将盲肠内容物置于离心管中,迅速至于-80 ℃冰箱备用。对所取样本进行编号,Ⅰ~Ⅵ组所取样本依次编号为a~f,每个组内所取样本依次编号为1~4。
1.6 肠道微生物基因组提取及测序肠道微生物基因组提取采用E.Z.N.ATM Mag-Bind Soil DNA Kit(Omega Bio-Tek公司),具体操作按说明书进行,提取完成后,将提取产物送往生工生物工程(上海)股份有限公司进行16S rDNA V3~V4区测序,分析饲喂不同花生秧添加量饲粮后伊拉兔肠道菌群的结构特征。
1.7 肠道微生物信息学分析Illumina MiseqTM测序平台得到的原始图像数据文件经CASAVA碱基识别(base calling)分析转化为原始测序序列(sequenced reads)。去除引物接头序列,再根据PE reads之间的overlap关系,将成对的reads拼接成一条序列,然后按照barcode标签序列识别并区分样本得到各样本数据,最后对各样本数据的质量进行质控过滤,得到各样本有效数据。
将所有样本序列按照序列间的距离进行聚类,然后根据序列之间的相似性将序列分成不同的操作分类单元(operational taxonomic units,OTU)。通常在97%的相似水平下对OTU进行生物信息统计分析。在OTU聚类结果的基础上,获取OTU聚类中的代表性序列,选择丰度最高的序列作为OTU的代表性序列,进行各类的OTU分析。
Alpha多样性指数包括Chao1指数、ACE指数、Shannon指数和Simpson指数等。参照祁晨等[8]的方法计算相关指数,Chao1指数和ACE指数可以估计群落中的OUT数目来反映物种丰富度,Shannon指数和Simpson指数可以估算样品中微生物的多样性。
1.8 数据处理与分析采用SPSS 21.0对数据进行处理和分析,P < 0.05为差异显著,结果以平均值±标准差表示。
2 结果与分析 2.1 DNA序列数据通过Illumina MiseqTM测序平台上的高通量测序,原始序列数和处理后的有效序列数如表 2所示。在高质量序列中,大多数长于400 bp,在400~440 bp之间(图 1)。Shannon指数稀释曲线(图 2)和物种累积曲线(图 3)表明,各组样本测序数据量合理,样品抽样充分,可以进行数据分析。
|
|
表 2 各样本数据信息统计 Table 2 Statistics of sample data |
|
图 1 质控后序列长度分布图 Fig. 1 Distribution map of quality control sequence length |
|
图 2 Shannon指数稀释曲线 Fig. 2 Rarefaction curve of Shannon index |
|
图 3 物种累积曲线 Fig. 3 Species cumulative curve |
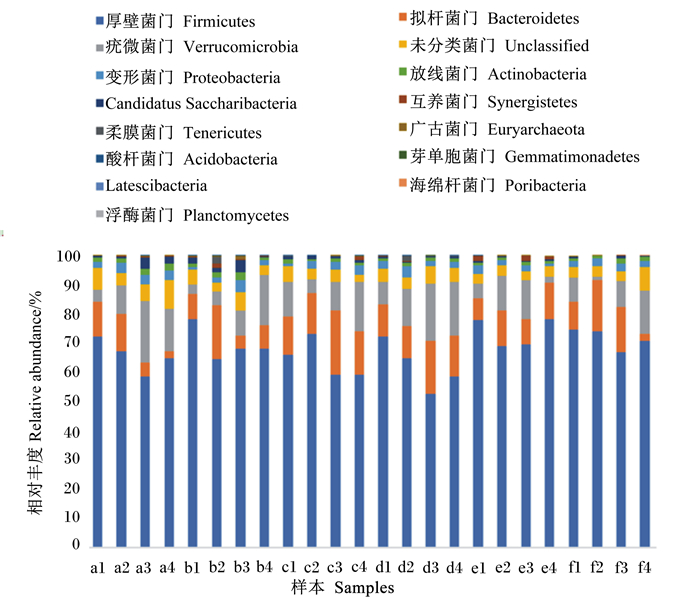
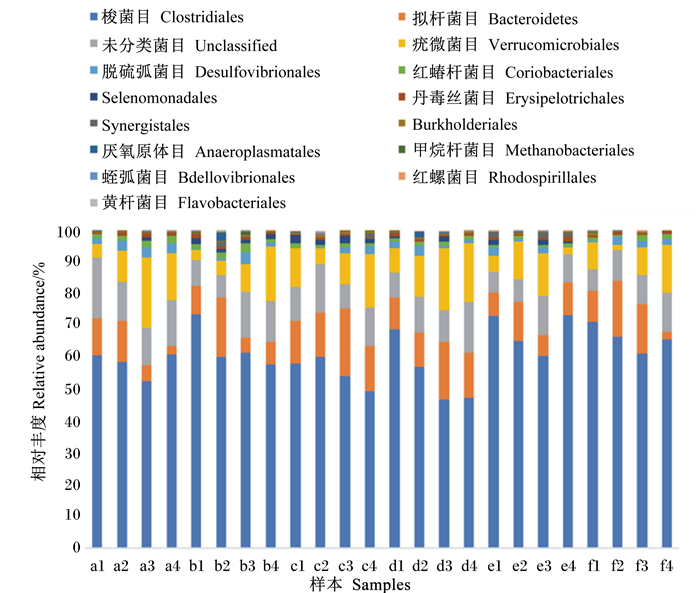
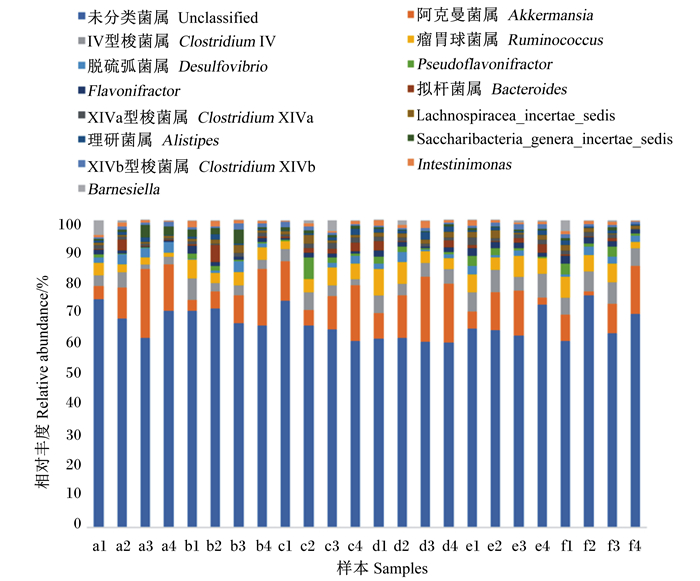
根据OTU分类和结果分类状态识别,利用R软件获得每个样本在门、纲、目、科和属水平上的细菌具体组成。每个样本的分类群丰度被鉴定为15个门、29个纲、35个目、50个科、50个属。细菌在门、目、属水平上的组成分别显示在图 4、图 5和图 6。在门水平上,每组中常见的3个门为厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)和疣微菌门(Verrucomicrobia),相对丰度分别为67.87%、11.50%和10.44%(图 4)。在目水平上,每组中常见的3个目为梭菌目(Clostridiales)、拟杆菌目(Bacteroidales)和疣微菌目(Verrucomicrobiales),相对丰度分别为60.99%、11.21%和10.56% (图 5)。在属水平上,阿克曼菌属(Akkermansia)、Ⅳ型梭菌属(Clostridium Ⅳ)、瘤胃球菌(Ruminococcus)属见于每组(图 6)。
|
颜色对应此分类学水平下各物种名称,不同色块宽度表示不同物种相对丰度。图 5、图 6同。 The color corresponds to the name of each species under this taxonomic level, and the different color block widths indicate the relative abundances of different species. The same as Fig. 5 and Fig. 6. 图 4 门水平上所有样本群落结构分布 Fig. 4 Community structure distribution of all samples at phylum level |
|
图 5 目水平上所有样本群落结构分布 Fig. 5 Community structure distribution of all samples at order level |
|
图 6 属水平上所有样本群落结构分布 Fig. 6 Community structure distribution of all samples at genus level |
如表 3所示,Shannon指数各组之间差异不显著(P > 0.05),说明饲粮中不同添加量的花生秧未显著改变肠道菌群多样性;ACE指数和Chao1指数均在花生秧添加量为17.5%时最高,表明此时肠道微生物总量最高;随着花生秧添加量的增加,Simpson指数呈先降后升的趋势。
|
|
表 3 Alpha多样性指数 Table 3 Alpha diversity indexes |
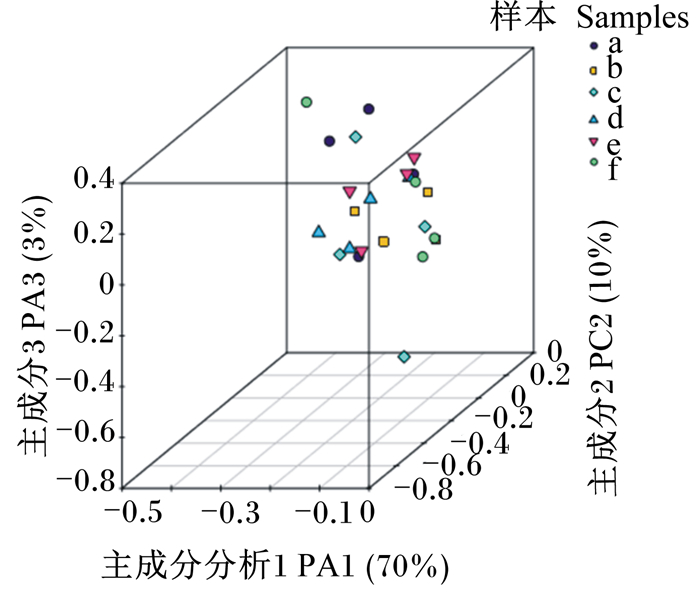
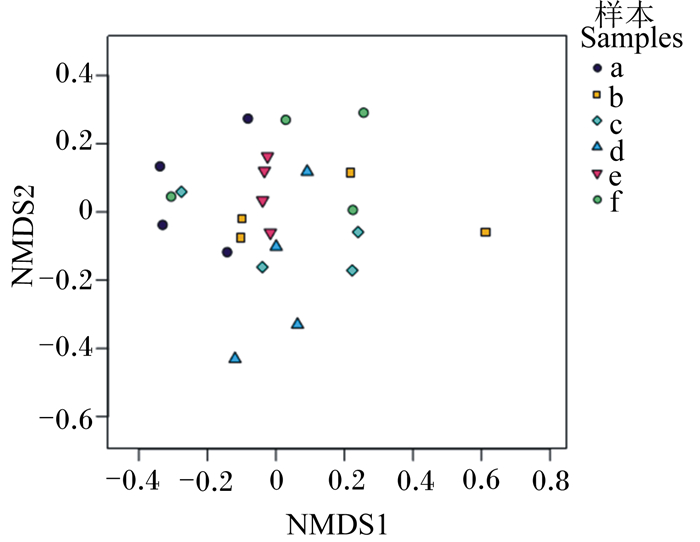
主成分分析(PCA)(图 7)和非度量多维尺度分析(NMDS)(图 8)表明,伊拉兔食用不同花生秧添加量的饲粮后,盲肠菌群多样性的相似性不同。各试验组内的几个重复都没有明显的聚类,说明样本间相似性比较高。Ⅴ组的组内间距小于其他组的组内间距,表明Ⅴ组伊拉兔盲肠菌群结构的稳定性高于其他组。
|
图中不同颜色代表不同组中的样本,样本间相似性越高则在图中越聚集。图 8同。 The different colors in the figure represent the samples in the different groups. The higher the similarity between samples, the more aggregated in the graph. The same as Fig. 8. 图 7 基于OTU的PCA图 Fig. 7 PCA diagram based on OTU |
|
图 8 基于OTU的NMDS图 Fig. 8 NMDS diagram based on OTU |
如表 4所示,在门水平上,与对照组相比,饲粮中添加不同水平花生秧后伊拉兔盲肠内容物中拟杆菌门、疣微菌门、厚壁菌门和变形菌门(Proteobacteria)的比例没有发生显著变化(P > 0.05),但拟杆菌门的比例均有所升高,变形菌门的比例均有所下降,Ⅳ、Ⅴ、Ⅵ组Candidatus Saccharibacteria的比例显著减少(P < 0.05),Ⅴ、Ⅵ组放线菌门(Actinobacteria)的比例显著减少(P < 0.05)。如表 5所示,在属的水平上,与对照组相比,Ⅴ、Ⅵ组Ⅳ型梭菌属的比例显著增加(P < 0.05)。
|
|
表 4 饲粮中添加花生秧对伊拉兔肠道菌群门水平细菌所占比例的影响 Table 4 Effects of peanut vine supplementation on proportions of intestinal phylum-level bacteria in Ira rabbits |
|
|
表 5 饲粮中添加花生秧对伊拉兔肠道菌群属水平细菌所占比例的影响 Table 5 Effects of peanut vine supplementation on proportions of intestinal genus-level bacteria in Ira rabbits |
众所周知,遗传、环境和饲料营养都会影响动物的生长和健康,同样的,这些因素也会影响着肠道菌群[9]。近10年来,分子生物学技术被广泛应用于动物肠道菌群的研究,宏基因组学技术和高通量测序技术成为研究肠道菌群结构的主要手段。
研究表明,一些霉菌毒素如黄曲霉霉菌毒素B1(AFB1)可以调节猪或大鼠的肠道细菌群落组成[10-13]。但是,目前没有不同花生秧添加量的颗粒饲料对断奶伊拉兔肠道菌群影响的报道。因此,本试验结果可以增加我们对花生秧对伊拉兔肠道菌群影响的认识。
本试验中,选择断奶的伊拉兔作为试验动物,是因为这个生理阶段的伊拉兔肠道菌群的组成趋于稳定[14-15],并且盲肠菌群从出生后的简单和不稳定的生物群逐渐发展成复杂和稳定的生物群[16]。因此,选择断奶后的伊拉兔进行动物试验,可以更准确、真实地反映饲粮中添加不同水平的花生秧对伊拉兔肠道菌群的影响。
本试验结果表明,在门水平上,伊拉兔盲肠菌群中相对丰度最高的是厚壁菌门,其次是拟杆菌门和疣微菌门,其结果与前人对兔盲肠菌群结构研究的结果一致[17-19]。与对照组相比,饲粮中添加花生秧后伊拉兔盲肠内容物中拟杆菌门、疣微菌门和厚壁菌门的比例没有显著变化,但是不同程度地降低了变形菌门的比例。虽然变形菌门在兔后肠道微生物群落中所占比例很小[20],但它包括许多致病菌,如大肠杆菌、沙门氏菌和霍乱弧菌;变形菌门比例的变化与严重的肠道炎症有关,如肠道疾病和坏死性小肠结肠炎[21]。据此我们可以推测,饲粮中添加花生秧可能会降低变形菌门中一些致病菌的数量,从而减少肠道炎症的发生,该推测需要进一步的试验来验证。随着饲粮中花生秧添加量的增加,Ⅳ、Ⅴ、Ⅵ组伊拉兔盲肠内容物中Candidatus Saccharibacteria的比例显著减少。与对照组相比,Ⅴ、Ⅵ组伊拉兔盲肠内容物中放线菌门的比例显著减少。放线菌一般存在于食草动物的消化道中,有促进消化的作用,本试验中,饲粮添加花生秧后,导致伊拉兔盲肠内容物中放线菌门细菌的数量减少,推测会影响伊拉兔的消化能力,但是需要进一步的研究来证明这一点。在属水平上,本试验结果表明,与对照组相比,Ⅴ、Ⅵ组伊拉兔内容物中Ⅳ型梭菌属的比例显著增加。Ⅳ型梭菌属细菌对于营养的吸收、肠道上皮细胞的成熟和维护具有一定的功效[22]。根据试验结果推测,饲粮中添加花生秧后,可以增加Ⅳ型梭菌属细菌的数量,提升肠道上皮细胞吸收营养的能力,但是需要进一步的试验来验证。
4 结论饲粮中添加花生秧后可改变一些具有重要代谢功能的细菌的丰度,影响盲肠微生物群落的平衡,表明饲粮中添加花生秧后会对伊拉兔肠道菌群产生影响。
| [1] |
陆小虹, 冯清泉. 花生秧粉代替苜蓿草粉对兔饲养效果的初步观察[J]. 上海实验动物科学, 1995, 15(4): 233-234. |
| [2] |
马佳, 郭东新, 田河, 等. 花生秧在肉兔中的表观消化能和主要养分消化率的评定[J]. 饲料工业, 2010, 31(21): 62-64. DOI:10.3969/j.issn.1001-991X.2010.21.017 |
| [3] |
田小蜜, 苏加义. 用花生藤粉饲喂繁殖獭兔的应用研究[J]. 安徽农学通报, 2009, 15(9): 189, 211. |
| [4] |
李海利.不同替代比例的花生秧、玉米和豆粕对生长獭兔营养物质表观消化率的影响[D].硕士学位论文.保定: 河北农业大学, 2015: 30-35. http://cdmd.cnki.com.cn/Article/CDMD-10086-1015392672.htm
|
| [5] |
史启杰, 霍自双, 任传帅, 等. 寒地獭兔肠道菌群定植的研究[J]. 黑龙江畜牧兽医, 2017(1): 238-243, 299. |
| [6] |
任传帅, 史启杰, 张震, 等. 软粪液和小肠液对寒地獭兔肠道菌群定植的影响[J]. 中国微生态学杂志, 2016, 28(1): 29-33. |
| [7] |
李福昌. 家兔营养需要研究进展[J]. 中国养兔, 2011(04): 41-44. DOI:10.3969/j.issn.1005-6327.2011.04.012 |
| [8] |
祁晨, 郭婧, 李蓓蓓, 等. 阴极菌群对MFC脱氮产电及动力学影响[J]. 工业水处理, 2018, 38(11): 33-36. |
| [9] |
GILBERT J A, QUINN R A, DEBELIUS J, et al. Microbiome-wide association studies link dynamic microbial consortia to disease[J]. Nature, 2016, 535(7610): 94-103. DOI:10.1038/nature18850 |
| [10] |
WANG J C, TANG L L, GLENN T C, et al. Aflatoxin B1 induced compositional changes in gut microbial communities of male F344 rats[J]. Toxicological Sciences, 2016, 150(1): 54-63. DOI:10.1093/toxsci/kfv259 |
| [11] |
WACHÉ Y J, VALAT C, POSTOLLEC G, et al. Impact of deoxynivalenol on the intestinal microflora of pigs[J]. International Journal of Molecular Sciences, 2009, 10(1): 1-17. |
| [12] |
LI F C, WANG J Q, HUANG L B, et al. Effects of adding Clostridium sp.WJ06 on intestinal morphology and microbial diversity of growing pigs fed with natural deoxynivalenol contaminated wheat[J]. Toxins, 2017, 9(12): 383. DOI:10.3390/toxins9120383 |
| [13] |
GUO M Z, HUANG K L, CHEN S Y, et al. Combination of metagenomics and culture-based methods to study the interaction between ochratoxin A and gut microbiota[J]. Toxicological Sciences, 2014, 141(1): 314-323. DOI:10.1093/toxsci/kfu128 |
| [14] |
MICHELLAND R J, COMBES S, MONTEILS V, et al. Molecular analysis of the bacterial community in digestive tract of rabbit[J]. Anaerobe, 2010, 16(2): 61-65. DOI:10.1016/j.anaerobe.2009.05.002 |
| [15] |
YANG C L, MI L, HU X L, et al. Investigation into host selection of the cecal acetogen population in rabbits after weaning[J]. PLoS One, 2016, 11(7): e0158768. DOI:10.1371/journal.pone.0158768 |
| [16] |
COMBES S, MICHELLAND R J, MONTEILS V, et al. Postnatal development of the rabbit caecal microbiota composition and activity[J]. FEMS Microbiology Ecology, 2011, 77(3): 680-689. DOI:10.1111/j.1574-6941.2011.01148.x |
| [17] |
ZHU Y L, WANG C Y, LI F C. Impact of dietary fiber/starch ratio in shaping caecal microbiota in rabbits[J]. Canadian Journal of Microbiology, 2015, 61(10): 771-784. DOI:10.1139/cjm-2015-0201 |
| [18] |
MONTEILS V, CAUQUIL L, COMBES S, et al. Potential core species and satellite species in the bacterial community within the rabbit caecum[J]. FEMS Microbiology Ecology, 2008, 66(3): 620-629. DOI:10.1111/fem.2008.66.issue-3 |
| [19] |
ZOU F Q, ZENG D, WEN B, et al. Illumina Miseq platform analysis caecum bacterial communities of rex rabbits fed with different antibiotics[J]. AMB Express, 2016, 6(1): 100. DOI:10.1186/s13568-016-0273-1 |
| [20] |
FU X C, ZENG B, WANG P, et al. Microbiome of total versus live bacteria in the gut of Rex rabbits[J]. Frontiers in Microbiology, 2018, 9: 733. DOI:10.3389/fmicb.2018.00733 |
| [21] |
SHIN N R, WHON T W, BAE J W. Proteobacteria:microbial signature of dysbiosis in gut microbiota[J]. Trends in Biotechnology, 2015, 33(9): 496-503. DOI:10.1016/j.tibtech.2015.06.011 |
| [22] |
LAPARRA J M, SANZ Y. Interactions of gut microbiota with functional food components and nutraceuticals[J]. Pharmacological Research, 2010, 61(3): 219-225. DOI:10.1016/j.phrs.2009.11.001 |




