2. 四川省畜牧科学研究院, 动物遗传育种四川省重点实验室, 成都 610066
2. Sichuan Academy of Agricultural Sciences, Sichuan Key Laboratory for Animal Genetic and Breeding, Chengdu 610066, China
由于温室气体排放造成全球性极端气候变化和生态环境危机是人类迄今面临的最复杂的挑战之一。自1750年起的工业化时代以来,快速增长的人口数量、大规模的密集型农业活动、加速的工业化进程及化石燃料源的能源消耗促使了大气中温室气体含量的持续增加[1]。甲烷(CH4)是一种比二氧化碳(CO2)危害更大的温室气体,在大气中存留时间超过10年,增温潜势是CO2的28倍[2]。
反刍动物养殖业是现代畜牧业生产中一个重要的组成部分,其绿色高效和可持续发展对于改善我国城乡居民的膳食结构、促进农村产业结构调整和带动国民经济产业发展具有重要意义[3]。我国是牛肉和乳制品生产和消费大国,2016年底中国奶牛和肉牛存栏量分别为1 507万和7 373万头,牛肉产量仅次于美国和巴西;2017年奶类产量达到3 655万t,居世界第3位[4]。作为重要的反刍动物畜种,牦牛和水牛在中国分布广,数量大。据统计,牦牛(2009年)和水牛(2004年)数量分别为1 400万和2 280万头[5-6]。持续增长的反刍动物产品消费量与由此而来的大规模养殖生产是重要的CH4排放源,其总量可达到人类活动CH4总排放的40%左右[7]。截止到2010年,中国奶牛胃肠道CH4排放量所占反刍动物CH4排放总量的比例由1990年的3.3%上升到了13.1%,且呈逐年上升的趋势[8]。更重要的是,以甲烷能的形式损失掉的能量可占到饲粮总能的2%~12%,制约了我国反刍动物生产的养殖效率和盈利能力[9-10]。饲粮有机物经瘤胃微生物降解作用生成挥发性脂肪酸,同时伴随有氢气(H2)和CO2的产生。H2作为反应底物经过一系列复杂的生物化学反应被瘤胃产甲烷菌利用生成CH4[11]。反刍动物品种和生理阶段等诸多因素的差异能够影响瘤胃内产甲烷菌的定植、组成和多样性特征,继而造成瘤胃CH4排放量的不同[12]。因此,深入研究和理解反刍动物瘤胃产甲烷菌菌群的组成及其多样性有助于明确CH4生成机理、为寻找合适的瘤胃CH4调控措施提供理论依据。为此,本文重点综述了近年来主要类型和品种反刍动物瘤胃内产甲烷菌的菌群结构和多样性特征,旨在为后期进一步研究产甲烷菌的基因功能和代谢途径、探索反刍动物瘤胃CH4减排机制和提高养殖效率提供思路和参考。
1 产甲烷菌分类产甲烷菌是一类区别于细菌且严格厌氧的原核生物,也是一类重要的代谢产甲烷的古菌。甲酸甲烷杆菌(Methanobacterium formicium)和巴氏甲烷八叠球菌(Methanosarcina barkeri)是最早鉴定出来,同时也是研究最为深入的产甲烷菌。与CH4为能量来源的嗜甲烷菌(Methanotrophs)不同,产甲烷菌(Methanogen)这个概念首次由Bryant提出并被广泛引用和研究[13]。随着分子生物学等技术的发展,越来越多存在于反刍动物瘤胃、海洋沉积物、厌氧发酵罐中的产甲烷菌菌株被鉴定出来。已知的产甲烷菌可以分为4个纲:甲烷杆菌纲(Methanobacteria)、甲烷球菌纲(Methanococci)、甲烷微菌纲(Methanomicrobia)和甲烷火菌纲(Methanopyri),7个目:甲烷杆菌目(Methanobacteriales)、甲烷球菌目(Methanococcales)、甲烷微菌目(Methanomicrobiales)、甲烷八叠球菌目(Methanosarcinales)、甲烷火球菌目(Methanopyrales)、甲烷胞菌目(Methanocellales)和甲烷原体目(Methanoplasmatales)。甲烷杆菌属(Methanobacterium)、甲烷短杆菌属(Methanobrevibacter)、甲烷微菌属(Methanomicrobium)和甲烷八叠球菌属(Methanosarcine)是广泛存在于反刍动物瘤胃内的4个产甲烷菌属[14]。目前有7个种的瘤胃产甲烷菌能够在体外培养基中存活,即:甲酸甲烷杆菌(Methanobacterium formicicum)、布氏产甲烷杆菌(Methanobacterium bryantii)、反刍兽甲烷短杆菌(Methanobrevibacter ruminantium)、米氏甲烷短杆菌(Methanobrevibacter millerae)、奥氏甲烷短杆菌(Methanobrevibacter olleyae)、可活动甲烷微菌(Methanomicrobium mobile)和奥兰汤基甲烷囊菌(Methanoculleus olentangyi)[15]。近年来,人们更多地利用系统发育学分类法,以物种间小亚基核糖体核苷酸差异性作为判断依据的方法被逐渐应用到产甲烷菌的鉴定和分类中[16];以16S rRNA/mcrA功能基因克隆文库等为代表的分子生物学技术也被逐渐应用到分析产甲烷菌的种群结构组成、群落多样性、菌群功能及其代谢机理的研究中。
2 产甲烷菌的甲烷合成途径瘤胃产甲烷菌生存在严格的厌氧环境中,通过CH4的生物合成作用形成维持细胞生存所需的能量,具有独特的生物化学代谢途径。产甲烷古菌能够将CO2、H2、甲酸等物质转化成CH4或者CH4和CO2。自然界中CH4生成的3种途径包括CO2还原途径、甲基营养型途径和乙酸发酵型途径。还原CO2是反刍动物瘤胃产CH4的重要途径(图 1),占到瘤胃总排放量的90%以上,而以挥发性脂肪酸和甲基化合物为底物的产甲烷途径较难在瘤胃内发生[17]。这3种途径中有众多关键酶和辅酶的参与[11, 18],但最终都形成甲基辅酶M(methyl-coenzyme M),其在甲基辅酶M还原酶(methyl-coenzyme reductase, mcr)的催化下生成CH4。甲基辅酶M还原酶是产甲烷菌特有的一种酶(除甲烷营养型古菌外),编码mcr其中的1个亚基基因(mcrA)可用于产甲烷菌的分类学研究。如图 1所示,产CH4过程伴随着细胞膜内外钠离子或质子跨膜梯度的形成和变化,能够借助位于细胞膜上的ATP合成酶将ADP转化为ATP。3种生物产CH4途径中产生的能量各有差异,以CO2还原途径所产生的能量最高,为-135△G kJ/mol CH4,而甲基营养型途径和乙酸发酵型产能较低[17-20]。
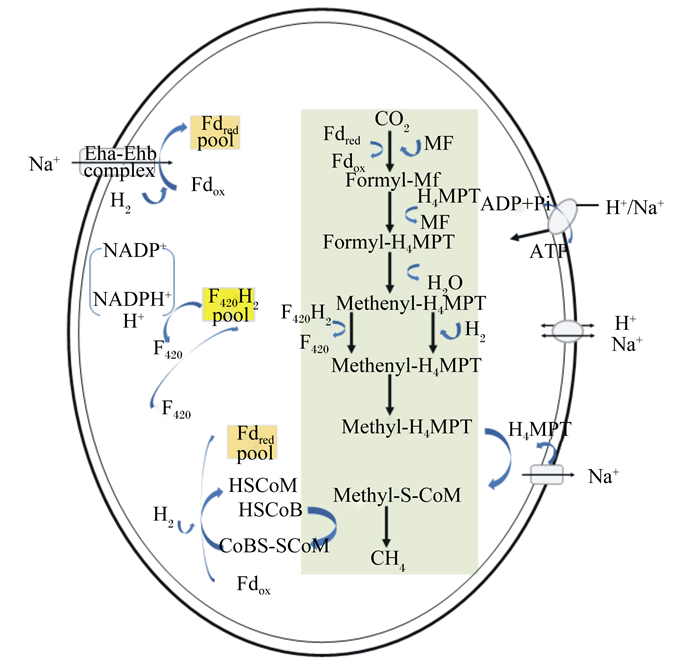
|
Fdred:还原的铁氧化还原蛋白ferredoxin-reduced;Fdox:氧化的铁氧化还原蛋白ferredoxin-oxygenated;MF:甲烷呋喃methanofuran;H4MPT:四氢甲烷蝶呤tetrahydromethanopterin;Methyl-S-CoM:甲基辅酶M methyl coenzyme M;F420:辅酶F420氧化态oxidized form of coenzyme F420;F420H2:辅酶F420还原态reduced form of coenzyme F420;HSCoM:辅酶M coenzyme M;HSCoB:辅酶B coenzyme B;CoMS-SCoB:异质二硫化物heterodisulfide of CoM and CoB;NADP:烟酰胺腺嘌呤二核苷磷酸nicotinamide adenine dinucleoside phosphate;NADPH:还原型烟酰胺腺嘌呤二核苷酸磷酸nicotinamide adenine dinucleotide phosphate。 图 1 产甲烷菌合成代谢通路(以反刍兽甲烷短杆菌为例) Fig. 1 Synthesis and metabolism pathway of methanogens (take Methanobrevibacter ruminantium as example)[17] |
动物品种、饲粮以及地理位置之间的复杂互作关系对奶牛瘤胃产甲烷菌区系具有明显的影响。King等[21]从荷斯坦和娟姗泌乳奶牛瘤胃液样品中提取微生物总DNA,分别从瘤胃产甲烷菌基因克隆文库得到了180和185个克隆子。试验得到的365条有效序列分属为55个操作分类单元(operational taxonomic units,OTU),其中荷斯坦和娟姗泌乳奶牛独有的OTU数量分别是23和12个,两者共有OTU数量为20个(占总序列的85%)。研究发现,娟姗牛瘤胃内米氏甲烷短杆菌相对丰度是荷斯坦奶牛的2倍以上,而反刍兽甲烷短杆菌相对丰度在2个品种之间没有显著差异。从香农指数的结果中发现,荷斯坦奶牛产甲烷菌的多样性显著高于娟姗牛。Skillman等[22]采用同样方法研究了放牧条件下娟姗牛瘤胃内产甲烷菌的分布特征,发现优势产甲烷菌分别为反刍兽甲烷短杆菌和unassigned Methanosphaera species,相对丰度都为33.3%,而居于第3位的unassigned Methanobrevibacter species相对丰度较低,为26.7%。同样的,Whitford等[23]发现舍饲条件下荷斯坦泌乳奶牛瘤胃优势产甲烷菌分别为unassigned Methanobrevibacter species和unassigned Methanosphaera species,相对丰度分别为36.6%和26.8%;反刍兽甲烷短杆菌和unassigned species of Methanosarcinales相对丰度则分别为21.9%和14.6%。De Mulder等[24]比较了荷斯坦和比利时蓝泌乳牛在相同饲粮组成和生理条件下瘤胃甲烷菌的多样性特征。基于16S rRNA扩增子测序的结果表明,甲烷杆菌科(Methanobacteriaceae)、甲烷热球菌科(Methanomassiliicoccaceae)和甲烷八叠球菌科(Methanosarcinaceae)是2个奶牛品种瘤胃内的优势菌群;但3种产甲烷菌的相对丰度在2个品种之间没有显著差异。以上结果与Cersosimo等[25]结果一致,认为荷斯坦和娟姗牛瘤胃内存在以甲烷短杆菌属为核心的产甲烷菌群。
反刍动物瘤胃内包括产甲烷菌在内的微生态系统对机体的生理活动非常重要,而奶牛所处的不同生理阶段反过来能够直接影响产甲烷菌的菌群组成和多样性[24]。Guzman等[26]采用16S rDNA和实时荧光定量PCR技术,发现荷斯坦犊牛出生20 min后瘤胃内就有Methanomicrobiales mobile、Methanobrevibacter spp.和Geobacter spp.的存在。出生后第2天沃氏球菌(Methanococcales votae)开始出现,但其相对丰度显著低于Methanomicrobiales mobile和Geobacter spp.。瘤胃内产甲烷菌相对丰度变化的原因可能与其他菌群的存在有关,如Geobacter spp.较早在瘤胃内存在,且能够通过直接的电子转移与产甲烷菌形成协同作用,减少有机物发酵产物的积累而促进产甲烷菌的定植。作为检测瘤胃产甲烷菌存在和相对丰度的重要标记物,Dong等[27]运用基于mcrA功能基因的高通量测序方法发现荷斯坦犊牛液体饲粮、固态饲粮和断奶时间的改变导致瘤胃内产甲烷菌菌群组成发生变化,在属水平上优势产甲烷菌分别是甲烷短杆菌和Methanosphaera。Cunha等[28]采用16S rRNA测序技术分析了处于青春前期、青春期和配种期的荷斯坦后备奶牛瘤胃中产甲烷菌菌群的多样性,结果发现3个不同生理阶段瘤胃内属水平上的优势菌群分别是甲烷短杆菌属和Methanosphaera,其相对丰度分别为95.03%和4.32%。但是随着生理阶段的递进,产甲烷菌的优势群落及数量上都存在较显著的差异。如青年前期后备奶牛瘤胃内甲烷短杆菌属的相对丰度显著低于青春期和配种期奶牛,但其瘤胃内Methanosphaera相对丰度最高。泌乳中期奶牛瘤胃产甲烷菌的组成与Cunha等[28]的结果相似,在属水平上甲烷短杆菌属相对丰度最高(88.8%),其次是Methanosphaera和甲烷热球菌科,相对丰度分别为4.8%和5.6%[29]。Wang等[30]采用基于功能基因的高通量测序方法对泌乳前期荷斯坦奶牛瘤胃内产甲烷菌进行分析,从6个mcrA克隆文库中得到了763条序列,分属于25个OTU。结果显示优势的产甲烷菌为未培养产甲烷菌(rumen cluster C,RCC),其相对丰度为71%,而甲烷杆菌相对丰度只有29%。Janssen等[15]发现RCC在瘤胃的含量较低,仅占到古菌总量的15.8%,而甲烷短杆菌属占古菌总量的比例超过60%以上。Li等[31]运用16S rRNA方法发现甲烷短杆菌属是泌乳前期荷斯坦奶牛瘤胃内的优势产甲烷菌,这与以往的研究结果一致,认为甲烷短杆菌属的相对丰度与瘤胃产甲烷能力密切相关。Cersosimo等[25]研究了荷斯坦、娟姗以及荷斯坦与娟姗杂交奶牛在不同泌乳阶段瘤胃产甲烷菌的特征。在泌乳的第3天,娟姗牛瘤胃内陶氏甲烷短杆菌(Methanobrevibacter thaueri)相对丰度(26.9%)显著低于荷斯坦奶(30.7%)和荷斯坦与娟姗杂交奶牛(30.3%)。3个品种奶牛在泌乳期间瘤胃内的主要产甲烷菌是史氏甲烷短杆菌(Methanobrevibacter smithii)、陶氏甲烷短杆菌、反刍兽甲烷短杆菌和米氏甲烷短杆菌。Jeyanathan等[32]研究发现反刍兽甲烷短杆菌、奥氏甲烷短杆菌、戈氏甲烷短杆菌(Methanobrevibacter gottschalkii)、陶氏甲烷短杆菌和米氏甲烷短杆菌,以及Methanosphaera相关的产甲烷菌共同存在于荷斯坦与娟姗杂交奶牛和绵羊的瘤胃内,属于核心的产甲烷菌群,不随着动物种类和饲粮类型而改变,这为下一步明确和寻找降低CH4产量的主导菌群提供了思路。
3.2 肉牛Wright等[33]从海福特杂交肉牛的瘤胃内容物中提取总DNA,利用产甲烷菌的特异性引物扩增建立了产甲烷菌基因文库。对241个克隆进行RDP分析之后共获得23个16S rRNA序列,60%以上的序列为反刍兽甲烷短杆菌。其中10个序列与可培养的3种产甲烷菌(甲烷杆菌目、甲烷微菌目和甲烷八叠球菌目)16S rRNA序列的相似性为89.8%~100.0%。13个序列与火山嗜热原体(Thermoplasma volcanium)和嗜酸热原体(Thermoplasma acidophilum)的相似性为74.1%~75.8%,表明该品种肉牛瘤胃产甲烷菌以反刍兽甲烷短杆菌为优势菌群。与上述结果一致,Daquiado等[34]采用基于mcrA功能基因的高通量测序手段研究了韩牛(Bos taurus coreanae)瘤胃液内产甲烷菌的多样性特征。试验共得到146个mcrA克隆,归属于6个OTU。研究发现反刍兽甲烷短杆菌(93个克隆)为优势产甲烷菌,占到了产甲烷菌总量的63.6%,Methanobrevibacter milerae和Methanobrevibacter mobile的相对丰度分别为21.2%和6.8%。Carberry等[35]研究也证实甲烷短杆菌属是利木赞杂交肉牛瘤胃内的优势菌属,主要包括史氏甲烷短杆菌和反刍兽甲烷短杆菌。Sirohi等[36]综合采用16S rRNA和功能基因mcrA构建了荷斯坦与塔帕卡杂交肉牛瘤胃产甲烷菌的基因文库。16S rRNA文库中的13个OTU都属于甲烷杆菌目,其中12个OTU属于Methanobrevibacter spp.,1个OTU属于斯氏甲烷球菌(Methanosphaera stadtmanae)。而mcrA文库18个OTU中有15个OTU属于甲烷杆菌目,2个OTU属于甲烷微菌目,只有1个OTU属于甲烷八叠球菌目。研究表明,荷斯坦杂交肉牛瘤胃中甲烷杆菌目是优势产甲烷菌,但基于功能基因的mcrA克隆文库能够更好地反映瘤胃内产甲烷菌的多样性。为了比较瘤胃古菌特异性引物对产甲烷菌群多样性的影响,裴彩霞等[37]基于3种特异性引物构建了中国晋南牛产甲烷菌的克隆文库,结果发现引物Arch f364/1386建立的克隆库中,100个克隆中有61个和15个克隆分别属于Methanobrevibacter sp. 1Y和Methanobrevibacter sp. SM9(相似性≥97%)。以1Af/1100Ar为引物的克隆文库中,所有16个OTU中只有JNB8和Methanosphaera stadtmanae DSM 3091的相似性大于97%,其他序列与奥胡斯甲烷杆菌(Methanobacterium aarhusense)或斯氏甲烷球菌的相似性较低。以Met86F/Met1340R为引物建立的产甲烷菌克隆文库中,属于Methanobrevibacter sp. 1Y的克隆子有47个,而只有26个克隆子属于Methanobrevibacter sp. SM9,两者的相似性都在98%以上。该结果与以Arch f364/1386为引物的菌群结构分析最为相似,表明晋南牛瘤胃内优势产甲烷菌为甲烷短杆菌属。以上结果表明,甲烷短杆菌属为肉牛瘤胃内的优势菌群,其组成和多样性结果不受遗传背景、杂交特征以及研究手段的影响。关于不同品种肉牛瘤胃内产甲烷菌区系的多样性及相关代谢通路的差异化机理还有待深入研究。
3.3 水牛和牦牛中国水牛以沼泽型品种为主,大多分布在淮河以南的水稻产区,由于水牛生活环境的特殊性和耐粗饲等特征,使其具有特殊的瘤胃功能;探索其瘤胃内产甲烷菌菌群结构和多样性对于提高水牛生产效率和降低温室气体排放具有重要意义。杨承剑等[38]基于Met86F/Met1340R引物研究了60月龄雌性德昌水牛瘤胃内产甲烷菌的多样性。在99条16S rRNA基因序列中属于甲烷短杆菌属的序列占到94%以上,该结果与以往关于其他反刍动物(肉牛和牦牛)上的研究结果[39-40]相一致。刘园园等[41]采用聚合酶链反应-变性梯度凝胶电泳(PCR-DGGE)技术对广西本地沼泽型水牛瘤胃产甲烷菌进行了研究,测序结果得到的5条古菌序列中,4条均属于甲烷短杆菌属,属于瘤胃内的优势产甲烷菌。Franzolin等[42]对比了3种(玉米秸秆、牧草和甘蔗稍叶)粗饲料组成对巴西水牛(Bubalus bubalis)瘤胃产甲烷菌菌群多样性的影响,从构建的3个克隆文库中共获得467个有效克隆子,可分为19个OTU,而甲烷短杆菌属是3个组中共有的优势产甲烷菌菌群。Singh等[43]基于mcrA功能基因发现印度苏替水牛瘤胃内的产甲烷菌以甲烷杆菌目为主。杨承剑等[44]发现中国摩拉水牛和德宏水牛瘤胃液中产甲烷菌以甲烷杆菌目中的甲烷短杆菌属为优势菌群,但德宏水牛瘤胃内发现较多未知的产甲烷菌序列。与以上结果不同,Kumar等[45]采用16S rRNA基因克隆文库的方法比较了印度4个地区摩拉水牛瘤胃内产甲烷菌的多样性分布,结果表明旁遮普、北方邦和拉贾斯坦邦地区水牛瘤胃优势产甲烷菌是甲烷短杆菌属,而甲烷微菌属是哈里亚纳邦地区水牛瘤胃内的优势菌群。Chaudhary等[46]采用Met86F/Met1340R为引物从印度摩拉水牛获得了17条16S rRNA基因序列,证明15条序列属于甲烷微菌属,属于瘤胃内的优势产甲烷菌。从目前的文献结果来看,尽管甲烷微菌属作为主导菌群存在于水牛瘤胃内,但不同品种和区域内水牛瘤胃内大多以甲烷短杆菌属为优势产甲烷菌群。由于水牛生存环境和饲养模式的特殊性,其瘤胃内产甲烷菌的分布和功能等特征还需进一步研究。
牦牛是高海拔地区重要的草食性反刍动物,能够把其他牛种无法利用的天然草地资源转化成人类需要的生产和生活资料。牦牛超强的环境适应能力和耐粗饲料的显著优势,使得其瘤胃具有独特的生物学研究价值。张学燕等[47]分析了青海省河南县放牧牦牛瘤胃食糜和瘤胃液中产甲烷菌的分布。基于产甲烷菌特异性引物Met86F/Met1340R扩增16S rRNA基因的研究发现,由2种样品构建的克隆文库中有200个序列,其中126个序列(食糜中68个、瘤胃液中58个)属于甲烷短杆菌属,占总序列的63%,12个序列(瘤胃食糜中7个、瘤胃液中5个)属于产甲烷球菌属(Methanococcus),占总序列6%。与此同时,Methanobrevibacter ruminantium strain Yak M3是瘤胃食糜中的优势序列,而Methanobrevibacter sp.1Y则为瘤胃液中的优势产甲烷菌序列。Xue等[48]发现西藏红原地区牦牛(Bos grunniens)瘤胃内优势产甲烷菌群分别是甲烷杆菌科(占总产甲烷菌含量的82.5%,下同)、甲烷热球菌科(13.0%)、甲烷八叠球菌科(3.0%)和甲烷微菌目(1.5%)。与以上结论不同,Huang等[49]发现青海牦牛瘤胃内的优势产甲烷菌是甲烷马赛球菌(Methanomassiliicoccales,80.9%)、甲烷杆菌目(12.4%)和甲烷微菌目(0.96%)。原因可能与采用的测定手段和动物品种有关。Wang等[50]采用高通量测序的方法比较了2个生理阶段(60和120月龄)西藏甘孜地区牦牛瘤胃内产甲烷菌的多样性,共获得2 033个OTU,其中974个OTU属于成年牦牛(60月龄),846个OTU属于老年牦牛(120月龄),而两者共有213个OTU。甲烷短杆菌属和热无胞壁单胞菌(Thermogymnomonas)是2个生理阶段牦牛瘤胃内的优势菌,甲烷短杆菌属在老年牦牛瘤胃内的相对丰度显著高于成年牦牛,而老年牦牛瘤胃内热无胞壁单胞菌的相对丰度比成年牦牛低34%,表明牦牛瘤胃内产甲烷菌的结构和相对丰度会随着日龄的增加而发生显著性改变。有研究发现,与肉牛瘤胃内优势产甲烷菌相比,牦牛瘤胃液里甲烷短杆菌属的相对丰度较高,尽管肉牛和牦牛的甲烷排放量与它们瘤胃内产甲烷菌组成和多样性的差异没有直接的相关关系,但肉牛瘤胃内较高丰度的甲烷马赛球菌有助于底物(H2)的利用和CH4的产生[51-52]。
4 小结CH4是一种造成全球性极端气候变化的重要的温室气体之一,是反刍动物瘤胃内产甲烷菌参与饲粮有机物厌氧发酵的终端产物,同时也是清洁能源的主要成分。因此,开展反刍动物瘤胃产甲烷菌生成甲烷的机制研究,对于提高畜牧养殖业生产效率、减缓全球变暖趋势和开发清洁能源具有重要的理论和实践意义。近年来,分子生物学技术的发展和应用克服了传统研究方法的局限,为深入了解瘤胃产甲烷菌的菌群组成和结构变化规律提供了强有力的手段,并取得了显著成效。尽管动物品种、遗传背景、生理阶段、地理背景和研究手段等因素能够影响反刍动物瘤胃内产甲烷菌的分布特征,但是越来越多的研究证实甲烷短杆菌属及其相关的产甲烷菌种能够在维持瘤胃发酵稳态和产甲烷方面发挥重要的主导作用。另外,随着现代宏基因组学技术在反刍动物瘤胃中的应用,人们能够更为全面和深入地研究产甲烷菌的代谢途径、营养调控,挖掘出更多以前尚未发现的关键性功能性基因,以及更多的不可培养的产甲烷菌菌群,为探索和开发甲烷减排和调控措施提供新的理论和科学指导。
致谢:
感谢塔里木大学叶疆凤同学参与该综述的文献搜集和整理工作,感谢四川农业大学动物营养研究所彭全辉副教授给予本文撰写和内容上的指导。
| [1] |
世界气象组织.温室气体公报[EB/OL].[2019-01-20].https://public.wmo.int/zh-hans/media.
|
| [2] |
IPCC.AR5 synthesis report: climate change 2014[R].Geneva, Switzerland: IPCC, 2014.
|
| [3] |
殷志扬, 袁小慧. 我国奶牛养殖业布局及生产组织模式现状[J]. 江苏农业科学, 2013, 41(8): 8-10. DOI:10.3969/j.issn.1002-1302.2013.08.003 |
| [4] |
中国农业年鉴编辑委员会. 中国农业年鉴[M]. 北京: 中国农业出版社, 2017.
|
| [5] |
牛春娥, 张利平, 孙俊锋, 等. 我国牦牛资源现状及其产品开发利用前景分析[J]. 安徽农业科学, 2009, 37(17): 8003-8005. DOI:10.3969/j.issn.0517-6611.2009.17.081 |
| [6] |
杨炳壮, 梁贤威, 曾庆坤, 等. 世界水牛发展趋势[J]. 中国牧业通讯, 2005(15): 70-71. |
| [7] |
IPCC.2006 IPCC guidelines for national greenhouse gas inventories[R].Kanagawa, Japan: Intergovernmental Panel on Climate Change, 2006.
|
| [8] |
王荣, 邓近平, 王敏, 等. 基于IPCC Tier 1层级的中国反刍家畜胃肠道甲烷排放格局变化分析[J]. 生态学报, 2015, 35(21): 7244-7254. |
| [9] |
MUÑOZ C, HUBE S, MORALES J M, et al. Effects of concentrate supplementation on enteric methane emissions and milk production of grazing dairy cows[J]. Livestock Science, 2015, 175: 37-46. DOI:10.1016/j.livsci.2015.02.001 |
| [10] |
周艳, 邓凯东, 董利锋, 等. 反刍家畜肠道甲烷的产生与减排技术措施[J]. 家畜生态学报, 2018, 39(4): 6-10, 54. DOI:10.3969/j.issn.1673-1182.2018.04.002 |
| [11] |
KNAPP J R, LAUR G L, VADAS P A, et al. Invited review:enteric methane in dairy cattle production:quantifying the opportunities and impact of reducing emissions[J]. Journal of Dairy Science, 2014, 97(6): 3231-3261. DOI:10.3168/jds.2013-7234 |
| [12] |
NIU M T, KEBREAB E, HRISTOV A N, et al. Prediction of enteric methane production, yield, and intensity in dairy cattle using an intercontinental database[J]. Global Change Biology, 2018, 24(8): 3368-3389. DOI:10.1111/gcb.14094 |
| [13] |
BRYANT M P.Methane-producing bacteria[M]//BUCHANAN R E, GIBBONS N E.Bergey's manual of determinative bacteriology.8th ed.Baltimore: Williams and Wilkins Co., 1974.
|
| [14] |
王保玉, 刘建民, 韩作颖, 等. 产甲烷菌的分类及研究进展[J]. 基因组学与应用生物学, 2014, 33(2): 418-425. |
| [15] |
JANSSEN P H, KIRS M. Structure of the archaeal community of the rumen[J]. Applied and Environmental Microbiology, 2008, 74(12): 3619-3625. DOI:10.1128/AEM.02812-07 |
| [16] |
张瑞, 雍晓雨, 周俊, 等. 分子生物学技术在产甲烷古菌多样性研究中的应用[J]. 江苏农业科学, 2015, 43(1): 16-20. |
| [17] |
LEAHY S C, KELLY W J, ALTERMANN E, et al. The genome sequence of the rumen methanogen Methanobrevibacter ruminantium reveals new possibilities for controlling ruminant methane emissions[J]. PLoS One, 2010, 5(1): e8926. DOI:10.1371/journal.pone.0008926 |
| [18] |
方晓瑜, 李家宝, 芮俊鹏, 等. 产甲烷生化代谢途径研究进展[J]. 应用与环境生物学报, 2015, 21(1): 1-9. |
| [19] |
祖波, 祖建, 周富春, 等. 产甲烷菌的生理生化特性[J]. 环境科学与技术, 2008, 31(3): 5-7, 51. |
| [20] |
王梦芝, 王曙, 潘晓花, 等. 4种油脂对瘤胃微生物体外产气及辅酶F420的影响[J]. 动物营养学报, 2011, 23(10): 1819-1825. |
| [21] |
KING E E, SMITH R P, ST-PIERRE B, et al. Differences in the rumen methanogen populations of lactating Jersey and Holstein dairy cows under the same diet regimen[J]. Applied and Environmental Microbiology, 2011, 77(16): 5682-5687. DOI:10.1128/AEM.05130-11 |
| [22] |
SKILLMAN L C, EVANS P N, STRÖMPL C, et al. 16S rDNA directed PCR primers and detection of methanogens in the bovine rumen[J]. Letters in Applied Microbiology, 2006, 42(3): 222-228. DOI:10.1111/j.1472-765X.2005.01833.x |
| [23] |
WHITFORD M F, TEATHER R M, FORSTER R J. Phylogenetic analysis of methanogens from the bovine rumen[J]. BMC Microbiology, 2001, 1: 5. DOI:10.1186/1471-2180-1-5 |
| [24] |
DE MULDER T, PEIREN N, VANDAELE L, et al. Impact of breed on the rumen microbial community composition and methane emission of Holstein Friesian and Belgian Blue heifers[J]. Livestock Science, 2018, 207: 38-44. DOI:10.1016/j.livsci.2017.11.009 |
| [25] |
CERSOSIMO L M, BAINBRIDGE M L, KRAFT J, et al. Influence of periparturient and postpartum diets on rumen methanogen communities in three breeds of primiparous dairy cows[J]. BMC Microbiology, 2016, 16: 78. DOI:10.1186/s12866-016-0694-7 |
| [26] |
GUZMAN C E, BEREZA-MALCOLM L T, DE GROEF B, et al. Presence of selected methanogens, fibrolytic bacteria, and proteobacteria in the gastrointestinal tract of neonatal dairy calves from birth to 72 hours[J]. PLoS One, 2015, 10(7). |
| [27] |
DONG L F, MA J N, TU Y, et al. Weaning methods affect ruminal methanogenic archaea composition and diversity in Holstein calves[J]. Journal of Integrative Agriculture, 2019, 18(5): 1080-1092. DOI:10.1016/S2095-3119(18)62120-3 |
| [28] |
CUNHA C S, MARCONDES M I, VELOSO C M, et al. Compositional and structural dynamics of the ruminal microbiota in dairy heifers and its relationship to methane production[J]. Journal of the Science of Food and Agriculture, 2019, 99(1): 210-218. DOI:10.1002/jsfa.9162 |
| [29] |
DANIELSSON R, DICKSVED J, SUN L, et al. Methane production in dairy cows correlates with rumen methanogenic and bacterial community structure[J]. Frontiers in Microbiology, 2017, 8: 226. |
| [30] |
WANG P P, ZHAO S G, WANG X W, et al. Ruminal methanogen community in dairy cows fed agricultural residues of corn stover, rapeseed, and cottonseed meals[J]. Journal of Agricultural and Food Chemistry, 2016, 64(27): 5439-5445. DOI:10.1021/acs.jafc.6b00708 |
| [31] |
LI X H, LIU C, CHEN Y X, et al. Effects of mineral salt supplement on enteric methane emissions, ruminal fermentation and methanogen community of lactating cows[J]. Animal Science Journal, 2017, 88(8): 1049-1057. DOI:10.1111/asj.12738 |
| [32] |
JEYANATHAN J, MARTIN C, MORGAVI D P. The use of direct-fed microbials for mitigation of ruminant methane emissions:a review[J]. Animal, 2014, 8(2): 250-261. DOI:10.1017/S1751731113002085 |
| [33] |
WRIGHT A D G, AUCKLAND C H, LYNN D H. Molecular diversity of methanogens in feedlot cattle from Ontario and Prince Edward Island, Canada[J]. Applied and Environmental Microbiology, 2007, 73(13): 4206-4210. DOI:10.1128/AEM.00103-07 |
| [34] |
DAQUIADO A R, CHO K M, KIM T Y, et al. Methanogenic archaea diversity in Hanwoo (Bos taurus coreanae) rumen fluid, rectal dung, and barn floor manure using a culture-independent method based on mcrA gene sequences[J]. Anaerobe, 2014, 27: 77-81. DOI:10.1016/j.anaerobe.2014.01.008 |
| [35] |
CARBERRY C A, WATERS S M, KENNY D A, et al. Rumen methanogenic genotypes differ in abundance according to host residual feed intake phenotype and diet type[J]. Applied and Environmental Microbiology, 2014, 80(2): 586-594. DOI:10.1128/AEM.03131-13 |
| [36] |
SIROHI S K, CHAUDHARY P P, SINGH N, et al. The 16S rRNA and mcrA gene based comparative diversity of methanogens in cattle fed on high fibre based diet[J]. Gene, 2013, 523(2): 161-166. DOI:10.1016/j.gene.2013.04.002 |
| [37] |
裴彩霞, 毛胜勇, 朱伟云. 晋南牛瘤胃中古菌分子多样性的研究[J]. 微生物学报, 2008, 48(1): 8-14. DOI:10.3321/j.issn:0001-6209.2008.01.003 |
| [38] |
杨承剑, 梁辛, 李丽莉, 等. 摩拉水牛及德宏水牛瘤胃产甲烷菌多样性比较[J]. 中国畜牧兽医, 2018, 45(2): 365-374. |
| [39] |
WRIGHT A D G, MA X L, OBISPO N E. Methanobrevibacter phylotypes are the dominant methanogens in sheep from Venezuela[J]. Microbial Ecology, 2008, 56(2): 390-394. DOI:10.1007/s00248-007-9351-x |
| [40] |
AN D D, DONG X Z, DONG Z Y. Prokaryote diversity in the rumen of yak (Bos grunniens) and Jinnan cattle (Bos taurus) estimated by 16S rDNA homology analyses[J]. Anaerobe, 2005, 11(4): 207-215. DOI:10.1016/j.anaerobe.2005.02.001 |
| [41] |
刘园园, 王士长.PCR-DGGE技术在水牛瘤胃产甲烷古菌多样性探索中的应用[C]//第四届第十次全国学术研讨会暨动物微生态企业发展战略论坛论文集(下册).沧州: 中国畜牧兽医学会动物微生态学分会, 2010: 180-186. http://cpfd.cnki.com.cn/Article/CPFDTOTAL-ZGXJ201008008049.htm
|
| [42] |
FRANZOLIN R, ST-PIERRE B, NORTHWOOD K, et al. Analysis of rumen methanogen diversity in water buffaloes (Bubalus bubalis) under three different diets[J]. Microbial Ecology, 2012, 64(1): 131-139. DOI:10.1007/s00248-012-0007-0 |
| [43] |
SINGH K M, PANDYA P R, PARNERKAR S, et al. Methanogenic diversity studies within the rumen of surti buffaloes based on methyl coenzyme m reductase a (mcrA) genes point to Methanobacteriales[J]. Polish Journal of Microbiology, 2010, 59(3): 175-178. |
| [44] |
杨承剑, 梁辛, 韦升菊, 等. 基于16S rRNA基因克隆文库技术分析广西富钟水牛瘤胃产甲烷菌组成及多样性[J]. 动物营养学报, 2014, 26(12): 3635-3642. |
| [45] |
KUMAR S, DAGAR S S, AGRAWAL R K, et al. Comparative diversity analysis of ruminal methanogens in Murrah buffaloes (Bubalus bubalis) in four states of North India[J]. Anaerobe, 2018, 52: 59-63. DOI:10.1016/j.anaerobe.2018.05.012 |
| [46] |
CHAUDHARY P P, SIROHI S K. Dominance of Methanomicrobium phylotype in methanogen population present in Murrah buffaloes (Bubalus bubalis)[J]. Letters in Applied Microbiology, 2009, 49(2): 274-277. DOI:10.1111/j.1472-765X.2009.02654.x |
| [47] |
张学燕, 刘书杰, 崔占鸿, 等. 应用16S rRNA基因序列技术分析青海高原放牧牦牛瘤胃产甲烷菌的多样性[J]. 青海大学学报, 2018, 36(1): 9-16, 46. |
| [48] |
XUE D, CHEN H, CHEN F, et al. Analysis of the rumen bacteria and methanogenic archaea of yak (Bos grunniens) steers grazing on the Qinghai-Tibetan Plateau[J]. Livestock Science, 2016, 188: 61-71. DOI:10.1016/j.livsci.2016.04.009 |
| [49] |
HUANG X D, TAN H Y, LONG R J, et al. Comparison of methanogen diversity of yak (Bos grunniens) and cattle (Bos taurus) from the Qinghai-Tibetan plateau, China[J]. BMC Microbiology, 2012, 12: 237. DOI:10.1186/1471-2180-12-237 |
| [50] |
WANG L Z, WANG Z S, XUE B, et al. Comparison of rumen archaeal diversity in adult and elderly yaks (Bos grunniens) using 16S rRNA gene high-throughput sequencing[J]. Journal of Integrative Agriculture, 2017, 16(5): 1130-1137. DOI:10.1016/S2095-3119(16)61454-5 |
| [51] |
HENDERSON G, COX F, GANESH S, et al. Rumen microbial community composition varies with diet and host, but a core microbiome is found across a wide geographical range[J]. Scientific Reports, 2015, 5: 14567. DOI:10.1038/srep14567 |
| [52] |
MI J D, ZHOU J W, HUANG X D, et al. Lower methane emissions from yak compared with cattle in Rusitec fermenters[J]. PLoS One, 2017, 12(1): e0170044. DOI:10.1371/journal.pone.0170044 |




