自噬是当前生命科学领域研究的热点及难点。在正常生理状态下,自噬可以保持在一个相对稳定的水平,从而调节蛋白质周转代谢的平衡。研究表明,诸多因素都会诱导细胞发生强烈的自噬反应,如营养缺乏、环境应激以及运动刺激等[1-4]。氨基酸作为蛋白质合成的基本单位,为动物细胞代谢和体组织生长发育所必需。当氨基酸不足时,则会诱导自噬等一系列不良反应[5],但不同氨基酸缺乏或供给与自噬的调控关系尚不完全清楚。
1 自噬概述营养缺乏可以导致细胞分解代谢途径的激活,这种激活途径是一个保守的过程,在许多细胞生长发育过程中都发挥着极其重要的作用[6-8]。自噬是细胞受到外部环境刺激时的一种应激反应[9],其标志是微管相关蛋白质1轻链3(microtubule-associated protein l light chain 3,LC3-Ⅰ)转化成微管相关蛋白质2轻链3(microtubule-associated protein 2 light chain 3,LC3-Ⅱ)。因此,LC3也被认为是自噬膜形成和自噬起始的标志物[10-11]。在哺乳动物中,有关细胞自噬的研究已十分深入,自噬对细胞代谢水平的维持、着床前胚胎的发育、新生儿的存活和器官的发生都是至关重要的[12];同时,自噬也被证明与许多疾病和癌症的发生及预防有着密切的关系[13-15]。
自噬有3种不同的类型,包括宏观自噬、微观自噬以及分子伴侣介导的自噬[16],虽然它们都会因营养缺乏而被激活,但它们在底物特异性、膜来源和向溶酶体传送的方式等方面都有所不同[17-18]。在这3种自噬类型中,宏观自噬(以下简称自噬)最具特色,也是本文的主要综述内容,它的发生主要包括4个阶段:起始阶段、伸长阶段、成熟阶段和完成阶段。在起始阶段细胞内首先会形成一个“隔离膜”,然后伸长、关闭,将细胞质中的线粒体等细胞器以及长寿命蛋白质包裹其中,最终形成一个完整的自噬体[19]。自噬体进而与溶酶体融合,形成自噬-溶酶体[20],然后发生降解,这种自噬体与溶酶体结合降解细胞内长寿命蛋白质以及损伤细胞器的途径称为自噬-溶酶体途径[21-23],但是其具体的分子调控机制尚不清楚。自噬-溶酶体的形成过程如图 1所示。
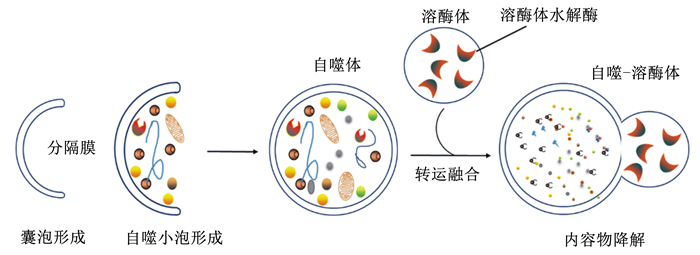
|
图 1 自噬-溶酶体的形成过程 Fig. 1 Formation process of autophagy-lysosome |
赖氨酸(lysine,Lys)作为一种必需氨基酸,不仅是蛋白质合成必不可少的成分,也是机体吸收利用其他营养物质的基本保证。Sato等[24]研究发现,Lys缺乏时,骨骼肌中蛋白质分解速率加快、合成速率减慢,导致肌肉质量减少,而正常水平Lys可抑制骨骼肌蛋白质的降解。原因可能是Lys不足激活自噬-溶酶体途径,导致骨骼肌细胞发生自噬,使得自身的蛋白质被降解用于抵抗由于外部环境等因素导致的胞内氨基酸缺乏,进而在短时间内维持自身氨基酸的平衡状态。
亮氨酸(leucine,Leu)属于中性氨基酸,它作为一种必需支链氨基酸,在动物体内不能合成,只能从食物中摄取来满足机体维持代谢平衡的需要。近年来的研究表明,Leu不仅可以调节哺乳动物体内蛋白质的代谢水平[25],而且在维持机体的免疫功能和抗氧化功能等方面也发挥着重要的作用[26]。许多研究已经将Leu剥夺引起的蛋白质分解与自噬联系在一起,Wu等[27]研究表明miR-17 microRNA家族的2个成员miR-20a和miR-106b可能通过抑制小鼠成肌细胞(C2C12)中Unc-51样自噬激活激酶1(Unc-51 like autophagy activating kinase 1,ULK1)的表达而参与调节Leu剥夺诱导的自噬。Sato等[24]的研究发现,摄入Leu可以通过激活自噬-溶酶体系统抑制肌原纤维蛋白质的降解。进一步的研究也发现,在用DMEM培养基培养的C2C12细胞中补充Leu 30 min后,LC3-Ⅱ与LC3-Ⅰ的比值显著降低,提示补充Leu对C2C12细胞自噬的发生具有显著的抑制作用。氨基酸缺乏可以诱导细胞发生自噬,Leu被认为是最有效的自噬抑制剂[28]。
精氨酸(arginine,Arg)是一氧化氮、多胺和蛋白质等生物合成的关键氨基酸,在肌肉蛋白质合成、机体免疫调控、组织损伤修复以及肠道健康维持等方面具有非常重要的作用[29]。正常细胞具有从瓜氨酸合成Arg的能力,因此,Arg被认为是半必需氨基酸[30]。Xia等[31]首次发现一般性调控阻遏蛋白质激酶2(general control nonderepressible 2,GCN2)可以介导干扰素-γ(interferon-γ,IFN-γ)响应Arg缺乏诱导的牛乳腺上皮细胞自噬。
谷氨酰胺(glutamine,Gln)是肌肉中丰富的游离氨基酸,约占人体游离氨基酸总量的60%。研究发现,Gln对维持人和动物的肠道完整性有着至关重要的作用[32-33]。朱玉华等[34]通过Gln缺乏的培养基处理猪肠道上皮细胞8 h后发现缺乏Gln可导致细胞数目显著减少,以及LC3-Ⅱ的表达显著升高,提示Gln缺乏可以诱导猪肠道上皮细胞自噬的发生。
综上所述,Lys、Leu、Arg和Gln这4种氨基酸关于自噬的研究较多,为了进一步阐述自噬的发生,对其机制解析发现,氨基酸对自噬的调控主要通过哺乳动物雷帕霉素靶蛋白质复合物1(mammalian target of rapamycin complex 1,mTORC1)信号通路来实现,但也有研究发现了一些其他的信号通路对自噬调控至关重要。
3 氨基酸依赖于mTORC1途径调控细胞自噬mTORC1是一种在进化上保守的丝氨酸/苏氨酸激酶,在调控蛋白质代谢、细胞生长和自噬等方面发挥着不可或缺的作用[35]。mTORC1可由氨基酸和胰岛素样生长因子等信号激活[36],通过磷酸化p70核糖体蛋白质S6激酶1(p70 ribosomal protein S6 kinase 1,S6K1)、真核起始因子4E结合蛋白质1(eukaryotic initiation factor 4E binding protein 1,4EBP1)和其他底物正向调节蛋白质翻译、核糖体生物发生以及细胞生长,通过磷酸化ULK1和自噬相关蛋白质13(autophagy-related protein 13,Atg13)对自噬起负向调控作用[37]。在细胞处于营养丰富的状态下,mTORC1支持细胞生长并抑制自噬。相反,在细胞处于营养饥饿的条件下,mTORC1就会被抑制,并诱导细胞发生自噬为其代谢提供能量[38]。mTORC1-自噬信号通路如图 2所示。
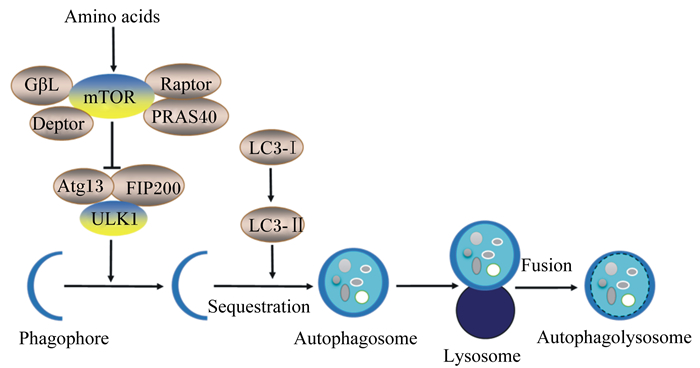
|
图 2 mTORC1-自噬信号通路 Fig. 2 mTORC1-autophagy signaling pathway |
mTOR:哺乳动物雷帕霉素靶蛋白质mammalian target of rapamycin;GβL:G蛋白质β亚基样蛋白质recombinant G protein beta subunit like protein;PRAS40:富脯氨酸AKT底物40 ku prolinerich AKT substrate of 40 ku;Raptor:mTOR调节相关蛋白质regulatory-associated protein of mTOR;Deptor:含有mTOR相互作用蛋白质的DEP结构域DEP domain-containing mTOR-interacting protein;Atg13:自噬相关蛋白质13 autophagy-related protein 13;FIP200:分子质量为200 ku的黏着斑激酶家族互作蛋白质focal adhesion kinase family interacting protein of 200 ku;ULK1:类Unc-51自噬激活激酶1 Unc-51 like autophagy activating kinase 1;LC3-Ⅰ:微管相关蛋白质1轻链3 microtubule-associated protein l light chain 3;LC3-Ⅱ:微管相关蛋白质2轻链3 microtubule-associated protein 2 light chain 3; Amino acids:氨基酸;Phagophore:吞噬泡;Sequestration:隔离;Autophagosome:自噬体;Lysosome:溶酶体; Fusion:融合;Autophagolysosome:自噬溶酶体。
3.1 赖氨酸依赖于mTORC1途径调控细胞自噬研究表明,自噬-溶酶体系统受mTORC1活性的调节[39]。Sato等[40]的研究中分析了Lys处理的C2C12肌管中mTORC1下游靶蛋白质S6K1和4EBP1的磷酸化水平,将C2C12肌管在含10 mmol/L Lys的培养基中处理30 min后,LC3-Ⅱ与LC3-Ⅰ的比值和对照组(培养基中不含氨基酸)相比显著降低了30%,S6K1和4EBP1的磷酸化水平也显著升高,表明Lys通过mTORC1依赖性途径调节C2C12肌管的自噬-溶酶体活性。当Lys供应充足时,Lys会通过激活mTORC1信号通路来抑制自噬-溶酶体系统的活性,并减少肌肉中肌原纤维蛋白质的降解。肌肉蛋白质的合成与mTORC1途径的激活有关,Lang等[41]的研究证明了补充Lys可以提高4EBP1的磷酸化水平,表明添加Lys可以通过激活肌细胞中的mTORC1信号通路来抑制自噬-溶酶体系统的活性,促进蛋白质的合成。有研究报道,Lys可以减缓大鼠肌原纤维蛋白质的降解,对每100 g体重的禁食大鼠灌喂114和570 mg Lys后发现肌原纤维蛋白质降解率明显降低,LC3-Ⅱ的表达量也显著降低,但是Lys抑制肌肉蛋白质降解的作用机制尚不清楚[24, 42]。
3.2 亮氨酸依赖于mTORC1途径调控细胞自噬Leu影响自噬主要是因为它能激活mTORC1信号通路,随后上调其下游靶蛋白质,对自噬产生抑制作用[43]。最近的研究表明,Leu对细胞自噬的抑制作用依赖于mTORC1信号通路,并且其上游靶蛋白质脑中丰富的Ras同系物(ras homologue enriched in brain,Rheb)和mTOR调节相关蛋白质(regulatory-associated protein of mTOR,Raptor)也是必需的[44]。Leu可以促进mTORC1的磷酸化,进而磷酸化其下游靶蛋白质4EBP1与S6K1[45],也能够激活mTORC1上游的靶蛋白质Rag,使得细胞内无活性且呈弥散分布的mTORC1聚合到其激活蛋白质周围。Lang等[41]和Yoshizaw等[46]的研究发现,补充Leu可以增强肌细胞mTORC1的活性,进而抑制细胞自噬的发生,将C2C12肌管细胞在含Leu的培养基中处理30 min,可显著提高4EBP1的磷酸化水平。吴浩等[47]的MicroRNA芯片结果表明,经过4 h Leu饥饿处理的C2C12肌管细胞中有84个microRNAs存在差异表达,对这些差异表达的microRNA靶基因预测分析得知Leu缺失诱导mTOR-自噬信号通路的变化可能是通过mmu-miR-20a及mmu-miR-106b的变化间接起效应的。廖宇龙[48]在Leu干预mTOR表达对局灶性脑缺血自噬的研究中发现,Leu可以在脑缺血发生后的6~24 h内促进mTOR的表达,在12 h时达到高峰,此时可一定程度上减弱自噬相关蛋白质LC3-Ⅱ的表达。
3.3 谷氨酰胺依赖于mTORC1途径调控细胞自噬Gln是肠道上皮细胞的主要能量来源, 其缺乏可导致肠道黏膜萎缩、绒毛稀疏、屏障功能下降,Zhu等[49]研究发现,培养基Gln缺乏会抑制猪肠道上皮细胞增殖,导致LC3-Ⅱ的表达上调,而mTORC1表达下调;用5 mmol/L Gln补足后发现LC3-Ⅱ的表达下调,mTORC1表达上调,研究结果支持了Gln缺乏诱导自噬并干扰肠道上皮细胞中的氨基酸代谢,以及减弱mTORC1信号通路以抑制蛋白质合成和细胞增殖的观点。Tan等[33]的研究中发现,在氨基酸饥饿时,Gln需要通过自噬重新激活mTORC1, 并且仅补充Gln就足以在长时间的氨基酸饥饿期间恢复mTORC1的活性和溶酶体定位。通过自噬抑制剂处理可使Gln的抑制作用消除,提示部分mTORC1的活化依赖自噬的发生。
4 氨基酸依赖于非mTORC1途径调控细胞自噬 4.1 赖氨酸依赖于非mTORC1途径调控细胞自噬尽管Lys对细胞自噬的调控作用主要是通过mTORC1信号通路介导的,但由于mTORC1通路的作用是有限的,Lys缺乏诱导细胞自噬也可能是通过mTORC1以外的信号分子介导实现的[50]。据报道,蛋白质激酶B(protein kinase B,Akt)通过自噬-溶酶体系统调节蛋白质的降解[51],之前也有研究表明Leu可以通过上调Akt的磷酸化水平来抑制自噬-溶酶体系统的活性[52],但Sato等[42]的研究已充分证实了Lys是另一个能够激活Akt信号通路的氨基酸。Zhao等[53]的研究表明,Akt作为一种调节因子,在肌管细胞自噬发生的过程中不可或缺。因此,Akt的激活可能在Lys调节自噬-溶酶体活性中起关键作用。Sato等[24]用Lys处理C2C12细胞发现Akt的Ser473和Thr308位点磷酸化水平显著升高;当用Akt的特异性抑制剂处理C2C12细胞抑制Akt磷酸化时,Lys对肌原纤维蛋白质降解的抑制作用被消除,证明了Lys可以通过激活Akt从而抑制肌原纤维蛋白质的降解。Sato等[24]还通过LC3-Ⅱ与总LC3的比值评估了Lys诱导的Akt激活对自噬-溶酶体活性的调节作用。在用Lys处理的C2C12细胞中,LC3-Ⅱ与总LC3的比值降低,用Akt抑制剂处理后可以消除这种降低的现象,表明Lys抑制自噬-溶酶体系统的活性依赖于Akt的激活。
4.2 亮氨酸依赖于非mTORC1途径调控细胞自噬尽管Leu对细胞自噬的影响主要是通过mTORC1信号通路实现的,其对细胞蛋白质代谢与周转的作用也依赖于其他信号通路[54],但相关研究报道甚少。为了确定Leu对肌肉细胞自噬的影响是否会通过mTORC1以外的信号通路发挥作用,Mordier等[55]进行了相关试验,将肌管细胞进行3 h的Leu饥饿处理,检测雷帕霉素的作用;将肌管细胞在含50 nmol/L雷帕霉素的培养基中孵育3 h,检测Leu饥饿的作用;结果发现当2种刺激同时施加时会有一个的累加效应。此外,通过8 h的试验对磷酸化S6核糖体蛋白质水平进行检测发现,其表达水平不受Leu饥饿的影响,从而进一步提供了Leu饥饿通过mTORC1非依赖性途径诱导肌肉细胞自噬的证据,表明Leu饥饿可能会通过与mTORC1无关的特定信号通路调节自噬,同时也调节蛋白质的合成。
4.3 精氨酸依赖于非mTORC1途径调控细胞自噬Arg是机体最大的供氮氨基酸, 也是机体脯氨酸、谷氨酸和多胺等物质合成的重要前体,Sarkar等[56]和Angcajas等[57]研究证实,Arg不仅可以通过mTORC1信号通路调节自噬,还可以通过一氧化氮(nitric oxide,NO)信号通路调控自噬。NO是心血管、神经和免疫系统中普遍存在的细胞信号分子,能够引发多种生理反应,如血流调节、组织对缺氧的反应等。Arg是人体内唯一的NO来源,在细胞内,Arg可以被一氧化氮合酶(NO synthases,NOS)降解为NO。Sarkar等[56]的研究表明,NO能够降低Jun氨基末端激酶(Jun N-terminal kinase 1,JNK1)的活性和B淋巴细胞瘤-2(B-cell lymphoma-2,Bcl-2)基因的磷酸化并以IkB激酶β(inhibitor of nuclear factor kappa-B kinase,IKKβ)和结节性硬化症基因2(tuberous sclerosis complex 2,TSC2)依赖性方式抑制哺乳动物细胞中的自噬通量。将大鼠肝癌细胞H4-Ⅱ-E在厄尔平衡盐溶液中饥饿处理,添加Arg后发现LC3-Ⅱ和LC3-Ⅰ比例下降了35%;用Arg和雷帕霉素共同处理后发现LC3-Ⅱ和LC3-Ⅰ比例下降33%,2种情况下对自噬抑制并没有显着性的差异,表明Arg还可能通过其他信号通路抑制自噬,而不完全依赖于mTORC1信号通路[57]。
5 氨基酸调控自噬相关研究中存在的不足尽管国内外诸多关于细胞自噬的研究已经充分表明,营养缺乏,特别是氨基酸缺乏,会导致细胞发生强烈的自噬反应。但关于单个氨基酸,如Lys、Leu、Arg和Gln等是如何响应细胞发生自噬的深层次机制尚不清楚,这也是研究氨基酸营养与细胞自噬的关系需要深入探究的地方。关于不同的氨基酸调控细胞自噬是否存在共性,已有的研究已经表明,Lys对自噬的调控机制可能与Leu相似,Lys和Leu都可以通过mTORC1信号通路调节蛋白质的合成,但是Leu对mTORC1信号传导的刺激作用要明显高于Lys[58],有关Lys和Leu在蛋白质代谢调节中的相似性和差异还需要进一步的研究来阐明。
关于营养物质,特别是氨基酸对自噬影响的研究,早些年就有证实,氨基酸缺乏会诱导细胞发生自噬,但是氨基酸进入胞内是如何发挥作用来缓解细胞自噬,以及在调控自噬的过程中氨基酸究竟是作为一种信号分子还是营养底物来发挥作用的尚不清楚。当细胞内营养物质缺乏时,细胞会启动自噬,降解自身的细胞成分,在短时间内维持细胞在特殊生理状态下合成代谢与分解代谢的平衡[59],但是细胞是如何感应氨基酸的缺乏,进而调控细胞自噬的问题仍然值得我们深入探究。
6 小结氨基酸作为基础营养物质和蛋白质合成代谢与分解代谢的信号分子,在提高机体的免疫力、增强机体的抗应激能力以及维持碳氮平衡等方面发挥着极其重要的作用。诸多关于氨基酸营养的研究多集中在其正向调控,即额外添加氨基酸或维持氨基酸水平对机体或细胞的促进作用,但其反向调控的机制研究却极易被忽视。自噬作为机体的一种重要的负向调控机制,是我们正确认知生命过程,完整阐述科学问题必不可少的一环。研究氨基酸,特别是必需氨基酸对细胞自噬的影响,将有助于我们更加全面地理解精准营养调控,为畜牧生产和医学研究提供理论依据。
| [1] |
ZHANG S J, LI X, LI L, et al. Autophagy up-regulation by early weaning in the liver, spleen and skeletal muscle of piglets[J]. British Journal of Nutrition, 2011, 106(2): 213-217. DOI:10.1017/S0007114511001000 |
| [2] |
KROEMER G, MARIÑO G, LEVINE B. Autophagy and the integrated stress response[J]. Molecular Cell, 2010, 40(2): 280-293. DOI:10.1016/j.molcel.2010.09.023 |
| [3] |
HE C C, KLIONSKY D J. Regulation mechanisms and signaling pathways of autophagy[J]. Annual Review of Genetics, 2009, 43: 67-93. DOI:10.1146/annurev-genet-102808-114910 |
| [4] |
VAINSHTEIN A, TRYON L D, PAULY M, et al. Role of PGC-1α during acute exercise-induced autophagy and mitophagy in skeletal muscle[J]. American Journal of Physiology-Cell Physiology, 2015, 308(9): C710-C719. DOI:10.1152/ajpcell.00380.2014 |
| [5] |
CARROLL B, KOROLCHUK V I, SARKAR S. Amino acids and autophagy:cross-talk and co-operation to control cellular homeostasis[J]. Amino Acids, 2015, 47(10): 2065-2088. DOI:10.1007/s00726-014-1775-2 |
| [6] |
NORRIS K L, YAO T P. Lysine modifications and autophagy[J]. Essays in Biochemistry, 2012, 52: 65-77. DOI:10.1042/bse0520065 |
| [7] |
KAUR J, DEBNATH J. Autophagy at the crossroads of catabolism and anabolism[J]. Nature Reviews Molecular Cell Biology, 2015, 16(8): 461-472. DOI:10.1038/nrm4024 |
| [8] |
MEI Y, THOMPSON M D, COHEN R A, et al. Autophagy and oxidative stress in cardiovascular diseases[J]. Biochimica et Biophysica Acta (BBA):Molecular Basis of Disease, 2015, 1852(2): 243-251. DOI:10.1016/j.bbadis.2014.05.005 |
| [9] |
LIU Y, LEVINE B. Autosis and autophagic cell death:the dark side of autophagy[J]. Cell Death and Differentiation, 2015, 22(3): 367-376. DOI:10.1038/cdd.2014.143 |
| [10] |
MIZUSHIMA N. Methods for monitoring autophagy[J]. International Journal of Biochemistry & Cell Biology, 2004, 36(12): 2491-2502. DOI:10.1016/j.biocel.2004.02.005 |
| [11] |
KLIONSKY D J, ABDALLA F C, ABELIOVICH H, et al. Guidelines for the use and interpretation of assays for monitoring autophagy[J]. Autophagy, 2012, 8(4): 445-544. DOI:10.4161/auto.19496 |
| [12] |
YIN Z Y, PASCUAL C, KLIONSKY D. Autophagy:machinery and regulation[J]. Microbial Cell, 2016, 3(12): 588-596. DOI:10.15698/mic2016.12.546 |
| [13] |
LORIN S, HAMAÏ A, MEHRPOUR M, et al. Autophagy regulation and its role in cancer[J]. Seminars in Cancer Biology, 2013, 23(5): 361-379. DOI:10.1016/j.semcancer.2013.06.007 |
| [14] |
JIANG P D, MIZUSHIMA N. Autophagy and human diseases[J]. Cell Research, 2014, 24(1): 69-79. DOI:10.1038/cr.2013.161 |
| [15] |
RUBINSZTEIN D C, CODOGNO P, LEVINE B. Autophagy modulation as a potential therapeutic target for diverse diseases[J]. Nature Reviews Drug Discovery, 2012, 11(9): 709-730. DOI:10.1038/nrd3802 |
| [16] |
丁渭, 张玉林, 陈德喜. 自噬——凋亡之后的新理念[J]. 中国生物化学与分子生物学报, 2012, 28(5): 419-423. |
| [17] |
SAHU R, KAUSHIK S, CLEMENT C C, et al. Microautophagy of cytosolic proteins by late endosomes[J]. Developmental Cell, 2011, 20(1): 131-139. DOI:10.1016/j.devcel.2010.12.003 |
| [18] |
ORENSTEIN S J, CUERVO A M. Chaperone-mediated autophagy:molecular mechanisms and physiological relevance[J]. Seminars in Cell & Developmental Biology, 2010, 21(7): 719-726. |
| [19] |
MIZUSHIMA N, KOMATSU M. Autophagy:renovation of cells and tissues[J]. Cell, 2011, 147(4): 728-741. DOI:10.1016/j.cell.2011.10.026 |
| [20] |
SHEN H M, MIZUSHIMA N. At the end of the autophagic road:an emerging understanding of lysosomal functions in autophagy[J]. Trends in Biochemical Sciences, 2014, 39(2): 61-71. DOI:10.1016/j.tibs.2013.12.001 |
| [21] |
KLIONSKY D J, CODOGNO P. The mechanism and physiological function of macroautophagy[J]. Journal of Innate Immunity, 2013, 5(5): 427-433. DOI:10.1159/000351979 |
| [22] |
YAO Q, KE Z Q, GUO S, et al. Curcumin protects against diabetic cardiomyopathy by promoting autophagy and alleviating apoptosis[J]. Journal of Molecular and Cellular Cardiology, 2018, 124: 26-34. DOI:10.1016/j.yjmcc.2018.10.004 |
| [23] |
LONG B S, YIN C, FAN Q W, et al. Global liver proteome analysis using iTRAQ reveals AMPK-mTOR-autophagy signaling is altered by intrauterine growth restriction in newborn piglets[J]. Journal of Proteome Research, 2016, 15(4): 1262-1273. DOI:10.1021/acs.jproteome.6b00001 |
| [24] |
SATO T, ITO Y, NAGASAWA T. Regulation of skeletal muscle protein degradation and synthesis by oral administration of lysine in rats[J]. Journal of Nutritional Science and Vitaminology, 2013, 59(5): 412-419. DOI:10.3177/jnsv.59.412 |
| [25] |
JEWELL J L, KIM Y C, RUSSELL R C, et al. Differential regulation of mTORC1 by leucine and glutamine[J]. Science, 2015, 347(6218): 194-198. DOI:10.1126/science.1259472 |
| [26] |
ABEDIN M J, WANG D, MCDONNELL M A, et al. Autophagy delays apoptotic death in breast cancer cells following DNA damage[J]. Cell Death & Differentiation, 2007, 14(3): 500-510. |
| [27] |
WU H, WANG F L, HU S L, et al. MiR-20a and miR-106b negatively regulate autophagy induced by leucine deprivation via suppression of ULK1 expression in C2C12 myoblasts[J]. Cellular Signalling, 2012, 24(11): 2179-2186. DOI:10.1016/j.cellsig.2012.07.001 |
| [28] |
RAUTOU P E, MANSOURI A, LEBREC D, et al. Autophagy in liver diseases[J]. Journal of Hepatology, 2010, 53(6): 1123-1134. DOI:10.1016/j.jhep.2010.07.006 |
| [29] |
REN W K, CHEN S, YIN J, et al. Dietary arginine supplementation of mice alters the microbial population and activates intestinal innate immunity[J]. The Journal of Nutrition, 2014, 144(6): 988-995. DOI:10.3945/jn.114.192120 |
| [30] |
KUNG H J, CHANGOU C A, LI C F, et al. Chromatophagy:autophagy goes nuclear and captures broken chromatin during arginine-starvation[J]. Autophagy, 2015, 11(2): 419-421. DOI:10.1080/15548627.2015.1009789 |
| [31] |
XIA X J, GAO Y, ZHANG J, et al. Autophagy mediated by arginine depletion activation of the nutrient sensor GCN2 contributes to interferon-γ-induced malignant transformation of primary bovine mammary epithelial cells[J]. Cell Death Discovery, 2016, 2: 15065. DOI:10.1038/cddiscovery.2015.65 |
| [32] |
WU G, BAZER F W, JOHNSON G A, et al. Triennial growth symposium:important roles for L-glutamine in swine nutrition and production[J]. Journal of Animal Science, 2011, 89(7): 2017-2030. DOI:10.2527/jas.2010-3614 |
| [33] |
TAN HAYDEN W S, SIM A Y L, LONG Y C. Glutamine metabolism regulates autophagy-dependent mTORC1 reactivation during amino acid starvation[J]. Nature Communications, 2017, 8(1): 338. DOI:10.1038/s41467-017-00369-y |
| [34] |
朱玉华, 薛欣合, 周天骄, 等.谷氨酰胺、精氨酸和亮氨酸缺乏诱导猪肠道上皮细胞自噬的研究[C]//中国畜牧兽医学会动物营养学分会第十一次全国动物营养学术研讨会论文集.长沙: 中国畜牧兽医学会, 2012.
|
| [35] |
YAN X H, SUN Q M, JI J, et al. Reconstitution of leucine-mediated autophagy via the mTORC1-Barkor pathway in vitro[J]. Autophagy, 2012, 8(2): 213-221. DOI:10.4161/auto.8.2.18563 |
| [36] |
LAPLANTE M, SABATINI D M. mTOR signaling in growth control and disease[J]. Cell, 2012, 149(2): 274-293. DOI:10.1016/j.cell.2012.03.017 |
| [37] |
MIZUSHIMA N. The role of the Atg1/ULK1 complex in autophagy regulation[J]. Current Opinion in Cell Biology, 2010, 22(2): 132-139. DOI:10.1016/j.ceb.2009.12.004 |
| [38] |
TAN V P, MIYAMOTO S. Nutrient-sensing mTORC1:integration of metabolic and autophagic signals[J]. Journal of Molecular and Cellular Cardiology, 2016, 95: 31-41. DOI:10.1016/j.yjmcc.2016.01.005 |
| [39] |
JUNG C H, RO S H, CAO J, et al. mTOR regulation of autophagy[J]. FEBS Letters, 2010, 584(7): 1287-1295. DOI:10.1016/j.febslet.2010.01.017 |
| [40] |
SATO T, ITO Y, NEDACHI T, et al. Lysine suppresses protein degradation through autophagic-lysosomal system in C2C12 myotubes[J]. Molecular and Cellular Biochemistry, 2014, 391(1/2): 37-46. |
| [41] |
LANG S M, KAZI A A, HONG-BROWN L, et al. Delayed recovery of skeletal muscle mass following hindlimb immobilization in mTOR heterozygous mice[J]. PLoS One, 2012, 7(6): e38910. DOI:10.1371/journal.pone.0038910 |
| [42] |
SATO T, ITO Y, NAGASAWA T. Lysine suppresses myofibrillar protein degradation by regulating the autophagic-lysosomal system through phosphorylation of Akt in C2C12 cells[J]. Springerplus, 2014, 3: 584. DOI:10.1186/2193-1801-3-584 |
| [43] |
DODD K M, TEE A R. Leucine and mTORC1:a complex relationship[J]. American Journal of Physiology-Endocrinology and Metabolism, 2012, 302(11): E1329-E1342. DOI:10.1152/ajpendo.00525.2011 |
| [44] |
晏向华.猪自噬发生及营养调控[C]//中国畜牧兽医学会动物营养分会第七届中国猪营养学术研讨会论文集.重庆: 中国畜牧兽医学会, 2015.
|
| [45] |
ESCOBAR J, FRANK J W, SURYAWAN A, et al. Physiological rise in plasma leucine stimulates muscle protein synthesis in neonatal pigs by enhancing translation initiation factor activation[J]. American Journal of Physiology-Endocrinology and Metabolism, 2005, 288(5): E914-E921. DOI:10.1152/ajpendo.00510.2004 |
| [46] |
YOSHIZAWA F. Regulation of protein synthesis by branched-chain amino acids in vivo[J]. Biochemical and Biophysical Research Communications, 2004, 313(2): 417-422. DOI:10.1016/j.bbrc.2003.07.013 |
| [47] |
吴浩, 晏向华.参与亮氨酸缺失诱导mTOR-自噬信号通路的microRNA的挖掘与功能分析[C]//第六次全国饲料营养学术研讨会论文集.杨凌: 中国畜牧兽医学会, 2010.
|
| [48] |
廖玉龙.亮氨酸干预mTOR表达对局灶性脑缺血自噬的影响[D].硕士学位论文.南昌: 南昌大学, 2016.
|
| [49] |
ZHU Y H, LIN G, DAI Z D, et al. L-glutamine deprivation induces autophagy and alters the mTOR and MAPK signaling pathways in porcine intestinal epithelial cells[J]. Amino Acids, 2015, 47(10): 2185-2197. DOI:10.1007/s00726-014-1785-0 |
| [50] |
SATO T, ITO Y, NAGASAWA T. Dietary L-lysine suppresses autophagic proteolysis and stimulates Akt/mTOR signaling in the skeletal muscle of rats fed a low-protein diet[J]. Journal of Agricultural and Food Chemistry, 2015, 63(37): 8192-8198. DOI:10.1021/acs.jafc.5b03811 |
| [51] |
MAMMUCARI C, MILAN G, ROMANELLO V, et al. FoxO3 controls autophagy in skeletal muscle in vivo[J]. Cell Metabolism, 2007, 6(6): 458-471. DOI:10.1016/j.cmet.2007.11.001 |
| [52] |
ZEANANDIN G, BALAGE M, SCHNEIDER S M, et al. Differential effect of long-term leucine supplementation on skeletal muscle and adipose tissue in old rats:an insulin signaling pathway approach[J]. AGE, 2012, 34(2): 371-387. DOI:10.1007/s11357-011-9246-0 |
| [53] |
ZHAO J H, BRAULT J J, SCHILD A, et al. FoxO3 coordinately activates protein degradation by the autophagic/lysosomal and proteasomal pathways in atrophying muscle cells[J]. Cell Metabolism, 2007, 6(6): 472-483. DOI:10.1016/j.cmet.2007.11.004 |
| [54] |
CROZIER S J, KIMBALL S R, EMMERT S W, et al. Oral leucine administration stimulates protein synthesis in rat skeletal muscle[J]. The Journal of Nutrition, 2005, 135(3): 376-382. DOI:10.1093/jn/135.3.376 |
| [55] |
MORDIER S, DEVAL C, BÉCHET D, et al. Leucine limitation induces autophagy and activation of lysosome-dependent proteolysis in C2C12 myotubes through a mammalian target of rapamycin-independent signaling pathway[J]. The Journal of Biological Chemistry, 2000, 275(38): 29900-29906. DOI:10.1074/jbc.M003633200 |
| [56] |
SARKAR S, KOROLCHUK V I, RENNA M, et al. Complex inhibitory effects of nitric oxide on autophagy[J]. Molecular Cell, 2011, 43(1): 19-32. DOI:10.1016/j.molcel.2011.04.029 |
| [57] |
ANGCAJAS A B, HIRAI N, KANESHIRO K, et al. Diversity of amino acid signaling pathways on autophagy regulation:a novel pathway for arginine[J]. Biochemical and Biophysical Research Communications, 2014, 446(1): 8-14. DOI:10.1016/j.bbrc.2014.01.117 |
| [58] |
MARTINET W, DE MEYER G R Y, HERMAN A G, et al. Amino acid deprivation induces both apoptosis and autophagy in murine C2C12 muscle cells[J]. Biotechnology Letters, 2005, 27(16): 1157-1163. DOI:10.1007/s10529-005-0007-y |
| [59] |
RUSSELL R C, YUAN H X, GUAN K L. Autophagy regulation by nutrient signaling[J]. Cell Research, 2014, 24(1): 42-57. DOI:10.1038/cr.2013.166 |




