长链非编码RNA(lncRNAs)是一类长度大于200 nt的转录本,主要由RNA聚合酶Ⅱ转录,一般具有5′端帽子和3′端多聚腺苷酸尾巴,能进行可变剪切[1-2]。lncRNAs广泛存在于哺乳动物中,其表达具有时空特异性和组织特异性,在动物几乎所有的生命过程中扮演重要角色。目前,lncRNAs的研究主要集中在人类和小鼠等模式生物中,在畜牧业和家禽研究中尚处于起步阶段。本文就lncRNAs的结构功能、调控机制及在家禽生产中的研究进展进行了综述。
1 lncRNAs分类2002年,lncRNAs的概念由日本学者Okazaki等在小鼠全长cDNA文库大规模测序中首次进行了描述[3-4]。DNA元素百科全书(ENCODE)的研究表明,人类基因组序列中约70%被转录,其中仅有不到2%的转录本具有蛋白质编码的能力,98%是没有编码能力的非编码RNA(ncRNAs)[5-7]。根据其功能,可将ncRNAs分为看家ncRNAs和调节ncRNAs[8]。看家ncRNAs通常稳定表达,对细胞存活发挥至关重要的功能,主要包括核糖体RNA、转运RNA及小核仁RNA等;调节ncRNAs主要分为lncRNAs和微小RNA(microRNAs)等[9]。其中,lncRNAs占ncRNAs的80%,能够在表观遗传、转录及转录后等多个层面调控基因的表达过程[10]。
lncRNAs具有数量多、种类多、作用方式多的特点[11]。目前,lncRNAs尚没有统一的分类标准。Laurent等[12]指出,lncRNAs分类的主要依据是分子大小、定位和功能。根据lncRNAs在细胞中的定位,可以分为细胞质lncRNAs和细胞核lncRNAs,有些既位于细胞核又存在于细胞质中,lncRNAs根据其不同的细胞定位可能发挥不同的调控功能[13]。根据lncRNAs在基因组中相对蛋白质编码基因的位置,可以分为5种类型,分别是正义lncRNAs(与具有编码能力的基因外显子区域重叠)、反义lncRNAs(转录本起始于蛋白质编码基因的逆转录)、双向lncRNAs(其表达起始位点与其反义链上相邻编码基因的表达起始位点十分靠近)、基因内lncRNAs(转录本来源于内含子区域)、基因间lncRNAs(位于基因组上2个基因的间隔区)[14]。根据分子功能,lncRNAs分为诱饵分子、引导分子和骨架分子等(图 1)。诱饵分子能够诱导转录因子与其相结合,抑制其下游基因的转录[15];引导分子能与其具有相同位点的分子(DNA或蛋白质)相结合,将其引导到作用位点,增强基因的转录活性[16];骨架分子作为蛋白质复合物的脚手架,与目的蛋白形成核酸-蛋白质复合物,将参与表观遗传的酶分子联合起来共同发挥作用,参与基因的转录调控[17-18]。
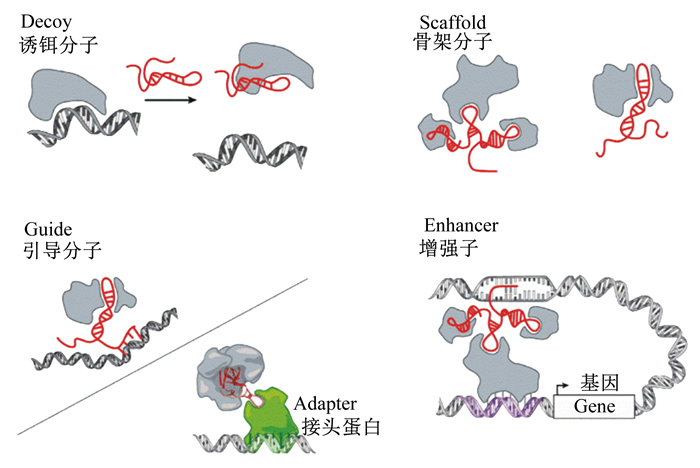
|
图 1 lncRNAs不同的作用机制模型 Fig. 1 Models of different mechanisms of action for lncRNAs[19] |
lncRNAs广泛分布在各种生物中,与信使RNA(mRNA)相比,lncRNAs表达水平较低且保守性极低,因此,lncRNAs最初被认为是基因组的转录“噪音”,没有任何功能[20-22]。但随后研究发现,lncRNAs具有特定的时空表达模式,仅在个体特定的生长发育阶段、特定的组织、特定的细胞类型中表达,表明lncRNAs在多细胞生物体的细胞生长分化过程中发挥不可替代的功能[14]。分子结构决定其功能,lncRNAs的序列特异性和高级结构的复杂性决定了它与DNA、RNA以及蛋白质分子间都存在互作关系,几乎能影响基因所有阶段的表达水平[23]。lncRNAs能够参与多种水平的调控,比如表观遗传修饰,转录及转录后水平的基因调控[10, 24]。
2.1 lncRNAs与表观遗传调控表观遗传是指基因组DNA序列不发生改变,而单基因的表达发生可遗传性改变,主要包括DNA甲基化、组蛋白修饰(甲基化、乙酰化、磷酸化等)、基因组印迹等。lncRNAs也可招募与染色质修饰相关的DNA或蛋白质复合物到相邻的基因,通过顺式或反式作用调控邻近基因的表达。lncRNAs调控染色质功能的经典例子是胚胎发育早期X染色体失活转录X染色体失活特异性转录本基因(Xist),它也是最早被发现的lncRNAs[25]。Xist的表达招募多梳抑制复合物2(PRC2)促进组蛋白H3的27位赖氨酸三甲基化(H3K27me3),从而导致X染色体的失活[26-28]。人lncRNA HOTAIR也通过类似的机制发挥调控作用,最终抑制同源基因(HOX)的表达[17-29]。lncRNAs也能够通过直接结合染色质从而影响其构象。同时,lncRNAs还能够参与基因组印迹[30]。
2.2 lncRNA与转录调控转录调控是真核基因表达调控的重要组成,也是基因表达最重要的调控模式。lncRNAs通过很多不同的机制调控转录过程,如图 2所示。
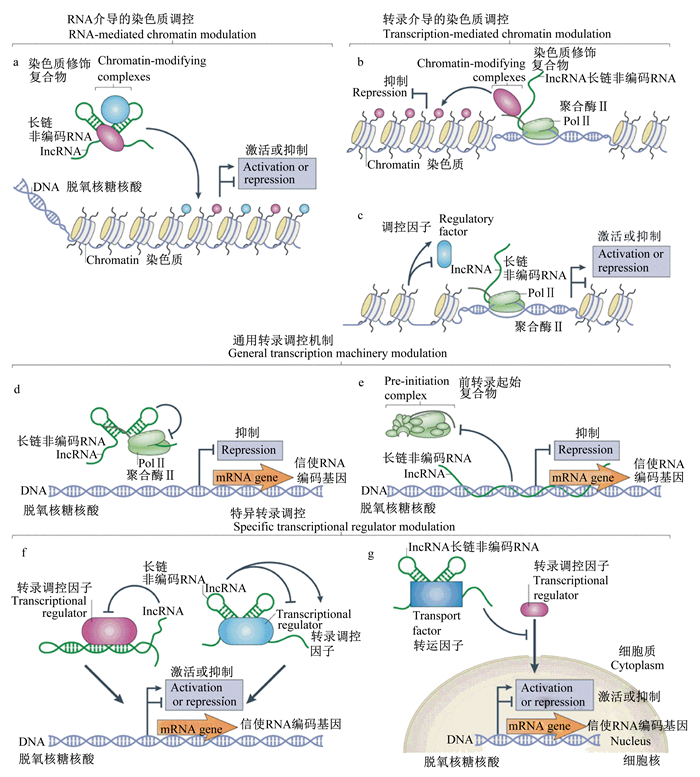
|
图 2 lncRNAs以不同的方式调控转录过程 Fig. 2 lncRNAs regulate transcription through different mechanisms[31] |
例如,lncRNAs能够结合调控序列形成稳定的DNA-RNA三重复合物从而抑制转录,为大家熟知的是人类二氢叶酸还原酶(DHFR)的例子。DH-FR基因的上游启动子能够转录出1条lncRNA,转录出的lncRNA通过与启动子的DNA序列形成lncRNA-DNA复合物抑制基因的转录[32]。
2.3 lncRNAs与转录后调控lncRNAs通过序列的互补结合调控基因的转录后过程,具体包括mRNA的降解或mRNA稳定性的维持;前体mRNA不同剪切位点的选择(可变剪切);作为分子“海绵”对miRNA的吸收及对翻译过程的调控等。lncRNAs可参与mRNA的稳定并调控翻译过程。mRNA的3′非编码区(3′UTR)可被miRNA降解,反义lncRNAs通过结合mRNA的3′UTR位点形成保护机制。例如,β-淀粉样蛋白转换酶1(BACE1)基因的反义转录本1-AS能够结合BACE1的mRNA形成稳定的RNA双链结构,从而阻断miRNA介导的对mRNA降解识别位点的结合,阻止其mRNA的降解,从而导致BACE1蛋白表达上调。mRNA的降解速度和转录本的数量决定了mRNA的含量,从而影响蛋白质的产量。lncRNAs可以和mRNA互补结合介导其降解。最广为熟知的例子是RNA结合蛋白(STAU1)介导的mRNA降解。STAU1通过招募无义介导的mRNA降解因子(NMD)到mRNA的3′UTR,导致mRNA的降解。细胞质lncRNAs可作为miRNA海绵和蛋白海绵发挥作用。作为miRNA海绵的lncRNAs称为内源竞争性RNA(ceRNA),ceRNA通过竞争性结合miRNA,降低miRNA对目标mRNA的影响,从而正向调控靶基因的表达。同样的,作为蛋白海绵的lncRNA通过与靶基因的mRNA竞争性结合某一类蛋白,反向调节靶基因的表达。
3 lncRNAs在家禽经济性状发育中的调节功能作为基因调控网络的重要调控因子,lncRNAs吸引了越来越多的关注。lncRNAs数量多、分布广,且在表观遗传、转录和转录后等多个水平参与基因表达的调控,已成为生命科学领域的研究热点之一[14]。但是,目前lncRNAs的研究主要集中于人类疾病、小鼠和植物等模式生物,在畜牧学领域与家养动物有关的lncRNAs研究尚处于刚起步阶段。相对于mRNA和microRNA,lncRNAs物种间保守性极低,很难从已有模式生物的研究中直接借鉴[33]。在模式物种中已建立的大多数生物信息学分析方法也不能很好的用于家养动物lncRNAs的分析。家养动物养殖成本高、个体差异大,开展动物机体或原代细胞水平的研究成本巨大。lncRNAs的表达具有很强的时空特异性,极易降解,研究其空间结构的工具匮乏,很难捕获其表达的动态变化过程,目前还没有家养动物的lncRNAs数据库[34]。在畜牧领域(家禽)lncRNAs的功能研究主要侧重于对家禽肌肉发育、脂肪沉积和繁殖等重要的经济性状的关注[35-37]。
3.1 lncRNAs调控家禽肌肉发育肉鸡是全世界饲养数量最多的农场动物,骨骼肌的发育是其最重要的经济性状之一[38-39]。在人类疾病的研究中,lncRNAs与杜氏肌肉营养不良症(DMD)和面肩肱型肌营养不良症(FSHD)这2种肌肉发育疾病的发生密不可分[40-42]。在鸡肌肉发育的研究中,第1个系统性的lncRNAs研究是Li等[43]采用高通量RNA-Seq对鸡骨骼肌转录组的检测,共鉴定到281个lncRNAs。随后有一些对鸡特定lncRNAs调控功能的研究报道,pouMU1是鸡骨骼肌组织最大量表达的lncRNA,而在其他组织中低表达或不表达。胸肌pouMU1的表达量在开始研究的第1天比第6周和第16周显著上升,这提示pouMU1基因在鸡的早期发育阶段发挥重要的调控作用[44]。有些研究对肌肉lncRNAs发挥作用的调控机制作了进一步研究,Cai等[45]在隐性白洛克鸡和杏花鸡差异表达的lncRNAs中发现了lncRNA-Six1,它在鸡胸肌中大量表达,可编码产生1个小肽,过表达lncRNA-Six1会促进靶基因Six1 mRNA和蛋白的表达,而敲除lncRNA-Six1会抑制Six1的表达,参与肌肉的生长发育的调控,但具体作用机制仍需深入的研究。
3.2 lncRNAs调控家禽脂肪沉积腹部脂肪是肉鸡的另一个重要的胴体性状。过去10多年,由于遗传育种和营养改良措施过度追求家禽生长速度,导致其腹部脂肪的沉积,胴体质量下降[46]。之前有研究表明,lncRNAs能够调控肉牛和猪的脂肪生成及与代谢相关的过程[47-48]。对于lncRNAs调控鸡前体脂肪细胞分化的机制研究处在起步阶段。在最近的研究中,Zhang等[49]采用RNA-Seq技术分析了鸡前体脂肪细胞lncRNAs的表达特性,发现了1 336个特异表达的lncRNAs,为进一步研究鸡的脂肪细胞分化提供了依据。Wang等[50]研究了高能量饲粮对鹅肝脏脂肪沉积的影响,与对照组相比,高能量饲粮组共筛选到302个差异表达的lncRNAs。其中,lncRNA XLOC_292762在高能量饲粮组显著下调,它位于fermitin家族同源蛋白2(FERMT2)基因下游,FERMT2基因的功能为参与脂代谢过程中3, 4, 5-三磷酸磷脂酰肌醇的结合。该结果表明,lncRNAs在鹅肝脏的脂代谢中发挥重要调控功能。
3.3 lncRNAs调控家禽繁殖在研究人类疾病和模式动物繁殖的研究中,已证实lncRNAs参与胚胎早期生殖细胞的形成,早期胚胎植入及相关的激素调控过程。精子活力是评价公鸡繁殖能力的最重要指示。研究人员研究采用RNA-Seq技术分析北京油公鸡(精子活力高、低2组)睾丸转录组,在公鸡的睾丸中共鉴定了2 597个lncRNAs(124个差异表达的lncRNAs)。动力蛋白重链5(DNAH5)是精子运动的一个重要马达蛋白,DNAH5基因突变可能引发精子的失活。通过软件预测,lncRNA MSTRG.4081作用的靶基因是LOC428510(LOC428510和DNAH5基因结构类似),与精子活力密切相关。另有研究发现,lncRNA MHM可调控鸡的胚胎发育和性腺发生,下调lncRNA MHM表达可引起母鸡卵巢发育不对称;双性基因(DMRT1)家族的主要功能是参与性别决定与分化,在雄性中,lncRNA MHM表达异常损坏睾丸DMRT1基因在性腺的表达,引发雄性胚胎致死率的增加[51]。
4 小结作为基因调控网络的主要调控因子,lncRNAs被越来越多的研究者所关注[52-53]。在畜牧学领域,鸡lncRNAs的研究相对滞后,大部分已开展的工作基本停留在特定组织或特定发育阶段lncRNAs的发现和鉴定上,对其作用机制缺乏更进一步研究。对已鉴定的特定lncRNAs,尤其与家禽重要经济性状相关的lncRNAs系统的研究和功能挖掘仍是一个亟待解决的科研问题[54-55]。因此,迫切需要构建家禽完整的lncRNAs数据库,同时需要分子生物学、动物营养学、环境卫生学、家禽遗传与分子育种学、生物信息学等多学科不断的交叉融合,将不断发展的生物学研究技术和手段应用于家禽经济性状的lncRNAs研究中。未来很长一段时间内,如何进一步研究lncRNAs在家禽重要经济性状中的功能将是畜牧工作者需要解决的问题。
| [1] |
GUTTMAN M, RINN L. Modular regulatory principles of large non-coding RNAs[J]. Nature, 2012, 482(7385): 339-346. DOI:10.1038/nature10887 |
| [2] |
CABILI M N, TRAPNELL C, GOFF L, et al. Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses[J]. Genes & Development, 2011, 25(18): 1915-1927. |
| [3] |
KAPRANOV P, CHENG J, DIKE S, et al. RNA maps reveal new RNA classes and a possible function for pervasive transcription[J]. Science, 2007, 316(5830): 1484-1488. DOI:10.1126/science.1138341 |
| [4] |
The FANTOM Consortium and the RIKEN Genome Exploration Research Group Phase Ⅰ & Ⅱ Team. Analysis of the mouse transcriptome based on functional annotation of 60, 770 full-length cDNAs[J]. Nature, 2002, 420(6915): 563-573. DOI:10.1038/nature01266 |
| [5] |
The ENCODE Project Consortium. Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project[J]. Nature, 2007, 447(7146): 799-816. DOI:10.1038/nature05874 |
| [6] |
FATICA A, BOZZONI I. Long non-coding RNAs:new players in cell differentiation and development[J]. Nature Reviews Genetics, 2014, 15(1): 7-21. DOI:10.1038/nrg3606 |
| [7] |
WILUSZ J E, SUNWOO H, SPECTOR D L. Long noncoding RNAs:functional surprises from the RNA world[J]. Genes & Development, 2009, 23(13): 1494-1504. |
| [8] |
CARTHEW R W, SONTHEIMER E J. Origins and mechanisms of miRNAs and siRNAs[J]. Cell, 2009, 136(4): 642-655. DOI:10.1016/j.cell.2009.01.035 |
| [9] |
MALONE C D, HANNON G J. Small RNAs as guardians of the genome[J]. Cell, 2009, 136(4): 656-668. DOI:10.1016/j.cell.2009.01.045 |
| [10] |
WAPINSKI O, CHANG H Y. Long noncoding RNAs and human disease[J]. Trends in Cell Biology, 2011, 21(6): 354-361. DOI:10.1016/j.tcb.2011.04.001 |
| [11] |
BOLHA L, RAVNIK-GLAVA Č M, GLAVA Č D. Long noncoding RNAs as biomarkers in cancer[J]. Disease Markers, 2017, 2017: 7243968. |
| [12] |
LAURENT G, WAHLESTEDT C, KAPRANOV P. The landscape of long noncoding RNA classification[J]. Trends in Genetics, 2015, 31(5): 239-251. DOI:10.1016/j.tig.2015.03.007 |
| [13] |
GHOSAL S, DAS S, CHAKRABARTI J. Long noncoding RNAs: new players in the molecular mechanism for maintenance and differentiation of pluripotent stem cells[J]. Stem Cells and Development, 2013, 22(16): 2240-2253. DOI:10.1089/scd.2013.0014 |
| [14] |
PONTING C P, OLIVER P L, REIK W. Evolution and functions of long noncoding RNAs[J]. Cell, 2009, 136(4): 629-641. DOI:10.1016/j.cell.2009.02.006 |
| [15] |
HUNG T, WANG Y L, LIN M F, et al. Extensive and coordinated transcription of noncoding RNAs within cell-cycle promoters[J]. Nature Genetics, 2011, 43(7): 621-629. DOI:10.1038/ng.848 |
| [16] |
GUPTA R A, NILAY S, WANG K C, et al. Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis[J]. Nature, 2010, 464(7291): 1071-1076. DOI:10.1038/nature08975 |
| [17] |
TSAI M C, MANOR O, WAN Y, et al. Long noncoding RNA as modular scaffold of histone modification complexes[J]. Science, 2010, 329(5992): 689-693. DOI:10.1126/science.1192002 |
| [18] |
SPITALE R C, MIAO-CHIH T, CHANG H Y. RNA templating the epigenome:long noncoding RNAs as molecular scaffolds[J]. Epigenetics, 2011, 6(5): 539-543. DOI:10.4161/epi.6.5.15221 |
| [19] |
RINN J L, CHANG H Y. Genome regulation by long noncoding RNAs[J]. Annual Review of Biochemistry, 2012, 81: 145-166. DOI:10.1146/annurev-biochem-051410-092902 |
| [20] |
PANG K C, FRITH M C, MATTICK J S. Rapid evolution of noncoding RNAs:lack of conservation does not mean lack of function[J]. Trends in Genetics, 2006, 22(1): 1-5. DOI:10.1016/j.tig.2005.10.003 |
| [21] |
KUNG J T Y, COLOGNORI D, LEE J T. Long noncoding RNAs:past, present, and future[J]. Genetics, 2013, 193(3): 651-669. DOI:10.1534/genetics.112.146704 |
| [22] |
ORGEL L E, CRICK F H. Selfish DNA:the ultimate parasite[J]. Nature, 1980, 284(5757): 604-607. |
| [23] |
BEVILACQUA P C, RITCHEY L E, SU Z, et al. Genome-wide analysis of RNA secondary structure[J]. Annual Review of Genetics, 2016, 50: 235-266. DOI:10.1146/annurev-genet-120215-035034 |
| [24] |
LI Z, REN T, LI W. Regulatory mechanism and application of lncRNAs in poultry[M]. [s.l.]: [s.n.], 2019.
|
| [25] |
GENDREL A V, HEARD E. Noncoding RNAs and epigenetic mechanisms during X-chromosome inactivation[J]. Annual Review of Cell and Developmental Biology, 2014, 30(1): 561-580. DOI:10.1146/annurev-cellbio-101512-122415 |
| [26] |
MIGEON B R. X-chromosome inactivation:molecular mechanisms and genetic consequences[J]. Trends in Genetics, 1994, 10(7): 230-235. DOI:10.1016/0168-9525(94)90169-4 |
| [27] |
MELLER V H, JOSHI S S, DESHPANDE N. Modulation of chromatin by noncoding RNA[J]. Annual Review of Genetics, 2015, 49: 673-695. DOI:10.1146/annurev-genet-112414-055205 |
| [28] |
DENG X X, BERLETCH J B, NGUYEN D K, et al. X chromosome regulation:diverse patterns in development, tissues and disease[J]. Nature Reviews Genetics, 2014, 15(6): 367-378. DOI:10.1038/nrg3687 |
| [29] |
RINN J L, KERTESZ M, WANG J K, et al. Functional demarcation of active and silent chromatin domains in human HOX loci by noncoding RNAs[J]. Cell, 2007, 129(7): 1311-1323. DOI:10.1016/j.cell.2007.05.022 |
| [30] |
BARLOW D P. Genomic imprinting:a mammalian epigenetic discovery model[J]. Annual Review of Genetics, 2011, 45: 379-403. DOI:10.1146/annurev-genet-110410-132459 |
| [31] |
GEISLER S, COLLER J. RNA in unexpected places:long non-coding RNA functions in diverse cellular contexts[J]. Nature Reviews Molecular Cell Biology, 2013, 14(11): 699-712. DOI:10.1038/nrm3679 |
| [32] |
MARTIANOV I, RAMADASS A, BARROS A S, et al. Repression of the human dihydrofolate reductase gene by a non-coding interfering transcript[J]. Nature, 2007, 445(7128): 666-670. DOI:10.1038/nature05519 |
| [33] |
EDDY S R. Computational genomics of noncoding RNA genes[J]. Cell, 2002, 109(2): 137-140. DOI:10.1016/S0092-8674(02)00727-4 |
| [34] |
ZHOU R, WANG Y X, LONG K R, et al. Regulatory mechanism for lncRNAs in skeletal muscle development and progress on its research in domestic animals[J]. Hereditas, 2018, 40(4): 292-304. |
| [35] |
魏彩虹, 吴明明, 刘瑞凿, 等. 肌肉发育相关LncRNA的研究进展[J]. 中国农业科学, 2014, 47(20): 4078-4085. DOI:10.3864/j.issn.0578-1752.2014.20.016 |
| [36] |
杨兵, 李晓凤, 王昕. 长链非编码RNA在畜禽经济性状中的研究进展[J]. 畜牧兽医学报, 2018, 49(10): 2063-2069. DOI:10.11843/j.issn.0366-6964.2018.10.001 |
| [37] |
占思远, 李利, 王林杰, 等. 骨骼肌发育调控相关lncRNAs研究进展[J]. 畜牧兽医学报, 2016, 47(4): 637-644. |
| [38] |
STERN C D. The chick:a great model system becomes even greater[J]. Developmental Cell, 2005, 8(1): 9-17. |
| [39] |
HAYES B J, LEWIN H A, GODDARD M E. The future of livestock breeding:genomic selection for efficiency, reduced emissions intensity, and adaptation[J]. Trends in Genetics, 2013, 29(4): 206-214. DOI:10.1016/j.tig.2012.11.009 |
| [40] |
CABIANCA D S, CASA V, BODEGA B, et al. A long ncRNA links copy number variation to a polycomb/trithorax epigenetic switch in FSHD muscular dystrophy[J]. Cell, 2012, 149(4): 819-831. DOI:10.1016/j.cell.2012.03.035 |
| [41] |
CESANA M, CACCHIARELLI D, LEGNINI I, et al. A long noncoding RNA controls muscle differentiation by functioning as a competing endogenous RNA[J]. Cell, 2011, 147(2): 358-369. DOI:10.1016/j.cell.2011.09.028 |
| [42] |
WATTS R, JOHNSEN V L, SHEARER J, et al. Myostatin-induced inhibition of the long noncoding RNA Malat1 is associated with decreased myogenesis[J]. American Journal of Physiology:Cell Physiology, 2013, 304(10): C995-C1001. DOI:10.1152/ajpcell.00392.2012 |
| [43] |
LI T T, WANG S Y, WU R M, et al. Identification of long non-protein coding RNAs in chicken skeletal muscle using next generation sequencing[J]. Genomics, 2012, 99(5): 292-298. DOI:10.1016/j.ygeno.2012.02.003 |
| [44] |
REN T H, ZHOU Y T, ZHOU Y, et al. Identification and association of novel lncRNA pouMU1 gene mutations with chicken performance traits[J]. Journal of Genetics, 2017, 96(6): 941-950. DOI:10.1007/s12041-017-0858-8 |
| [45] |
CAI B L, LI Z H, MA M T, et al. LncRNA-Six1 encodes a micropeptide to activate Six1 in cis and is involved in cell proliferation and muscle growth[J]. Frontiers in Physiology, 2017, 8: 230. DOI:10.3389/fphys.2017.00230 |
| [46] |
DONG J Q, ZHANG H, JIANG X F, et al. Comparison of serum biochemical parameters between two broiler chicken lines divergently selected for abdominal fat content[J]. Journal of Animal Science, 2015, 93(7): 3278-3286. DOI:10.2527/jas.2015-8871 |
| [47] |
SUN L, GOFF L A, TRAPNELL C, et al. Long noncoding RNAs regulate adipogenesis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(9): 3387-3392. DOI:10.1073/pnas.1222643110 |
| [48] |
LI M X, SUN X M, CAI H F, et al. Long non-coding RNA ADNCR suppresses adipogenic differentiation by targeting miR-204[J]. Biochimica et Biophysica Acta:Gene Regulatory Mechanisms, 2016, 1859(7): 871-882. DOI:10.1016/j.bbagrm.2016.05.003 |
| [49] |
ZHANG T, ZHANG X Q, HAN K P, et al. Analysis of long noncoding RNA and mRNA using RNA sequencing during the differentiation of intramuscular preadipocytes in chicken[J]. PLoS One, 2017, 12(2): e0172389. DOI:10.1371/journal.pone.0172389 |
| [50] |
WANG G S, JIN L, LI Y, et al. Transcriptomic analysis between normal and high-intake feeding geese provides insight into adipose deposition and susceptibility to fatty liver in migratory birds[J]. BMC Genomics, 2019, 20: 372. DOI:10.1186/s12864-019-5765-3 |
| [51] |
ROESZLER K N, ITMAN C, SINCLAIR A H, et al. The long non-coding RNA, MHM, plays a role in chicken embryonic development, including gonadogenesis[J]. Developmental Biology, 2012, 366(2): 317-326. DOI:10.1016/j.ydbio.2012.03.025 |
| [52] |
CHAN W L, HUANG H D, CHANG J G. lncRNA map:a map of putative regulatory functions in the long non-coding transcriptome[J]. Computational Biology and Chemistry, 2014, 50: 41-49. DOI:10.1016/j.compbiolchem.2014.01.003 |
| [53] |
IWAKIRI J, HAMADA M, ASAI K. Bioinformatics tools for lncRNA research[J]. Biochimica et Biophysica Acta:Gene Regulatory Mechanisms, 2016, 1859(1): 23-30. DOI:10.1016/j.bbagrm.2015.07.014 |
| [54] |
MCCLOSKEY J A, CRAIN P F. The RNA modification database-1998[J]. Nucleic Acids Research, 1998, 26(1): 196-197. DOI:10.1093/nar/26.1.196 |
| [55] |
BLYTHE A J, FOX A H, BOND C S. The ins and outs of lncRNA structure:how, why and what comes next?[J]. Biochimica et Biophysica Acta:Gene Regulatory Mechanisms, 2016, 1859(1): 46-58. DOI:10.1016/j.bbagrm.2015.08.009 |




