定殖于哺乳动物胃肠道中数量庞大且组成多样的微生物菌群具有广泛的生物学活性,可与宿主形成宿主-微生物代谢轴,对动物机体营养物质代谢和免疫稳态产生重要的影响[1]。肠道微生物区系受多种因素影响,如抗生素使用与否、饲粮组成、年龄或病原微生物感染等。肠道微生物在胃肠道中的增殖主要依赖于饲粮所提供的营养成分,因而饲粮是影响其组成的最主要因素之一。饲粮中各组分的含量或类型的改变均可快速改变肠道微生物种类、数量及其代谢活性[2-3]。
淀粉作为谷物饲料中最主要的碳水化合物来源,是动物生长必不可少的能量物质[4]。在自然界中,淀粉主要有2种存在形式:支链淀粉和直链淀粉,其中支链淀粉属于快速消化淀粉,在肠道中易被快速消化吸收;而直链淀粉属于慢速消化淀粉,动物机体小肠中的酶对其降解较慢,可到达后肠部位作为微生物发酵的底物而被大量利用[5],并产生大量的短链脂肪酸(short-chain fatty acid,SCFA),广泛参与后肠的代谢调控并改善机体健康[6]。因此,不同淀粉类型饲粮(含有不同的直链淀粉/支链淀粉)对动物后肠微生物菌群结构和代谢功能可产生不同的影响。给断奶仔猪饲喂含有高直链的玉米淀粉饲粮21 d后,可显著增加其结肠前端和粪样中乳酸杆菌(Lactobacilli)和双歧杆菌(Bifidobacteria)的数量[7]。在生长猪上的研究发现,与蜡质玉米淀粉相比,饲喂含有高直链的非蜡质玉米淀粉饲粮未改变平均日增重,但可改变回肠和盲肠食糜中微生物的发酵模式,增加有益菌如耐酸乳杆菌(Lactobacillus acetotolerans)的相对丰度,而降低潜在致病菌——丙型变形菌纲(Gammaproteobacteria)的相对丰度和部分SCFA的含量[8]。另有研究发现,给猪长期饲喂含有高直链的生土豆淀粉饲粮,显著增加了其平均日采食量,改变了结肠的微生物区系,并有利于维系肠道健康[9]。本试验前期研究结果也表明,给育肥猪饲喂不同淀粉类型饲粮显著改变其生长性能以及结肠微生物组成和代谢,与木薯淀粉饲粮组相比,摄食含有高直链的豌豆淀粉饲粮组的平均日增重增加了14.12%,料重比降低了9.88%;此外,增加结肠食糜中产SCFA和乳酸相关菌的丰度[如乳杆菌属(Lactobacillus)、粪杆菌属(Faecalibacterium)等]和有机酸、糖类和长链脂肪酸等代谢产物的含量,降低潜在致病菌——大肠杆菌(Escherichia coli)的丰度和一些氨基酸类代谢产物的含量[10]。除结肠外,猪盲肠食糜中也含有大量的微生物,它们对动物机体的营养物质代谢和肠道健康也存在极大的影响。到目前为止,尽管不同淀粉类型饲粮对猪肠道微生物组成的影响已有一定的报道,但其对机体“后肠营养”具有不可忽视作用的盲肠微生物组成和代谢的调控还有待进一步研究。基于此,本试验旨在探讨不同淀粉类型饲粮对育肥猪盲肠食糜主要微生物数量和代谢产物含量的影响,研究结果将为在配制育肥猪饲粮时合理应用淀粉原料提供科学依据和理论指导。
1 材料与方法 1.1 试验动物、分组与试验饲粮本试验采用单因素完全随机区组试验设计,选取72头健康且初始体重相近[(77.00±0.52) kg]的“杜×长×大”三元杂交阉公猪,按照体重随机分为3组,以栏为单位,每组8栏(重复),每栏3头猪。3组猪分别饲喂含有木薯淀粉(TS组)、玉米淀粉(CS组)和豌豆淀粉(PS组)作为唯一淀粉来源配制的3种饲粮,饲粮的直链淀粉与支链淀粉的比例分别为0.11、0.25和0.44。饲粮根据NRC(2012)[11]75~100 kg阶段育肥猪营养需要配制,3种饲粮配方的代谢能、粗蛋白质、总淀粉以及4种主要必需氨基酸(赖氨酸、蛋氨酸、苏氨酸和色氨酸)的含量基本一致,饲粮组成及营养水平见表 1。
|
|
表 1 饲粮组成及营养水平(饲喂基础) Table 1 Composition and nutrient levels of diets (as-fed basis) |
饲养试验于2018年1—3月在广东省农业科学院动物科学研究所试验场进行,试验为期46 d,其中包括预试期4 d。所有试验猪均在同一猪舍内并进行分栏饲养。试验期间,所有猪自由采食并自由饮水。每天不定期冲洗笼舍,并保持猪舍内清洁、通风和干燥。试验期间记录每天的采食量,并观察试验猪的健康状况。于正式试验的第42天,所有试验猪禁食12 h后,从每栏内选取1头体重接近平均体重的猪(每组8头,共24头猪),经电击晕后进行放血屠宰,迅速分离出盲肠,收集所有盲肠食糜经混匀后立即测定食糜的pH,之后取10 g左右样品置于-20 ℃保存待测食糜中总淀粉和直链淀粉的含量并计算直链淀粉/支链淀粉。另取10 g左右样品保存于-80 ℃冰箱,用于后续微生物和细菌代谢产物分析。
1.3 测定指标与方法 1.3.1 饲粮及盲肠食糜总淀粉、直链淀粉含量以及直链淀粉/支链淀粉的测定饲粮和盲肠食糜中总淀粉、直链淀粉含量以及直链淀粉/支链淀粉按照试剂盒(Megazyme,爱尔兰)说明书,使用酶标仪进行测定。
1.3.2 食糜pH的测定采用便携式pH计(HI 9024C;HANNA Instruments,美国)测定盲肠食糜的pH,每个样品测定3次,取平均值。
1.3.3 乳酸含量的测定食糜中乳酸含量测定参照试剂盒(南京建成生物工程研究所)说明书,使用酶标仪进行测定。
1.3.4 SCFA含量的测定盲肠食糜中SCFA含量的测定参照Yu等[12]的方法,称取0.4 g盲肠食糜,经处理后采用气相色谱仪(7890B,Agilent Technologies Inc.,美国)进行测定。SCFA的含量利用外标法通过标准曲线计算得出。
1.3.5 氨态氮(NH3-N)含量的测定盲肠食糜中NH3-N的含量测定参照Chaney等[13]的方法,以氯化铵为标品,采用比色法进行测定。
1.3.6 生物胺含量的测定盲肠食糜中生物胺含量的测定参照Yang等[14]的方法,称取0.5 g样品,经处理后使用Waters Alliance高效液相色谱仪(e2695)进行测定。各类生物胺的含量采用外标法通过标准曲线计算得出。
1.3.7 酚和吲哚类物质含量的测定称取0.1 g盲肠食糜样品,参照Yu等[15]的方法进行处理后,用Waters Alliance高效液相色谱仪(e2695)配备Zorbax Extend-C18分离柱(Agilent Technologies, 美国)进行测定。酚和吲哚类物质含量利用外标法通过标准曲线计算得出。
1.3.8 盲肠食糜总菌DNA的提取和real-time PCR检测称取0.25 g盲肠食糜,根据MOBIO试剂盒中的说明书提取细菌的总DNA(Qiagen,德国)。采用核酸蛋白检测仪Nanodrop 1100(Thermo Fisher Scientific,美国)检测所提取的细菌DNA浓度和质量,样品合格的标准为:吸光度值(OD)260/280在1.80~2.00,OD260/230在2.00左右。
采用Bio-Rad CFX Connect系统对盲肠食糜中主要微生物的16S rRNA基因拷贝数进行定量分析,所定量微生物种类以及引物见表 2。使用10倍梯度稀释的标准质粒作为模板建立标准曲线,每个样品3个重复。PCR采用20 μL的反应体系:10 μL的TB GreenTM Premix Ex TaqTM(TaKaRa,大连),DNA模板2.0 μL,上、下游引物各0.4 μL,灭菌的超纯水7.2 μL。PCR的扩增程序具体参照Yu等[15]的方法。数据均表示为每克盲肠食糜中16S rRNA基因的拷贝数。
|
|
表 2 定量微生物种类以及引物 Table 2 Quantitative microbial species and primers |
所有试验数据均采用SPSS 20.0软件包中的one-way ANOVA模块进行单因素方差分析,差异显著者再用Tukey’s进行多重比较。数据均表示为平均值±标准误,P < 0.05表示差异显著。为了评估肠道中微生物数量的改变是否伴随着代谢产物含量的改变,本试验采用GraphPad Prism 5.0软件(GraphPad Software,美国)对盲肠有差异的微生物数量和代谢产物含量进行Pearson相关性分析,当Pearson的绝对相关系数大于0.5,且P < 0.05时,认为二者存在相关性。为了评估不同组间微生物数量与代谢产物之间的相似性,本试验分别将主要微生物数量、pH和代谢产物(pH、SCFA、乳酸、NH3-N、生物胺、酚和吲哚类物质)含量导入SIMCA-P 13.0 (Umetrics, Umeả, 瑞典)中采用偏最小二乘分类分析(partial least-squares discriminant analysis,PLS-DA)模型进行分析。
2 结果与分析 2.1 不同淀粉类型饲粮对育肥猪盲肠食糜中总淀粉含量、直链淀粉/总淀粉和直链淀粉/支链淀粉的影响由表 3可知,与TS组相比,PS组育肥猪盲肠食糜中总淀粉含量和直链淀粉/支链淀粉显著增加(P < 0.05);同时,CS组育肥猪盲肠食糜中总淀粉含量也显著提高(P < 0.05)。此外,不同组间育肥猪盲肠食糜中直链淀粉/总淀粉无显著差异(P>0.05)。
|
|
表 3 不同淀粉类型饲粮对育肥猪盲肠食糜中总淀粉含量、直链淀粉/总淀粉和直链淀粉/支链淀粉的影响 Table 3 Effects of different starch type diets on total starch content, amylose starch to total starch ratio and amylose starch to amylopectin starch ratio in cecal digesta of finishing pigs |
由表 4可知,不同淀粉类型饲粮显著影响了育肥猪盲肠食糜pH(P < 0.05),其中PS组的pH显著低于TS组(P < 0.05),而CS组的pH与TS组和PS组相比无显著差异(P>0.05)。与TS组相比,CS组和PS组育肥猪食糜中乳酸含量显著增加(P < 0.05)。对于SCFA含量而言,与TS组相比,PS组育肥猪食糜中乙酸、丁酸和总SCFA的含量显著增加(P < 0.05),而CS组上述SCFA的含量与TS组和CS组相比差异不显著(P>0.05)。此外,不同组间育肥猪食糜中丙酸、戊酸、异丁酸、异戊酸和支链脂肪酸的含量无显著差异(P>0.05)。
|
|
表 4 不同淀粉类型饲粮对育肥猪盲肠食糜pH、乳酸和SCFA含量的影响 Table 4 Effects of different starch type diets on pH and contents of lactate and SCFA in cecal digesta of finishing pigs |
由表 5可知,与TS组相比,CS组和PS组育肥猪盲肠食糜中NH3-N的含量显著降低(P < 0.05),而CS组和PS组NH3-N的含量差异不显著(P>0.05)。对于生物胺而言,与TS组相比,PS组育肥猪盲肠食糜中尸胺和色胺的含量显著降低(P < 0.05),而CS组尸胺和色胺含量与TS组和PS组相比无显著差异(P>0.05)。同时,CS组和PS组育肥猪盲肠食糜中腐胺和总生物胺的含量显著低于TS组(P < 0.05),而CS组与PS组腐胺和总生物胺含量无显著差异(P>0.05)。此外,不同组间育肥猪盲肠食糜中甲胺、酪胺、精胺和亚精胺的含量无显著差异(P>0.05)。对于酚和吲哚类物质而言,与TS组相比,CS组和PS组育肥猪盲肠食糜中吲哚和粪臭素的含量显著降低(P < 0.05),而CS组与PS组吲哚和粪臭素含量无显著差异(P>0.05)。此外,不同组间育肥猪盲肠食糜中甲酚和苯酚的含量无显著差异(P>0.05)。
|
|
表 5 不同淀粉类型饲粮对育肥猪盲肠食糜微生物氮代谢产物含量的影响 Table 5 Effects of different starch type diets on microbial nitrogen metabolite contents in cecal digesta of finishing pigs |
由图 1可知,不同组间育肥猪盲肠食糜中微生物总数和2个主要细菌门[厚壁菌们(Firmicutes)和拟杆菌门(Bacteroidetes)]的数量无显著差异(P>0.05)。在属水平上,与TS组相比,CS组和PS组育肥猪盲肠食糜中普雷沃氏菌属(Prevotella)的数量显著增加(P < 0.05),大肠杆菌的数量显著降低(P < 0.05),而CS组和PS组间普雷沃氏菌属和大肠杆菌的数量无显著差异(P>0.05)。此外,PS组育肥猪食糜中乳酸杆菌、双歧杆菌、梭菌Ⅳ(Clostridium cluster Ⅳ)和梭菌ⅩⅣ(Clostridium cluster ⅩⅣ)的数量显著高于TS组(P < 0.05)。育肥猪盲肠食糜中瘤胃球菌属(Ruminococcus)的数量在各组间差异不显著(P>0.05)。
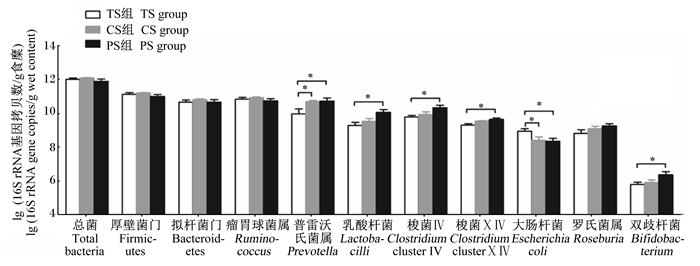
|
数据柱间标注*表示差异显著(P < 0.05)。 Marked * between data columns indicated significant difference (P < 0.05). 图 1 不同淀粉类型饲粮对育肥猪盲肠食糜主要微生物数量的影响 Fig. 1 Effects of different starch type diets on main microbial population in cecal digesta of finishing pigs |
为了直观显示不同类型淀粉饲粮处理对育肥猪盲肠食糜微生物菌群和代谢产物的差异,我们采用SIMCA-P 13.0中的PLS-DA方法进行多元变量分析发现,PLS-DA建立的多元分析模型能分别将PS组和TS组的微生物(图 4-A)和代谢产物(图 4-B)明显区分开,表明给育肥猪饲喂PS组和TS组饲粮显著改变了其盲肠微生物数量和代谢产物的组成。
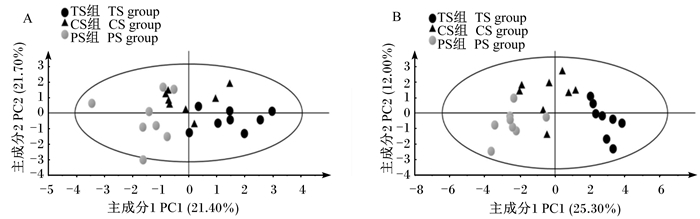
|
图 2 育肥猪盲肠食糜中微生物(A)和代谢产物(B)PLS-DA得分图 Fig. 2 PLS-DA score plots of microbes (A) and metabolites (B) in cecal digesta of finishing pigs |
为了探索肠道微生物组成与其代谢产物变化之间的潜在关系,我们采用Pearson分析方法对盲肠有差异的微生物数量和代谢产物含量进行相关性分析。由图 3可知,pH与乳酸、乙酸、丁酸和总SCFA的含量显著负相关(P < 0.05),而与NH3-N、腐胺、尸胺、总生物胺、吲哚和粪臭素的含量显著正相关(P < 0.05)。大肠杆菌的数量与乙酸、丁酸和总SCFA的含量显著负相关(P < 0.05),而与NH3-N、腐胺、尸胺、吲哚和粪臭素的含量显著正相关(P < 0.05);梭菌Ⅳ的数量与乳酸、乙酸、丁酸和总SCFA的含量显著正相关(P < 0.05),而与NH3-N含量和pH呈显著负相关(P < 0.05);梭菌ⅩⅣ的数量与丁酸和总SCFA的含量呈显著正相关(P < 0.05),而与总生物胺含量和pH呈显著负相关(P < 0.05);乳酸杆菌的数量与乳酸和乙酸的含量呈显著正相关(P < 0.05),而与腐胺、尸胺、总生物胺和吲哚的含量呈显著负相关(P < 0.05);双歧杆菌的数量与乳酸、乙酸、丁酸和总SCFA的含量显著正相关(P < 0.05),而与腐胺、尸胺、总生物胺含量和pH呈显著负相关(P < 0.05)。此外,普雷沃氏菌属的数量与乙酸和总生物胺的含量呈显著正相关(P < 0.05),而与NH3-N含量和pH呈显著负相关(P < 0.05)。
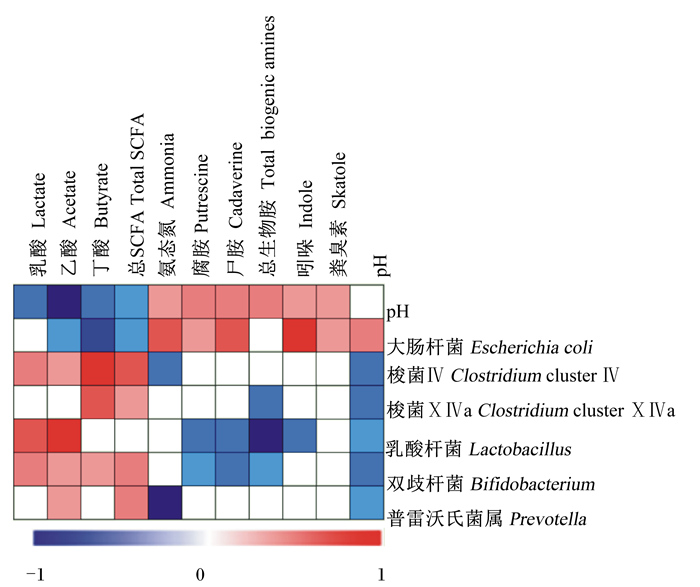
|
将有差异的微生物(数量)和代谢产物(含量)进行了Pearson相关性分析,相关系数范围为-1~1,其绝对值大于0.5且P < 0.05时视为显著相关。单元格红色表示显著正相关(P < 0.05),蓝色表示显著负相关(P < 0.05),白色表示无显著相关性(P>0.05)。颜色的深浅表明相关强度,并随着绝对相关系数的增大由浅向深渐变。 Pearson's correlation analysis between the significantly changed bacteria (numbers) and metabolites (contents). The correlation coefficient ranged from-1 to 1, and a correlation was considered significant when the absolute Pearson's correlation coefficient was >0.5 and significant at P < 0.05. Red represented a significant positive correlation (P < 0.05), blue represented significantly negative correlation (P < 0.05), and white showed that the correlation was not significant (P>0.05). The intensity of the colors represented the degree of association, and with the increasing of absolute correlation coefficient, the color gradually changed from shallow to deep. 图 3 育肥猪盲肠食糜中主要微生物数量和代谢产物含量的相关性分析 Fig. 3 Correlation analysis between the number of main microbial population and metabolite contents in cecal digesta of finishing pigs |
饲粮结构和组成是影响肠道微生物种类、数量及其代谢活性的主要因素之一。通过定量PCR检测,本试验研究发现,不同淀粉类型饲粮对育肥猪盲肠食糜主要微生物数量有不同的影响,表现为摄食含有高直链的豌豆淀粉显著增加了产SCFA或乳酸相关细菌的数量,如乳酸杆菌、普雷沃氏菌属、双歧杆菌、梭菌Ⅳ和梭菌ⅩⅣ,而降低潜在致病菌——大肠杆菌的数量,这与我们前期在结肠中的发现[10]一致。在生长猪上的研究也发现,饲喂含有高直链的非蜡质玉米淀粉增加其回肠食糜中耐酸乳杆菌的相对丰度,而减少了潜在致病菌丙型变形菌纲的相对丰度[8]。而在断奶仔猪上的研究发现,饲粮中含有63%直链淀粉可显著增加结肠食糜中双歧杆菌的数量,但减少了梭菌Ⅳ和梭菌ⅩⅣ的数量[26],这可能与育肥猪对高直链淀粉消化能力更强有关,但具体原因还有待进一步研究。乳酸杆菌和双歧杆菌是存在于肠道内的主要有益微生物,可通过竞争性抑制肠道病原菌的感染和定殖,并调控宿主的免疫反应,从而促进宿主的肠道和机体健康[27]。普雷沃氏菌属细菌属于淀粉利用菌,可降解淀粉产生短链脂肪酸等物质[28]。梭菌Ⅳ和梭菌ⅩⅣ是肠道中主要的产丁酸菌,可通过代谢淀粉或含氮化合物产生大量丁酸,进而促进机体肠道上皮细胞能量代谢和调节肠道黏膜免疫系统的发育来维系肠道内环境的稳态[29-30]。大肠杆菌属于蛋白降解菌,且对酸敏感[31]。在本试验中,含有高直链的豌豆淀粉增加了食糜SCFA的含量,进而降低了食糜的pH,相关性分析也发现大肠杆菌的数量与pH呈显著负相关。因此,豌豆淀粉降低大肠杆菌数量可能是由于其降低了食糜中的pH,进而抑制了大肠杆菌在肠道中的生长繁殖。大肠杆菌属于一种潜在致病菌,与结肠炎或者克罗恩等疾病的发生密切相关[32]。良好的肠道内环境可维护动物的肠道健康状况,进而改善动物的生长性能,进一步提高生产效益。因此,本试验中产乳酸菌和产SCFA菌数量的增加,以及潜在致病菌数量的降低,表明摄入高直链的豌豆淀粉可维系肠道菌群的稳态并对肠道健康具有益生作用。这一研究结果从肠道健康层面进一步解释我们的前期研究结果,即豌豆淀粉饲粮提高了育肥猪的生产性能[10]。
3.2 不同淀粉类型饲粮改变育肥猪盲肠食糜中微生物对碳水化合物的代谢在肠道中,可被微生物发酵的底物不同也会导致其发酵代谢过程和代谢产物的不同。本试验结果显示,给育肥猪饲喂不同淀粉类型饲粮显著改变了盲肠微生物对碳水化合物的代谢,表现为摄食豌豆淀粉显著增加食糜中乳酸、乙酸、丁酸和总SCFA的含量,这与我们前期在结肠中的研究结果[10]相一致。如前言所述,直链淀粉属于不易被单胃动物前肠消化酶分解,但易被发酵的碳水化合物,当其进入后肠可作为微生物发酵的底物而被优先利用[33]。本试验也发现,PS组育肥猪盲肠食糜中的总淀粉和直链淀粉含量均高于TS组。因此,本试验中乳酸和SCFA含量增加的一个原因可能是由于豌豆淀粉中含有较高的直链淀粉,未被前肠消化吸收的淀粉进入后肠后作为微生物的发酵底物而被大量分解产生。此外,肠道中微生物代谢产物含量的变化与产这些代谢物的细菌数量或丰度的变化息息相关[34]。如前面所述,给育肥猪饲喂豌豆淀粉显著增加了其盲肠食糜中主要产乳酸细菌——乳酸杆菌和双歧杆菌和产短链脂肪酸菌——普雷沃氏菌属、梭菌Ⅳ和梭菌ⅩⅣ的数量,相关性分析结果也表明乳酸、乙酸和丁酸含量与这些细菌存在显著的正相关作用。因此,本试验中育肥猪盲肠食糜产乳酸和SCFA这些细菌数量的增加可能是导致乳酸和SCFA含量升高的另一原因。
在肠道中的微生物代谢产物,如SCFA,与微生物的功能、肠道健康状态和宿主营养物质代谢密切相关[35]。乳酸可被大肠内的乳酸利用菌代谢生成乙酸和丁酸等物质,进一步降低肠腔内的pH,抑制肠道内对酸敏感的潜在致病菌的过度生长(如大肠杆菌),进而维持宿主肠道健康[36]。乙酸可迅速被肠上皮细胞吸收并进入外周血液循环组织,作为能量基底物质为宿主提供能量[5]。丁酸除了可为结肠上皮细胞提供能量促进肠上皮细胞增殖和发育外,还具有抗炎作用和降低上皮细胞的凋亡[5]。因此,本试验中饲喂含高直链的豌豆淀粉增加食糜中乳酸、乙酸和丁酸含量,提示摄食豌豆淀粉有利于维持宿主肠道健康,并为宿主生长提供更多的能量物质,进而促进育肥猪的生长性能。
3.3 不同淀粉类型饲粮改变育肥猪盲肠食糜中微生物对含氮化合物的代谢前肠未被消化吸收的含氮物质进入后肠可被微生物发酵生成NH3-N、生物胺、酚和吲哚类化合物。本研究结果显示,给育肥猪饲喂含有高直链的豌豆淀粉显著降低了食糜中NH3-N的含量,这与本试验在结肠中的研究结果[10]不一致,表明给育肥猪饲喂不同淀粉类型饲粮,其盲肠和结肠微生物的脱氨基反应不同。在动物机体的不同肠段中,其微生物在组成上存在明显的差异,不同肠道微生物对饲粮的代谢可能具有不同的影响。因此,不同淀粉类型饲粮对盲肠和结肠食糜中NH3-N含量具有的不同影响,可能是由于盲肠和结肠食糜中本身微生物组成不同所引起。高含量的NH3-N可抑制肠上皮细胞增殖和分化以及线粒体的氧化利用,增加腹泻率[37]。因此,食糜中NH3-N含量降低可能有益于机体肠道健康。对于生物胺而言,本研究发现含有高直链的豌豆淀粉降低了尸胺、腐胺、色胺和总生物胺的含量,表明微生物对这些代谢物相对应的前体氨基酸——赖氨酸、鸟氨酸和色氨酸的脱羧反应受到抑制。高含量的生物胺对肠道有毒性作用,尤其是尸胺、腐胺和色胺,当超过一定含量,可引起DNA损伤和氧化应激,从而增加肿瘤发生的风险[37]。本研究还发现含高直链的豌豆淀粉显著降低了食糜中吲哚和粪臭素的含量。吲哚和粪臭素是肠道微生物发酵代谢色氨酸而产生的代谢物,且具有遗传毒性、致突变性和致癌性[33]。因此,本试验中吲哚和粪臭素含量的降低进一步表明摄食豌豆淀粉降低了有害发酵产物的生成,有利于维系育肥猪的肠道健康。
改变微生物所处肠道内环境的pH,可改变微生物的代谢途径[33]。微生物分解代谢氨基酸所分泌的蛋白酶活性在中性或碱性条件下优于酸性条件,高pH条件有利于生物胺和吲哚类物质的生成[38]。此外,pH还会调节NH3-N和铵根离子(NH4+)二者之间的平衡,当pH低于7以下,主要以NH4+形式存在,从而减少NH3-N的生成。在本试验中,豌豆淀粉显著降低了盲肠食糜的pH(6.11±0.16)。因此,本试验中豌豆淀粉降低含氮物质的腐败发酵的一个原因可能是降低了肠道内环境的pH,进而降低微生物所分泌的蛋白酶活性,最终抑制微生物对这些含氮物质的腐败发酵。此外,大肠杆菌具有代谢含氮化合物生成生物胺和吲哚类物质的能力,是肠道中主要的蛋白降解菌[37]。如前面相关性分析结果所示,食糜中NH3-N、生物胺和吲哚类物质的含量与大肠杆菌的数量呈显著正相关的关系。因此,本试验中豌豆淀粉降低大肠杆菌的数量可能是减少这些氮代谢分解代谢物的另一个主要原因,但其具体作用机制还有待进一步研究。
4 结论综上所述,在本试验条件下,与木薯淀粉相比,给育肥猪饲喂含有高直链的豌豆淀粉改变了其盲肠食糜中微生物的组成和代谢活性;促进肠道乳酸杆菌、普雷沃氏菌属、双歧杆菌、梭菌Ⅳ和梭菌ⅩⅣ等有益微生物在后肠中竞争获得碳源而增殖,并竞争性抑制潜在致病菌大肠杆菌的生长。同时,豌豆淀粉饲粮可通过改变育肥猪肠道微生物的发酵模式,使肠道微生物偏向于碳水化合物型发酵,产生对肠道有益的代谢物——乳酸、乙酸和丁酸,而减少微生物对含氮化合物的腐败发酵,从而减少NH3-N、生物胺和酚类物质等物质的产生。这些结果表明摄食含有高直链的淀粉可作为预防保护性物质,维系宿主肠道健康。
| [1] |
BÄCKHED F, LEY R E, SONNENBURG J L, et al. Host-bacterial mutualism in the human intestine[J]. Science, 2005, 307(5717): 1915-1920. |
| [2] |
朱伟云, 余凯凡, 慕春龙, 等. 猪的肠道微生物与宿主营养代谢[J]. 动物营养学报, 2014, 26(10): 3046-3051. |
| [3] |
MARSUMOTO M, KIBE R, OOGA T, et al. Impact of intestinal microbiota on intestinal luminal metabolome[J]. Scientific Reports, 2012, 2: 233. |
| [4] |
NOFRARÍAS M, MARTÍNEZ-PUIG D, PUJOLS J, et al. Long-term intake of resistant starch improves colonic mucosal integrity and reduces gut apoptosis and blood immune cells[J]. Nutrition, 2007, 23(11/12): 861-870. |
| [5] |
TREMAROLI V, BÄCKHED F. Functional interactions between the gut microbiota and host metabolism[J]. Nature, 2012, 71(7415): 242-249. |
| [6] |
KNUDSEN K E B, HEDEMANN M S, LÆRKE H N. The role of carbohydrates in intestinal health of pigs[J]. Animal Feed Science and Technology, 2012, 173(1/2): 41-53. |
| [7] |
BIRD A R, VUARAN M, BROWN I, et al. Two high-amylose maize starches with different amounts of resistant starch vary in their effects on fermentation, tissue and digesta mass accretion, and bacterial populations in the large bowel of pigs[J]. British Journal of Nutrition, 2007, 97(1): 134-144. |
| [8] |
LUO Y H, YANG C, WRIGHT A D G, et al. Responses in ileal and cecal bacteria to low and high amylose/amylopectin ratio diets in growing pigs[J]. Applied Microbiology and Biotechnology, 2015, 99(24): 10627-10638. |
| [9] |
SUN Y, ZHOU L P, FANG L D, et al. Responses in colonic microbial community and gene expression of pigs to a long-term high resistant starch diet[J]. Frontiers in Microbiology, 2015, 6: 877. |
| [10] |
YU M, LI Z M, CHEN W D, et al. Microbiome-metabolomics analysis investigating the impacts of dietary starch types on the composition and metabolism of colonic microbiota in finishing pigs[J]. Frontiers in Microbiology, 2019, 10: 1143. |
| [11] |
N RC. Nutrient requirements of swine[M]. 2012.
|
| [12] |
YU M, ZHANG C J, YANG Y X, et al. Long-term effects of early antibiotic intervention on blood parameters, apparent nutrient digestibility, and fecal microbial fermentation profile in pigs with different dietary protein levels[J]. Journal of Animal Science and Biotechnology, 2017, 8: 60. |
| [13] |
CHANEY A L, MARBACH E P. Modified reagents for determination of urea and ammonia[J]. Clinical Chemistry, 1962, 8: 130-132. |
| [14] |
YANG Y X, MU C L, ZHANG J F, et al. Determination of biogenic amines in digesta by high performance liquid chromatography with precolumn dansylation[J]. Analytical Letters, 2014, 47(8): 1290-1298. |
| [15] |
YU M, LI Z M, CHEN W D, et al. Hermetia illucens larvae as a potential dietary protein source altered the microbiota and modulated mucosal immune status in the colon of finishing pigs[J]. Journal of Animal Science and Biotechnology, 2019, 10: 50. |
| [16] |
SUZUKI M T, TAYLOR L T, DELONG E F. Quantitative analysis of small-subunit rRNA genes in mixed microbial populations via 5'-nuclease assays[J]. Applied and Environment Microbiology, 2000, 66(11): 4605-4614. |
| [17] |
GUO X, XIA X, TANG R, et al. Development of a real-time PCR method for Firmicutes and Bacteroidetes in faeces and its application to quantify intestinal population of obese and lean pigs[J]. Letters in Applied Microbiology, 2008, 47(5): 367-373. |
| [18] |
HUIJSDENS X W, LINSKENS R K, MAK M, et al. Quantification of bacteria adherent to gastrointestinal mucosa by real-time PCR[J]. Journal of Clinical Microbiology, 2002, 40(12): 4423-4427. |
| [19] |
LAN Y, XUN S, TAMMINGA S, et al. Real-time PCR detection of lactic acid bacteria in cecal contents of Eimeria tenella-infected broilers fed soybean oligosaccharides and soluble soybean polysaccharides[J]. Poultry Science, 2004, 83(10): 1696-1702. |
| [20] |
WALKER A W, INCE J, DUNCAN S H, et al. Dominant and diet-responsive groups of bacteria within the human colonic microbiota[J]. The ISME Journal, 2011, 5(2): 220-230. |
| [21] |
MATSUKI T, WATANABE K, FUJIMOTO J, et al. Use of 16S rRNA gene-targeted group-specific primers for real-time PCR analysis of predominant bacteria in human feces[J]. Applied and Environmental Microbiology, 2004, 70(12): 7220-7228. |
| [22] |
BARTOSCH S, FITE A, MACFARLANE G T, et al. Characterization of bacterial communities in feces from healthy elderly volunteers and hospitalized elderly patients by using real-time PCR and effects of antibiotic treatment on the fecal microbiota[J]. Applied and Environmental Microbiology, 2004, 70(6): 3575-3581. |
| [23] |
VERMA R, VERMA A K, AHUJA V, et al. Real-time analysis of mucosal flora in patients with inflammatory bowel disease in India[J]. Journal of Clinical Microbiology, 2010, 48(11): 4279-4282. |
| [24] |
LAYTON A, MCKAY L, WILLIAMS D, et al. Development of Bacteroides 16S rRNA gene TaqMan-based real-time PCR assays for estimation of total, human, and bovine fecal pollution in water[J]. Applied and Environment Microbiology, 2006, 72(6): 4214-4224. |
| [25] |
WALKER A W, DUNCAN S H, LEITCH E C M, et al. pH and peptide supply can radically alter bacterial populations and short-chain fatty acid ratios within microbial communities from the human colon[J]. Applied and Environment Microbiology, 2005, 71(7): 3692-3700. |
| [26] |
FOUHSE J M, GÄNZLE M G, REGMI P R, et al. High amylose starch with low in vitro digestibility stimulates hindgut fermentation and has a bifidogenic effect in weaned pigs[J]. The Journal of Nutrition, 2015, 145(11): 2464-2470. |
| [27] |
WANG W, CHEN L P, ZHOU R, et al. Increased proportions of Bifidobacterium and the Lactobacillus group andloss of butyrate-producing bacteria in inflammatory bowel disease[J]. Journal of Clinical Microbiology, 2014, 52(2): 398-406. |
| [28] |
FLINT H J, BAYER E A, RINCON M T, et al. Polysaccharide utilization by gut bacteria:potential for new insights from genomic analysis[J]. Nature Reviews Microbiology, 2008, 6(2): 121-131. |
| [29] |
WANG X, CONWAY P L, BROWN I L, et al. In vitro utilization of amylopectin and high-amylose maize (amylomaize) starch granules by human colonic bacteria[J]. Applied and Environment Microbiology, 1999, 65(11): 4848-4854. |
| [30] |
LOUIS P, HOLD G L, FLINT H J. The gut microbiota, bacterial metabolites and colorectal cancer[J]. Nature Reviews Microbiology, 2014, 12(10): 661-672. |
| [31] |
CHEN X Y, SONG P X, FAN P X, et al. Moderate dietary protein restriction optimized gut microbiota and mucosal barrier in growing pig model[J]. Frontiers in Cellular and Infection Microbiology, 2018, 8: 246. |
| [32] |
BARNICH N, CARVALHO F A, GLASSER A L, et al. CEACAM6 acts as a receptor for adherent-invasive E. coli, supporting ileal mucosa colonization in Crohn disease[J]. Journal of Clinical Investigation, 2007, 117(6): 1566-1574. |
| [33] |
余苗, 陈卫东, 邓盾, 等. 日粮淀粉对猪肠道微生物和臭气排放调控的研究进展[J]. 家畜生态学报, 2019, 39(9): 1-6. |
| [34] |
GAO K, PI Y, PENG Y, et al. Time-course responses of ileal and fecal microbiota and metabolite profiles to antibiotics in cannulated pigs[J]. Applied Microbiology and Biotechnology, 2018, 102(5): 2289-2299. |
| [35] |
DEN BESREN G, VAN EUNEN K, GROEN A K, et al. The role of short-chain fatty acids in the interplay between diet, gut microbiota, and host energy metabolism[J]. Journal of Lipid Research, 2013, 54(9): 2325-2340. |
| [36] |
TAN J, MCKENZIE C, VUILLERMIN P J, et al. Dietary fiber and bacterial SCFA enhance oral tolerance and protect against food allergy through diverse cellular pathways[J]. Cell Reports, 2016, 15(12): 2809-2824. |
| [37] |
RIST V T S, WEISS E, EKLUND M, et al. Impact of dietary protein on microbiota composition and activity in the gastrointestinal tract of piglets in relation to gut health:a review[J]. Animal, 2013, 7(7): 1067-1078. |
| [38] |
PI Y, GAO K, PENG Y, et al. Antibiotic-induced alterations of the gut microbiota and microbial fermentation in protein parallel the changes in host nitrogen metabolism of growing pigs[J]. Animal, 2018, 13(2): 262-272. |




