2. 中国农业科学院北京畜牧兽医研究所, 农业部奶及奶制品质量安全控制重点实验室, 北京 100193
2. Key Laboratory of Quality and Safety Control for Milk and Dairy Products, Institute of Animal Sciences, Chinese Academy of Agricultural Sciences, Beijing 100193, China
反刍动物瘤胃中产脲酶菌水解尿素产生氨的速率过快,导致大量的氨无法及时与碳架结合。过量的氨被释放到环境中,造成营养流失与环境污染[1]。产脲酶菌产生的脲酶是造成这些危害的原因,细菌脲酶由结构基因UreA、UreB、UreC分别编码的α亚基、β亚基、γ亚基组成的(αβγ)3三聚体构成,分子质量在222~333 ku[2]。细菌脲酶的活性中心是其催化尿素的关键功能位点,通常其活性中心位于UreC编码的蛋白区域,由2个氨基甲酰化赖氨酸桥接的2个镍离子构成[3]。其中1个镍离子进一步由2个组氨酸和1个末端水分子配位;另1个镍与另外2个组氨酸、天冬氨酸以及水配位[4-5]。此外,脲酶活性中心外的柔性片状区域是抑制脲酶的重要变构位点[6]。该区域的半胱氨酸位点能调控柔性片状区域的构象,从而影响底物与脲酶活性中心的结合[7-8]。利用脲酶抑制剂,以脲酶活性中心及柔性片状区域为靶标,能有效抑制细菌脲酶活性,达到控制氨的产生速率的目的。
目前仅有的药用脲酶抑制剂为乙酰氧肟酸,但长期使用乙酰氧肟酸会导致微生物产生适应性,降低抑制效果[9]。因此,亟需开发生物效率高的新型脲酶抑制剂。
近年来,基于分子对接(molecular docking)技术筛选蛋白靶标化合物的报道较为多见。分子对接是在充分解析靶标蛋白三维结构的基础上,通过计算靶标受体与配体化合物的结合分值,以寻找可以作用于目标结构的候选化合物[10-12]。Macomber等[6]以产气克雷伯氏菌脲酶(PDB_ID 1EJX)为靶标,通过基于分子对接技术虚拟筛选ZINC化合物库,结合刀豆脲酶活性抑制试验构建筛选、评价模型,最终获得了具有刀豆脲酶抑制作用的表没食子儿茶素及槲皮素2种天然产物化合物,同时利用基因定点突变技术证实了通过分子对接技术预测的脲酶抑制剂的结合位点,虽然这2种化合物的脲酶抑制活性在该研究前已知,但该研究证实了分子对接技术在预测脲酶活性位点及虚拟筛选中应用的可行性。Azizian等[13]以幽门螺旋杆菌脲酶(PDB_ID 1E9Y)为靶标, 结合幽门螺旋杆菌脲酶活性抑制试验构建筛选、评价模型,从ZINC化合物库中筛选获得了一些具有幽门螺旋杆菌脲酶抑制效果的巴比妥酸盐类化合物。
迄今,鲜见虚拟筛选技术与瘤胃脲酶活性抑制试验结合作为筛选、评价模型以筛选化合物库的报道。随着化合物合成技术及化合物提取技术的发展,天然产物化合物库及人工合成化合物库均有很大的发展。例如ZINC、ChemDiv、DrugBank、TCM。特别是人工合成化合物的数量和化合物结构的多样性增加。其中ChemDiv化合物数据库含有约106万个化合物,其中未知活性的小分子化合物种类丰富,其生物活性有待发掘。因此,本研究以细菌脲酶蛋白结构为靶标,运用分子对接技术对ChemDiv化合物库进行虚拟筛选,以期筛选获得新型脲酶抑制剂。
1 材料与方法 1.1 试剂与仪器 1.1.1 试剂通过虚拟筛选获得的化合物购自上海某生化科技有限公司。试验中用到的试剂均为分析纯,具体有尿素、苯酚、亚硝基铁氰化钠、氢氧化钠、次氯酸钠、氯化铵、4-羟乙基哌嗪磺酸钠。试验用水均为双蒸水。
1.1.2 仪器服务器1台、超纯水仪(Milli Q,MERCK,美国)、酶标仪(G-282,Thermo Scientific,美国)、电热恒温培养箱(DRP-9082型,上海森信实验仪器有限公司)、厌氧操作箱(855-AC & 855-ACB, PLAS-LAB,美国)、高压细胞破碎机(JG-1A, 宁波新芝生物科技股份有限公司)、台式高速冷冻离心机(G170,SIGMA,德国)、恒温水浴锅(GFL-1083,Gesell Schafe, 美国)。
1.2 脲酶蛋白结构的准备搜索PDB蛋白结构库(http://www.rcsb.org/)中获取高分辨率且结构完整的脲酶蛋白结构作为分子对接的靶标。
在对接前利用sybyl-X2.1软件的“Prepare Protein Structure”模块对脲酶蛋白结构进行预处理。选择产气克雷伯氏菌(Klebsiella aerogenes)脲酶蛋白晶体结构(PDB_ID 4EP8)的C链,利用sybyl-X2.1的“Prepare Protein Structure”模块对蛋白结构进行处理。选择从Thr308~Arg336的氨基酸柔性片状区域与脲酶的活性中心。在“Remove Substructures”选项中选择晶体结构中所有的水分子,并予以去除。然后,利用“Analyze Selected Structure”来对蛋白进行分析与修改,完成对蛋白的加氢原子的操作。
1.3 基于分子对接虚拟筛选化合物库ChemDiv(https://www.chemdiv.com/)中包含约106万个化合物,其中大部分为小分子未知活性化合物。使用sybyl-X2.1软件的“Compound Filtering”模块基于五倍率法则[14]进行小分子数据库的虚拟筛选。
1.4 脲酶抑制候选化合物的瘤胃脲酶抑制活性评价 1.4.1 瘤胃液的采集与瘤胃脲酶溶液的制备晨饲前通过瘤胃瘘管采集荷斯坦奶牛瘤胃内容物,按赵圣国等[15]的方法提取瘤胃脲酶溶液。
1.4.2 脲酶抑制剂对瘤胃脲酶活性的影响 1.4.2.1 脲酶抑制候选化合物的抑制活性排序采用酚次氯酸法[16]测定脲酶活性。在96孔板中加入15 μL的脲酶抑制候选化合物溶液(终浓度为500 μmol/L),加入70 μL尿素缓冲液[50 mmol/L尿素, 10 μmol/L NiCl2,50 mmol/L 4-羟乙基哌嗪磺酸钠(HEPES),pH=7.5],再向96孔板中加入15 μL粗酶液启动反应,在37 ℃下孵育反应30 min。加入50 μL酚-硝普纳溶液,混匀,再加入50 μL碱性次氯酸钠溶液。混匀后37 ℃温浴30 min。在625 nm下测定吸光度,每个样品3个重复。空白对照的反应体系中不添加脲酶抑制候选化合物,阴性对照反应体系中添加未溶解尿素的HEPES缓冲液。根据抑制率对脲酶抑制候选化合物进一步筛选,选择抑制效果高的脲酶抑制候选化合物Y041-7877进行半抑制浓度(IC50)值的测定及脲酶抑制动力学抑制试验。抑制率的计算公式如下:

|
选取脲酶抑制效果好的候选化合物Y041-7877测定其IC50值。具体操作为:在96孔板中分别加入15 μL梯度浓度的候选瘤胃脲酶抑制剂Y041-7877,每个浓度3个重复,其梯度浓度见表 1。
|
|
表 1 脲酶抑制剂梯度浓度 Table 1 Gradient concentration of urease inhibitor |
各浓度下脲酶抑制剂的抑制率测定方法同“脲酶抑制候选化合物的抑制活性排序”部分,通过Graphpad prism软件中非线性拟合在不同化合物浓度下的脲酶活性,计算化合物的IC50值。
1.5 瘤胃脲酶抑制化合物的酶动力学评价取18支试管,各管按表 2加入不同浓度的尿素缓冲液(pH 7.5)70 μL,摇匀。再加入不同浓度的化合物15 μL,确保各管内液体体积相同,摇匀。对于未加化合物的组需要添加与加化合物组等量的溶剂。于37 ℃恒温水浴预热2 min。各管加入稀释好的酶液15 μL,充分摇匀,37 ℃精确反应10 min后,用酚次氯酸法测定溶液中的氨氮浓度,继续保温显色10 min。
|
|
表 2 各管化合物及尿素浓度 Table 2 Compound and urea concentrations for each tube |
厌氧培养基及尿素厌氧稀释液的配制参见文献[17]。
1.6.2 瘤胃微生物体外厌氧培养评价化合物对氨氮产生量的影响在厌氧培养箱内,向厌氧培养基中加入100 μL尿素厌氧稀释液。将200 μL瘤胃液和100 μL化合物溶液混合后,接种到厌氧培养基中,培养基中抑制剂的终浓度为100 μmol/L,同时设置甲醇作为阴性对照组,每组3个重复。将厌氧培养管放入培养箱内39 ℃培养24 h,作为第1代。每代24 h时吸取200 μL培养物和100 μL受试脲酶抑制剂或甲醇溶液混合后接种到新厌氧培养基,传代培养,传至第6代。每1代在培养0、6、12、24 h分别取200 μL样品, 4 ℃,12 000×g离心5 min,取200 μL上清液加入2.0 μL HCL(8 mol/L)后冻存, 用于测定氨氮浓度,测定方法为酚次氯酸法[16]。
1.7 新型脲酶抑制剂与瘤胃脲酶的结合模式分析将化合物Y041-7877与脲酶蛋白(PDB_ID 4EP8)活性中心的分子对接结果输入pymol软件作图,显示化合物Y041-7877与脲酶活性中心的结合模式。
1.8 数据分析采用Graphpad prism软件对不同抑制剂浓度条件下瘤胃脲酶的活性进行非线性拟合,获得抑制剂的IC50值与脲酶的动力学曲线。采用SAS 9.4统计软件一般线性模型(GLM)对不同化合物处理和处理时间的主效应及其交互效应进行双因素方差分析(two-way ANOVA),如存在交互效应,则对各处理进行Duncan氏法多重比较。P < 0.05为差异显著判断标准,P < 0.01为差异极显著判断标准。
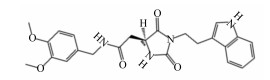
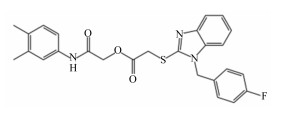
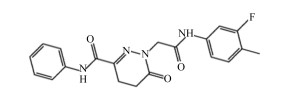
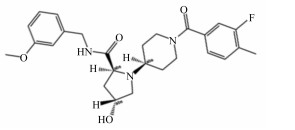
2 结果与分析 2.1 基于分子对接虚拟筛选化合物库使用sybyl-X2.1软件的“Compound Filtering”模块进行小分子数据库筛选,筛选共分为3步:初步筛选、第1轮筛选和第2轮筛选。首先,从ChemDiv化合物库中的100万个化合物中初步筛选出87万个符合类药规则的化合物。随后进行第1轮筛选,通过计算获得了8 753个化合物。接着进行第2轮筛选,最终筛选出5个化合物作为脲酶抑制候选化合物进行下一步的验证研究,这些化合物打分及化学结构信息见表 3。
|
|
表 3 化合物打分及化学结构信息 Table 3 Docking score and chemical structures information of compounds |
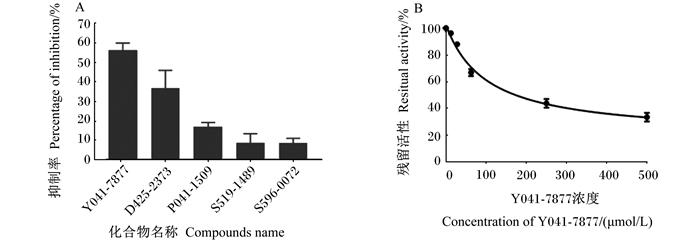
用终浓度为500 μmol/L的脲酶抑制候选化合物处理细菌脲酶,经酚次氯酸法检测脲酶抑制活性,结果见图 1。其中化合物Y041-7877的脲酶抑制活性高于其他候选化合物(图 1-A)。通过Graphpad prism非线性拟合(图 1-B)计算其IC50值为101.4 μmol/L。
|
图 1 候选化合物对瘤胃脲酶的抑制率(A)和不同浓度化合物Y041-7877下瘤胃脲酶活性的非线性拟合曲线(B) Fig. 1 Inhibition percentage of candidate compounds against ruminal urease (A) and nonlinear fitting curve of rumen urease activity under different concentrations of compound Y041-7877 (B) |
通过筛选获得了瘤胃脲酶抑制效果较好的化合物Y041-7877。通过酶动力学曲线进一步评价该化合物。在不同底物浓度条件下化合物Y041-7877对脲酶的酶促反应速度的影响见表 4。
|
|
表 4 不同浓度底物及化合物Y041-7877作用下脲酶的酶促反应速度 Table 4 Enzymatic reaction rate of urease under different concentrations of substrate and compound Y041-7877 |
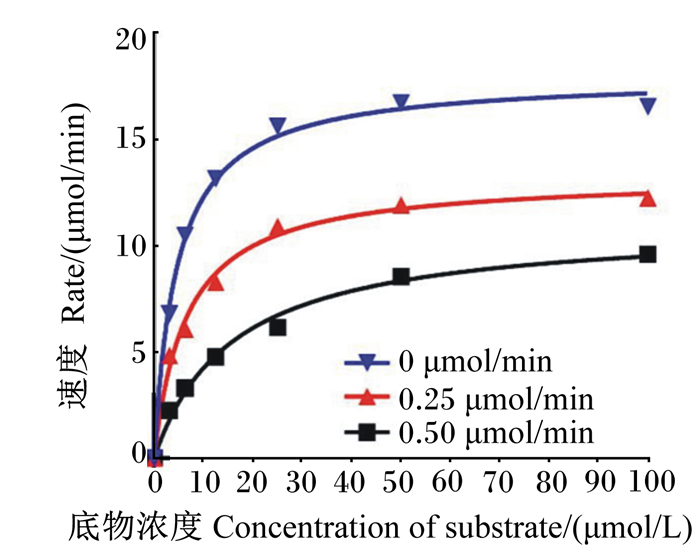
通过Graphpad prism 8数据分析功能分析非线性拟合过程,经拟合的不同浓度的化合物Y041-7877作用下的底物浓度与瘤胃脲酶的酶促反应速度的动力学曲线如图 2所示,获得了与该酶动力学曲线对应的Km值及Vmax值。化合物Y041-7877对瘤胃脲酶的动力学参数有影响,在未添加化合物Y041-7877时瘤胃脲酶的动力学曲线的参数为Km=4.630 μmol/L、Vmax=18.00 μmol/(min·L)、Vmax/Km=3.887 7;添加0.25 mmol/L化合物Y041-7877时的Km=6.574 μmol/L、Vmax=13.30 μmol/(min·L)、Vmax/Km=2.023 1;添加0.5 mmol/L化合物Y041-7877时的Km=15.930 μmol/L、Vmax=11.03 μmol/(min·L)、Vmax/Km=0.692 4。鉴于不同浓度抑制剂Y041-7877造成瘤胃脲酶动力学曲线的Km值与Vmax值同时发生变化, 且在不同浓度的化合物Y041-7877作用下的酶动力学曲线参数Vmax/Km亦发生相应变化。因此,认定化合物Y041-7877为混合型抑制剂。
|
图 2 不同浓度的化合物Y041-7877作用下脲酶的酶促反应动力学曲线 Fig. 2 Kinetics curves of urease under different concentrations of compound Y041-7877 |
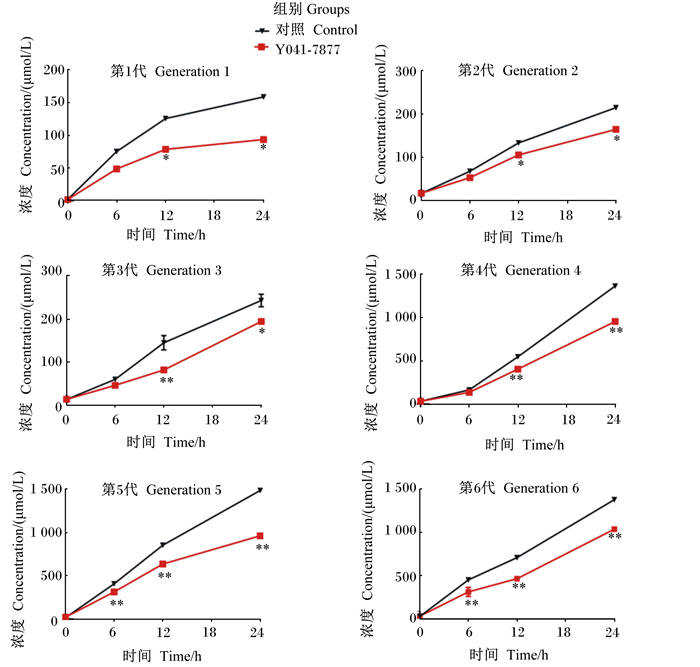
体外瘤胃微生物厌氧传代培养液中氨氮浓度随时间的变化见图 3。Y041-7877组培养液中氨氮浓度及积累速度(斜率)均小于对照组,表明化合物Y041-0047能抑制体外厌氧培养体系中瘤胃微生物的氮代谢,且抑制效果在传代培养各代间稳定。统计结果表明,在0 h时,各代中Y041-7877组与对照组间氨氮浓度均无显著差异(P>0.05)。在6 h时,传代培养的第1~4代Y041-7877组与对照组间无显著差异(P>0.05),第5~6代Y041-7877组培养体系中的氨氮浓度与对照组差异极显著(P < 0.01)。在12 h时,各代中2组间氨氮浓度差异均显著(P < 0.05),其中第3~6代中2组间氨氮浓度差异极显著(P < 0.01)。在第24 h时,各代中2组间氨氮浓度差异均显著(P < 0.05),其中第4~6代中2组间氨氮浓度差异极显著(P < 0.01)。
|
*表示Y041-7877组与对照组差异显著(P < 0.05), * *表示Y041-7877组与对照组差异极显著(P < 0.01). * indicated that the difference between Y041-7877 group and control group was significant (P < 0.05), and * * indicated that the difference between Y041-7877 group and control group was extremely significant (P < 0.01). 图 3 化合物Y041-7877对体外瘤胃微生物厌氧传代培养(共6代)体系中氨 Fig. 3 Effects of compound Y041-7877 on ammonia nitrogen concentration in anaerobic culture of ruminal bacteria in vitro (6 generations) |
分子对接后,用pymol分析化合物Y041-7877与目标脲酶蛋白结构的结合模式(图 4)。结果表明,化合物Y041-7877能与His246、His321形成氢键,与His166、Ala363、Asp360、Cys319、Glu220形成碳氢键。接近活性中心的苯环结构能与Asp221、Arg336形成Pi-Anion作用力。此外,化合物Y041-7877尾部的杂环结构还能与His320、Phe332形成Pi-Sigma、Pi-Pi stacked作用力。从二维对接图来看,化合物的形状能很好地吻合活性中心口袋,从而阻止底物尿素与脲酶的结合。
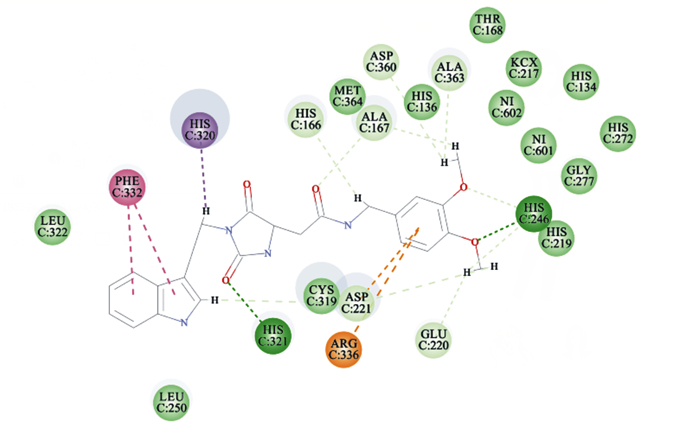
|
图中圆形表示脲酶活性中心中氨基酸残基,灰色实线表示化合物Y041-7877的化学结构。图中深绿色虚线表示氢键形成位置;浅绿色虚线表示碳氢键形成的位置;黄色虚线表示Pi-Anion作用力形成位置;紫色、粉色虚线分别表示Pi-Sigma、Pi-Pi stacked作用力形成位置。 The amino acid residues in the active site were shown as round objects, while the compounds were shown as gray lines. The hydrogen bonds were shown in the dark green dot lines. The carbon hydrogen bonds were shown in the light green dot lines. The Pi-Anion interactions were shown in yellow dot lines. The Pi-Sigma and Pi-Pi stacked interactions were shown in purple and pink dot lines, respectively. 图 4 化合物Y041-7877与产气克雷伯氏菌脲酶活性中心的二维对接图 Fig. 4 2D docking mode of compound Y041-7877 against Klebsiella aerogenes urease active center |
细菌脲酶蛋白结构、活性中心中关键的氨基酸残基具有保守性[8]。因此,以脲酶蛋白结构为靶标进行分子对接筛选脲酶抑制剂具有可行性[13, 18]。通过搜索PDB蛋白结构库,对比各已知结构分辨率及完整性,发现产气克雷伯氏菌脲酶(PDB_ID 4EP8)结构分辨率高于其他脲酶蛋白结构,为1.55 [19];同时,此脲酶蛋白结构分辨率较与之接近的其他脲酶蛋白结构更加完整,如脲酶蛋白晶体结构(PDB_ID 1EJX)在活性中心外部区域具有氨基酸残基缺失[20],这部分蛋白结构对于脲酶发挥活性十分重要[21],该区域缺失对于基于分子对接筛选脲酶抑制剂的效果影响很大。因此,最终确定使用产气克雷伯氏菌脲酶(PDB_ID 4EP8)作为靶标脲酶蛋白结构。基于此脲酶结构进行分子对接筛选ChemDiv化合物库,通过脲酶活性抑制试验及体外微生物厌氧培养验证,最终得到了1个脲酶抑制效果在微摩尔级的脲酶抑制小分子化合物,与以脲酶蛋白晶体结构1EJX为靶标进行分子对接筛选脲酶抑制剂的其他文献报道相比[6],本试验筛选获得了原本未知脲酶抑制活性的化合物,从侧面反映了靶标结构完整性对于分子对接结果的准确性影响很大。
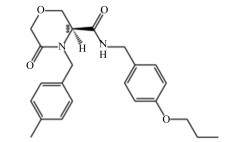
3.2 化合物Y041-7877的结构分析通过虚拟筛选及评价试验,获得了1个具有较高的脲酶抑制效果的小分子化合物Y041-7877,该化合物名称为N-(3, 4-二甲氧基苄基)-2-{(4S)-1-[2-(1H-吲哚-3-基)乙基]-2, 5-二氧杂咪唑啉-4-烃基}乙酰胺。该化合物结构由前部的二甲氧基苯基、中部的乙酰基及丙基咪唑基、尾部的苯并吡咯杂环基团构成,其中含有多个已报道具有重要脲酶抑制活性的结构。
化合物Y041-7877中含有的甲氧基是一种与脲酶抑制活性相关的重要基团,其脲酶抑制效果已在多篇报道中被证实,如Taha等[22]筛选一系列化合物,其中具有脲酶抑制效果且无毒性的化合物均含有甲氧基,表明甲氧基对于化合物的脲酶抑制活性具有重要作用。其他化合物筛选试验也证实了甲氧基对于化合物脲酶抑制效果的重要性[23-25]。
化合物Y041-7877中含有苯并吡咯杂环基团,为含氮杂环基团。近年来,有关含氮杂环类化合物的脲酶抑制活性的报道较多。含有该基团的化合物除与活性中心内部位点结合之外,还能与活性中心外的柔性片状区域结合,从而抑制脲酶活性,并且通常为混合性抑制剂[26]。因此,化合物Y041-7877很可能也具有此种作用模式。通过对该化合物的结合模式分析,发现其亦能与活性中心外柔性片状区域中Cys319位点相互作用,从而证明了此前的推测。
有关化合物Y041-7877的前、中、后部化学结构的组合模式亦多见报道[27-29],如Noreen等[28]报道,N-{[5-(4-氯苯基)噻吩-2-基]磺酰基}乙酰胺具有良好的脲酶抑制效果。此外,化合物芳环上的吸电子类取代基团亦能够增强化合物的脲酶抑制效果[30];化合物的化学结构中含有甲氧基有利于化合物的脲酶抑制效果[22]。对比化合物Y041-7877与N-{[5-(4-氯苯基)噻吩-2-基]磺酰基}乙酰胺的化学结构,发现与苯基相连的基团均为2个甲氧基而非卤族元素这类吸电子基团。虽然前人研究表明,甲氧基及芳环均有利于增强化合物的脲酶抑制效果,但甲氧基与苯环直接相连时对化合物脲酶抑制效果的影响还有待进一步研究。Pervez等[29]报道,将化合物中甲氧基的氢用卤族元素(如氟)取代所合成的化合物比取代前的化合物的脲酶抑制效果要增强5倍之多。因此,下一步可以以化合物Y041-7877作为先导化合物,优化其苯环上甲氧基中卤族元素的取代数量,以进一步挖掘其脲酶抑制潜力。
3.3 化合物Y041-7877瘤胃微生物体外培养评价Ludden等[31]研究了不同浓度正丁基磷酸三胺对瘤胃氨氮含量的影响,发现在瘤胃灌注该化合物的第2天,各浓度组的氨氮浓度随时间变化曲线均低于对照组,而在第15天,灌注该化合物后,各浓度组氨氮浓度随时间变化曲线均趋向对照组,部分较低浓度组氨氮浓度随时间变化曲线与对照组重合,表明随着抑制剂处理天数的增加,瘤胃微生物对正丁基磷酸三胺产生了适应性。因此,在筛选新的瘤胃脲酶抑制剂时,要评价瘤胃微生物是否对该化合物产生适应性。本研究通过基于分子对接的虚拟筛选技术从ChemDiv的未知活性化合物中筛选获得了1个瘤胃脲酶抑制效果较好的化合物Y041-7877,通过体外传代培养试验的方法来研究瘤胃微生物对该化合物是否存在适应性。从各代的氨氮浓度随时间变化曲线来看,化合物Y041-7877组的氨氮浓度随时间变化曲线没有趋近对照组曲线的趋势,初步表明化合物Y041-7877对瘤胃微生物氮代谢的抑制效果是稳定的。下一步可通过反刍动物研究模型,进一步研究该化合物对瘤胃氨氮代谢及其他发酵指标的影响。
3.4 化合物Y041-7877与脲酶的结合模式产气克雷伯氏菌脲酶的活性中心是由1个氨基甲酰化的赖氨酸(KCX217)作为桥键连接2个镍离子,镍离子间距离通常在3.5~3.7 [32]。镍离子会与活性中心的一些关键的氨基酸残基配对,组成脲酶的活性中心[33-34]。本试验中脲酶蛋白活性中心中关键的氨基酸残基为:His219、His246、His272、Gly277、His134、His136、Asp360[19]。活性中心外柔性片状区域中Cys319也是关键的氨基酸残基,能够控制柔性片状区域的开闭状态从而影响脲酶与底物的作用[21]。由二维对接图可知,化合物Y041-7877能与His246、Cys319结合,不仅能够通过竞争脲酶活性中心中有效位点来防止底物尿素与活性中心结合,还能通过固定活性中心外柔性片状区域的构象的方式来防止底物尿素进入活性中心,从而抑制脲酶活性。
4 结论① 通过基于分子对接的虚拟筛选及脲酶活性抑制试验构建脲酶抑制候选化合物筛选模型,获得了1个脲酶抑制效果较强的新型脲酶抑制化合物(ChemDiv_ID:Y041-7877),该化合物的IC50值为101.4 μmol/L,是混合性脲酶抑制剂。
② 体外微生物厌氧培养试验结果表明,化合物Y041-7877能显著降低微生物培养体系中氨氮浓度。
③ 分子对接结果表明,化合物Y041-7877在与脲酶蛋白活性中心的多个关键氨基酸残基结合的同时还能与活性中心外控制柔性片状区域开闭的半胱氨酸氨基酸残基结合。基于该化合物与脲酶的结合模式对该化合物相应部位进行结构优化,有望进一步提高该化合物的脲酶抑制效果。
| [1] |
PATRA A K, ASCHENBACH J R. Ureases in the gastrointestinal tracts of ruminant and monogastric animals and their implication in urea-N/ammonia metabolism:a review[J]. Journal of Advanced Research, 2018, 13: 39-50. DOI:10.1016/j.jare.2018.02.005 |
| [2] |
李晓姣, 赵圣国, 郑楠, 等. 细菌脲酶蛋白复合物及其活化机制[J]. 生物工程学报, 2019, 35(2): 204-215. |
| [3] |
ZAMBELLI B, MUSIANI F, BENINI S, et al. Chemistry of Ni2+ in urease:sensing, trafficking, and catalysis[J]. Accounts of Chemical Research, 2011, 44(7): 520-530. DOI:10.1021/ar200041k |
| [4] |
赵圣国, 王加启, 卜登攀, 等. 细菌尿素酶的生化和分子生物学特点[J]. 微生物学通报, 2008, 35(7): 1146-1152. DOI:10.3969/j.issn.0253-2654.2008.07.028 |
| [5] |
MOBLEY H L, ISLAND M D, HAUSINGER R P. Molecular biology of microbial ureases[J]. Microbiological Reviews, 1995, 59(3): 451-480. DOI:10.1128/MMBR.59.3.451-480.1995 |
| [6] |
MACOMBER L, MINKARA M S, HAUSINGER R P, et al. Reduction of urease activity by interaction with the flap covering the active site[J]. Journal of Chemical Information and Modeling, 2015, 55(2): 354-361. DOI:10.1021/ci500562t |
| [7] |
PEARSON M A, MICHEL L O, HAUSINGER R P, et al. Structures of Cys319 variants and acetohydroxamate-inhibited Klebsiella aerogenes urease[J]. Biochemistry, 1997, 36(26): 8164-8172. DOI:10.1021/bi970514j |
| [8] |
KRAJEWSKA B. Ureases Ⅰ.Functional, catalytic and kinetic properties:a review[J]. Journal of Molecular Catalysis B:Enzymatic, 2009, 59(1/2/3): 9-21. |
| [9] |
CHAN C C L, JONES G A. Effect of acetohydroxamic acid on growth and volatile fatty acid production by rumen bacteria[J]. Canadian Journal of Microbiology, 1973, 19(1): 27-33. DOI:10.1139/m73-004 |
| [10] |
ABBASI M A, HASSAN M, AZIZ-UR-REHMAN R, et al. Synthesis, in vitro and in silico studies of novel potent urease inhibitors:N-[4-({5-[(3-Un/substituted-anilino-3-oxopropyl)sulfanyl]-1, 3, 4-oxadiazol-2-yl}methyl)-1, 3-thiazol-2-yl]benzamides[J]. Bioorganic & Medicinal Chemistry, 2018, 26(13): 3791-3804. |
| [11] |
ABDEL-BAKY R M, ALI M A, ABUO-RAHMA G E D A A, et al.Inhibition of urease enzyme production and some other virulence factors expression in Proteus mirabilis by n-acetyl cysteine and dipropyl disulphide[M]//DONELLI G.Advances in microbiology.Cham: Springer, 2017: 99-113.
|
| [12] |
AHMED M, QADIR M A, HAMEED A, et al. Azomethines, isoxazole, N-substituted pyrazoles and pyrimidine containing curcumin derivatives:urease inhibition and molecular modeling studies[J]. Biochemical and Biophysical Research Communications, 2017, 490(2): 434-440. DOI:10.1016/j.bbrc.2017.06.059 |
| [13] |
AZIZIAN H, NABATI F, SHARIFI A, et al. Large-scale virtual screening for the identification of new Helicobacter pylori urease inhibitor scaffolds[J]. Journal of Molecular Modeling, 2012, 18(7): 2917-2927. DOI:10.1007/s00894-011-1310-2 |
| [14] |
LIPINSKI C A. Drug-like properties and the causes of poor solubility and poor permeability[J]. Journal of Pharmacological and Toxicological Methods, 2000, 44(1): 235-249. DOI:10.1016/S1056-8719(00)00107-6 |
| [15] |
赵圣国, 王加启, 刘开朗, 等. 荷斯坦奶牛瘤胃微生物脲酶的分离与鉴定[J]. 畜牧兽医学报, 2010, 41(6): 692-696. |
| [16] |
WEATHERBURN M W. Phenol-hypochlorite reaction for determination of ammonia[J]. Analytical Chemistry, 1967, 39(8): 971-974. DOI:10.1021/ac60252a045 |
| [17] |
王加启. 反刍动物营养学研究方法[M]. 北京: 现代教育出版社, 2011.
|
| [18] |
RASHID U, BATOOL I, WADOOD A, et al. Structure based virtual screening-driven identification of monastrol as a potent urease inhibitor[J]. Journal of Molecular Graphics and Modelling, 2013, 43: 47-57. DOI:10.1016/j.jmgm.2013.04.006 |
| [19] |
WARKENTIN M, BADEAU R, HOPKINS J B, et al. Spatial distribution of radiation damage to crystalline proteins at 25-300 K[J]. Acta Crystallographica Section D:Biological Crystallography, 2012, 68(9): 1108-1117. DOI:10.1107/S0907444912021361 |
| [20] |
PEARSON M A, KARPLUS P A.Crystal structure of wild-type klebsiella aerogenes urease at 100K[J/OL]. Protein Data Bank.2003, [2020-04-05] https://www.rcsb.org/structure/1EJX
|
| [21] |
ROBERTS B P, Miller ⅢB R, ROITBERG A E, et al. Wide-open flaps are key to urease activity[J]. Journal of the American Chemical Society, 2012, 134(24): 9934-9937. DOI:10.1021/ja3043239 |
| [22] |
TAHA M, ISMAIL N H, IMRAN S, et al. Synthesis of potent urease inhibitors based on disulfide scaffold and their molecular docking studies[J]. Bioorganic & Medicinal Chemistry, 2015, 23(22): 7211-7218. |
| [23] |
RAHIM F, ALI M, ULLAH S, et al. Development of bis-thiobarbiturates as successful urease inhibitors and their molecular modeling studies[J]. Chinese Chemical Letters, 2016, 27(5): 693-697. DOI:10.1016/j.cclet.2015.12.035 |
| [24] |
MUHAMMAD M T, KHAN K M, ARSHIA, et al. Syntheses of 4, 6-dihydroxypyrimidine diones, their urease inhibition, in vitro, in silico, and kinetic studies[J]. Bioorganic Chemistry, 2017, 75: 317-331. DOI:10.1016/j.bioorg.2017.08.018 |
| [25] |
IFTIKHAR F, ALI Y, KIANI F A, et al. Design, synthesis, in vitro evaluation and docking studies on dihydropyrimidine-based urease inhibitors[J]. Bioorganic Chemistry, 2017, 74: 53-65. DOI:10.1016/j.bioorg.2017.07.003 |
| [26] |
HAMEED A, AL-RASHIDA M, UROOS M, et al. A patent update on therapeutic applications of urease inhibitors (2012—2018)[J]. Expert Opinion on Therapeutic Patents, 2019, 29(3): 181-189. DOI:10.1080/13543776.2019.1584612 |
| [27] |
SAIFY Z, SULTANA N, KHAN A, et al. (1H-pyrrolo[2, 3-b]pyridine)7-azaindole derivatives and their antiurease, phosphodiesterase and β-glucuronidase activity[J]. International Journal of Biochemistry Research & Review, 2015, 8(1): 1-12. |
| [28] |
NOREEN M, RASOOL N, GULL Y, et al. Synthesis, density functional theory (DFT), urease inhibition and antimicrobial activities of 5-aryl thiophenes bearing sulphonylacetamide moieties[J]. Molecules, 2015, 20(11): 19914-19928. DOI:10.3390/molecules201119661 |
| [29] |
PERVEZ H, CHOHAN Z H, RAMZAN M, et al. Synthesis and biological evaluation of some new N4-substituted isatin-3-thiosemicarbazones[J]. Journal of Enzyme Inhibition and Medicinal Chemistry, 2009, 24(2): 437-446. DOI:10.1080/14756360802188420 |
| [30] |
TAHA M, ISMAIL N H, IMRAN S, et al. Hybrid benzothiazole analogs as antiurease agent:synthesis and molecular docking studies[J]. Bioorganic Chemistry, 2016, 66: 80-87. DOI:10.1016/j.bioorg.2016.03.010 |
| [31] |
LUDDEN P A, HARMON D L, HUNTINGTON G B, et al. Influence of the novel urease inhibitor N-(n-butyl) thiophosphoric triamide on ruminant nitrogen metabolism:Ⅱ.Ruminal nitrogen metabolism, diet digestibility, and nitrogen balance in lambs[J]. Journal of Animal Science, 2000, 78(1): 188-198. DOI:10.2527/2000.781188x |
| [32] |
CARTER E L, FLUGGA N, BOER J L, et al. Interplay of metal ions and urease[J]. Metallomics, 2009, 1(3): 207-221. DOI:10.1039/b903311d |
| [33] |
PEARSON M A, PARK I S, SCHALLER R A, et al. Kinetic and structural characterization of urease active site variants[J]. Biochemistry, 2000, 39(29): 8575-8584. DOI:10.1021/bi000613o |
| [34] |
赵圣国.牛瘤胃脲酶基因多样性分析与酶活性调控[D].博士学位论文.北京: 中国农业科学院, 2012.
|