2. 四川省草原科学研究院, 成都 611731
2. Sichuan Academy of Grassland Sciences, Chengdu 611731, China
瘤胃微生物与纤维饲料的消化[1-2]、营养物质的吸收[3]和瘤胃壁绒毛的发育相关[4],在反刍动物中发挥着重要的作用[5-6]。瘤胃通过微生物发酵可以利用大量纤维饲料,这对反刍动物的维持生长和生长性能至关重要[7],瘤胃微生物的研究可为反刍动物提供一定的饲养策略[8-9]。瘤胃生态系统中微生物群落的组成和代谢功能是瘤胃微生物领域的研究重点[10]。从宏基因组学的角度探讨瘤胃微生物[11],可进一步了解瘤胃微生物代谢功能及对反刍动物的调控机理[12]。在不同饲养条件下瘤胃微生物的动态变化可为研究人员提供有利的信息,有助于了解瘤胃微生物的作用机理。
牦牛生长在高原海拔地区,主要以高山牧草为食,有较高的植物纤维利用率。牦牛瘤胃微生物变化受其自身的遗传、瘤胃调节和年龄等因素的影响,还受到牦牛采食的种类、季节和环境变化的影响[13]。为提高牦牛的生长性能,解决因高原地区冬春气候恶劣、牧草消耗殆尽造成的牦牛掉膘和死亡等养殖问题,研究者提出了用舍饲养殖进行高效饲养管理[14-16]。在牦牛的舍饲养殖中,养殖场地的选择、养殖期饲养管理和养殖时间的把握等都对牦牛的生长性能产生着不同程度的影响[17]。目前,基于新的测序技术对反刍动物瘤胃微生物的建立过程已有一定的研究,但牦牛瘤胃微生物的研究较少[18]。为了研究舍饲养殖对牦牛生长性能和瘤胃微生物的影响,本研究采用宏基因组测序方法分析麦洼牦牛瘤胃微生物的变化及牦牛生长性能与瘤胃微生物之间的关系,旨在为麦洼牦牛的健康养殖提供参考依据。
1 材料与方法 1.1 试验动物与饲养管理选取10只2~3岁的健康雄性麦洼牦牛为试验动物,体重在150~180 kg,采用完全随机试验设计,将其分为舍饲组和放牧组,每组5只。放牧组牦牛于海拔高、气候严寒的红原县牦牛科技园区放牧,以天然牧草为食。舍饲组牦牛于四川省广汉市进行舍饲养殖,饲喂基础饲粮,基础饲粮参照《肉牛饲养标准》(NY/T 815—2004)配制,以水稻干草、黑麦干草、燕麦干草和小麦酒糟为粗料,具体组成及营养水平见表 1,基础饲粮预饲30 d,每日07:00和18:00饲喂。正试期为4个月。预试期和正试期为冬、春季节。
|
|
表 1 基础饲粮组成及营养水平(风干基础) Table 1 Composition and nutrient levels of the basal diet (air-dry basis) |
试验期间每隔30 d对牦牛体重进行称重,计算试验第1~30天、第31~60天、第61~90天、第91~120天、第121~150天的平均日增重,计算试验第1~150天平均增重。养殖结束后,每组5只牦牛进行屠宰,并采集瘤胃液各50 mL,4层灭菌纱布过滤之后冻存于液氮中。
1.3 测序文库的构建和高通量测序用DNA提取试剂盒(天根生化科技北京有限公司)提取基因组DNA,检测合格的DNA样品用超声破碎仪打断成约350 bp的小片段,这些片段经末端修复、加A尾、加测序接头、纯化、PCR扩增等过程对文库进行构建。构建的文库进行检测,合格后按有效浓度进行Illumina PE150测序,测序由北京诺禾致源生物信息科技有限公司完成。
1.4 瘤胃微生物宏基因组数据分析使用Readfq对测序获得的原始数据进行质控和过滤,获得有效数据,对其进行Metagenome组装,组装后采用MetaGeneMark进行基因预测,综合有效数据获得样品中的丰度信息。从gene catalogue出发,用DIAMOND软件与MicroNR库进行比对,比对结果采用LCA算法确定物种的注释信息,结合基因丰度表,获得不同分类层级的物种丰度表。用DIAMOND软件与功能数据库进行比对,从比对结果统计不同功能层级的相对丰度,从而进一步对代谢通路(KEGG)、同源基因簇(eggNOG)、碳水化合物酶(CAZy)进行功能注释和丰度分析。
1.5 数据处理试验数据采用Excel 2016进行初步处理,用SPSS 26.0统计软件对牦牛生长性能数据进行单因素方差分析,并用Duncan氏法进行多重比较,P < 0.05为差异显著。用统计学软件中的独立样本t检验过程来完成KEGG、eggNOG和CAZy相对丰度的差异显著性检验,并用Levene检验程序进行方差齐性检验。
2 结果与分析 2.1 牦牛体重变化由表 2可知,在整个试验期间,舍饲组牦牛平均增重和第1~30天、第31~60天、第61~90天平均日增重与放牧组相比均显著增加(P < 0.05)。
|
|
表 2 舍饲养殖对牦牛生长性能的影响 Table 2 Effects of confined farming on growth performance of yaks |
本试验测序数据组装后共得到1 484 705 776 bp的Scaftigs。基因预测共得到2 065 638个开放阅读框(ORFs),去冗余后,完整基因数为333 371个,所占比例为21.21%。非冗余基因集注释到属和门的比例分别为51.99%和69.89%,有63 377个ORFs比对到CAZy数据库,780 249个ORFs比对到KEGG数据库,770 270个ORFs比对到eggNOG数据库。基因注释结果显示舍饲组与放牧组之间基因数目具有差异,舍饲组的基因数目比放牧组的基因数目少53 320个,在注释到的基因中,舍饲组与放牧组有255 882个基因具有差异。
2.3 物种相对丰度分析由表 3可知,在门水平上,放牧组拟杆菌门(Bacteroidetes)的相对丰度最高(27.9%),其次为厚壁菌门(Firmicutes,20.6%)、纤维杆菌门(Fibrobacteres,2.5%)、壶菌门(Chytridiomycota,1.9%);舍饲组厚壁菌门的相对丰度最高(36.8%),其次为拟杆菌门(31.3%)、变形菌门(Proteobacteria,0.9%)。与放牧组相比,舍饲组厚壁菌门、拟杆菌门、软壁菌门(Tenericutes)、浮霉菌门(Candidatus)的相对丰度均增加(P>0.05),纤维杆菌门相对丰度显著下降(P < 0.05),壶菌门、子囊菌门(Ascomycota)、变形菌门、毛霉门(Mucoromycota)、螺旋体门(Spirochaetes)相对丰度均下降(P>0.05)。
|
|
表 3 舍饲养殖对牦牛瘤胃菌群门水平相对丰度的影响 Table 3 Effects of confined farming on relative abundance of phylum level in rumen flora of yaks |
由表 4可知,在属水平上,放牧组普雷沃氏菌属(Prevotella)的相对丰度最高(12.3%),其次为拟杆菌属(Bacteroides,3.0%)、丁酸弧菌属(Butyrivibrio,2.5%)、丝状杆菌属(Fibrobacter,2.5%);舍饲组普雷沃氏菌属的相对丰度最高(14.3%),其次为梭菌属(Clostridium,6.3%)、拟杆菌属(3.5%)、丁酸弧菌属(2.6%)。与放牧组相比,舍饲组普雷沃氏菌属、梭菌属、拟杆菌属、丁酸弧菌属、假丁酸弧菌属(Pseudobutyrivibrio)、另枝菌属(Alistipes)、产琥珀酸菌属(Succiniclasticum)相对丰度均增加(P>0.05),丝状杆菌属(Fibrobacter)相对丰度显著下降(P < 0.05),瘤胃球菌属(Ruminococcus)、密螺旋体属(Treponema)相对丰度均下降(P>0.05)。
|
|
表 4 舍饲养殖对牦牛瘤胃菌群属水平相对丰度的影响 Table 4 Effects of confined farming on relative abundance of genus level in rumen flora of yaks |
由表 5可知,与放牧组比较,舍饲组糖苷水解酶(GH)、糖基转移酶(GT)相对丰度增加,而碳水化合物结合模块(CBM)、碳水化合物酯酶(CE)、多聚体裂解酶(PL)、辅助活性酶系(AA)相对丰度均下降(P>0.05)。这表明牦牛瘤胃微生物中降解食物中植物纤维素最主要的CAZy是GH家族,舍饲养殖对GT相对丰度的上调和CBM相对丰度的下调影响最大。
|
|
表 5 舍饲养殖对牦牛瘤胃微生物CAZy酶类相对丰度的影响 Table 5 Effects of confined farming on relative abundance of CAZy enzymes in rumen microbes of yaks |
为了筛选组间具有显著差异的功能Biomarker,首先通过秩和检验的方法检测不同组间的差异功能并通过线性判别分析(LDA)实现降维,并评估差异功能的影响大小,即得到LDA值。结果显示,舍饲组较放牧组,糖苷水解酶家族中CH36、CH38的功能具有显著差异,差异功能的LDA值分布如图 1所示。
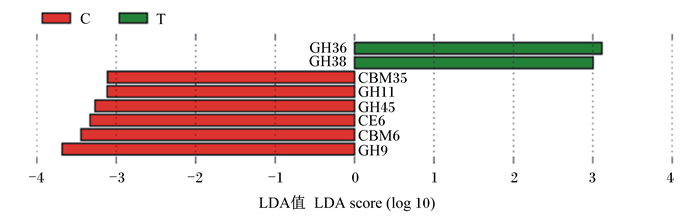
|
C:放牧组grazing group; T:舍饲组feeding group;GH36\38\11\45\9:糖苷水解酶36\38\11\45\9 glucoside hydrolase 36\38\11\45\9;CBM35\6:碳水化合物结合模块35\6 carbohydrate-binding module 35\6;CE6:碳水化合物酯酶6 carbohydrate esterase 6。 图 1 差异功能的LDA值分布图 Fig. 1 LDA value distribution of difference function |
由表 6可知,舍饲组较放牧组微生物复制、重组和修复功能蛋白相对丰度增加最为明显(P>0.05),其次为细胞壁/膜/包膜生物发生蛋白;氨基酸转运和代谢等功能蛋白、信号转导机制等功能类蛋白相对丰度下降(P>0.05)。
|
|
表 6 舍饲养殖对牦牛瘤胃微生物eggNOG功能蛋白相对丰度的影响 Table 6 Effects of confined farming on relative abundance of eggNOG functional protein in rumen microbes of yaks |
通过KEGG数据库,对六大代谢通路进行分析,进一步在Level 2层级上相对丰度最高的10个通路进行组间差异比较。由表 7可知,与放牧组相比,舍饲组中碳水化合物代谢、氨基酸代谢、核苷酸代谢、能量代谢、辅助因子和维生素的代谢、膜运输、复制和修复通路的相对丰度增加,翻译、信号转导、运输和分解代谢通路的相对丰度减少(P>0.05)。
|
|
表 7 舍饲养殖对牦牛瘤胃微生物KEGG代谢通路相对丰度的影响 Table 7 Effects of confined farming on relative abundance of KEGG metabolic pathway in rumen microbes of yaks |
本研究结果显示,厚壁菌门和拟杆菌门均是瘤胃微生物中的优势菌门,这与安雅静等[19]、闫小婷[20]对牦牛瘤胃进行的宏基因组测序结果一致。本试验发现,与放牧组相比,舍饲组厚壁菌门和拟杆菌门相对丰度均增加,这与徐晓锋等[21]对奶牛研究中高精粗比饲粮可降低瘤胃厚壁菌门和拟杆菌门丰度的结果有所差异。由于本试验中牦牛舍饲养殖过程中仍然以粗料为主,且舍饲过程中仍有大量的牧草投喂,虽然拟杆菌门相对丰度没有降低,但改变了组成结构,使厚壁菌门相对丰度作为优势菌群高于拟杆菌门。梭菌属是厚壁菌门中的一类有益细菌,舍饲养殖使其相对丰度增加,这与舍饲养殖为牦牛提供能量饲料,被梭菌属利用并产生短链脂肪酸为机体提供能量有关。梭菌属还可能通过代谢产酶降解膳食纤维方式在猪的机体能量过度摄入中发挥重要作用[22]。舍饲养殖牦牛与相对以牧草为主要食物的高原放牧牦牛获得更丰富的膳食纤维,提示梭菌属在牦牛瘤胃对食物的消化吸收中起着重要的作用。拟杆菌门具有促进脂肪沉积的作用,能够促进宿主吸收和储存能量[23]。牦牛体重在舍饲养殖模式下有所增加,这与拟杆菌门等可在瘤胃中促进食物的消化吸收,从而促进脂肪的沉积相关;另外,还与舍饲养殖的环境有关,舍饲养殖在圈舍内进行饲养,牦牛的活动范围受到限制,能量消耗减少,所以其体重增加。高原地区的寒冷环境,牧草缺乏,需要消耗自身的能量在相对恶劣的环境下生存,而舍饲环境温和[24],所以舍饲养殖的牦牛瘤胃微生物属水平相对丰度的变化可能由于舍饲环境变化所引起的。此外,舍饲养殖组中琥珀酸菌属的相对丰度比放牧组高,由于琥珀酸是三羧酸循环(TCA)的中间产物,是一些微生物厌氧代谢的发酵产物之一[25-26],所以产琥珀菌属相对丰度的增加对于瘤胃中的代谢活动起着重要的作用。
3.2 舍饲养殖对瘤胃微生物CAZy酶类相对丰度的影响复合碳水化合物的多样性由参与生物体或生物体之间组装和分解的一组酶控制,这组酶统称为CAZy[27]。头孢喹肟(CEF)[19]、黄曲霉毒素B1(AFB1)[28]、二氟沙星[29]均能够导致瘤胃微生物CAZy的丰度发生改变。瘤胃微生物是CAZy的丰富来源,其可用于转化纤维素,包括转化为生物燃料,并参与许多生物过程,如碳水化合物代谢、蛋白质糖基化和植物生物量合成以及不同生态系统中的降解[26]。由于对牦牛进行舍饲养殖使瘤胃菌群发生了改变,从而导致其产生的碳水化合物酶类各家族基因丰度发生变化,因此可以推断舍饲养殖对牦牛瘤胃微生物降解纤维素产生了影响。李晓等[30]的试验表明,反刍动物瘤胃微生物区系存在明显差异,而且饲粮类型影响糖苷水解酶基因的转录和表达,舍饲牦牛由于营养水平较高,对糖苷水解酶基因的表达有所提高。天然产物糖基转移酶对糖基底物具有高度的特异性[31-32],在舍饲养殖牦牛瘤胃中,其糖基化水平会有所提高。CBM是CAZy的重要组成部分,其功能是识别并结合到多糖底物上以增加催化模块在底物中的浓度并提高活性[33],在舍饲养殖牦牛瘤胃中,由于营养成分较高,有利于提高多模块CAZy的活性。
3.3 舍饲养殖对蛋白代谢通路相对丰度的影响由于舍饲养殖对微生物的影响,其中微生物中的碳水化合物的运输代谢、能源生产、辅酶转运等蛋白的相对丰度均增加,舍饲养殖改变了微生物的蛋白功能,这在一定程度上可能影响微生物在牦牛体内所起的功能作用,微生物蛋白质参与能量的生产和运输过程是相当重要的[34]。此外,KEGG通路不仅提供生化物质相互转化所有可能的代谢途径,还包含对催化各步反应的酶的全面注解[35],舍饲养殖导致部分通路的丰度发生改变,即在一定程度上也对酶的丰度和相关功能作用有影响,但在一定程度上基于通路来探讨酶的相互作用还需要进一步研究。
4 结论舍饲养殖显著增加了麦洼牦牛的体重,影响了牦牛瘤胃微生物的组成结构,改变其门和属的物种丰度,对拟杆菌属-普雷沃氏菌属相对丰度影响最大。牦牛瘤胃微生物中降解食物中植物纤维素最主要的CAZy是GH家族,舍饲养殖对GT相对丰度的上调和CBM相对丰度的下调影响最大。此外,还增加了牦牛瘤胃微生物转录、辅酶转运和代谢等功能蛋白的相对丰度,增加了碳水化合物代谢、氨基酸代谢和核苷酸代谢通路的相对丰度。
| [1] |
JAMI E, ISRAEL A, KOTSER A, et al. Exploring the bovine rumen bacterial community from birth to adulthood[J]. The ISME Journal, 2013, 7(6): 1069-1079. DOI:10.1038/ismej.2013.2 |
| [2] |
雷海波, 王杰. 不同处理方法提高反刍动物对粗饲料纤维消化力的机理研究[J]. 畜牧兽医杂志, 2019, 38(4): 50-52, 54. |
| [3] |
MCGRATH J, DUVAL S M, TAMASSIA L F M, et al. Nutritional strategies in ruminants:a lifetime approach[J]. Research in Veterinary Science, 2018, 116: 28-39. DOI:10.1016/j.rvsc.2017.09.011 |
| [4] |
BRULC J M, ANTONOPOULOS D A, MILLER M E B, et al. Gene-centric metagenomics of the fiber-adherent bovine rumen microbiome reveals forage specific glycoside hydrolases[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(6): 1948-1953. DOI:10.1073/pnas.0806191105 |
| [5] |
LEAN I J, GOLDER H M, HALL M B. Feeding, evaluating, and controlling rumen function[J]. Veterinary Clinics of North America:Food Animal Practice, 2014, 30(3): 539-75. DOI:10.1016/j.cvfa.2014.07.003 |
| [6] |
米见对, 廖新俤, 周建伟, 等. 反刍动物瘤胃微生物的建立过程与调控研究进展[J]. 家畜生态学报, 2019, 40(7): 1-8. |
| [7] |
ZHANG J, SHI H T, WANG Y J, et al. Effect of dietary forage to concentrate ratios on dynamic profile changes and interactions of ruminal microbiota and metabolites in Holstein heifers[J]. Frontiers in Microbiology, 2017, 8: 2206. DOI:10.3389/fmicb.2017.02206 |
| [8] |
SASSON G, BEN-SHABAT S K, SEROUSSI E, et al. Heritable bovine rumen bacteria are phylogenetically related and correlated with the cow's capacity to harvest energy from its feed[J]. mBio, 2017, 8(4): e00703-17. DOI:10.1128/mBio.00703-17 |
| [9] |
MORGAVI D P, KELLY W J, JANSSEN P H, et al. Rumen microbial (meta) genomics and its application to ruminant production[J]. Animal, 2013, 7(Suppl.1): 184-201. |
| [10] |
牛化欣, 常杰, 胡宗福, 等. 基于组学技术研究反刍动物瘤胃微生物及其代谢功能的进展[J]. 畜牧兽医学报, 2019, 50(6): 1113-1122. |
| [11] |
LI R W, CONNOR E E, LI C J, et al. Characterization of the rumen microbiota of pre-ruminant calves using metagenomic tools[J]. Environmental Microbiology, 2012, 14(1): 129-39. DOI:10.1111/j.1462-2920.2011.02543.x |
| [12] |
彭帅.应用宏基因组方法检测猪致病微生物及分析牛胃菌群组成[D].博士学位论文.长春: 吉林大学, 2015. http://cdmd.cnki.com.cn/Article/CDMD-10183-1015590923.htm
|
| [13] |
梁泽毅, 张剑搏, 李晨, 等. 牦牛瘤胃微生物研究进展[J]. 中国草食动物科学, 2019, 39(2): 52-55. |
| [14] |
马国军, 马进寿. 育肥牦牛高效饲养管理技术[J]. 畜牧兽医科学(电子版), 2019(5): 95-96. |
| [15] |
官久强, 安添午, 谢荣清, 等. 农区牦牛、犏牛舍饲短期育肥效益分析[J]. 中国奶牛, 2019(7): 17-20. |
| [16] |
杨昌福, 柏雪, 高彦华, 等. 全放牧与舍饲育肥对牦牛肉品质及安全性的影响[J]. 畜牧与兽医, 2019, 51(1): 23-28. |
| [17] |
张成祥. 浅谈牦牛育肥的技术要点[J]. 养殖与饲料, 2019(6): 32-33. |
| [18] |
徐俊.不同牧草来源的NDF在瘤胃中降解特性及其对细菌群落结构的影响[D].博士学位论文.扬州: 扬州大学, 2014. http://d.wanfangdata.com.cn/Thesis_Y2631872.aspx
|
| [19] |
安雅静, 文勇立, 赵佳琦, 等. 头孢喹肟(CEF)影响牦牛瘤胃微生态的宏基因组学分析[J]. 黑龙江畜牧兽医, 2019(5): 136-141, 182. |
| [20] |
闫小婷.缓释尿素对牦牛瘤胃细菌和真菌群落组成的影响[D].硕士学位论文.兰州: 兰州大学, 2019. http://cdmd.cnki.com.cn/Article/CDMD-10730-1019874445.htm
|
| [21] |
徐晓锋, 胡丹丹, 郭婷婷, 等. 不同精粗比饲粮条件下奶牛瘤胃细菌菌群结构变化的研究[J]. 动物营养学报, 2019, 31(12): 5541-5550. |
| [22] |
杨华.金华猪肠道微生物结构解析及其与脂肪沉积的关联分析[D].博士学位论文.北京: 中国农业大学, 2018. http://cdmd.cnki.com.cn/Article/CDMD-10019-1018069223.htm
|
| [23] |
郭秀兰.猪肠道硬壁菌门和拟杆菌门数量的检测及其相对丰度与脂肪沉积的相关性研究[D].博士学位论文.雅安: 四川农业大学, 2009. http://cdmd.cnki.com.cn/Article/CDMD-10626-2009258864.htm
|
| [24] |
柏雪, 文勇立, 刘少文, 等. 青藏高原部分地区自由放牧与舍饲育肥牦牛食源性致病微生物污染的差异[J]. 食品科学, 2017, 38(17): 66-71. |
| [25] |
林日辉, 黄新林, 黄文勤, 等. 产琥珀酸菌的快速筛选及鉴定[J]. 食品科技, 2012, 37(1): 13-16. |
| [26] |
LIU C, LI X H, CHEN Y X, et al. Age-related response of rumen microbiota to mineral salt and effects of their interactions on enteric methane emissions in cattle[J]. Microbial Ecology, 2017, 73(3): 590-601. DOI:10.1007/s00248-016-0888-4 |
| [27] |
LOMBARD V, RAMULU H G, DRULA E, et al. The carbohydrate-active enzymes database (CAZy) in 2013[J]. Nucleic Acids Research, 2014, 42(D1): D490-D495. DOI:10.1093/nar/gkt1178 |
| [28] |
安雅静, 文勇立, 赵佳琦, 等. 宏基因组学揭示AFB1对牦牛瘤胃微生物多样性及CAZy谱影响[J]. 家畜生态学报, 2019, 40(2): 13-20. |
| [29] |
赵佳琦, 文勇立, 安雅静, 等. 宏基因组学揭示二氟沙星对牦牛瘤胃微生态及CAZy谱影响[J]. 畜牧与兽医, 2019, 51(3): 62-69. |
| [30] |
李晓, 杨玉霞, 汪艳, 等. 不同日粮类型对绵羊瘤胃微生物木聚糖酶基因多样性的影响[J]. 新疆农业大学学报, 2014, 37(3): 173-180. |
| [31] |
林钦恒, 肖吉, 陈瑞东, 等. 微生物糖苷类抗生素糖基转移酶的体外研究进展[J]. 中国抗生素杂志, 2012, 37(12): 881-895. |
| [32] |
EGOROVA K S, TOUKACH P V. CSDB_GT:a new curated database on glycosyltransferases[J]. Glycobiology, 2016, 27(4): 285-290. |
| [33] |
张雨, 王晓燕, 张鑫宇, 等. CBM的结构、应用及与多糖的相互作用研究进展[J]. 纤维素科学与技术, 2019, 27(2): 66-73, 84. |
| [34] |
王炳.饲喂秸秆日粮奶牛泌乳性能低下的消化吸收与代谢机制研究[D].博士学位论文.杭州: 浙江大学, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10335-1016267422.htm
|
| [35] |
李向真, 刘子朋, 李娟, 等. KEGG数据库的进展及其在生物信息学中的应用[J]. 药物生物技术, 2012, 19(6): 535-539. |




