近年来,饲用抗生素因具有促进生长、提高饲料利用率、降低死亡率和发病率等特点,而被广泛应用于集约化养猪生产中。但由于抗生素不规范使用,从而造成猪体内细菌耐药性增强[1],畜产品和畜禽废弃物中抗生素残留超标[2],严重危害了健康和环境。对此世界各国均出台了一系列的法律法规,减少或者禁止在养殖环节中使用抗生素。至此,在自然界广泛存在、安全高效的益生菌进入到了人们的视野,逐渐成为研究热点。贝莱斯芽孢杆菌具有促进植物生长和抵御病原微生物的作用,是一种新型的生物防治菌[3-4]。目前该菌的主要研究方向为:植物病害、生物防控及水产动物肉质保鲜等方面。本试验前期研究显示,贝莱斯芽孢杆菌具有良好的大肠杆菌K88、金黄色葡萄球菌和鼠伤寒沙门氏菌拮抗效果[5],其生长过程产生絮凝物质,能吸附絮凝微小物质,但其对猪的生长、粪菌微生物多样性和代谢产物的影响目前未见报道。因此,本试验旨在进一步挖掘和验证该菌的使用范围和效果,了解和掌握其对猪生长性能、代谢产物和粪便中微生物多样性的影响,以期为贝莱斯芽孢杆菌替代抗生素在生产上的应用提供理论依据。
1 材料与方法 1.1 试验材料试验所用贝莱斯芽孢杆菌B13为本试验室从迪庆藏猪肠道内容物中分离所得。
1.2 试验动物从云南省畜牧兽医科学院云上飘香猪杂交选育猪群中,选择无病史、体重为(34.00±0.25)kg、去势的生长猪(含滇南小耳猪、迪庆藏猪、太湖猪和杜洛克血缘均为25%)20头,按照完全随机区组设计,根据猪的日龄和体重随机分为2组,每组10头,对照组饲喂基础饲粮,试验组饲喂基础饲粮+107 CFU/kg贝莱斯芽孢杆菌B13。
1.3 试验饲粮及管理参照肉脂型猪饲养标准(NY/T 65—2004)进行基础饲粮配方设计,云南东道饲料有限公司根据配方采购优质原料生产加工成颗粒饲料,基础饲粮组成及营养水平见表 1。饲养试验在云南省畜牧兽医科学院种猪质量检验测试中心种猪测定舍内进行,全阶段自由采食和自由饮水,免疫,驱虫、消毒均按照管理程序进行,预试期为7 d,试验期为21 d。
|
|
表 1 基础饲粮组成及营养水平(风干基础) Table 1 Composition and nutrient levels of the basal diet (air-dry basis) |
试验期开始和结束前禁食12 h,对试验猪只称重并记录,用全自动采食测定系统(ACEMO64)进行采食量测定,试验结束后计算平均日增重、平均日采食量和料重比。试验结束前1天,每组随机选择6头猪收集粪便样品,取刚排泄的粪便中间部分,液氮中速冻后取出,-80 ℃保存,用于后续分析。
1.5 16S rRNA PCR扩增总DNA抽提使用试剂盒(Omega Bio-Tek,Norcross,美国)完成,DNA浓度和纯度利用NanoDrop2000检测;用338F(5’-ACTCCTACGGGAGGCAGCAG-3’)和806R(5’-GGACTACHVGGGTWTCTAAT-3’)引物对V3~V4可变区进行PCR扩增,回收PCR产物并纯化、洗脱和检测。利用QuantiFluorTM-ST (Promega, 美国)进行检测定量。
1.6 高通量测序和数据分析使用Trimmomatic软件对原始测序序列质控,FLASH软件进行拼接。根据Illumina MiSeq平台构建PE300的文库。根据97%的相似度对序列进行操作分类单位(OTU)聚类,并去除单序列和嵌合体,通过I-Sanger云平台数据库(https://cloud.majorbio.com/)对每条序列进行物种分类注释。
1.7 代谢组学分析样品通过前处理去除杂质、提取代谢物之后,使用液质联用技术(LC-MS、ABSCIEX、UPLC-TripleTOF)检测,采用Progenesis QI(waters corporation, Milford, 美国)软件进行代谢物注释、数据预处理等,最终得到代谢物列表和数据矩阵,结合t检验和VIP(OPLS-DA)筛选出差异的代谢物,进一步分析并对差异代谢物的生物学信息进行挖掘。
1.8 数据分析采用SPSS 17.0统计软件进行单因素方差分析(one-way ANOVA)和LSD法多重比较,P < 0.05为差异显著,P < 0.01为差异极显著。数据以平均值±标准差表示。
2 结果与分析 2.1 贝莱斯芽孢杆菌对猪生长性能影响由表 2可知,试验组的终末体重和平均日采食量均高于对照组,料重比低于对照组,但差异均不显著(P>0.05);试验组平均日增重高于对照组,且差异显著(P < 0.05),说明添加贝莱斯芽孢杆菌具有潜在提高猪生长性能的作用。
|
|
表 2 贝莱斯芽孢杆菌对猪生长性能的影响 Table 2 Effects of Bacillus velezensis on growth performance of pigs |
12个样品共获得570 656条高质量序列,平均长度为418.43 nt。OTU聚类分析,12个粪便样品共获得1 124个OTU,聚类为19个门,25个纲,38个目,71个科,207个属,376个种。从表 3中可知,2组间Chao1指数、Ace指数、Shannon指数和Simpson指数均无显著差异(P>0.05)。
|
|
表 3 Alpha多样性指数分析 Table 3 Analysis of α diversity index |
如图 1所示,基于Unweighted unifrac和Weighted unifrac加权距离主坐标分析(PCoA),其中主成分(PC)1和PC2对样品差异的贡献值分别为59.04%、18.58%和59.57%、22.26%。2组的样品并未明显分开,有重叠部分,Weighted unifrac综合主要物种进化关系、种类和其相对丰度分析,结果显示2组样品菌群组成有相似部分,又有不同部分,Unweighted unifrac主要对物种的存在和进化关系分析,结果显示试验组的物种比对照组更为丰富。综上所述,添加贝莱斯芽孢杆菌后,样品中菌群群落组成发生改变且菌群物种更为丰富。
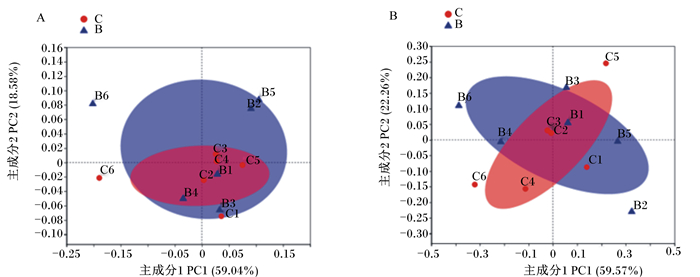
|
C:对照组样品 samples in control group;B:试验组样品 samples in experimental group。下图同 the same as below. 图 1 基于Unweighted unifrac(A)和Weighted unifrac(B)距离主坐标分析 Fig. 1 PCoA based on Unweighted unifrac distance (A) and Weighted unifrac distance (B) |
由表 4可知,样品中细菌主要归于厚壁菌门、拟杆菌门和螺旋菌门三大类。本试验中相对丰度大于1%的菌门共3个。试验结果显示,2组样品中厚壁菌门的相对丰度最高,但组间无显著差异(P>0.05),其次为拟杆菌门,试验组的拟杆菌门和柔膜菌门相对丰度高于对照组(P>0.05),添加了贝莱斯芽孢杆菌后,降低了厚壁菌门/拟杆菌门,且差异显著(P < 0.05)。
|
|
表 4 门水平下的优势菌群分布 Table 4 Distribution of dominant microflora at phylum level |
OTU分类分析结果中相对丰度大于1%的菌属见表 5,试验组和对照组优势菌中的相对丰度最高的菌属分别为普雷沃氏菌科_NK3B31群和链球菌属。试验组的乳杆菌属(P < 0.05)、普雷沃氏菌属_9(P < 0.05)、未分类的毛螺菌科(P < 0.01)和瘤胃球菌属_1(P < 0.01)的相对丰度显著或极显著高于对照组,链球菌属和瘤胃球菌科_UCG-002相对丰度则显著低于对照组(P < 0.05)。结果显示试验猪在饲养过程中可能感染链球菌,但未显示出明显的表观病症,添加了贝莱斯芽孢杆菌后,显著降低链球菌的相对丰度,并提高具有直接或间接抗病菌(乳杆菌属、普雷沃氏菌属_9、未分类的毛螺菌科和瘤胃球菌属_1)的相对丰度,对其抗病能力具有潜在的促进作用。
|
|
表 5 属水平下的优势菌群分布 Table 5 Distribution of dominant microflora at genus level |
物种丰度聚类分析见图 2,对照组和试验组的物种丰度聚类明显分开。试验组添加了贝莱斯芽孢杆菌后,提高了未分类的毛螺菌科、瘤胃球菌科_UCG-005、未明确的F082科、理研菌科RC9肠群、产粪甾醇真细菌属群、未分类的普雷沃氏菌科、普雷沃氏菌科_UCG-001、克里斯滕森菌科_RC9群、毛螺菌科_NK4B1336群、瘤胃球菌科_NK4A214群、瘤胃球菌科_UCG-014、2534-18B5科_肠群、瘤胃球菌科_UCG-002、毛螺菌科_XPB1014群、未明确的p_251_o5科、瘤胃球菌属_1的相对丰度,从而改变了菌群的结构。
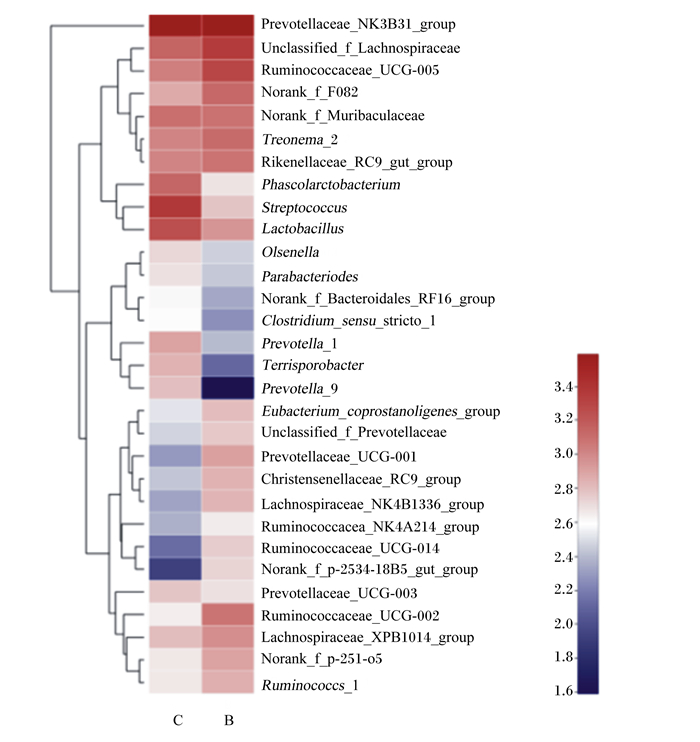
|
Prevotellaceae_NK3B31_group:普雷沃氏菌科_NK3B31群;Unclassified_f_Lachnospiraceae:未分类的毛螺菌科;Ruminococcaceae_UCG-005:瘤胃球菌科_UCG-005;Norank_f_F082:未明确的F082科;Treonema_2:密螺旋体属_2;Rikenellaceae_RC9_gut_group:理研菌科_RC9肠群;Phascolarctobacterium:考拉杆菌属;Streptococcus:链球菌属;Lactobacillus:乳杆菌属;Olsenella:欧氏菌属;Parabacteriodes:副拟杆菌属;Norank_f_Bacteroidales_RF16_group:未明确的拟杆菌科_RF16群;Clostridium_sensu_stricto_1:狭义梭菌属_1;Prevotella_1:普雷沃氏菌属_1;Terrisporobacter:土孢杆菌属;Prevotella_9:普雷沃氏菌属_9;Eubacterium_coprostanoligenes_group:产粪甾醇真细菌属群;Unclassified_f_Prevotellaceae:未分类的普雷沃氏菌科;Prevotellaceae_UCG-001:普雷沃氏菌科_UCG-001;Christensenellaceae_RC9_group:克里斯滕森菌科_RC9群:Lachnospiraceae_NK4B1336_group:毛螺菌科_NK4B1336群;Ruminococcacea_NK4A214_group:瘤胃球菌科_NK4A214群;Ruminococcaceae_UCG-014:瘤胃球菌科_UCG-014;Norank_f_p-2534-18B5_gut_group:未明确的p-2534-18B5肠群;Prevotellaceae_UCG-003:普雷沃氏菌科_UCG-003;Ruminococcaceae_UCG-002:瘤胃球菌科_UCG-002;Lachnospiraceae_XPB1014_group:毛螺菌科_XPB1014群;Norank_f_p-251-o5:未明确的p-251-o5科;Ruminococcs_1:瘤胃球菌属_1。 图 2 物种丰度聚类图 Fig. 2 Heat map of species abundance |
为了能更好地研究添加贝莱斯芽孢杆菌对猪粪便菌群组成的影响,本研究采用了多级物种差异判别分析方法(LEfSe)进行了差异分析,研究结果显示试验组和对照组细菌群落内的相对丰度差异显著(图 3-A),LDA值大于2.0时(图 3-B),2组共获得56个生物标记物,其中对照组获得36个生物标记物,说明2组的物种组成差异显著(P < 0.05)。对照组内LDA值最大的是Selenomonadales,试验组LDA值最大的是瘤胃球菌科_UCG-014,二者是相对丰度差异最大的细菌。纲水平上,试验组中放线菌纲相对丰度显著高于对照组(P < 0.05),试验组Negativicutes、γ-变形菌纲、丹毒丝菌纲的相对丰度则显著低于对照组(P < 0.05)。属水平上,与对照组相比,试验组瘤胃球菌科_UCG-014、未分类的普雷沃氏菌科、未分类的瘤胃球菌科、未明确的柔膜菌纲_RF95、瘤胃球菌科_UCG-004、未明确的红椿菌目、未分类的红椿菌目、未分类的梭菌目、Lachnoclostidium_10、半乳糖己酸菌、瘤胃梭菌属_1、GCA_900066225型菌的相对丰度显著提高(P < 0.05),土孢杆菌属、巨型球菌属、颤螺菌属、副拟杆菌属、毛螺菌科_AC2044群、未分类的拟杆菌目、普雷沃氏菌属_2、琥珀酸弧菌属、Tyzzerella_3、艰难杆菌属、韦荣氏球菌属、未分类的拟杆菌科、未分类的丹毒丝菌科、优杆菌属群、狭义梭菌属_6、Intestinibacter、厌氧原体属、霍尔德曼氏菌和未分类的硒单胞菌目相对丰度显著下降(P < 0.05)。
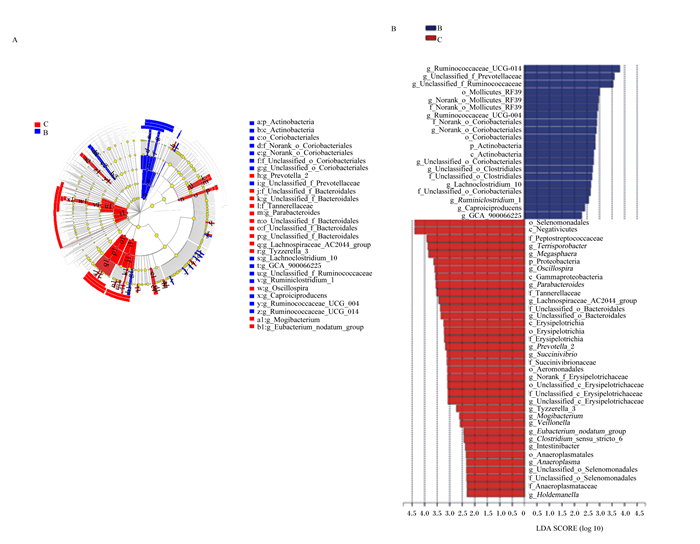
|
A:基于LDA的进化分支图taxonomic cladogram based on LDA;B:56个生物标记物56 biomarkers。Ruminococcaceae_UCG-014:瘤胃球菌科_UCG-014;Unclassified_f_Prevotellaceae:未分类的普雷沃氏菌科;Unclassified_o_Ruminococcaceae:未分类的瘤胃球菌目;O_Mollicutes_RF95:柔膜菌目_RF95;norank_o_Mollicutes_RF95:未明确的柔膜菌目_RF95;Ruminococcaceae_UCG-004:瘤胃球菌科_UCG-004;Ruminococcaceae_UCG-002:瘤胃球菌科_UCG-002;Norank_o_Coriobacteriales:未明确的红椿菌目;O_Coriobacteriales:红椿菌目;Actinobacteria:放线菌门;Unclassified_o_Coriobacteriales:未分类的红椿菌目;Unclassified_o_Clostridiales:未分类的梭菌目;Ruminiclostridium_1:瘤胃梭菌属_1;Caproiciproducens:半乳糖己酸菌;GCA_900066225:GCA_900066225菌属;Peptostreptococcaceae:消化链球菌科;Terrisporobacter:土孢杆菌属;Megasphaera:巨型球菌属;Proteobacteria:变形菌门;Oscillospira:颤螺菌属;Gammaproteobacteria:γ-变相菌纲;Parabacteriodes:副拟杆菌属;Lachnospiraceae_AC2044_group:毛螺菌科_AC2044群;Prevotella_2:普雷沃氏菌属_2;Succinivibrio:琥珀酸弧菌属;Succinivibrionaceae:琥珀酸弧菌科;Aeromonadales:气单胞菌目;Unclassified_c_Bacteroidia:未分类的拟杆菌纲;Unclassified_o_Bacteroidales:未分类的拟杆菌目;Erysipelotrichales:丹毒丝菌目;Erysipelotrichia:丹毒丝菌纲;Erysipelotrichaceae:丹毒丝菌科;Succinivibrio:琥珀酸弧菌属;Norank_f_Erysipelotrichaceae:未分类的丹毒丝菌科;Unclassified_o_Bacteroidia:未分类的拟杆菌目;Mogibacterium:艰难杆菌属;Veillonella:韦荣氏球菌属;Eubacterium_nudatum_group:真杆菌属nudatum群;Clostridium_sensu_stricto_6:狭义梭菌属_6;Anaeroplasmatales:厌氧原体目;Anaeroplasma:厌氧原体属;Anaeroplasmataceae:厌氧原体科;Unclassified_o_Selenomonadales:未分类的硒单胞菌目;Holdemanella:霍尔德曼氏菌属。 图 3 群落组成差异 Fig. 3 Differences of microbiota composition |
PLS-DA是一种有监督的判别分析方法。由图 4可知,正负离子模式下,R2Y分别为0.849和0.778。图中PLS-DA建立的多元分析模型在2种模式下,能将2组样品完全分开,且分离度大,说明分类效果显著,饲粮添加贝莱斯芽孢杆菌B13后,显著改变了代谢物的组成。
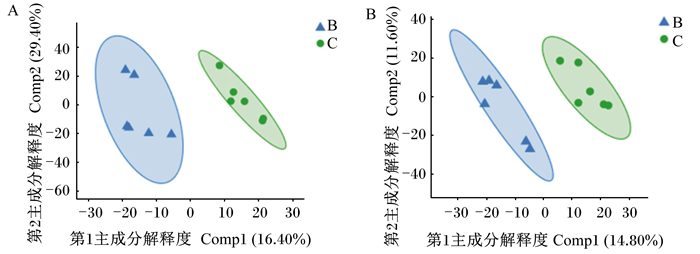
|
图 4 正(A)、负(B)离子模式下的PLS-DA Fig. 4 PLS-DA chart of positive (A) and negative (B) ion modes |
采用非靶向代谢组学方法分析样本中的差异代谢产物,结果见表 6,试验组和对照组的粪便样品在正负离子模式下分别检测出4 748和5 074种代谢物,其中阳离子条件下试验组差异代谢产物为237种,与对照组比较,102种产物相对含量上升,135种产物的相对含量下降;阴离子条件下试验组的差异代谢产物为254种,其中112种产物相对含量上升,142种产物的相对含量下降,其中差异表达前10位的代谢物见表 6,由表可以看出,添加贝莱斯芽孢杆菌后,试验组中天竺葵素-3-槐糖苷和Nigellic acid分别是正、负离子模式下相对含量最大的差异代谢产物,均属于极显著上调物质(P < 0.01)。与对照组相比,试验组猪粪便中的化学药物中间体,如4-乙酰对氨基苯甲醛和对乙酰氨基苯甲醛,其相对含量均显著下调(P < 0.05),人参炔醇相对含量极显著下调(P < 0.01),黑麦酮酸、甲基-10-过氧氢-8E,12Z,15Z-十八碳三烯酸甲酯和羟吲哚相对含量均显著下调(P < 0.05)。
|
|
表 6 正、负离子模式下差异表达前10位的差异代谢产物 Table 6 Top 10 significant different metabolites of positive and negative ion modes |
为了挖掘和探究粪便中菌群与代谢物之间的潜在关系,发掘其潜在的生物标志物,采用皮尔逊(Person)分析方法将差异菌群和差异代谢产物进行相关性分析,本研究将分析菌群相对丰度与关联性最高的2种代谢产物相对含量之间的关系。由图 5可知,链球菌属相对丰度与前列腺衍生物相对含量呈极显著正相关(P < 0.01),与羟喹啉相对含量呈极显著负相关(P < 0.01);乳杆菌属相对丰度与寡肽和肠内脂相对含量呈显著负相关(P < 0.05);p-251-o5、密螺旋体_2和瘤胃球菌科_UCG-005相对丰度均与前列腺素衍生物相对含量呈显著负相关(P < 0.05),与寡肽相对含量呈正相关(P>0.05);理研氏菌科_RC9肠群相对丰度与7-脱氢胆固醇(P < 0.05)和羟喹啉相对含量呈正相关(P>0.05);未分类的毛螺菌科相对丰度与PC(16:0/0:0)相对含量呈正相关(P>0.05),与肠内脂相对含量呈负相关(P>0.05);普雷沃氏菌属_NK3B31群相对丰度与2-吲哚酮和9(S)-HpOTrE相对含量呈正相关(P>0.05);未明确的Muribaculaceae相对丰度与2-吲哚酮相对含量呈正相关(P>0.05),与羟喹啉相对含量呈负相关(P>0.05);考拉杆菌属相对丰度与前列腺衍生物相对含量呈正相关(P>0.05),与寡肽相对含量呈负相关(P>0.05)。其中链球菌属、未分类的毛螺菌科和乳杆菌属为粪菌中优势菌,相对丰度占前10,添加贝莱斯芽孢杆菌后,未分类的毛螺菌科和乳杆菌属相对丰度均显著上升(P < 0.05),且均与肠内脂相对含量呈负相关,链球菌属相对丰度显著下降(P < 0.05),且与前列腺衍生物相对含量呈显著相正相关(P < 0.05)。
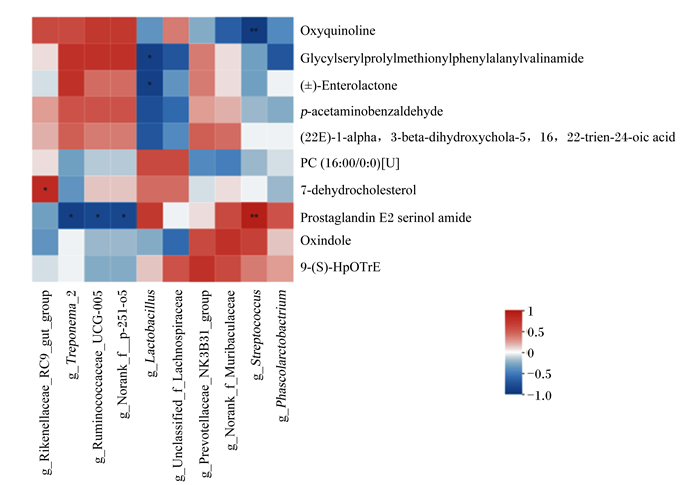
|
*表示差异显著(P < 0.05);* *表示差异极显著(P < 0.01)。* mean significant difference (P < 0.05);* *mean extremely significant (P < 0.01). Phascolarctobacterium:考拉杆菌属;Rikenellaceae_RC9_gut_group:理研氏菌科_RC9肠群;Treponema_2:密螺菌属_2;Ruminococcaceae_UCG-005:瘤胃球菌科_UCG-005;Norank_f_p-251-o5:未分类的p-251-o5;Lactobacillus:乳杆菌属;Unclassified_f_Lachnospiraceae:未分类的毛螺菌科;Prevotellaceae_NK3B31_group:普雷沃氏菌科_NK3B31群;Streptococcus:链球菌属;Oxyquinoline:羟喹啉;Glycylserylprolylmethionylphenylalanylvalinamide:一种寡肽;(±)-Enterolactone:肠内脂;p-acetaminobenzaldehyde:对乙酰氨基苯甲醛;(22E)-1-alpha,3-beta-dihydroxychola-5,16,22-trien-24-oic acid:油酸;7-dehydrocholesterol:7-脱氢胆固醇;Prostaglandin E2 serinol amide:前列腺衍生物;Oxindole:2-吲哚酮。 图 5 关联分析 Fig. 5 Correlation analysis |
在动物饲粮中添加一定剂量的益生菌,具有改善动物消化吸收、促进生长的作用。常见益生菌有乳酸菌、酵母菌、芽孢杆菌、粪球菌等。研究结果显示,由于添加菌的品种和添加剂量、试验动物的品种、日龄和发育阶段等的不同,试验结果也不同。丁浩等[6]的研究发现,在饲粮中添加枯草芽孢杆菌能显著提高猪的平均日增重和降低料重比;邢帅兵等[7]在断奶仔猪饲粮中添加107 CFU/kg的枯草芽孢杆菌后,则发现其对日增重、采食量和料重比均无显著影响。本试验研究结果表明,添加107 CFU/kg的贝莱斯芽孢杆菌后,试验组中猪的平均日增重显著高于对照组,其他生长性能指标均有提高的趋势,但差异不显著。链球菌属为机会致病菌,与猪的发病率显著相关,添加贝莱斯芽孢杆菌后,肠道内链球菌的相对丰度显著下降,具有潜在降低发病促进生长性能的潜能;本试验结果与前人研究结果具有一定的差异性,但趋势一致,结果的不同可能与菌种的选择、菌的添加量和试验动物的选择等因素相关。综上,添加贝莱斯芽孢杆菌具有潜在提高猪生长性能的趋势。
3.2 贝莱斯芽孢杆菌对猪粪便中微生物菌群的影响肠道微生物菌群是宿主体内营养代谢和免疫功能的重要参与者[8],与宿主的健康与疾病显著相关[9-11]。刚排泄的粪便,其微生物菌群能够间接地反映肠道微生物组成,并能节约试验经费、减少应激因素和进行连续性跟踪试验,故在肠道微生物研究方面,越来越多的学者通过关注粪菌来研究肠道微生物[12-14]。
厚壁菌门和拟杆菌门为肠道内优势微生物,参与体内营养物质的代谢,如编码多糖分解的酶,参与糖的代谢,协同参与氨基酸代谢。厚壁菌门/拟杆菌门与脂肪的贮存呈正相关[15-16],通过调控脂代谢相关基因表达与脂肪贮存和代谢,从而调节脂肪酸的合成和脂肪贮存。薛春旭等[17]研究发现,高谷物饲粮能提高山羊回肠内厚壁菌门与拟杆菌门细菌的数量,但对其比值无显著影响。高糖、高脂饲粮能显著降低粪菌中厚壁菌门/拟杆菌门[18],而在高血压[19]、辐射[20]、脂肪肝[21]的粪菌中则显著上升,碳水化合物代谢、生物降解和DNA损伤通路为特征的菌群功能改变,同时可作为坏死性小肠结肠炎(NEC)的生物标记物[22]。本研究发现添加贝莱斯芽孢杆菌后,粪菌中厚壁菌门/拟杆菌门显著下降,但其与脂肪沉积和营养代谢之间的关系还待进一步研究。
短链脂肪酸在肠道内发挥重要作用[23],如抗炎、防癌、清除耐药性致病菌和调控基因表达等作用[24-26]。短链脂肪酸的含量与保育猪的腹泻呈显著负相关,与平均采食量和平均日增重呈显著正相关[27]。短链脂肪酸可以作为信号分子,在细胞外激活G蛋白偶联受体(GPCRs),在细胞内抑制组蛋白脱乙酰化酶(HDAC)作用,从而引起宿主免疫应答,降低肠道炎症,增强肠道屏障功能[28-29],与抵抗肠道致病菌具有显著的相关性。肠道中短链脂肪酸主要受肠道菌群(如梭菌属、瘤胃球菌属、粪杆菌属、毛螺菌科和拟杆菌属等)、益生元等的影响[30-31],如饲粮中添加枯草芽孢杆菌能显著提高粪便中短链脂肪酸的含量和乳杆菌的相对丰度[32]。瘤胃球菌属是猪肠道内一种常见菌,属于厚壁菌门,其相对丰度与宿主的饲料利用率(如蛋白水解、淀粉消化吸收)和短链脂肪酸的代谢呈正相关[33-34],且受饲粮中淀粉[35-36]、粗纤维[37]、含氮饲粮部分[38]和肠道炎症[39-40]等因素的影响。本研究发现,添加贝莱斯芽孢杆菌B13后,显著提高了试验组中短链脂肪酸产生菌瘤胃球菌属_1和未分类的毛螺菌科的相对丰度,对炎症的抵抗具有潜在促进意义。
链球菌属为机会致病菌[41],对猪的发病具有潜在诱导作用[42],其相对丰度与乳杆菌相对丰度呈显著负相关[43]。本试验结果显示,试验过程中2组猪均感染链球菌,但并未发病,可能与试验猪具有抗逆性有关,还有待进一步研究,添加贝莱斯芽孢杆菌后显著降低了链球菌属的相对丰度,分析原因,可能因为:1)添加菌贝莱斯芽孢杆菌具有病原菌拮抗性;2)添加贝莱斯芽孢杆菌后,部分抗病菌的相对丰度升高,如乳杆菌能直接或者间接抑制链球菌的生长[44];瘤胃球菌能产生具有抗菌活性物质——瘤胃球菌素C1(RumC1),通过抑制核酸合成对多重耐药菌和致病性梭菌等病原菌发挥杀菌作用[45];毛螺菌科对病原菌具有拮抗作用[46]。故而添加贝莱斯芽孢杆菌具有潜在抑制链球菌和促进有益菌相对丰度的趋势。
3.3 贝莱斯芽孢杆菌对猪粪便中代谢产物的影响在肠道中,底物相同的前提下,由于微生物的种类不同从而导致其发酵产物和代谢产物的不同。本试验结果显示,饲粮添加贝莱斯芽孢杆菌后,在提高了粪便中具有抗氧化功能的物质的相对含量的同时,也降低了药物中间体的含量,如显著上调了天竺葵素-3-槐糖苷的相对含量,天竺葵素-3-槐糖苷是花青素的一种,主要作用为抗氧化剂。猪粪便中的化学药物中间体,如4-乙酰对氨基苯甲醛和对乙酰氨基苯甲醛的相对含量均呈现显著下调状态,黑麦酮酸的相对含量呈现下调状态,黑麦酮酸是曲霉菌和草酸青霉菌等霉菌的代谢产物,已报道贝莱斯芽孢杆菌具有抗植物病害如疮痂链霉菌作用,说明添加贝莱斯芽孢杆菌后,有潜在促进机体降低粪便中药物残留和体内抗霉菌作用的趋势。
吲哚及其衍生物是由色氨酸经肠道菌群发酵产生[47],通过降低核因子-κB(NF-κB)信号通路中关键蛋白的表达和下游促炎症基因的上调,抑制脂多糖(LPS)引起的胆固醇代谢变化[48];降低肠道通透性,影响黏膜稳态,诱导胰高血糖素样肽-1(GLP-1)的释放,从而抑制食欲,降低胃排空速度[49],某些来源于大肠杆菌代谢物的吲哚类化合物具有促进耐药型大肠杆菌持留菌的生长的作用[50],同时吲哚也与肉中的膻味成正比[51]。羟吲哚则具有促进大鼠迷走神经c-Fos蛋白表达,增加焦虑行为的作用[52]。而本试验中添加贝莱斯芽孢杆菌后,平均日增重显著提高,平均日采食量有上升趋势,但差异不显著,羟吲哚的相对含量显著下调。添加贝莱斯芽孢杆菌后是否能通过吲哚类代谢物而间接影响其采食量,且通过什么通路来影响等问题,还待进一步研究。
4 结论① 添加贝莱斯芽孢杆菌提高了试验猪的平均日增重。
② 添加贝莱斯芽孢杆菌对猪粪菌微生物多样性没有显著的影响,但降低了粪菌中厚壁菌门/拟杆菌门,属水平上菌群结构发生显著变化,显著提高了未分类的毛螺菌科和瘤胃球菌属_1等短链脂肪酸产生菌群和乳杆菌属等益生菌的相对丰度,显著降低了链球菌属等机会致病菌的相对丰度。
③ 添加贝莱斯芽孢杆菌提高了天竺葵素-3-槐糖苷等抗氧化物质的相对含量,具有潜在降低肠道炎症的功能;降低了4-乙酰对氨基苯甲醛、对乙酰氨基苯甲醛等药物中间体物质、羟吲哚和黑麦酮酸等霉菌代谢产物的相对含量。
| [1] |
李威, 李佳熙, 李吉平, 等. 我国不同环境介质中的抗生素污染特征研究进展[J]. 南京林业大学学报(自然科学版), 2020, 44(1): 205-214. |
| [2] |
李昕, 曾洁, 王岱, 等. 细菌耐药耐受性机制的最新研究进展[J]. 中国抗生素杂志, 2020, 45(2): 113-121. |
| [3] |
康星星.贝莱斯芽孢杆菌CC09防治小麦全蚀病菌侵染的机制[D].博士学位论文.南京: 南京大学, 2019.
|
| [4] |
申永瑞, 向君亮, 王佳琦, 等. 疮痂链霉菌拮抗菌株BU396的分离鉴定与抗菌性质分析[J]. 微生物学通报, 2019, 46(10): 2601-2611. |
| [5] |
刘韶娜, 张斌, 相德才, 等. 芽孢杆菌B13的分离鉴定及其抑菌作用研究[J]. 中国畜牧兽医, 2019, 46(2): 573-581. |
| [6] |
丁浩, 黄攀, 章文明, 等. 饲粮添加枯草芽孢杆菌对保育猪生长性能和血浆生化参数的影响[J]. 动物营养学报, 2020, 32(2): 605-612. |
| [7] |
邢帅兵, 陈代文, 余冰, 等. 枯草芽孢杆菌对断奶仔猪生长性能和肠道形态、黏膜免疫及菌群数量的影响[J]. 动物营养学报, 2020, 32(5): 2066-2073. |
| [8] |
SENGHOR B, SOKHNA C, RUIMY R, et al. Gut microbiota diversity according to dietary habits and geographical provenance[J]. Human Microbiome Journal, 2018(7/8): 1-9. |
| [9] |
KIM H B, ISAACSON R E. The pig gut microbial diversity:understanding the pig gut microbial ecology through the next generation high throughput sequencing[J]. Veterinary Microbiology, 2015, 177(3/4): 242-251. |
| [10] |
PAJARILLO E A B, CHAE J P, BALOLONG M P, et al. Effects of probiotic Enterococcus faecium NCIMB 11181 administration on swine fecal microbiota diversity and composition using barcoded pyrosequencing[J]. Animal Feed Science and Technology, 2015, 201: 80-88. DOI:10.1016/j.anifeedsci.2015.01.011 |
| [11] |
SONNENBURG J L, BÄCKHED F. Diet-microbiota interactions as moderators of human metabolism[J]. Nature, 2016, 535(7610): 56-64. DOI:10.1038/nature18846 |
| [12] |
孙东方, 王晨, 刘颖, 等. 双乙酸钠对小鼠肠道菌群结构和功能的影响[J]. 微生物学杂志, 2020, 40(2): 57-65. |
| [13] |
NAKAYAMA J, ZHANG H P, LEE Y K. Asian gut microbiome[J]. Science Bulletin, 2017, 62(12): 816-817. DOI:10.1016/j.scib.2017.04.001 |
| [14] |
RAIMONDI M T, ALBANI D, GIORDANO C. An organ-on-a-chip engineered platform to study the microbiota-gut-brain axis in neurodegeneration[J]. Trends in Molecular Medicine, 2019, 25(9): 737-740. DOI:10.1016/j.molmed.2019.07.006 |
| [15] |
TURNBAUGH P J, LEY R E, MAHOWALD M A, et al. An obesity-associated gut microbiome with increased capacity for energy harvest[J]. Nature, 2006, 444(7122): 1027-1031. DOI:10.1038/nature05414 |
| [16] |
ZHU J Q, KONG Y, YU J, et al. Consumption of drinking water N-nitrosamines mixture alters gut microbiome and increases the obesity risk in young male rats[J]. Environmental Pollution, 2019, 248: 388-396. DOI:10.1016/j.envpol.2019.02.012 |
| [17] |
薛春旭, 叶慧敏, 冯泮飞, 等. 高谷物日粮对山羊小肠发酵、肠道结构和微生物菌群数量的影响研究[J]. 草业学报, 2016, 25(5): 175-183. |
| [18] |
谷莉.不同鼠种的肠道菌群在不同饮食结构干预中的组成改变[D].博士学位论文.长沙: 中南大学, 2014.
|
| [19] |
ADNAN S, NELSON J W, AJAMI N J, et al. Alterations in the gut microbiota can elicit hypertension in rats[J]. Physiological Genomics, 2017, 49(2): 96-104. DOI:10.1152/physiolgenomics.00081.2016 |
| [20] |
LAVRINIENKO A, MAPPES T, TUKALENKO E, et al. Environmental radiation alters the gut microbiome of the bank vole Myodes glareolus[J]. The ISME Journal, 2018, 12(11): 2801-2806. DOI:10.1038/s41396-018-0214-x |
| [21] |
LEE N Y, YOON S J, HAN D H, et al. Lactobacillus and Pediococcus ameliorate progression of non-alcoholic fatty liver disease through modulation of the gut microbiome[J]. Gut Microbes, 2020, 11(4): 882-899. DOI:10.1080/19490976.2020.1712984 |
| [22] |
RUSCONI B, GOOD M, WARNER B B. The microbiome and biomarkers for necrotizing enterocolitis:are we any closer to prediction?[J]. The Journal of Pediatrics, 2017, 189: 40-47. DOI:10.1016/j.jpeds.2017.05.075 |
| [23] |
SERINO M. SCFAs-the thin microbial metabolic line between good and bad[J]. Nature Reviews Endocrinology, 2019, 15(6): 318-319. DOI:10.1038/s41574-019-0205-7 |
| [24] |
FERNÁNDEZ J, REDONDO-BLANCO S, GUTIÉRREZ-DEL-RÍO I, et al. Colon microbiota fermentation of dietary prebiotics towards short-chain fatty acids and their roles as anti-inflammatory and antitumour agents:a review[J]. Journal of Functional Foods, 2016, 25: 511-522. DOI:10.1016/j.jff.2016.06.032 |
| [25] |
SMITH P M, HOWITT M R, PANIKOV N, et al. The microbial metabolites, short-chain fatty acids, regulate colonic Treg cell homeostasis[J]. Science, 2013, 341(6145): 569-573. DOI:10.1126/science.1241165 |
| [26] |
SORBARA M T, DUBIN K, LITTMANN E R, et al. Inhibiting antibiotic-resistant Enterobacteriaceae by microbiota-mediated intracellular acidification[J]. Journal of Experimental Medicine, 2019, 216(1): 84-98. DOI:10.1084/jem.20181639 |
| [27] |
吕存, 孙玉亭, 胡传炯, 等. 合生元对保育猪生长性能、粪样菌群结构和短链脂肪酸含量的影响[J]. 南京农业大学学报, 2019, 42(4): 721-728. |
| [28] |
ZHANG J C, GUO Z, XUE Z S, et al. A phylo-functional core of gut microbiota in healthy young Chinese cohorts across lifestyles, geography and ethnicities[J]. The ISME Journal, 2015, 9(9): 1979-1990. DOI:10.1038/ismej.2015.11 |
| [29] |
王可鑫, 姜宁, 张爱忠. 短链脂肪酸介导的宿主肠道免疫调控机制[J]. 动物营养学报, 2020, 32(4): 1544-1550. |
| [30] |
赵秀英, 县怡涵, 李晨博, 等. 灌喂植物乳杆菌和干酪乳杆菌增加仔猪肠道菌群多样性及短链脂肪酸生成[J]. 微生物学报, 2016, 56(8): 1291-1300. |
| [31] |
CRAIG A D, KHATTAK F, HASTIE P, et al. Xylanase and xylo-oligosaccharide prebiotic improve the growth performance and concentration of potentially prebiotic oligosaccharides in the ileum of broiler chickens[J]. British Poultry Science, 2020, 61(1): 70-78. DOI:10.1080/00071668.2019.1673318 |
| [32] |
王汉星, 虎千力, 杨建涛, 等. 饲粮中添加粪链球菌与枯草芽孢杆菌对断奶仔猪生长性能和肠道健康的影响[J]. 动物营养学报, 2020, 32(5): 2074-2086. |
| [33] |
LI F Y, LI C X, CHAN Y H, et al. Host genetics influence the rumen microbiota and heritable rumen microbial features associate with feed efficiency in cattle[J]. Microbiome, 2019, 7(1): 92. DOI:10.1186/s40168-019-0699-1 |
| [34] |
DERAKHSHANI H, TUN H M, CARDOSO F C, et al. Linking peripartal dynamics of ruminal microbiota to dietary changes and production parameters[J]. Frontiers in Microbiology, 2017, 7: 2143. |
| [35] |
SUN Y, SU Y, ZHU W Y. Microbiome-metabolome responses in the cecum and colon of pig to a high resistant starch diet[J]. Frontiers in Microbiology, 2016, 7: 779. |
| [36] |
FERRARIO C, STATELLO R, CARNEVALI L, et al. How to feed the mammalian gut microbiota:bacterial and metabolic modulation by dietary fibers[J]. Frontiers in Microbiology, 2017, 8: 1749. DOI:10.3389/fmicb.2017.01749 |
| [37] |
HEALEY G, MURPHY R, BUTTS C, et al. Habitual dietary fibre intake influences gut microbiota response to an inulin-type fructan prebiotic:a randomised, double-blind, placebo-controlled, cross-over, human intervention study[J]. British Journal of Nutrition, 2018, 119(2): 176-189. DOI:10.1017/S0007114517003440 |
| [38] |
曹宁坤, 杨新宇, 吴峰洋, 等.无氮饲粮对家兔肠道消化酶和微生物区系的影响[C]//中国畜牧兽医学会养兔学分会第二届学术交流大会论文集.北京: 中国畜牧兽医学会, 2018.
|
| [39] |
VAN DEN MUNCKHOF I C L, KURILSHIKOV A, TER HORST R, et al. Role of gut microbiota in chronic low-grade inflammation as potential driver for atherosclerotic cardiovascular disease:a systematic review of human studies[J]. Obesity Reviews, 2018, 19(12): 1719-1734. DOI:10.1111/obr.12750 |
| [40] |
FU B C, HULLAR M A J, RANDOLPH T W, et al. Associations of plasma trimethylamine N-oxide, choline, carnitine, and betaine with inflammatory and cardiometabolic risk biomarkers and the fecal microbiome in the Multiethnic Cohort Adiposity Phenotype Study[J]. The American Journal of Clinical Nutrition, 2020, 111(6): 1226-1234. DOI:10.1093/ajcn/nqaa015 |
| [41] |
GU S L, CHEN Y F, WU Z J, et al. Alterations of the gut microbiota in patients with coronavirus disease 2019 or H1N1 Influenza[J]. Clinical Infectious Diseases, 2020. |
| [42] |
郑成坤.猪链球菌2型Spx调节因子和NADH氧化酶的致病性研究[D].博士学位论文.武汉: 华中农业大学, 2017.
|
| [43] |
CHIUMENTO S, ROBLIN C, KIEFFER-JAQUINOD S, et al. Ruminococcin C, a promising antibiotic produced by a human gut symbiont[J]. Science Advances, 2019, 5(9): eaaw9969. DOI:10.1126/sciadv.aaw9969 |
| [44] |
KRZYŚCIAK W, KOŚCIELNIAK D, PAPIEŻ M, et al. Effect of a Lactobacillus salivarius probiotic on a double-species Streptococcus mutans and Candida albicans caries biofilm[J]. Nutrients, 2017, 9(11): 1242. DOI:10.3390/nu9111242 |
| [45] |
CHIUMENTO S, ROBLIN C, KIEFFER-JAQUINOD S, et al. Ruminococcin C, a promising antibiotic produced by a human gut symbiont[J]. Science Advances, 2019, 5(9): eaaw9969. DOI:10.1126/sciadv.aaw9969 |
| [46] |
SURANA N K, KASPER D L. Moving beyond microbiome-wide associations to causal microbe identification[J]. Nature, 2017, 552(7684): 244-247. DOI:10.1038/nature25019 |
| [47] |
MARSLAND B J. Regulating inflammation with microbial metabolites[J]. Nature Medicine, 2016, 22(6): 581-583. DOI:10.1038/nm.4117 |
| [48] |
BEAUMONT M, NEYRINCK A M, OLIVARES M, et al. The gut microbiota metabolite indole alleviates liver inflammation in mice[J]. The FASEB Journal, 2018, 32(12): 6681-6693. DOI:10.1096/fj.201800544 |
| [49] |
ROAGER H M, LICHT T R. Microbial tryptophan catabolites in health and disease[J]. Nature Communications, 2018, 9: 3294. DOI:10.1038/s41467-018-05470-4 |
| [50] |
KIM C S, LI J H, BARCO B, et al. Cellular stress upregulates indole signaling metabolites in Escherichia coli[J]. Cell Chemical Biology, 2020, 27(6): 698-707. DOI:10.1016/j.chembiol.2020.03.003 |
| [51] |
JAGLIN M, RHIMI M, RHIMI C, et al. Indole, a signaling molecule produced by the gut microbiota, negatively impacts emotional behaviors in rats[J]. Frontiers in Neuroscience, 2018, 12: 216. DOI:10.3389/fnins.2018.00216 |
| [52] |
JAGLIN M, RHIMI M, PHILIPPE C, et al. Indole, a signaling molecule produced by the gut microbiota, negatively impacts emotional behaviors in rats[J]. Frontiers in Neuroscience, 2018, 12: 216. DOI:10.3389/fnins.2018.00216 |




