2. 仲恺农业工程学院, 动物科技学院, 广州 510225;
3. 广东省水环境与水产品安全工程技术研究中心, 广州 510225;
4. 广州市水产病害与水禽养殖重点实验室, 广州 510225
2. College of Animal Sciences and Technology, Zhongkai University of Agriculture and Engineering, Guangzhou 510225, China;
3. Guangdong Provincial Key Laboratory of Waterfowl Healthy Breeding, Guangzhou 510225, China;
4. Guangzhou Key Laboratory of Aquatic Animal Diseases and Waterfowl Breeding, Guangzhou 510225, China
鱼粉是不可或缺的优质饲料原料,饲料行业对其具有高度的依赖性。我国是全球最大的鱼粉需求国,自2013年起,每年鱼粉的消费量都超过100万t,其中70%~80%源自进口[1]。近年来,由于秘鲁、智利等鱼粉生产大国出口配额的限制,我国不断拓展进口鱼粉采购渠道,越南、泰国、美国、印度、南非等国的鱼粉相继进入中国市场,其原料鱼组成各不相同,且批次变化较大,商品名不标准,产品标签仅标注产地或仅提及1~2种原料鱼名称,仅凭营养指标难以对其品质作出准确判断。
另外,由于鱼粉价格昂贵,市场上充斥着各种假冒伪劣鱼粉,包括在鱼粉中掺入各种高蛋白质原料(豆粕、玉米蛋白粉、肉骨粉、鸡肉粉、皮革粉等)或是在高值鱼粉中掺入低值鱼粉,如在海水鱼粉中掺入淡水鱼粉、下杂鱼粉,在进口蒸汽鱼粉中掺入国产鱼粉等。前者通过镜检,结合某些物理、化学分析方法,较易快速鉴别[2-5];而鱼粉互掺,则鉴定难度较大,主要通过在显微镜下识别不同鱼类的镜检特征,如耳石、鳞片或骨骼等组织来进行形态鉴定[6-8],存在着鉴定种类少、专业性要求高、重复性差、灵敏度低等缺点,给鱼粉的掺假鉴定带来了严峻的挑战。因此,急需开发一种准确、快速、灵敏鉴定鱼粉组成的方法,对饲料行业的鱼粉采购、质量判断与控制、科学配方都具有重要的意义。
利用基于DNA序列分析的分子标记技术鉴定生物样品的物种组成,具有不易受样品形态和数量影响、准确性和灵敏度高、操作简便等诸多优点,目前已广泛应用于食品、生物制品、中药等复杂基质的生物组成鉴定以及环境生物多样性检测等领域[9-15]。近年来也开展了饲料原料生物成分的DNA鉴定技术研究,例如:开发疑似掺假物的分子标记,确定目的物种的存在[16];利用限制性片段长度多态性聚合酶链反应(PCR-RFLP)法建立酶切图谱,鉴定混合生物样本中的某些物种[17-19];利用多重PCR法鉴定更复杂的混合生物样本[20-21]等,以上方法各有优缺点。
鱼粉的原料鱼组成取决于渔获物组成。由于每种鱼粉的原料鱼产地不同、每批次渔获物存在不同程度的变动,或者掺假鱼种类的变动,导致鱼粉组成的不确定性增加。因此,难以针对鱼粉中的每种原料鱼分别设计引物或制作酶切图谱。同时,由于鱼粉是经过高温、高压等工艺制作的深加工动物产品,DNA破坏较严重,而多重PCR对引物的设计、扩增条件有着较高的要求,易产生非特异性扩增产物,导致检测结果出现假阳性[22]。针对以上问题,本研究拟通过筛选1种特异性良好的鱼类通用引物,结合分子克隆的方法,开发一种灵敏、准确、简便的鱼粉中原料鱼组成的鉴定方法,以期为鱼粉等混合性饲料原料生物组成的准确鉴定提供可行的解决方案。
1 材料与方法 1.1 样品收集与处理 1.1.1 鱼类样品从国内水产市场收集9种制作鱼粉常用的新鲜原料鱼,其商品名分别为:圆鲹、沙丁鱼、鲭鱼、马鲛鱼、秋刀鱼、鳕鱼、鲽鱼、巴沙鱼、罗非鱼。分别取以上鱼类的背肌约2.0 g,无菌剪刀剪碎后,保存于-80 ℃冰箱中待用。
1.1.2 商业鱼粉样品从饲料原料市场上收集7种常见的商业鱼粉样品,其商品标签分别名为:①海水鱼排粉;②淡水鱼粉;③国产半脱脂鱼粉;④国产普通鱼粉;⑤秘鲁鱼粉;⑥阿根廷鱼粉;⑦美国鱼粉。其中①~④为国产鱼粉,⑤~⑦为进口鱼粉。将以上鱼粉样品分别粉碎并通过40目筛,保存于-80 ℃冰箱中待用。
1.1.3 自配鱼粉样品为验证分子鉴定方法的结果,自配2种鱼粉。①自配鱼粉一:在秘鲁鱼粉中掺入10%(质量比)的罗非鱼粉[原料鱼为吉富罗非鱼(Oreochromis niloticus)],混合均匀;②自配鱼粉二:海水鱼排粉中掺入0.1%(质量比)的巴沙鱼粉[原料鱼为低眼巨鲶(Pangasianodon hypophthalums),俗称巴沙鱼],混合均匀。将以上鱼粉样品分别粉碎并通过40目筛,保存于-80 ℃冰箱。
1.2 DNA提取及PCR扩增将鱼类及鱼粉样品用天根生化科技有限公司的DNA试剂盒提取总DNA。根据文献[23-24],选取2对引物作为候选引物,并由广州天一辉远生物科技有限公司合成(表 1)。用这2对引物分别对提取的鱼类和鱼粉样品DNA进行扩增,反应体系50 μL,包括:PCR缓冲液5 μL,无菌水30.75 μL,正、反向引物(10 μmol/L)各2 μL,DNA模板2 μL,dNTP(2.0 mmol/L)5 μL,MgCl2(25 mmol/L)3 μL,Taq聚合酶(5 U/μL)0.25 μL。反应条件为:95 ℃预变性5 min,95 ℃变性30 s,53 ℃退火30 s,72 ℃复性45 s,35个循环,72 ℃延伸10 min。反应结束后,分别取5 μL扩增产物,用1.0%琼脂糖凝胶电泳检测扩增结果。挑选出对鱼类和鱼粉样品DNA都能够良好扩增的引物,对其PCR扩增产物进行纯化、回收。
|
|
表 1 PCR所用的2对引物 Table 1 Two pairs of primers for PCR |
将经过纯化回收的鱼粉样品扩增产物连接到载体pMD® 19-T(TaKaRa公司)上,转化到大肠杆菌DH5α感受态细胞(北京博迈德基因技术有限公司),在含有氨苄青霉素的LB平板上培养14 h。具体操作参照pMD® 19-T Vector说明书进行。
挑取单克隆,经PCR鉴定后选择阳性克隆,送广州天一辉远生物科技有限公司进行测序。引物、反应体系及反应条件同上。将测序结果去除载体序列后,在GenBank数据库中进行BlastN比对分析。比对序列长度不小于180 bp,物种相似度不低于97%。根据比对结果确定样品的物种组成。
2 结果与分析 2.1 PCR扩增结果分别使用2对引物,对9种鱼类及7种商业鱼粉的DNA进行了PCR扩增,扩增产物的凝胶电泳结果见图 1、图 2。
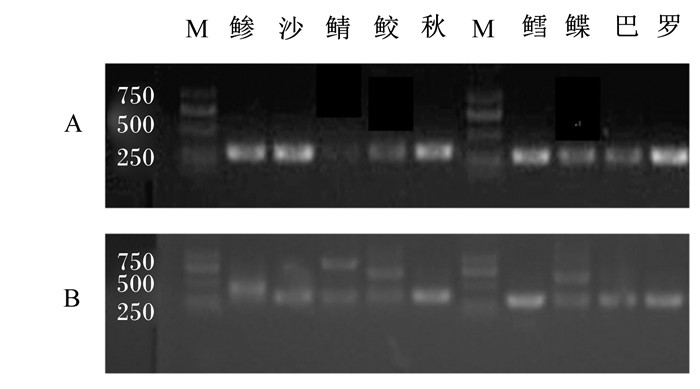
|
A:cytb引物扩增结果the results of fish samples amplified by cytb;B:16S rDNA引物扩增结果the results of fish samples amplified by 16S rDNA。M:Marker D2000;鲹:圆鲹round scad;沙:沙丁鱼sardine;鲭:鲭鱼mackerel;鲛:马鲛鱼Spanish mackerel;秋:秋刀鱼Pacific saury;鳕:鳕鱼cod fish;鲽:鲽鱼plaice;巴:巴沙鱼Basa catfish;罗:罗非鱼tilapia。 图 1 cytb和16S rDNA引物扩增鱼类样品的结果 Fig. 1 Results of fish samples amplified by primers of cytb and 16S rDNA |
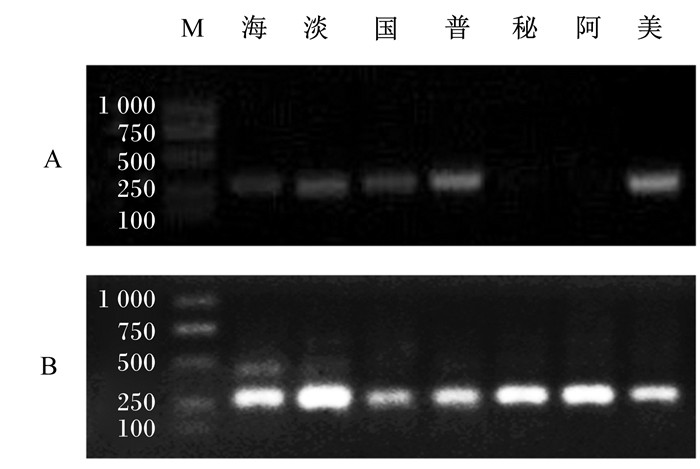
|
A:第1次扩增结果the amplification results for the first time;B:第2次扩增结果the amplification results for the second time。M:Marker D2000;海:海水鱼排粉marine fish steak meal;淡:淡水鱼粉freshwater fish meal;国:国产半脱脂鱼粉domestic semi-skimmed fish meal;普:国产普通鱼粉domestic common fish meal;秘:秘鲁鱼粉fish meal from Peru;阿:阿根廷鱼粉fish meal from Argentina;美:美国鱼粉fish meal from America。图 3同the same as Fig. 3。 图 2 cytb引物第1、2次扩增7种商业鱼粉样品的结果 Fig. 2 Results of 7 commercial fish meal samples amplified by primer cytb for the first and second time |
cytb引物扩增的9种鱼类PCR条带较亮(图 1-A),条带位置和目的片段长度吻合;7种商业鱼粉PCR条带位置也和目的片段长度吻合,但颜色较暗,其中秘鲁鱼粉和阿根廷鱼粉的条带几乎看不见(图 2-A),推测可能是加工过程中鱼粉DNA被破坏,导致目的基因浓度较低。为此,以所有鱼粉的PCR产物为模板进行第2次扩增,反应条件不变。结果表明,第2次扩增后,大多数鱼粉的目的条带比第1次明亮、清晰,且没有非特异性的杂带,无拖尾,表明扩增产物浓度升高(图 2-B)。
16S rDNA引物的扩增结果表明,鱼类样品中的沙丁鱼、秋刀鱼、鳕鱼、巴沙鱼、罗非鱼出现了目的条带,其他鱼类的条带位置和目的条带偏差较大,且大多出现了杂带(图 1-B);7种商业鱼粉样品中仅有淡水鱼粉出现了目的条带,且条带颜色较暗。进行第2次扩增后,淡水鱼粉的目的条带亮度有所增强,但其他鱼粉依然没有出现目的条带(图 3)。

|
图 3 16S rDNA引物第1、2次扩增7种商业鱼粉样品的结果 Fig. 3 Results of 7 commercial fish meal samples amplified by primer 16S rDNA for the first and second time |
由此可见,2种引物相比较,cytb引物能特异性扩增出所有受试鱼类和鱼粉样品的目的基因,可进行后续基因克隆试验。为验证cytb引物的有效性,再次使用cytb引物扩增2种自配鱼粉的DNA,也能获得清晰明亮的条带,且第2次扩增的目的条带亮度明显增加(图 4)。
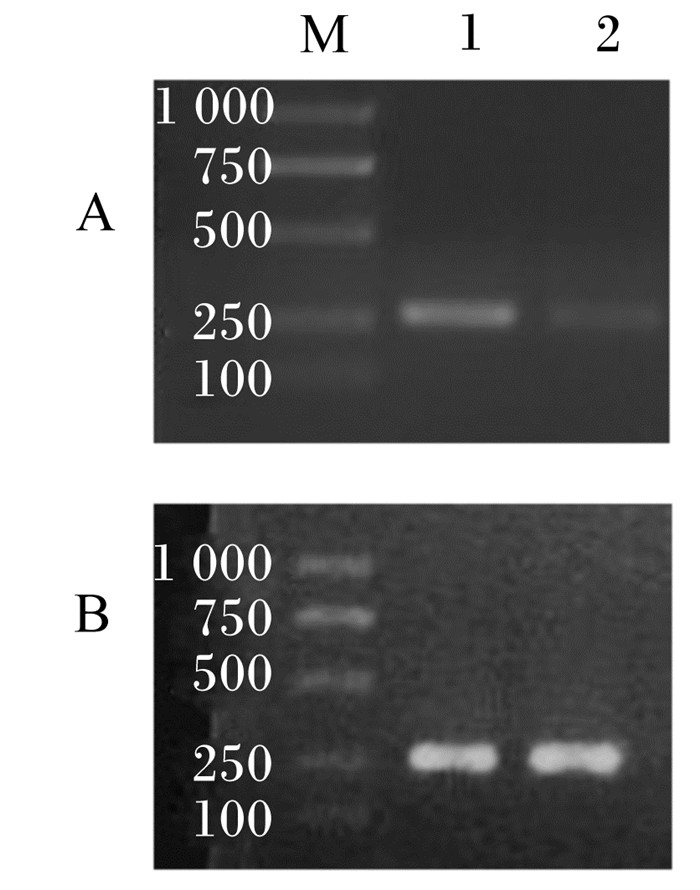
|
A:第1次扩增结果the amplification results for the first time;B:第2次扩增结果the amplification results for the second time。M:Marker D2000;1:自配鱼粉一self-formulated fish meal 1;2:自配鱼粉二self-formulated fish meal 2。 图 4 cytb引物第1、2次扩增2种自配鱼粉样品的结果 Fig. 4 Results of 2 self-formulated fish meal samples amplified by the primer cytb for the first and second time |
将所有鱼粉cytb引物的二次扩增产物纯化回收、连接、转化、培养后,每个样品从LB平板中随机选择30个单克隆,PCR鉴定后,分别获得阳性克隆13~21个,阳性克隆率为43.3%~70.0%。将得到的阳性克隆进行测序和BlastN比对分析,确定样品中原料鱼的种类。
结果显示,不同鱼粉的原料鱼组成差别较大。以海水鱼排粉为例,16个阳性克隆中,共检出9个蓝圆鲹(Decapterus maruadsi)、2个二长棘鲷(Parargyrops edita)、3个隆背小沙丁鱼(Sardinella gibbosa)、1个日本竹筴鱼(Trachurus japonicus)以及1个青背竹筴鱼(Trachurus declivis),共5种鱼(表 2)。将这5种鱼的基因序列通过DNAman软件进行多重比对发现,其序列的同源性为79.05%,5种鱼2段共同的保守序列分别与本研究所使用的cytb基因上、下游引物序列匹配,说明cytb引物在此作为通用引物是可靠的(图 5)。
|
|
表 2 海水鱼排粉的测序结果及比对结果 Table 2 Sequencing and blasting results of marine fish steak meal |

|
图中上、下方框内的基因序列分别对应cytb基因的正、反引物序列。1、2、3、5、12号物种分别与表 2中同一编号的物种对应,即分别为二长棘鲷、蓝圆鲹、隆背小沙丁鱼、青背竹筴鱼和日本竹荚鱼。 The gene sequences in the upper and lower boxes correspond to those of positive and negative primers of cytb gene. Species of No.1, 2, 3, 5 and 12 correspond to those in Table 2, they are Parargyrops edita, Decapterus maruadsi, Sardinella gibbosa, Trachurus declivis and Trachurus japonicas, respectively. 图 5 海水鱼排粉中5种原料鱼基因序列的多重比较图 Fig. 5 Multiple aligment map of gene sequences of 5 raw fish species in marine fish steak meal |
7种商业鱼粉原料鱼组成的鉴定结果见表 3。淡水鱼粉检出尼罗罗非鱼(Oreochromis niloticus)和杜氏绫鯷(Thryssa dussumieri),但后者的检出频次只有1/14;国产半脱脂鱼粉检出隆背小沙丁鱼、蓝圆鲹、丽叶鲹(Alepes kleinii)、小沙丁鱼(Sardinella fijiensis)和杜氏绫鳀(Thryssa dussumieri);国产普通鱼粉检出尼罗罗非鱼、欧洲鳀(Engraulis ringen)、大西洋油鲱(Brevoortia tyrannus)及似鳚镖鲈(Etheostoma blennioides);秘鲁鱼粉检出秘鲁鳀(Engraulis ringens)、绿鳍马面鲀(Thamnaconus modestu)、巴西海猪鱼(Halichoeres brasiliensis)及五棘渊八角鱼(Bathyagonus pentacanthus);阿根廷鱼粉检出日本鳀(Engraulis japonicus)、隆背小沙丁鱼、秋刀鱼(Cololabis saira)、方氏云鳚(Pholis fangi)、智利竹筴鱼(Trachurus murphyi)、赤鼻棱鳀(Thryssa kammalensis)、太平洋沙丁鱼(Sardinops melanostictus)及驼背大马哈鱼(Oncorhynchus gorbuscha);美国鱼粉检出巴西油鲱(Brevoortia aurea)、大西洋油鲱(Brevoortia tyrannus)、岩栖真鳂(Holocentrus adscensionis)、蓝圆鲹和青背竹筴鱼。从以上鉴定结果来看,组成各鱼粉的原料鱼种类众多,远超鱼粉标签所示的1~2种主要鱼种。
|
|
表 3 7种商业鱼粉原料鱼组成鉴定结果 Table 3 Identification results of raw fish species composition of 7 commercial fish meals |
自配鱼粉的鉴定结果如表 4所示。自配鱼粉一的12个阳性克隆共检出8次秘鲁鳀、3次尼罗罗非鱼和1次澳洲鳀(Engraulis australis),即包括秘鲁鱼粉的主要种类和罗非鱼;自配鱼粉二检出隆背小沙丁鱼、秋刀鱼、蓝圆鲹以及巴沙鱼等,即包括海水鱼排粉的主要种类及巴沙鱼,均与试验设计吻合。
|
|
表 4 2种自配鱼粉原料鱼组成鉴定结果 Table 4 Identification results of raw fish species composition of 2 self-formulated fish meal samples |
本研究中的9种受试鱼类包括了制作商品鱼粉的常见鱼类种属:制作红鱼粉常用的鳀鱼、沙丁鱼、秋刀鱼、鲭鱼、蓝鲹等;制作白鱼粉常用的鳕鱼、鲽鱼;制作淡水鱼粉常用的罗非鱼、巴沙鱼。成功扩增这9种鱼类的特定基因序列,是其混合物(鱼粉)组成鉴定的前提。
本研究分别使用了线粒体cytb和16S rDNA引物作为鱼类通用引物的筛选对象,它们作为重要的DNA分子标记,广泛应用于包括鱼类在内的多种真核生物的鉴定和分类。Cytb基因的进化速度适中,其中一个基因片段就包含了从种内到种间的遗传信息,多用于物种种属鉴定[25];线粒体16S rDNA基因结构既具有保守性,又具有高变性,可为水生动物群体遗传多样性和系统进化提供线索,又是种属鉴定的分子基础[26-27]。
从研究结果来看,cytb引物显然较16S rDNA引物对各种鱼类及鱼粉样品有更好的通用性,能扩增出全部受试鱼类和鱼粉的DNA,且从鱼粉中鉴定到的鱼类种属还包括多种非受试鱼类,如竹荚鱼、马面鲀、大马哈鱼等,这表明基于cytb引物的分子鉴定,将有可能获得鱼粉中更多原料鱼的信息。Thomsen等[28]曾用cytb基因片段上设计的通用引物扩增出了丹麦埃尔西诺港口常见的10种海鱼的基因片段,但只能扩增到池塘水样中的4种淡水鱼类。刘军等[13]用线粒体D-loop区、16S rDNA、COⅠ以及cytb引物对千岛湖48种鱼类的DNA进行扩增后发现,发现16S rDNA和COⅠ引物的扩增效果优于其他几对引物,分析原因可能是该引物的扩增条件差异造成的。张爱菊等[29]使用5种引物对钱塘江鱼类的cytb基因、16Sr DNA和COⅠ基因片段进行扩增,发现cytb和16S rDNA引物都可作为鱼类群落结构研究的通用引物,但是前者扩增产物注释的鱼类物种数高于后者,并推测可能长度较小的目的基因引物更有利于获得较高的扩增成功率。综上可知,引物在鱼类中的通用性受到目的基因片段长度、鱼的种类及扩增反应条件的影响,确定某种引物作为通用引物时,需要综合考虑以上因素。
3.2 鱼粉中原料鱼组成的鉴定本研究以cytb为通用引物,结合分子克隆的方法,开发了一种灵敏、准确、简便的鱼粉中原料鱼组成的鉴定方法,利用该方法,从7种鱼粉中共鉴定到33种鱼类,包括市场上大部分常见的原料鱼物种,以及几种非原料鱼物种,这是传统的镜检技术无法达到的,体现了基因鉴定技术无可比拟的优点。
33种鱼类包括制作红鱼粉常见的鳀鱼、沙丁鱼、鲱鱼、竹荚鱼、鲹等科或属鱼类的多种鱼。这些鱼种在不同鱼粉样品中检出的频次各不相同,反映了鱼粉原料鱼组成的差异。秘鲁鳀是一种小型中上层鱼类,是世界上重要的商业性捕捞鱼类之一,也是制作红鱼粉最大的单种鱼[30]。它在秘鲁鱼粉、阿根廷鱼粉和国产普通鱼粉中分别检出了10、9和4次,分别占检测总次数的76.9%、42.9%和30.8%,证实了秘鲁鳀是制作这些鱼粉的主要原料鱼。值得注意的是,国产普通鱼粉除了秘鲁鳀等海水鱼,还多次地检测到尼罗罗非鱼。作为制作淡水鱼粉的常见原料鱼,罗非鱼本不应在该鱼粉样品中大量检出,说明该鱼粉可能高比例掺入了以罗非鱼为原料的淡水鱼粉。本实验室对该鱼粉样品进行了镜检,在5次抽样中均检出了罗非鱼特征骨,证实了分子检测结果的可靠性。反之,在淡水鱼粉中,16个阳性克隆中检出15个尼罗罗非鱼,这是由于我国淡水鱼粉的主要原料是罗非鱼下脚料[30];此外,在淡水鱼粉中还检出1个杜氏绫鳀,推测可能是在转运或加工过程中偶然混入,只是常规方法通常难以检出,在此被检出体现了分子检测技术的高灵敏度。
在其他红鱼粉如海水鱼排粉、国产半脱脂鱼粉、美国鱼粉中均未检出鳀属鱼类,而是以鲹、沙丁鱼、竹荚鱼或油鲱等属的鱼种为主。海水鱼排粉和半脱脂鱼粉通常由经济价值较高的鲹科鱼类制作鱼片后,其头、骨、内脏等下脚料混合价值较低的沙丁鱼、油鲱等制成。在这些样品中多次检出蓝圆鲹、隆背小沙丁鱼、日本竹荚鱼也证实了这些鱼粉的原料鱼组成。除此之外,以上鱼粉中低频次检出的物种如似鳚镖鲈、绿鳍马面鲀、巴西海猪鱼、五棘渊八角鱼等并非制作鱼粉的原料鱼,但也真实地反映了渔获物的偶然性和捕捞水域的物种多样性。
美国鱼粉的主要原料为油鲱鱼等小型中上层鱼类,它们主要分布于大西洋西海岸的加拿大至南美海域,以大西洋中部产量为最多[31]。本研究中,20个阳性克隆中检测到5个巴西油鲱和7个大西洋油鲱,两者合计占比达60%,证实了美国鱼粉的主要原料为油鲱。值得注意的是,在美国鱼粉中还多次检测到岩栖真鳂,这种鱼属于鳂科真鳂属,主要栖息于近海水域,广泛分布于大西洋西部、大西洋东部岛屿,甚至整个西印度群岛和加勒比海地区,并非制作鱼粉的捕捞对象[32],推测可能是偶然原因进入捕捞水域,导致在该鱼粉中被检出。同样,蓝圆鲹及青背竹筴鱼在此鱼粉中的检出,也显示了渔获物组成的不确定和多样性。
由于缺乏标签信息,以上鉴定结果虽然与鱼粉的常识相符,且详细呈现了每种鱼粉鱼种组成的特点,但无法逐一证实。因此,又用添加已知鱼种的自配鱼粉验证本方法的可靠性。结果表明,自配鱼粉的物种鉴定结果与试验设计相符,即2种自配鱼粉中均检出了配方鱼种。在自配鱼粉一中掺入了10%罗非鱼粉,罗非鱼的检出次数占阳性克隆的25%;在自配鱼粉二中仅掺入0.1%巴沙鱼粉,结果检出频次为8.3%。据此推测提高阳性克隆的个数到达一定数量时,该数值可能更接近实际的比例。
综上所述,本研究运用基于cytb引物的分子鉴定技术,获得了7种商业鱼粉的原料鱼组成信息,为包括鱼粉在内等复杂饲料原料生物组成的鉴定提供了可行的技术方案。今后,有必要对以上方法进行进一步的优化,如提高目的基因的转化效率、简化阳性克隆鉴定方法、开发定量检测技术,为分子检测技术在饲料行业中的推广应用奠定基础。
4 结论cytb引物可作为鱼粉中原料鱼组成鉴定的通用引物。通过获得鱼粉中该基因的阳性克隆子并进行测序,可鉴定出7种商品鱼粉中的共33种鱼类。用已知鱼种的鱼粉样品对以上方法进行了验证,确定了该方法的可靠性。
| [1] |
杜玉雯.我国鱼粉市场供求分析[D].硕士学位论文.上海: 上海海洋大学, 2016: 1-9.
|
| [2] |
LI Y P, HUANG F R, XIAN R Y, et al. Qualitative identification of fish meal and meat bone meal via fluorescence spectral imaging[J]. Food Analytical Methods, 2015, 8(8): 2150-2156. DOI:10.1007/s12161-015-0109-7 |
| [3] |
郑丽明. 使用氨基酸指标鉴别掺假鱼粉研究概况[J]. 广东饲料, 2015, 24(5): 38-40. |
| [4] |
吕程序.基于二维相关NIRS/NIRM的蛋白饲料原料判别方法研究[D].博士学位论文.北京: 中国农业大学, 2014: 33-63.
|
| [5] |
刘佳佳, 何芬, 张松, 等.一种快速检验鱼粉掺假的方法: 中国, 201810394111.3[P].2018-11-13.
|
| [6] |
YEDIER S, BOSTANCI D. Aberrant otoliths in four marine fishes from the Aegean Sea, Black Sea, and Sea of Marmara (Turkey)[J]. Regional Studies in Marine Science, 2020, 34: 101011. DOI:10.1016/j.rsma.2019.101011 |
| [7] |
VAN RAAMSDONK L W D, PRINS T W, VAN DE RHEE N, et al. Microscopic recognition and identification of fish meal in compound feeds[J]. Food Additives and Contaminants:Part A, 2017, 34(8): 1364-1376. DOI:10.1080/19440049.2017.1283711 |
| [8] |
IBÁÑEZA A, O'HIGGINS P. Identifying fish scales:the influence of allometry on scale shape and classification[J]. Fisheries Research, 2011, 109(1): 54-60. |
| [9] |
金雨婷, 蒋日进, 赵进, 等. 鱼卵和仔稚鱼鉴定技术研究进展[J]. 安徽农业科学, 2019, 47(15): 18-20. |
| [10] |
柳楠, 刘积凤, 赵金山, 等. 基于线粒体16S rRNA基因扩增检测动物源性成分的方法研究[J]. 中国畜牧杂志, 2016(15): 74-78. |
| [11] |
高梓童, 王晓玥, 刘杨, 等. 基于ITS2序列的川贝母中成药的鉴定[J]. 中国科学(生命科学), 2018, 48(4): 482-489. |
| [12] |
李青娇.利用分子生物学技术对几种水产加工品原料的鉴定研究[D].硕士学位论文.南京: 南京农业大学, 2013.
|
| [13] |
刘军, 赵良杰, 凡迎春, 等. 鱼类环境DNA研究中通用引物的筛选验证[J]. 淡水渔业, 2016, 46(1): 9-17. |
| [14] |
JERDE C L, MAHON A R, CHADDERTON W L, et al. "Sight-unseen" detection of rare aquatic species using environmental DNA[J]. Conservation Letters, 2011, 4(2): 150-157. DOI:10.1111/j.1755-263X.2010.00158.x |
| [15] |
TAKAHARA T, MINAMOTO T, DOI H. Using environmental DNA to estimate the distribution of an invasive fish species in ponds[J]. PLoS One, 2013, 8(2): e565584. |
| [16] |
NATONEK-WIŚNIEWSKA M, KRZY Ś CIN P, PIESTRZYŃSKA-KAJTOCH A. The species identification of bovine, porcine, ovine and chicken components in animal meals, feeds and their ingredients, based on COX Ⅰ analysis and ribosomal DNA sequences[J]. Food Control, 2013, 34(1): 69-78. |
| [17] |
KUMAR D, SINGH S, KARABASANAVAR N, et al. Authentication of beef, carabeef, chevon, mutton and pork by a PCR-RFLP assay of mitochondrial cytb gene[J]. Journal of Food Science and Technology, 2014, 51(11): 3458-3463. DOI:10.1007/s13197-012-0864-z |
| [18] |
SHINODA N, HASHIMOTO Y, TAKAGI M, et al. PCR-RFLP identification of prohibited animal derived DNA in animal feed[J]. Food Hygiene and Safety Science, 2011, 52(1): 24-27. |
| [19] |
闫平平, 齐欣, 黄大亮, 等. 中国北方沿海14种经济鱼类和7种鲽科鱼类的PCR-RFLP分析[J]. 检验检疫学刊, 2015, 25(6): 13-16. |
| [20] |
BALLIN N Z, VOGENSEN F K, KARLSSON A H. Species determination-can we detect and quantify meat adulteration?[J]. Meat Science, 2009, 83(2): 165-174. DOI:10.1016/j.meatsci.2009.06.003 |
| [21] |
ZHANG C. Semi-nested multiplex PCR enhanced method sensitivity of species detection in further-processed meats[J]. Food Control, 2013, 31(2): 326-330. |
| [22] |
宋岱松. 多重PCR技术在食品安全检测中的应用[J]. 山东畜牧兽医, 2009, 30(5): 57-58. |
| [23] |
REA S, STORANI G, MASCARO N, et al. Species identification in anchovy pastes from the market by PCR-RFLP technique[J]. Food Control, 2009, 20(5): 515-520. DOI:10.1016/j.foodcont.2008.08.001 |
| [24] |
EVANS N T, OLDS B P, RENSHAW M A, et al. Quantification of mesocosm fish and amphibian species diversity via environmental DNA metabarcoding[J]. Molecular Ecology Resources, 2016, 16(1): 29-41. DOI:10.1111/1755-0998.12433 |
| [25] |
BECKER R A, SALES N G, SANTOS G M, et al. DNA barcoding and morphological identification of neotropical ichthyoplankton from the Upper Paraná and São Francisco[J]. Journal of Fish Biology, 2015, 87(1): 159-168. DOI:10.1111/jfb.12707 |
| [26] |
孙超, 刘志鸿, 杨爱国, 等. 4种河蓝蛤线粒体COⅠ和16S rRNA基因序列的种间遗传分析[J]. 渔业科学进展, 2014, 35(1): 82-90. |
| [27] |
陈文炳, 缪婷玉, 彭娟, 等. 基于16S rRNA基因DNA条形码鉴定美洲鳗、欧洲鳗、日本鳗[J]. 食品科学, 2017, 38(4): 283-289. |
| [28] |
THOMSEN P F, KIELGAST J, IVERSEN L L, et al. Monitoring endangered freshwater biodiversity using environmental DNA[J]. Molecular Ecology, 2012, 21(11): 2565-2573. DOI:10.1111/j.1365-294X.2011.05418.x |
| [29] |
张爱菊, 郝雅宾, 郭爱环, 等. 基于高通量测序技术的鱼类环境DNA研究中通用引物的筛选验证[J]. 浙江农业学报, 2019, 31(10): 1615-1623. |
| [30] |
朱华平, 卢迈新, 黄樟翰, 等. 我国罗非鱼加工的现状、产业化发展的优势和提高出口竞争力的措施[J]. 水产科技, 2009(5): 11-14. |
| [31] |
FRANKLIN H B.The most important fish in the sea: Menhaden and America[M].Washington, D.C.: Island Press, 2007.
|
| [32] |
SHINOZAKI-MENDES R, HAZIN F, DE OLIVEIRA P G, et al. Reproductive biology of the squirrelfish, Holocentrus adscensionis (Osbeck, 1765), caught off the coast of Pernambuco, Brazil[J]. Scientia Marina, 2007, 71(4): 715-722. DOI:10.3989/scimar.2007.71n4715 |




