2. 西北农林科技大学林学院, 杨凌 712100;
3. 凤县逢春济民可信科技养殖有限公司, 凤县 721000
2. College of Forestry, Northwest A & F University, Yangling 712100, China;
3. Fengxian Fengchun Jimin Credible Science and Technology Breeding Co., Ltd., Fengxian 721000, China
林麝(Moschus berezovskii)属于麝科(Moschidae)麝属(Moschus),目前已被列为我国一级保护动物,其所产麝香是一种药用价值极高的动物产品。林麝在野生状态下属于泛食、嫩食动物,即使在食物匮乏期仍会回避高纤维植物[1]。对野生林麝消化道微生物进行研究有助于我们了解自然状态下林麝消化道微生物组成,对野生林麝保护及圈养林麝饲养具有借鉴和参考价值。
近年来对圈养反刍动物瘤胃微生物的研究,摸清了牛、羊等动物瘤胃中微生物群落组成,发现其对食物发酵与初步消化具有重要意义,消化道微生物活动会影响动物产品质量[2-3]。林麝属于反刍动物,这使它们容易受到消化道微生物群落活动的影响[4]。在圈养条件下,由环境问题和饲喂不当引起的菌群失衡已经成为影响麝健康的重要因素[5]。目前因采样受限,对林麝消化道微生物的研究进展缓慢[6],仅有的林麝粪便研究表明,环境、食物、健康状态等因素都会影响林麝消化道微生物组成,其中与营养分解相关的核心菌群(拟杆菌门和厚壁菌门细菌)丰度变化不大,而非核心菌群的种类和丰度变化明显[6-8],这些微生物在免疫反应和营养消化方面发挥着重要作用[9]。
目前林麝消化道微生物研究样本以粪便为主,常用的方法是16S rRNA高通量测序,这种方法的缺点是测序深度有限,而宏基因组测序不仅能获得全面的微生物类群信息,还能提供基因功能层面的预测,近年来被广泛用于微生物群落研究[10]。粪便采样具有集合了贯穿胃肠道分散微生物群落的优势,但其与大肠微生物相似度较高,不能完全反映除大肠外其他消化道部位微生物群落结构的真实情况[11]。为全面了解野生林麝消化道微生物结构及其抗生素抗性基因(ARGs)种类,对其消化道分部位取样并进行宏基因组测序是十分必要的。
鉴于此,本试验采用宏基因组测序的方法,对1只珍贵的野生林麝消化道不同区段微生物群落结构和ARGs进行研究,旨在更全面地分析野生林麝消化道不同部位微生物组成及所携带ARGs的特点和差异,为人工养麝防治胃肠道疾病提供深入的数据支持与参考。
1 材料与方法 1.1 试验材料试验样本取自陕西省宝鸡市扶风县一柿园内遭家犬追咬死亡的野生雄性林麝,年龄1.5岁,经解剖后观察,样本发育良好,器官均无病变,表明样本在死亡前健康状况良好。采样时间为初夏(5月中旬),样本死亡后及时取瘤胃、小肠和大肠内容物加50%甘油于-40 ℃保存。
1.2 试验方法 1.2.1 DNA提取与宏基因组建库采用十二烷基苯磺酸钠(SDS)法对样本基因组DNA提取[12],采用琼脂糖凝胶电泳(AGE)和Qubit 2.0荧光计对DNA质量进行检测,然后用Covaris超声波破碎仪将样品DNA随机打断成长度为350 bp的片段,经末端修复、加A尾、加测序接头、纯化、PCR扩增等步骤完成文库制备。DNA文库构建完成后,使用Qubit 2.0荧光计进行初步定量,稀释文库浓度至2 ng/μL,Agilent 2100分析仪对文库的插入片段进行检测。符合预期后使用定量PCR(q-PCR)法对DNA文库有效浓度进行准确定量,不同文库按照有效浓度及目标下机数据量需求合并,然后用Illumina PE150测序仪测序,最终得到原始数据。
1.2.2 生物信息学分析对下机的原始数据进行质控,过滤掉低质量片段和宿主片段后得到有效数据,然后对有效数据进行宏基因组装,为保证数据可靠,将各样品未被利用的片段放在一起进行混合组装。从单样品和混合组装样品中不含N端的片段出发,采用MetaGeneMark软件进行基因预测,将各单样品和混合组装样品预测产生的基因放在一起进行去冗余,去冗余后的片段用于构建基因目录,综合各样品有效数据获得基因目录在各样品中的丰度信息。将基因目录与MicroNR库进行比对,获得每个基因的物种注释信息,然后与KEGG数据库、Card数据库进行比对得到相关基因注释结果。
2 结果与分析 2.1 宏基因组测序经质控过滤,小肠、瘤胃和大肠中分别获得了11 354.99、13 246.22和14 315.52 Mbp有效数据。有效数据中测序错误率小于1%(Q20)和1‰(Q30)的碱基数目比例分别达到97%和92%以上,表明测序数据可靠性较高(表 1)。基因预测共得到11 461 517条开放阅读框(ORFs)用于后续物种注释和基因功能分析。
|
|
表 1 数据预处理统计 Table 1 Data preprocessing statistics |
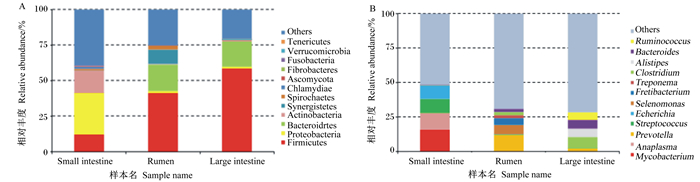
消化道3个区段中优势菌门组成存在一定差异,瘤胃中优势菌门为厚壁菌门(51%)、拟杆菌门(23%)和互养菌门(12%);小肠中优势菌门为变形菌门(43%)、放线菌门(23%)和厚壁菌门(17%);大肠中优势菌门为厚壁菌门(73%)和拟杆菌门(22%),两者比值明显高于瘤胃(图 1-A)。属水平上瘤胃和小肠细菌组成差异十分明显,3个区段之间没有共享的优势菌属(图 1-B)。瘤胃中主要菌属是与碳水化合物、蛋白质分解相关的厌氧菌属:普雷沃氏菌属(16%)、月形单胞菌属(8%)、依赖杆菌属(6%)、拟杆菌属(3%);而小肠中多为致病菌属和条件致病菌属:分枝杆菌属(23%)、链球菌属(15%)、埃希氏菌属(14%)、无形体属(12%);大肠中多为与营养物质分解相关的菌属:梭菌属(10%)、拟杆菌属(8%)、另枝菌属(8%)。
|
Small intestine:小肠;Rumen:瘤胃;Large intestine:大肠;Firmicutes:厚壁菌门;Proteobacteria:变形菌门;Bacteroidrtes:拟杆菌门;Actinobacteria:放线菌门;Synergistetes:互养菌门;Spirochaetes:螺旋体门;Chlamydiae:衣原体门;Ascomycota:子囊菌门; Tenericutes:柔膜菌门;Fibrobacteres:纤维杆菌门;Fusobacteria:梭杆菌门;Verrucomicrobia:疣微菌门;Mycobacterium:分枝杆菌属;Anaplasma:无形体属;Prevotella:普雷沃氏菌属;Streptococcus:链球菌属;Echerichia:埃希氏菌属;Selenomonas:月形单胞菌属;Fretibacterium:依赖杆菌属;Treponema:密螺旋体属;Alistipes:另枝菌属;Bacteroides:拟杆菌属;Clostridium:梭菌属;Ruminococcus:瘤胃球菌属;Others:其他。 图 1 林麝瘤胃、小肠、大肠细菌在门(A)和属(B)水平上的相对丰度 Fig. 1 Relative abundance of bacteria on phylum (A) and genus (B) levels in rumen, small intestine and large intestine of wild forest musk deer |
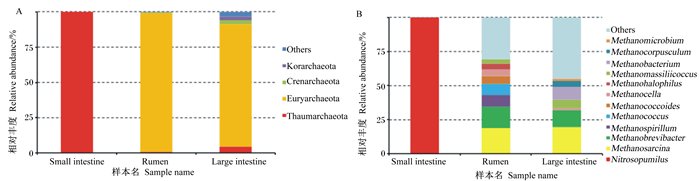
消化道3个区段主要古菌为广古菌门和奇古菌门,大肠中有少部分属于泉古菌门(1%)和初古菌门(1%)(图 2-A)。在属水平上,瘤胃和大肠优势古菌属相似,相对丰度最大的2个古菌属均为甲烷八叠球菌属、甲烷短杆菌属,小肠中的古菌都属于亚硝化侏儒菌属(图 2-B)。
|
Small intestine:小肠;Rumen:瘤胃;Large intestine:大肠;Euryarchaeota:广古菌门;Thaumarchaeota:奇古菌门;Crenarchaeota:泉古菌门;Korarchaeota:初古菌门;Methanosarcina:甲烷八叠球菌属;Methanobrevibacter:甲烷短杆菌属;Methanospirillum:甲烷螺菌属;Methanococcus:产甲烷球菌属;Nitrosopumilus:亚硝化侏儒菌属;Methanococcoides :拟甲烷球菌属;Methanocella:甲烷胞菌属;Methanohalophilus:甲烷嗜盐菌属;Methanobacterium :甲烷杆菌属;Methanocorpusculum :甲烷粒菌属;Methanomicrobium:甲烷微菌属;Others:其他。 图 2 野生林麝瘤胃、小肠和大肠中古菌在门(A)和属(B)水平上的相对丰度 Fig. 2 Relative abundance of archaea on phylum (A) and genus (B) levels in rumen, small intestine and large intestine of wild forest musk deer |
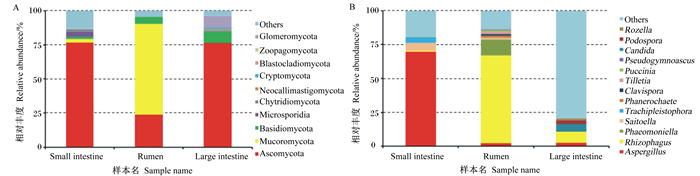
瘤胃、小肠和大肠中共有的优势真菌属于担子菌门、子囊菌门(图 3-A)。在属水平上三者共有的优势真菌为曲霉属和根内球囊霉属,瘤胃中优势真菌还有Phaeomoniella(12%),小肠还有Saitoella(6%),大肠中还有假丝酵母菌属(5%)、柄孢壳菌属(3%)(图 3-B)。
|
Small intestine:小肠;Rumen:瘤胃;Large intestine:大肠;Basidiomycota:担子菌门;Ascomycota:子囊菌门;Mucoromycota:毛霉亚门;Microsporidia:微孢子门;Chytridiomycota:壶菌门;Neocallimastigomycota:新丽鞭毛菌门;Cryptomycota:隐真菌门;Blastocladiomycota:芽枝霉门;Zoopagomycota:捕虫霉门;Glomeromycota:球囊菌门;Aspergillus:曲霉属;Rhizophagus:根内球囊霉属;Saitoella:齐藤酵母属;Phanerochaete:平革菌属;Clavispora:棒孢酵母菌属;Tilletia:腥黑粉菌属;Puccinia:柄锈菌属;Pseudogymnoascus:假裸囊菌属;Candida:假丝酵母菌属;Podospora:柄孢壳菌属;Others:其他。 图 3 野生林麝瘤胃、小肠和大肠中真菌在门(A)和属(B)水平上的相对丰度 Fig. 3 Relative abundance of fungus on phylum (A) and genus (B) levels in rumen, small intestine and large intestine of wild forest musk deer |
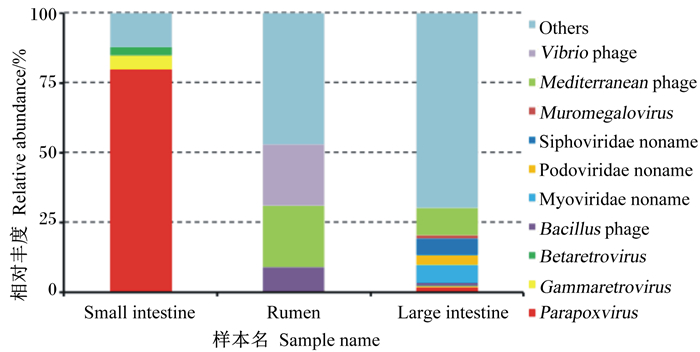
瘤胃中主要病毒种类是弧菌噬菌体(22%)、地中海噬菌体(22%)和芽孢杆菌噬菌体(9%);小肠中相对丰度最高的是副痘病毒属(80%),其次是丙型反录病毒属(5%)、乙型反录病毒属(3%);大肠中优势病毒种类为无害的地中海噬菌体(10%)、肌尾噬菌体(7%)和长尾噬菌体(6%)和(图 4)。
|
Small intestine:小肠;Rumen:瘤胃;Large intestine:大肠;Vibrio phage:弧菌噬菌体;Mediterranean phage:地中海噬菌体;Muromegalovirus:鼠巨细胞病毒属;Siphoviridae noname:长尾噬菌体科未命名;Podoviridae noname:短尾噬菌体科未命名;Myoviridae noname:肌尾噬菌体科未命名;Bacillus phage:芽孢杆菌噬菌体;Betaretrovirus:乙型反转录病毒属;Gammaretrovirus:丙型反转录病毒属;Parapoxvirus:副痘病毒属;Others:其他。 图 4 野生林麝瘤胃、小肠和大肠中病毒的相对丰度 Fig. 4 Relative abundance of virus in rumen, small intestine and large intestine of wild forest musk deer |
去冗余后的ORFs经过KEGG数据库注释,结果如图 5所示。消化道中微生物在数据库中注释数量最多的通路是新陈代谢。对注释数据进行富集差异分析发现,瘤胃、小肠、大肠中微生物在不饱和脂肪酸生物合成、灵杆菌素生物合成以及新霉素、庆大霉素、卡那霉素生物合成这些方面差异显著(P < 0.05),还有一些通路上富集的差异基因数量很多,如淀粉和蔗糖代谢、其他抗生素生物合成、氨基糖和核苷酸糖代谢等。
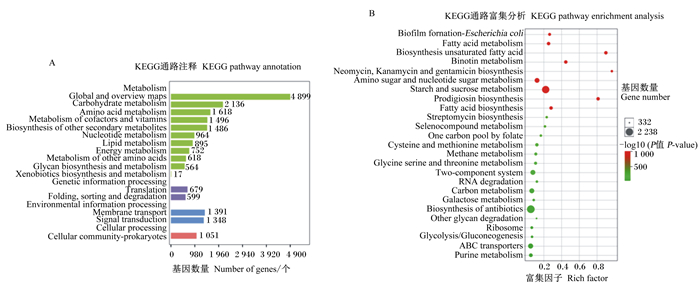
|
图A展示了注释在不同通路中的基因的数量;图B展示了消化道不同区段差异基因的富集分析,点的大小代表差异基因个数,富集因子是当前通路差异基因数与注释在这个通路中所有基因数的比值,颜色代表-log10(P值)的大小,值越大代表差异越显著。The figure A shows the number of genes annotated in different pathways; the figure B shows the enrichment analysis of genes at different digestive tract segments. The size of the dots represents the number of differentially annotated genes. The rich factor is the ratio of the number of differentially annotated genes in the current pathway to the number of all genes in the pathway, and the color represents P-value, the larger value means the more significant the difference. Metabolism:新陈代谢;Global and overview maps:全局概览图;Carbohydrate metabolism:碳水化合物代谢;Amino acid metabolism:氨基酸代谢;Metabolism of cofactors and vitamins:辅酶因子和维生素代谢;Biosynthesis of other secondary metabolites:其他次生代谢物生物合成;Nucleotide metabolism:核苷酸代谢;Lipid metabolism:脂代谢;Energy metabolism:能量代谢;Metabolism of other amino acids:其他氨基酸代谢;Glycan biosynthesis and metabolism:多糖生物合成与代谢;Xenobiotics biosynthesis and metabolism:外源物质生物合成与代谢;Genetic information processing:遗传信息处理;Translation:翻译;Folding, sorting and degradation:折叠、分类和降解;Environmental information processing:环境信息处理;Membrane transport:膜运输;Signal transduction:信号转导;Cellular processing:细胞调控;Cellular community-prokaryotes:原核生物细胞群落;Biosynthesis unsaturated fatty acid:不饱和脂肪酸生物合成;Prodigiosin biosynthesis:灵杆菌素生物合成;Neomycin, kanamycin and gentamicin biosynthesis:新霉素、卡那霉素和庆大霉素生物合成;Starch and sucrose metabolism:淀粉和蔗糖代谢;Biosynthesis of antibiotic:抗生素生物合成;Amino sugar and nucleotide sugar metabolism:氨基糖和核苷酸糖代谢。 图 5 KEGG数据库注释与差异性分析 Fig. 5 KEGG database annotation and variance analysis |
消化道细菌的ARGs对野生林麝的耐药性贡献较大,细菌ARGs注释结果显示,野生林麝瘤胃、小肠和大肠ARGs种类存在明显差异(图 6)。绝对丰度最大的3种ARGs都来自大肠细菌,分别是macB、sav1866和bcrA。大肠中绝对丰度最高的ARGs是macB,瘤胃中绝对丰度最高的ARGs是Aminocoumarin_resistant_alaS,小肠中绝对丰度最高的ARGs是adeG。小肠微生物对adeG、TaeA绝对丰度的贡献较高,分别达到50%和30%。
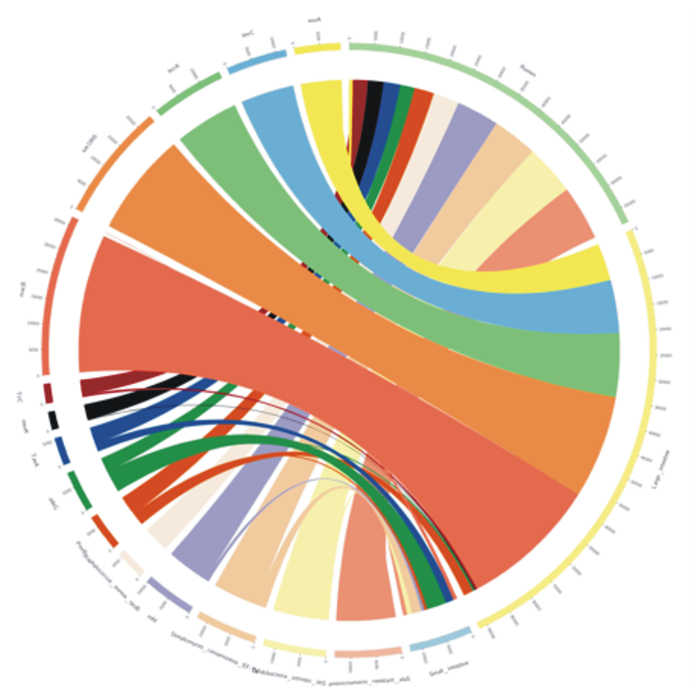
|
Small intestine:小肠;Rumen:瘤胃;Large intestine大肠。 内圈不同颜色表示不同的样品和抗生素抗性基因(ARGs); 外圈刻度表示绝对丰度,左侧为各个ARGs的绝对丰度之和,右侧为各ARGs在样本中的绝对丰度之和。Different colors in the inner circle represent different samples and antibiotic resistance genes (ARGs). The scale of the outer circle is absolute abundance, the left side is the sum of absolute abundance of ARGs in each sample, and the right side is the sum of relative abundance of ARGs in a sample. 图 6 抗生素抗性基因Overview圈图 Fig. 6 Overview graph of ARGs in different sample |
瘤胃中细菌ARGs主要来源于厚壁菌门(44%)和拟杆菌门(26%);小肠中细菌ARGs主要来源于变形菌门(93%),厚壁菌门和拟杆菌门中ARO所占比例较少(共3%);大肠中细菌ARGs主要来源于厚壁菌门(58%)和拟杆菌门(18%)。(图 7)
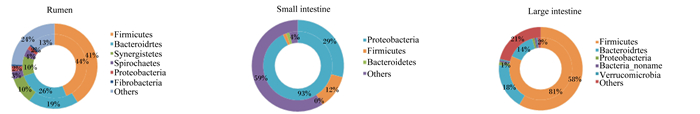
|
Small intestine:小肠;Rumen:瘤胃;Large intestine:大肠;Firmicutes:厚壁菌门;Proteobacteria:变形菌门;Bacteroidrtes:拟杆菌门;Synergistetes:互养菌门;Verrucomicrobia:疣微菌门;Spirochaetes:螺旋体门;Others:其他。 外圈为各菌门基因所占比例,内圈为各菌门抗生素抗性基因所占比例。The outer circle is the proportion of genes in each phylum, and the inner circle is the proportion of ARGs in each phylum. 图 7 野生林麝瘤胃、小肠和大肠中抗生素抗性基因物种归属分析 Fig. 7 Species identification analysis of ARGs in rumen, small intestine and large intestine of wild forest musk deer |
本研究对1只野生林麝的瘤胃、小肠和大肠内容物进行了宏基因组测序,研究野生林麝肠道微生物组成。虽然样本量少,但是关键在于该野生林麝在自然界中自由采食,成长过程中未接触人工抗生素,研究该林麝肠道微生物组成和ARGs种类,能为野生林麝保护和圈养林麝养殖提供重要参考依据。
3.1 野生林麝瘤胃、小肠和大肠中细菌组成差异通过对消化道3个区段微生物组成分析发现,厚壁菌门和拟杆菌门为瘤胃的优势菌门,厚壁菌门能够分解纤维素并产生机体可以直接吸收利用的短链脂肪酸(SCFA),对维持肠道稳态和减少病原菌定植有重要作用[13],拟杆菌门主要作用是降解蛋白质、碳水化合物和脂肪[14];大肠中拟杆菌门和厚壁菌门相对丰度达75%以上,与Li等[6-7]、Hu等[8]对林麝粪便中微生物测序结果一致。相较于瘤胃,大肠中厚壁菌门相对丰度较高,小肠中厚壁菌门相对丰度明显下降,拟杆菌门几乎消失,大肠和小肠中厚壁菌门、拟杆菌门的比值大幅上升。Islam等[15]指出,用含胆酸的食物饲喂大鼠会导致回肠厚壁菌门、拟杆菌门的比值大幅增加。林麝等反刍动物胆汁中胆酸含量较高[16],这可能是导致小肠中拟杆菌门相对丰度下降的原因。
变形菌门在小肠中的相对丰度明显偏高,这与赵文静等[11]对小鼠小肠细菌观察到的结果相似。小肠中的优势菌属多为致病菌,其中大肠杆菌属于γ变形菌门中的一种条件致病菌,在宿主免疫系统健全的情况下能刺激小肠免疫系统发育和免疫应答,促进肠上皮细胞分裂并分泌杀菌物质[17-18]。麝在管理不当或受到强烈刺激时会导致免疫力下降,易受到大肠杆菌影响导致腹泻[19]。林麝小肠细菌中多为病原菌的原因可能是小肠中充满营养丰富的食糜,是病菌与机体竞争吸收营养物质的主要体外场所。至今为止,对麝消化道中病菌的研究较少,这些病菌在麝免疫系统和消化系统中发挥的作用还需要进一步探究。
本研究结果显示,互养菌门是瘤胃的优势菌门(相对丰度达到10%),在小肠和大肠样本中没有检测到,在先前以粪便为样本的研究[6-8]中也未发现。有报道称互养菌门在陕北白绒山羊瘤胃中主要负责降解氨基酸和丙酮酸,在幼年山羊瘤胃中与母乳消化有关[20]。林麝属于嫩食动物,嫩叶中蛋白质含量丰富,互养菌门细菌可能是林麝为适应高蛋白质食物所保有的共生菌,在氨基酸消化中起重要作用。
在属水平上,普雷沃氏菌属、月形单胞菌属、依赖杆菌属等厌氧有益菌在瘤胃中可以分解纤维素和糖,产生营养物质,改善胃肠道环境的代谢物[21],对维持消化道稳态起到非常重要的作用。有益菌还能通过刺激免疫系统、消耗空间和氧气等方法抑制多数需氧致病菌的繁殖[22]。基于以上有助于消化、免疫的野生林麝消化道微生物,后续可以开展麝用益生菌制剂的研究和开发,对于改善圈养林麝的健康状态有重要意义。
一些细菌在消化道中丰度较低,但对消化道稳态影响较大,如艾克曼嗜黏蛋白菌,通过吞噬胃肠道黏膜中的黏蛋白调整黏液层厚度,其在消化道分布是由黏液层厚度决定的[23]。本试验仅在瘤胃和大肠中检测到少量艾克曼嗜黏蛋白菌,这与Li等[6]认为该菌只存在于圈养林麝的推断不同。Li等[6]推测由于圈养林麝与人接触多,艾克曼嗜黏蛋白菌可能通过人传给了圈养林麝,通过本研究结果推测,艾克曼嗜黏蛋白菌可以正常定植在野生林麝的消化道中,但该菌过量繁殖可能会对宿主产生不利影响,例如艾克曼嗜黏蛋白菌繁殖会干扰宿主黏膜平衡,从而加剧伤寒沙门氏菌诱发的肠道炎症[24]。艾克曼嗜黏蛋白菌在麝消化道中的作用还不清楚,其定植与人为活动、麝胃肠炎之间的关系还有待进一步验证。
3.2 野生林麝瘤胃、小肠和大肠中古菌、真菌和病毒组成差异瘤胃中真菌的主要作用是提高细菌对纤维素的利用率并参与细胞壁多糖降解后的发酵[25]。林麝属于嫩食性动物,其食物中纤维素含量低,所以其瘤胃中真菌数量也低于牛、马等粗饲动物[26]。麝瘤胃中优势真菌是根内球囊霉,它是一种植物共生菌,能提高植物抗旱性能和对微量元素的获取[27],其可能是伴随植物一起进入瘤胃的。小肠中优势真菌是黄曲霉,其在林麝小肠中占比较大,这种真菌在无氧条件下不产生黄曲霉素,对牦牛消化道微生物的研究表明其主要抑制拟杆菌门中普雷沃氏菌属的增殖[28],这从另一个角度解释了小肠中拟杆菌门相对丰度下降的现象。
瘤胃和大肠中优势古菌属是甲烷八叠球菌属、甲烷短杆菌属,小肠中优势古菌属是亚硝化侏儒菌属。已有研究显示圈养林麝粪便中的古菌主要有甲烷短杆菌属和热裸单胞菌属[29],这与本试验所得大肠结果有差距。甲烷八叠球菌属是一种可以利用多种途径产甲烷的古菌,通过消耗氢气保证消化道正常发酵的进行[30]。造成这种差异的原因可能是由于野生林麝和圈养林麝生存环境不同导致,后续还需要多样本验证。
瘤胃和大肠中的病毒多为弧状噬菌体、地中海噬菌体、肌尾噬菌体这类对宿主无害的噬菌体,它们主要侵染细菌,其中一些会导致细菌裂解。而小肠中多为对宿主有害的致病性病毒,如副痘病毒属,其能引起动物组织病变、增生[31],也能被肠上皮树突状细胞吞噬,将其呈递给T细胞,进而引发机体的免疫反应[32],对维持正常免疫反应有一定作用。
3.3 野生林麝瘤胃、小肠和大肠中ARGs差异本研究发现小肠微生物ARGs主要是adeG和TaeA,多编码膜内转运体和外排泵,TaeA赋予小肠细菌对泰妙菌素的耐药性;瘤胃中微生物ARGs主要为Aminocoumarin_resistant_alaS,赋予大肠杆菌对新生霉素的抗性;大肠中微生物ARGs主要编码外排泵,其赋予大肠对大环内酯类抗生素、维拉帕米、四苯基氯化磷等抗生素的耐药性。先前对圈养林麝消化道微生物ARGs的研究表明,其对阿莫西林、青霉素、四环素等常见抗生素具有耐药性[33],这与本研究所得野生林麝消化道ARGs种类差距较大,导致差异的原因可能是生长环境不同。外排泵相关基因的表达是导致细菌多重耐药性的主要原因[34], 小肠和大肠中微生物外排泵相关基因表达会导致宿主多重耐药性,这提示消化道后半部分中微生物与宿主耐药性关系更密切,在治疗消化道疾病用药时,应注意避开已经有抗性的药物以达到最佳治疗效果。野生林麝耐药性主要来自于消化道微生物所携带的ARGs,这些微生物的ARGs可能来自于土壤微生物基因的水平交换。本试验中野生林麝消化道ARGs数据可为圈养林麝消化道微生物ARGs研究做参考。
4 结论① 本试验所采样的这只野生林麝瘤胃、小肠和大肠中细菌组成差异较大,共有优势菌门为厚壁菌门和变形菌门。
② 古菌、真菌等在野生林麝瘤胃、小肠和大肠中组成差距较大,占比均相对较少;此外,一些病毒和真菌会对细菌的相对丰度产生影响。
③ 野生林麝大肠和小肠中微生物与宿主的多重耐药性关系密切,表明这些部位的一些特殊菌群可能在麝免疫反应和耐药性中起重要作用。
| [1] |
吴家炎, 王伟. 中国麝类[M]. 北京: 中国林业出版社, 2006: 75-78. WU J Y, WANG W. The musk deer of China[M]. Beijing: China Forestry Publishing House, 2006: 75-78 (in Chinese). |
| [2] |
王婷.甜高粱饲草和葡萄籽对肉羊养分利用、瘤胃微生物区系及肉质性能的影响[D].硕士学位论文.兰州: 兰州大学, 2019: 5-7. WANG T.Effects of sweet sorghum forage grass and grape seed on nutrient utilization, rumen microflora and meat quality of mutton sheep[D]. Master's Thesis.Lanzhou: Lanzhou University, 2019: 5-7.(in Chinese) |
| [3] |
李俊霞. 提高奶山羊瘤胃发酵特性的技术研究进展[J]. 中国饲料, 2020(6): 10-13. LI J X. Advances in improving rumen fermentation characteristics of dairy goats[J]. China Feed, 2020(6): 10-13 (in Chinese). |
| [4] |
RUSSELL J B, RYCHLIK J L. Factors that alter rumen microbial ecology[J]. Science, 2001, 292(5519): 1119-1122 (in Chinese). DOI:10.1126/science.1058830 |
| [5] |
闫敏, 颜其贵, 杨光友. 圈养麝的群发性疾病[J]. 经济动物学报, 2016, 20(2): 112-117. YAN M, YAN Q G, YANG G Y. The mass diseases of captive musk deer[J]. Journal of Economic Animal, 2016, 20(2): 112-117 (in Chinese). |
| [6] |
LI Y M, HU X L, YANG S, et al. Comparative analysis of the gut microbiota composition between captive and wild forest musk deer[J]. Frontiers in Microbiology, 2017, 8: 1705. DOI:10.3389/fmicb.2017.01705 |
| [7] |
LI Y M, HU X L, YANG S, et al. Comparison between the fecal bacterial microbiota of healthy and diarrheic captive musk deer[J]. Frontiers in Microbiology, 2018, 9: 300. DOI:10.3389/fmicb.2018.00300 |
| [8] |
HU X L, LIU G, LI Y M, et al. High-throughput analysis reveals seasonal variation of the gut microbiota composition within forest musk deer (Moschus berezovskii)[J]. Frontiers in Microbiology, 2018, 9: 1674. DOI:10.3389/fmicb.2018.01674 |
| [9] |
叶勇, 全建平, 吴杰, 等. 家畜肠道微生物代谢组学研究进展[J]. 家畜生态学报, 2020, 41(3): 1-8. YE Y, QUAN J P, WU J, et al. Advances of study on livestock intestinal microbial metabolites[J]. Journal of Domestic Animal Ecology, 2020, 41(3): 1-8 (in Chinese). |
| [10] |
何静, 海勒, 斯仁达来, 等. 宏基因组学在哺乳动物肠道微生物中的应用[J]. 中国畜牧兽医, 2018, 45(12): 3438-3446. HE J, HAI L, SIRENDALAI, et al. Application of metagenomics in mammals gut microbiom[J]. China Animal Husbandry & Veterinary Medicine, 2018, 45(12): 3438-3446 (in Chinese). |
| [11] |
赵文静, 刘书云, 丁金梅, 等. 小鼠不同肠道段内容物和粪便中微生物的宏基因组测序和比较分析[J]. 上海交通大学学报(农业科学版), 2016, 34(3): 15-21. ZHAO W J, LIU S Y, DING J M, et al. Metagenomic sequencing of gut microbiota along the intestinal tracts and feces in mice[J]. Journal of Shanghai Jiaotong University (Agricultural Science), 2016, 34(3): 15-21 (in Chinese). DOI:10.3969/J.ISSN.1671-9964.2016.03.003 |
| [12] |
单志, 吴宏亮, 李成磊, 等. 改良SDS法提取多种植物基因组DNA研究[J]. 广东农业科学, 2011, 38(8): 113-115. SHAN Z, WU H L, LI C L, et al. Improved SDS method for general plant genomic DNA extraction[J]. Guangdong Agricultural Sciences, 2011, 38(8): 113-115 (in Chinese). DOI:10.3969/j.issn.1004-874X.2011.08.046 |
| [13] |
DUNCAN S H, LOUIS P, THOMSON J M, et al. The role of pH in determining the species composition of the human colonic microbiota[J]. Environmental Microbiology, 2009, 11(8): 2112-2122. DOI:10.1111/j.1462-2920.2009.01931.x |
| [14] |
JAMI E, ISRAEL A, KOTSER A, et al. Exploring the bovine rumen bacterial community from birth to adulthood[J]. The ISME Journal, 2013, 7(6): 1069-1079. DOI:10.1038/ismej.2013.2 |
| [15] |
ISLAM K B M S, FUKIYA S, HAGIO M, et al. Bile acid is a host factor that regulates the composition of the cecal microbiota in rats[J]. Gastroenterology, 2011, 141(5): 1773-1781. DOI:10.1053/j.gastro.2011.07.046 |
| [16] |
张能荣. 牛羊胆汁质量的研究[J]. 中草药, 1983, 14(7): 15-17. ZHANG N R. Study on the quality of cattle and sheep bile[J]. Chinese Traditional and Herbal Drugs, 1983, 14(7): 15-17 (in Chinese). |
| [17] |
杨事达, 赵鸿梅. 大肠埃希菌细菌膜外蛋白A对小肠上皮细胞增殖的影响及其作用机制[J]. 中国生物制品学杂志, 2018, 31(11): 1207-1209. YANG S D, ZHAO H M. Effect of outer-membrane protein A on proliferation of intestinal epithelial cells and relevant mechanism[J]. Chinese Journal of Biologicals, 2018, 31(11): 1207-1209 (in Chinese). |
| [18] |
张明昱, 杜肖彦, 赵鹏伟. 大肠埃希菌诱导小肠上皮细胞产生β防御素-3的机制[J]. 中国生物制品学杂志, 2018, 31(11): 1219-1221. ZHANG M Y, DU X Y, ZHAO P W. Mechanism of induction of β-defensin-3 production in intestinal epithelial cells by E.coli[J]. Chinese Journal of Biologicals, 2018, 31(11): 1219-1221 (in Chinese). |
| [19] |
CHUNG L K, RAFFATELLU M. G.I.pros:antimicrobial defense in the gastrointestinal tract[J]. Seminars in Cell & Developmental Biology, 2019, 88: 129-137. |
| [20] |
HAN X F, YANG Y X, YAN H L, et al. Rumen bacterial diversity of 80 to 110-day-old goats using 16S rRNA sequencing[J]. PLoS One, 2015, 10(2): e0117811. DOI:10.1371/journal.pone.0117811 |
| [21] |
MATSUI H, OGATA K, TAJIMA K, et al. Phenotypic characterization of polysaccharidases produced by Four Prevotella type strains[J]. Current Microbiology, 2000, 41(1): 45-49. DOI:10.1007/s002840010089 |
| [22] |
MOAYYEDI P. The epidemiology of obesity and gastrointestinal and other diseases:an overview[J]. Digestive Diseases and Sciences, 2008, 53(9): 2293-2299. DOI:10.1007/s10620-008-0410-z |
| [23] |
冯泽猛, 包显颖, 印遇龙. 胃肠道黏液层中Akkermansia muciniphila的定殖研究及其与宿主的相互作用[J]. 中国农业科学, 2016, 49(8): 1577-1584. FENG Z M, BAO X Y, YIN Y L. The Interaction of colonization of Akkermansia muciniphila in gastrointestinal tract and its host[J]. Scientia Agricultura Sinica, 2016, 49(8): 1577-1584 (in Chinese). |
| [24] |
GANESH B P, KLOPFLEISCH R, LOH G, et al. Commensal Akkermansia muciniphila exacerbates gut inflammation in Salmonella typhimurium-infected gnotobiotic mice[J]. PLoS One, 2013, 8(9): e74963. DOI:10.1371/journal.pone.0074963 |
| [25] |
THAREJA A, PUNIYA A K, GOEL G, et al. In vitro degradation of wheat straw by anaerobic fungi from small ruminants[J]. Archives of Animal Nutrition, 2006, 60(5): 412-417. |
| [26] |
GRIFFITH G W, OZKOSE E, THEODOROU M K, et al. Diversity of anaerobic fungal populations in cattle revealed by selective enrichment culture using different carbon sources[J]. Fungal Ecology, 2009, 2(2): 87-97. |
| [27] |
BIDELLAOUI B, SEGARRA G, HAKKOU A, et al. Beneficial effects of Rhizophagus irregularis and Trichoderma asperellum strain T34 on growth and fusarium wilt in tomato plants[J]. Journal of Plant Pathology, 2018, 101(1): 121-127. |
| [28] |
安雅静, 文勇立, 赵佳琦, 等. 宏基因组学揭示AFB1对牦牛瘤胃微生物多样性及CAZy谱影响[J]. 家畜生态学报, 2019, 40(2): 13-20. AN Y J, WEN Y L, ZHAO J Q, et al. Metagenomics reveals the effect of AFB1 on rumen microbial diversity and CAZy of yak[J]. Journal of Domestic Animal Ecology, 2019, 40(2): 13-20 (in Chinese). |
| [29] |
王立志, 徐谊英, 蔡永华. 高通量测序分析人工养殖成年林麝粪便古菌菌群多样性[J]. 动物营养学报, 2016, 28(2): 477-484. WANG L Z, XU Y Y, CAI Y H. Fecal archaeal diversity of artificial breeding adult forest musk deer analyzed by high-throughput sequencing technologies[J]. Chinese Journal of Animal Nutrition, 2016, 28(2): 477-484 (in Chinese). |
| [30] |
田彦.基于高通量测序的中国荷斯坦奶牛瘤胃宏转录组研究[D].博士学位论文.北京: 中国科学院北京基因组研究所, 2015: 21-23. TIAN Y.Metatranscriptomic study of Chinese Holstein dairy cow rumen based on high-throughput sequencing[D]. Ph.D.Thesis.Beijing: Beijing Institute of Genomics Chinese Academy of Sciences, 2015: 21-23.(in Chinese) |
| [31] |
VILA A R, BRICEÑO C, MCALOOSE D, et al. Putative parapoxvirus-associated foot disease in the endangered huemul deer (Hippocamelus bisulcus) in Bernardo O'Higgins National Park, Chile[J]. PLoS One, 2019, 14(4): e0213667. DOI:10.1371/journal.pone.0213667 |
| [32] |
GÓRSKI A, WAŻNA E, DABROWSKA B K, et al. Bacteriophage translocation[J]. FEMS Immunology & Medical Microbiology, 2006, 46(3): 313-319. |
| [33] |
LUO X, WANG P, CHENG J G, et al. Characterization of virulence genes and antimicrobial resistance of lung pathogenic Escherichia coli isolates in forest musk deer (Moschus berezovskii)[J]. Journal of Zoo and Wildlife Medicine, 2016, 47(2): 540-550. |
| [34] |
周黎明, 王正荣, 王浴生. 大肠杆菌主动外排泵与抗生素多重耐药性研究进展[J]. 四川生理科学杂志, 2003, 25(4): 163-164. ZHOU L M, WANG Z R, WANG Y S. Research progress on active efflux pump of Escherichia coli and antibiotic multi-resistance[J]. Sichuan Journal of Physiological Sciences, 2003, 25(4): 163-164 (in Chinese). |




