2. 中国科学院西北生态环境资源研究院, 兰州 730030;
3. 甘肃省寒区旱区逆境生理与生态重点实验室, 兰州 730030;
4. 宁夏大学生命科学学院, 银川 750021
2. Northwest Institute of Eco-Environment and Resources, Chinese Academy of Sciences, Lanzhou 730030, China;
3. Key Laboratory of Stress Physiology and Ecology of Gansu Province, Lanzhou 730030, China;
4. Life Sciences College of Ningxia University, Yinchuan 750021, China
随着我国居民消费水平的提高和膳食结构的改变,对反刍动物肉和奶制品的需求持续增加。在畜牧业的发展中,降低饲养成本、提高饲料利用效率、减少环境污染是核心的科学问题。饲养饲料利用效率高的动物不仅可以降低成本,还可以产生更少的甲烷排放,减少对大气环境的污染,缓解温室效应[1-2]。胃肠道菌群作为反刍动物的“第二基因组”[3]受到广泛关注,基因组、转录组、代谢组等多组学技术的发展促进了反刍动物胃肠道菌群的组成和作用机制的认知,大量研究表明反刍动物瘤胃和肠道菌群作为中间介质共同影响着宿主对饲料的消化吸收。其中,反刍动物的瘤胃发酵产生的挥发性脂肪酸(VFAs)提供每日能量的70%,盲肠也可以发酵纤维物质影响宿主的饲料利用效率[4]。目前,反刍动物肠道菌群的研究较少,本文就反刍动物肠道菌群的结构、对饲料利用效率的调节效应以及调控措施等方面的相关研究进行总结分析,为未来调控反刍动物饲料利用效率和肠道菌群的研究提供一定思路。
1 饲料利用效率饲料转化率(FCR)常用来表示饲料利用效率,与饲料利用效率呈负相关,被广泛用于畜牧生产和研究中。剩余采食量(RFI)是另一个常用于评估反刍动物营养生理的概念,RFI定义为动物实际采食量和预测采食量(基于体重和生长情况计算)之间的差异。RFI值越低,动物的饲料利用效率就越高。RFI受到食欲、进食行为、瘤胃发酵和胃肠道菌群组、肠道吸收和细胞形态、线粒体功能和生理应激、基因组成与环境互作等因素的综合影响[5]。在绵羊的相关研究中,RFI与FCR具有显著相关性,与干物质采食量(DMI)极显著相关,与FCR相比更能准确地反映饲料利用效率[6]。
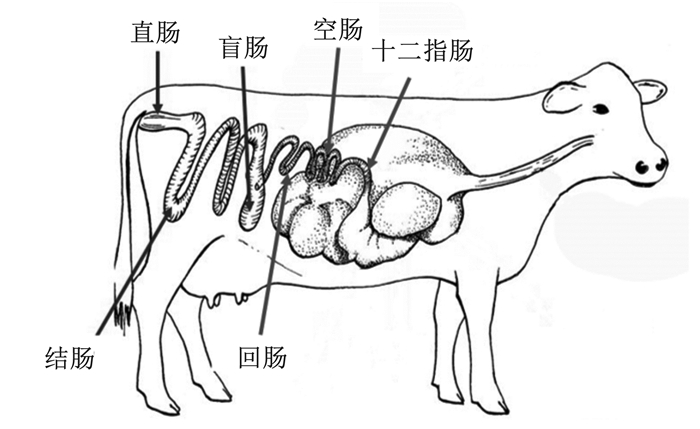
2 反刍动物肠道菌群的多样性反刍动物肠道结构如图 1所示。肠道作为反刍动物的后消化道,是消化吸收营养物质的重要场所,其中定植的微生物具有重要作用。反刍动物肠道pH的差异、肠蠕动、氧化还原电位、营养物质供应和宿主分泌物等的不同造就了肠道不同区域间细菌丰富度和多样性的不同[8]。Wang等[9]根据小尾寒羊菌群组成的相似性,将其肠道菌群分为小肠(十二指肠、空肠和回肠)菌群和大肠(盲肠、结肠和直肠)菌群2大部分。研究发现,小肠中的优势菌属为埃希氏菌属(Escherichia)、未分类毛螺菌科(unclassified Lachnospiraceae)和瘤胃球菌属(Ruminococcus)。大肠中主要的菌属是瘤胃球菌属、未分类瘤胃菌科(unclassified Ruminococcaceae)和普氏菌属(Prevotella)。
|
图 1 牛的肠道结构 Fig. 1 Gut structure of cattle[7] |
在牛的肠道中,十二指肠菌群主要由厚壁菌门(Firmicutes)和放线菌门(Actinobacteria)组成。厚壁菌门主要包括瘤胃菌科、毛螺菌科和韦荣氏球菌科(Veillonellaceae)等。与小肠的其余部分相比,十二指肠中毛螺菌科的相对丰度较高,这可能与该区域存在的瘤胃液有关。牛空肠菌群与回肠菌群结构类似,在门水平上厚壁菌门皆占大多数,其次是拟杆菌门(Bacteroidetes)。厚壁菌门主要由梭菌科(Clostridiaceae)、苏黎世杆菌科(Turicibacteraceae)和瘤胃菌科组成。盲肠是反刍动物发酵的第2个主要部位。在盲肠中厚壁菌门、拟杆菌门和变形菌门(Proteobacteria)是主要的菌门,在厚壁菌门中梭菌科和瘤胃菌科是主要菌科,其次是毛螺菌科和苏黎世杆菌科。与小肠相比,盲肠中菌群多样性和丰度增加,与盲肠中消化物的停留时间和养分发酵时间加长有关。结肠的菌群在门和科水平上都与盲肠相似,拟杆菌门以帕拉普氏菌科(Paraprevotellaceae)、普氏菌科(Prevotellaceae)和拟杆菌科(Bacteroidaceae)为主[10-11]。
在其他反刍动物中也有类似发现。He等[12]对双峰驼的肠道菌群测序分析发现,其以厚壁菌门、疣微菌门(Verrucomicrobia)和拟杆菌门为主,肠道中的优势菌属为阿克曼氏菌属(Akkermansia)、纤维杆菌属(Fibrobacter)、普氏菌属和未分类瘤胃菌科。而在斯瓦尔巴德群岛驯鹿的肠道菌群中,最丰富的门是厚壁菌门和拟杆菌门,梭菌目(Clostridiales)和拟杆菌目(Bacteroidales)为优势菌群[13]。骆驼和驯鹿的肠道菌群组成与牛、羊有一定的相似性,但不同的物种、生存环境和采食等造就了菌群组成的明显差异。
3 反刍动物肠道菌群与饲料利用效率的相关性 3.1 肠道菌群与宿主新陈代谢反刍动物的肠道菌群能以多种形式参与宿主新陈代谢的调节,并与宿主相互作用。在反刍动物的多糖、蛋白质消化吸收,氨基酸、脂多糖、维生素合成等代谢途径中有重要作用[14],并参与短链脂肪酸、胆汁酸、多酚代谢等多种代谢途径的调节,通过信号传导,调节多个器官组织的活动,从而影响反刍动物的健康和生产性能[4, 15]。
3.2 反刍动物肠道菌群与饲料利用效率牛、羊的肠道菌群组成上有很大的相似性,饲料利用效率不同的牛、羊肠道主要菌群间的相对丰度和多样性差异较小,但在少数关键菌群间存在明显差异。
在十二指肠中,艰难杆菌科(Mogibacteriaceae)、芽孢杆菌属(Bacillus)和普氏菌属的丰度与饲料利用效率均呈负相关[11]。由于瘤胃和十二指肠位置的邻近性,艰难杆菌科、普氏菌属与其在瘤胃中的作用相同,其丰度均与饲料利用效率显著相关[16-17]。反刍动物中艰难杆菌科的相关研究较少,小鼠肠道菌群中艰难杆菌科丰度升高会减少脂肪增加量,减轻体重[18]。芽孢杆菌属的丰度与饲料利用效率呈负相关,芽孢杆菌的丰度与饲料利用效率的关系尚未有研究报告,潜在机制可能是芽孢杆菌降低了促进机体吸收养分微生物的活性。在低平均日增重(ADG)阉牛的十二指肠中,普氏菌属丰度更高;普氏菌属可降解多糖和蛋白质含量,并参与多种维生素的合成,影响宿主的饲料利用效率[11]。
空肠是氨基酸、葡萄糖和脂肪酸吸收的主要部位。在空肠中,菌群丰度变化对饲料利用效率有重要作用,艰难杆菌属(Mogibacterium)、丁酸弧菌属(Butyrivibrio)、乳酸杆菌属(Lactobacillus)、棒状杆菌科(Corynebacteriaceae)、红蝽菌科(Coriobacteriaceae)、YS2目、脱硫弧菌属(Desulfovibrio)、根瘤菌目(Rhizobiales)等的丰度与饲料利用效率间有显著的相关性[11, 19]。其中,艰难杆菌属的丰度与饲料利用效率呈负相关。艰难杆菌属是糖酵解性细菌,与氨同化有关,可利用氨合成苯丙氨酸的前体苯乙酸,增加牛的氮消化,减轻体重[20]。丁酸弧菌属是毛螺菌科中的革兰氏阳性细菌,可分解纤维物质、果胶、淀粉等多糖产生丁酸盐,在空肠中可用糖类作为底物[19, 21]。其产物丁酸盐可以在转录水平上调节牛的脂肪细胞中瘦素的表达,减少食物摄入量并增加能量消耗[22]。乳酸杆菌常作为益生菌使用,其在空肠中其丰度与ADG呈负相关的具体原因尚不明确。但有研究发现,乳酸杆菌丰度在肉牛的瘤胃菌群中与RFI呈负相关[23],可能是该属的某些菌种与胆汁酸解偶联导致脂质吸收减少,从而降低宿主ADG[24]。棒状杆菌属(Corynebacterium)丰度与ADG呈负相关,反刍动物肠道的相关研究较少,在肥胖人群的肠道菌群中也有类似发现[25]。红蝽菌科在高饲料利用效率的猪盲肠中也有较高的丰度,具有调节宿主动物脂质代谢的作用[26]。YS2是蓝细菌门(Cyanobacteria)中唯一的非光合蓝细菌,其在肠道中的作用尚未有报道。在奶牛瘤胃菌群中,YS2的相对丰度与产奶量、乳品质以及饲料利用效率都存在正相关性[27]。脱硫弧菌属也大量存在于牛的瘤胃中,但尚不清楚与饲料利用相关的潜在机制[28]。大鼠肠道脱硫弧菌属丰度降低可减少高脂饮食引起的肥胖,与ADG呈正相关[29]。根瘤菌科(Rhizobiaceae)常被认为是土壤中的固氮细菌[30],空肠中的可能来自于饲粮中豆类植物,其所包含的菌种可能在促进反刍动物消化吸收方面有潜在的作用机制。
在回肠中,艰难杆菌科和红蝽菌科丰度与饲料利用效率呈正相关[11],空肠中也有类似结果;芽孢杆菌属丰度与饲料利用效率呈负相关,这与其在牛十二指肠和瘤胃菌群中效果类似。回肠中布雷德菌属(Bulleidia)的丰度与ADG呈正相关。布雷德菌属与脂质代谢有关,在生物氢化中有潜在作用[31]。在反刍动物回肠中与其在人十二指肠中的功能相似,高丰度的布雷德菌属与肥胖显著相关[32]。
盲肠是反刍动物肠道发酵的主要部位。在盲肠中,普氏菌属、瘤胃球菌属、拟杆菌属(Bacteroides)、布劳特氏菌属(Blautia)、丹毒丝菌科(Erysipelotrichaceae)、萨特氏菌属(Sutterella)和RF39的丰度均与饲料利用效率有显著相关性[11, 33-34]。普氏菌属、瘤胃球菌属、布劳特氏菌属均参与盲肠的发酵,能分泌纤维素酶、木聚糖酶、α-葡萄糖苷酶等多种碳水化合物活性酶,分解纤维素、半纤维素和淀粉等[35]。由于盲肠与瘤胃功能的相似性,其中定植菌群的结构和功能也存在一定的相似性。据报道,拟杆菌属也是牛瘤胃中的优势菌群,可以分解纤维素等多糖,与饲料利用效率相关[23, 36]。布劳特氏菌属在牛瘤胃菌群中的丰度与普氏菌属丰度呈正相关,与饲料利用效率呈负相关[37]。在高饲料利用效率的肉牛盲肠中,丹毒丝菌科相对丰度较高。丹毒丝菌科与胆固醇代谢、肝脏脂肪变化有相关性,在肥胖仓鼠的肠道中丰度较高[38]。盲肠中的萨特氏菌属和RF39的丰度也均与饲料利用效率呈正相关。萨特氏菌属的丰度与葡萄糖代谢具有相关性,反刍动物相关研究较少。与其在大鼠结肠中的功能类似,在体重减少的大鼠结肠中萨特氏菌属的丰度显著降低[39]。RF39属于柔膜菌纲(Mollicutes),在肉牛盲肠中与其在大鼠盲肠中的作用相似,其丰度与脂肪含量增加呈正相关[40]。
在结肠菌群中,拟杆菌属、布劳特氏菌属、丁酸弧菌属、粪球菌属(Coprococcus)、颤螺菌属(Oscillospira)和普氏菌属的丰度都与饲料利用效率存在显著相关性[11, 41]。普氏菌属、拟杆菌属和布劳特氏菌属都与多糖分解有关,由于位置邻近,均与在盲肠菌群中的作用相似。丁酸弧菌属在不同效率的瘤胃、空肠中也存在差异。粪球菌属能发酵碳水化合物,在饲喂高抗性淀粉的猪结肠和盲肠中丰度显著增加,与麦芽糖、6-磷酸葡萄糖、谷氨酸和谷氨酰胺等代谢产物含量呈正相关[42]。颤螺菌属(Oscilliospira)在反刍动物肠道中的研究很少,其也存在于人肠道菌群中,但未被培养过,可以代谢多糖产生丁酸盐,与促进形成瘦肉的小克里斯滕森氏菌(Christensenella minuta)高度相关,且在非肥胖人群肠道中丰度较高,与人的身体质量指数(BMI)呈负相关[43]。
在粪便菌群中,不同饲料利用效率的牛在毛螺菌科、瘤胃菌科、普氏菌属和新美鞭菌科(Neocallimastigaceae)的丰度上存在显著差异[33, 44]。这几种细菌的丰度也在反刍动物肠道其他肠段与饲料利用效率有相关性。新美鞭菌科是反刍动物的消化道中大量存在的真菌,可分泌降解细胞壁的酶和细胞外多酶复合物,在纤维的分解中发挥关键作用[45]。
羔羊的饲料利用效率与肠道菌群也存在相关性。Perea等[46]对2组饲料利用效率差异显著的公羔羊的十二指肠、空肠、回肠、结肠和直肠菌群测序分析,发现肠道中的差异菌群为在纤维降解中发挥作用的纤维杆菌门(Fibrobacteres)、瘤胃菌科和Saccharofermentans,与健康有关的双歧杆菌科(Bifidobacteriaceae)和克里斯滕氏菌科(Christensenellaceae)以及与生态失调相关的变形菌门。有研究发现,林麝的肠道菌群中瘤胃球菌属、阿克曼氏菌属、颤螺菌属、粪球菌属的丰度与ADG存在正向调节作用[47]。这些菌群也在牛和羊的肠道消化吸收中发挥重要作用,进而调节宿主生长。
3.3 肠道菌群对反刍动物营养物质吸收的影响肠道是反刍动物营养物质吸收的主要场所,不同的肠道菌群也会造成物质吸收效率的差异。饲料利用效率高的动物也具有高的氮利用率和低的甲烷排放量[48]。Wang等[49]通过高通量16S rRNA基因测序研究发现努比亚山羊肠道细菌结构与氮利用率之间存在显著的相关性。氮的利用率与空肠细菌中的瘤胃球菌属、Candidatus_Saccharimonas和粪球菌属丰度呈负相关;与结肠中的丹毒丝菌UCG-004属(Erysipelotrichaceae_UCG-004)、阿克曼氏菌属和甲烷粒菌属(Methanocorpusculum)丰度呈正相关;与盲肠中的瘤胃球菌UCG-002属(Ruminococcaceae_UCG-002)、厌氧弧菌属(Anaerovibrio)和瘤胃球菌UCG-007属(Ruminococcaceae_UCG-007)丰度呈正相关。进入宿主小肠的蛋白质中大约70%是瘤胃合成的微生物蛋白,但小肠和大肠在蛋白质代谢中也有重要作用。瘤胃菌科可以分解纤维素,促进氮的吸收利用,在盲肠中其丰度与氮利用率呈正相关,而空肠中的瘤胃球菌属的功能可能与其他菌种不同。结肠中的甲烷粒菌属可以促进纤维素分解和含氮物质的消化吸收,从而减少了甲烷排放[50]。
肠道菌群还影响反刍动物对矿物质元素的吸收。Liu等[51]对高钙消化率和低钙消化率的努比亚山羊肠道微生物进行16S rRNA扩增子测序分析发现,结肠中纤维杆菌属和盲肠中丁酸弧菌属的丰度与钙的真消化率呈负相关。肠道菌群代谢产生的短链脂肪酸形成酸性环境可能会提高钙的吸收[52]。丁酸弧菌属可能通过产生丁酸盐影响钙的吸收。小鼠和人相关研究发现,产丁酸盐细菌的丰度增加,特定丁酸盐的类型和浓度都可以提高肠道对钙的吸收利用[53-54]。山羊菌群中的丁酸弧菌属则相反,这可能与宿主差异及细菌的分类注释水平有关,种水平上不同的微生物具有不同的生物学功能。高磷消化率和低磷消化率的山羊肠道菌群也存在差异,磷的真消化率与空肠菌群中的脱硫弧菌属、纤维杆菌属、普氏菌属的丰度呈正相关;与盲肠中的脱硫弧菌属的丰度呈正相关[55]。肠道细菌能够分泌植酸酶水解植酸来提高饲料中磷的真消化率,也有研究发现这可能是相关菌群通过代谢物石胆酸,以肠细胞维生素D受体依赖的方式增加了反刍动物肠道对磷和钙的吸收[56]。
4 反刍动物肠道菌群的调控措施随着现代生物技术的发展,特别是多组学技术在动物营养学和饲料学领域的运用,更深入地揭示了不同营养素与肠道相关菌群及其代谢物间的作用和机理。饲粮中添加益生菌制剂和菌群移植是最直接有效调节肠道菌群结构、菌群组成和功能的措施。
4.1 益生菌益生菌不仅在调节反刍动物的瘤胃和肠道菌群结构,提高生产性能,而且在增强机体免疫力和健康恢复等方面均具有益生作用,因此被广泛用于畜牧生产中。常用作饲料添加剂的益生菌有乳酸菌、双歧杆菌、芽孢杆菌、丁酸梭菌、酵母菌、黑曲霉等。
益生菌制剂可以调节动物肠道消化吸收,提高生产性能。Apas等[57]用羊源的乳杆菌(Lactobacillus reuteri DDL 19, Lactobacillus alimentarius DDL 48)、粪肠球菌(Enterococcus faecium DDE 39)、双歧杆菌(Bifidobacterium bifidum DDBA)制成的混合菌剂饲喂山羊后,肠道菌群平衡改变,沙门氏菌(Salmonella)丰度减少,有益菌双歧杆菌和乳酸菌增加,山羊的体重显著增加,粪便中腐胺含量显著减少。益生菌在提高宿主生产力的同时,也能增加其免疫力。在萨能奶山羊饲粮中添加酿酒酵母(Saccharomyces cerevisiae)、枯草芽孢杆菌(Bacillus subtilis)和粪肠球菌(Enterococcus faecalis),肠道中的拟杆菌、埃希氏菌属-志贺氏菌属(Escherichia-Shigella)和克里斯滕氏菌科(Christensenellaceceae)的丰度降低,琥珀酸弧菌科(Succinivibrionaceae)丰度增加,羊奶产量提高,乳蛋白率、乳糖率、乳脂率显著提高[58]。在荷斯坦奶牛饲粮中添加乳杆菌(Lactobacillus case zhang和Lactobacillus plantarum P-8),奶牛肠道中瘤胃球菌属等有益菌的丰度增加,蜡样芽孢杆菌等条件致病菌丰度显著下降,奶牛的产奶量增加,乳品质也得到提高[59]。
4.2 菌群移植粪菌移植(FMT)是通过将健康供体的粪便移植到受体肠道以恢复其肠道菌群正常功能的一种技术手段。FMT作为传统中医治疗手段的改良,被广泛用于艰难梭菌感染(CDI)、溃疡性结肠炎(UC)、克罗恩病(CD)、肠易激综合征(IBS)、放射性肠炎等人类疾病的治疗,并对肠脑轴疾病、肝病、糖尿病、癌症等有良好的治疗效果[60]。
在动物生产中,FMT可以有效改善被动免疫失败的犊牛肠道菌群结构和肠道上皮的屏障功能,对犊牛的生长和健康状况产生积极影响[61]。FMT还可以增加早产猪肠道菌群多样性和少数菌群丰度,改善了免疫细胞聚集和黏膜抵抗力,减少了肠炎患病率[62]。但也有研究发现,高饲料利用效率猪的肠道菌群移植到妊娠母猪和仔猪体内后,结果并没有改善饲料利用效率,还造成肠道损伤,并降低了ADG[63]。这可能与所选供体、所移植的菌群以及移植方法有关。因此,反刍动物FMT需要参照人医,建立标准化制备流程,严格筛选供体,改善菌群移植方法,以提高菌群的移植效率和安全性。
除FMT技术外,反刍动物生产中常用的菌群移植方法是喂服瘤胃液。幼龄反刍动物的瘤胃和胃肠道菌群不成熟并且对微环境敏感,直接通过喂服瘤胃液进行菌群移植的有效性高。在羔羊断奶前反复喂服新鲜瘤胃液可加速瘤胃发育和肠道菌群的建立,增加菌群的多样性,改善菌群组成,调节羔羊肠道微生态[64]。给羔羊喂服瘤胃液还能增强肠道中Toll样受体的表达,释放更多的免疫相关因子,维持肠道黏膜局部免疫应答[65]。另外,喂服瘤胃液还可以减少羔羊的断奶休克,在8周后可以显著提高平均日采食量(ADFI)和ADG,促进其生长发育[66]。
5 小结虽然瘤胃菌群一直是反刍动物微生物组研究的重点,但肠道菌群与饲料利用效率间也存在显著相关性,在实际生产中可通过粪便菌群监测宿主表型和健康状况,调控肠道菌群提高反刍动物的生产效率,反刍动物肠道菌群具有重要的研究意义。
目前,针对反刍动物的肠道菌群研究较少,且主要集中于细菌种属上,肠道中的古菌、真菌、原虫、病毒等微生物对宿主的调节作用有待深入研究。反刍动物菌群的研究还应考虑整体与部分相结合,既要探索口腔、胃、肠道的菌群结构功能,又要对消化道菌群进行整体研究。由于微生物的研究受多种因素影响,在试验设计、操作方法和生物信息学的数据分析等方面都需要严格的操作程序才能保证试验的准确性。将微生物培养组学、宏基因组、转录组、蛋白质组、代谢组等多种技术相结合以及多种生理系统(如血液、瘤胃和肠道内容物以及尿液、粪便等)的研究将提供有关宿主与其微生物组之间关系的更多信息。除营养调控外,在未来的研究中,将瘤胃和肠道微生物组与宿主饲料利用效率以及其他重要的生产参数(如遗传型)进行关联,对提高畜牧业的整体效率和可持续性具有重要意义。
| [1] |
BEN SHABAT S K, SASSON G, DORON-FAIGENBOIM A, et al. Specific microbiome-dependent mechanisms underlie the energy harvest efficiency of ruminants[J]. The ISME Journal, 2016, 10(12): 2958-2972. DOI:10.1038/ismej.2016.62 |
| [2] |
XUE M Y, SUN H Z, WU X H, et al. Multi-omics reveals that the rumen microbiome and its metabolome together with the host metabolome contribute to individualized dairy cow performance[J]. Microbiome, 2020, 8: 64. DOI:10.1186/s40168-020-00819-8 |
| [3] |
CELLUZZI A, MASOTTI A. How our other genome controls our epi-genome[J]. Trends in Microbiology, 2016, 24(10): 777-787. DOI:10.1016/j.tim.2016.05.005 |
| [4] |
O'HARA E, NEVES A L A, SONG Y, et al. The role of the gut microbiome in cattle production and health: driver or passenger?[J]. Annual Review of Animal Biosciences, 2020, 8: 199-220. DOI:10.1146/annurev-animal-021419-083952 |
| [5] |
KENNY D A, FITZSIMONS C, WATERS S M, et al. Invited review: improving feed efficiency of beef cattle the current state of the art and future challenges[J]. Animal, 2018, 12(9): 1815-1826. DOI:10.1017/S1751731118000976 |
| [6] |
莫负涛. 不同RFI育肥羔羊生产性能和体组成及消化代谢研究[D]. 硕士学位论文. 兰州: 甘肃农业大学, 2016: 15-24. MO F T. The Effect of supplement with four kinds of ruminal fluid preparations on the intestinal mucosal immune of newborn lambs[D]. Master's Thesis. Lanzhou: Gansu Agricultural University, 2016: 15-24. (in Chinese) |
| [7] |
李胜利. 奶牛营养学[M]. 北京: 科学出版社, 2020: 1-13. LI S L. Nutriology of dairy cattle[M]. Beijing: Science Press, 2020: 1-13 (in Chinese). |
| [8] |
MAO S Y, ZHANG M L, LIU J H, et al. Characterising the bacterial microbiota across the gastrointestinal tracts of dairy cattle: membership and potential function[J]. Scientific Reports, 2015, 5: 16116. DOI:10.1038/srep16116 |
| [9] |
WANG J, FAN H, HAN Y, et al. Characterization of the microbial communities along the gastrointestinal tract of sheep by 454 pyrosequencing analysis[J]. Asian-Australasian Journal of Animal Sciences, 2017, 30(1): 100-110. |
| [10] |
MYER P R, FREETLY H C, WELLS J E, et al. Analysis of the gut bacterial communities in beef cattle and their association with feed intake, growth, and efficiency[J]. Journal of Animal Science, 2017, 95(7): 3215-3224. |
| [11] |
FREETLY H C, DICKEY A, LINDHOLM-PERRY A K, et al. Digestive tract microbiota of beef cattle that differed in feed efficiency[J]. Journal of Animal Science, 2020, 98(2): skaa008. DOI:10.1093/jas/skaa008 |
| [12] |
HE J, YI L, HAI L, et al. Characterizing the bacterial microbiota in different gastrointestinal tract segments of the Bactrian camel[J]. Scientific Reports, 2018, 8: 654. DOI:10.1038/s41598-017-18298-7 |
| [13] |
ZIELIŃSKA S, KIDAWA D, STEMPNIEWICZ L, et al. New insights into the microbiota of the Svalbard reindeer Rangifer tarandus platyrhynchus[J]. Frontiers in Microbiology, 2016, 7: 170. |
| [14] |
ZHANG J C, XU C B, HUO D X, et al. Comparative study of the gut microbiome potentially related to milk protein in Murrah buffaloes (Bubalus bubalis) and Chinese Holstein cattle[J]. Scientific Reports, 2017, 8(7): 42189. |
| [15] |
SANZ-FERNANDEZ M V, DANIEL J, SEYMOUR D J, et al. Targeting the hindgut to improve health and performance in cattle[J]. Animals, 2020, 10(10): 1817. DOI:10.3390/ani10101817 |
| [16] |
LI Y Q, XI Y M, WANG Z D, et al. Combined signature of rumen microbiome and metabolome in dairy cows with different feed intake levels[J]. Journal of Animal Science, 2020, 98(3): skaa070. DOI:10.1093/jas/skaa070 |
| [17] |
LI F Y, HITCH T C A, CHEN Y H, et al. Comparative metagenomic and metatranscriptomic analyses reveal the breed effect on the rumen microbiome and its associations with feed efficiency in beef cattle[J]. Microbiome, 2019, 7: 6. DOI:10.1186/s40168-019-0618-5 |
| [18] |
ZIETAK M, KOVATCHEVA-DATCHARY P, MARKIEWICZ L H, et al. Altered microbiota contributes to reduced diet-induced obesity upon cold exposure[J]. Cell Metabolism, 2016, 23(6): 1216-1223. DOI:10.1016/j.cmet.2016.05.001 |
| [19] |
MYER P R, WELLS J E, SMITH T P L, et al. Microbial community profiles of the jejunum from steers differing in feed efficiency[J]. Journal of Animal Science, 2016, 94(1): 327-338. DOI:10.2527/jas.2015-9839 |
| [20] |
NAKAZAWA F, SATO M, POCO S E, et al. Description of Mogibacterium pumilum gen.nov., sp.nov.and Mogibacterium vescum gen. nov., sp. nov., and reclassification of Eubacterium timidum (Holdeman et al.1980) as Mogibacterium timidum gen.nov., comb.nov[J]. International Journal of Systematic and Evolutionary Microbiology, 2000, 50(2): 679-688. DOI:10.1099/00207713-50-2-679 |
| [21] |
PALEVICH N, KELLY W J, LEAHY S C, et al. Comparative genomics of rumen Butyrivibrio spp.uncovers a continuum of polysaccharide-degrading capabilities[J]. Applied and Environmental Microbiology, 2019, 86(1): e01993-19. |
| [22] |
SOLIMAN M, KIMURA K, AHMED M, et al. Inverse regulation of leptin mRNA expression by short- and long-chain fatty acids in cultured bovine adipocytes[J]. Domestic Animal Endocrinology, 2007, 33(4): 400-409. DOI:10.1016/j.domaniend.2006.08.005 |
| [23] |
MYER P R, SMITH T P L, WELLS J E, et al. Rumen microbiome from steers differing in feed efficiency[J]. PLoS One, 2015, 10(6): e0129174. DOI:10.1371/journal.pone.0129174 |
| [24] |
MOSER S A, SAVAGE D C. Bile salt hydrolase activity and resistance to toxicity of conjugated bile salts are unrelated properties in lactobacilli[J]. Applied and Environmental Microbiology, 2001, 67(8): 3476-3480. DOI:10.1128/AEM.67.8.3476-3480.2001 |
| [25] |
PALOMO-BUITRAGO M E, SABATER-MASDEU M, MORENO-NAVARRETE J M, et al. Glutamate interactions with obesity, insulin resistance, cognition and gut microbiota composition[J]. Acta Diabetologica, 2019, 56(5): 569-579. DOI:10.1007/s00592-019-01313-w |
| [26] |
HE B B, LI T T, WANG W, et al. Metabolic characteristics and nutrient utilization in high-feed-efficiency pigs selected using different feed conversion ratio models[J]. Science China Life Sciences, 2019, 62(7): 959-970. DOI:10.1007/s11427-018-9372-6 |
| [27] |
JIANG Y, OGUNADE I M, PECH-CERVANTES A A, et al. Effect of sequestering agents based on a Saccharomyces cerevisiae fermentation product and clay on the ruminal bacterial community of lactating dairy cows challenged with dietary aflatoxin B1[J]. Journal of Dairy Science, 2020, 103(2): 1431-1447. DOI:10.3168/jds.2019-16851 |
| [28] |
REN Q M, SI H Z, YAN X T, et al. Bacterial communities in the solid, liquid, dorsal, and ventral epithelium fractions of yak (Bos grunniens) rumen[J]. Microbiology Open, 2020, 9(2): e963. |
| [29] |
ZHAO L, ZHANG Q, MA W N, et al. A combination of quercetin and resveratrol reduces obesity in high-fat diet-fed rats by modulation of gut microbiota[J]. Food & Function, 2017, 8(12): 4644-4656. |
| [30] |
ALEXANDRE A, OLIVEIRA S. Response to temperature stress in rhizobia[J]. Critical Reviews in Microbiology, 2012, 39(3): 219-228. |
| [31] |
PITTA D W, INDUGU N, VECCHIARELLI B, et al. Alterations in ruminal bacterial populations at induction and recovery from diet-induced milk fat depression in dairy cows[J]. Journal of Dairy Science, 2018, 101(1): 295-309. DOI:10.3168/jds.2016-12514 |
| [32] |
ANGELAKIS E, ARMOUGOM F, CARRIÈRE F, et al. A metagenomic investigation of the duodenal Microbiota reveals links with obesity[J]. PLoS One, 2015, 10(9): e0137784. DOI:10.1371/journal.pone.0137784 |
| [33] |
WELCH C B, LOURENCO J M, DAVIS D B, et al. The impact of feed efficiency selection on the ruminal, cecal, and fecal microbiomes of Angus steers from a commercial feedlot[J]. Journal of Animal Science, 2020, 98(7): skaa230. DOI:10.1093/jas/skaa230 |
| [34] |
MYER P R, WELLS J E, SMITH T P L, et al. Cecum microbial communities from steers differing in feed efficiency[J]. Journal of Animal Science, 2015, 93(11): 5327-5340. DOI:10.2527/jas.2015-9415 |
| [35] |
BIDDLE A, STEWART L, BLANCHARD J, et al. Untangling the genetic basis of fibrolytic specialization by lachnospiraceae and ruminococcaceae in diverse gut communities[J]. Diversity, 2013, 5(3): 627-640. DOI:10.3390/d5030627 |
| [36] |
STANTON T, CANALE-PAROLA E. Treponema bryantii sp. nov., a rumen spirochete that interacts with cellulolytic bacteria[J]. Archives of Microbiology, 1980, 127(2): 145-156. DOI:10.1007/BF00428018 |
| [37] |
LI F Y, LI C X, CHEN Y H, et al. Host genetics influence the rumen microbiota and heritable rumen microbial features associate with feed efficiency in cattle[J]. Microbiome, 2019, 7: 92. DOI:10.1186/s40168-019-0699-1 |
| [38] |
KAAKOUSH N O. Insights into the role of Erysipelotrichaceae in the human host[J]. Frontiers in Cellular and Infection Microbiology, 2015, 5: 84. |
| [39] |
WANG C, ZHANG H, LIU H J, et al. The genus Sutterella is a potential contributor to glucose metabolism improvement after Roux-en-Y gastric bypass surgery in T2D[J]. Diabetes Research and Clinical Practice, 2020, 162: 108116. DOI:10.1016/j.diabres.2020.108116 |
| [40] |
THOMAZ F S, TOMSETT K I, PANCHAL S K, et al. Wasabi supplementation alters the composition of the gut microbiota of diet-induced obese rats[J]. Journal of Functional Foods, 2020, 67: 103868. DOI:10.1016/j.jff.2020.103868 |
| [41] |
MYER P R, WELLS J E, SMITH T P L, et al. Microbial community profiles of the colon from steers differing in feed efficiency[J]. Springer Plus, 2015, 4: 454. DOI:10.1186/s40064-015-1201-6 |
| [42] |
SUN Y, SU Y, ZHU W Y. Microbiome-metabolome responses in the cecum and colon of pig to a high resistant starch diet[J]. Frontiers in Microbiology, 2016, 7: 779. |
| [43] |
KONIKOFF T, GOPHNA U. Oscillospira: a central, enigmatic component of the human gut microbiota[J]. Trends in Microbiology, 2016, 24(7): 523-524. DOI:10.1016/j.tim.2016.02.015 |
| [44] |
LOPES D R G, LA REAU A J, DUARTE M D S, et al. The bacterial and fungal microbiota of nelore steers is dynamic across the gastrointestinal tract and its fecal-associated microbiota is correlated to feed efficiency[J]. Frontiers in Microbiology, 2019, 10: 1263. DOI:10.3389/fmicb.2019.01263 |
| [45] |
CHENG Y F, SHI Q C, SUN R L, et al. The biotechnological potential of anaerobic fungi on fiber degradation and methane production[J]. World Journal of Microbiology and Biotechnology, 2018, 34(10): 155. DOI:10.1007/s11274-018-2539-z |
| [46] |
PEREA K, PERZ K, OLIVO S K, et al. Feed efficiency phenotypes in lambs involve changes in ruminal, colonic, and small-intestine-located microbiota[J]. Journal of Animal Science, 2017, 95(6): 2585-2592. |
| [47] |
LIU X, ZHAO W, YU D, et al. Effects of compound probiotics on the weight, immunity performance and fecal microbiota of forest musk deer[J]. Scientific Reports, 2019, 9: 19146. DOI:10.1038/s41598-019-55731-5 |
| [48] |
RIUS A G, KITTELMANN S, MACDONALD K A, et al. Nitrogen metabolism and rumen microbial enumeration in lactating cows with divergent residual feed intake fed high-digestibility pasture[J]. Journal of Dairy Science, 2012, 95(9): 5024-5034. DOI:10.3168/jds.2012-5392 |
| [49] |
WANG L Z, LIU K Z, WANG Z S, et al. Bacterial community diversity associated with different utilization efficiencies of nitrogen in the gastrointestinal tract of goats[J]. Frontiers in Microbiology, 2019, 10: 239. DOI:10.3389/fmicb.2019.00239 |
| [50] |
ZHOU M, HERNANDEZ-SANABRIA E, GUAN L L. Characterization of variation in rumen methanogenic communities under different dietary and host feed efficiency conditions, as determined by PCR-denaturing gradient gel electrophoresis analysis[J]. Applied and Environmental Microbiology, 2010, 76(12): 3776-3786. DOI:10.1128/AEM.00010-10 |
| [51] |
LIU Y H, SHAH A M, WANG L Z, et al. Relationship between the true digestibility of dietary calcium and gastrointestinal microorganisms in goats[J]. Animals, 2020, 10(5): 875. DOI:10.3390/ani10050875 |
| [52] |
WHISNER C M, MARTIN B R, NAKATSU C H, et al. Soluble corn fiber increases calcium absorption associated with shifts in the gut microbiome: a randomized dose-response trial in free-living pubertal females[J]. The Journal of Nutrition, 2016, 146(7): 1298-1306. DOI:10.3945/jn.115.227256 |
| [53] |
ZENG H L, HUANG C C, LIN S, et al. Lotus seed resistant starch regulates gut microbiota and increases short-chain fatty acids production and mineral absorption in mice[J]. Journal of Agricultural and Food Chemistry, 2017, 65(42): 9217-9225. DOI:10.1021/acs.jafc.7b02860 |
| [54] |
WHISNER C M, MARTIN B R, NAKATSU C H, et al. Soluble maize fibre affects short-term calcium absorption in adolescent boys and girls: a randomised controlled trial using dual stable isotopic tracers[J]. British Journal of Nutrition, 2014, 112(3): 446-456. DOI:10.1017/S0007114514000981 |
| [55] |
WANG L Z, SHAH A M, LIU Y H, et al. Relationship between true digestibility of dietary phosphorus and gastrointestinal bacteria of goats[J]. PLoS One, 2020, 15(5): e0225018. DOI:10.1371/journal.pone.0225018 |
| [56] |
HASHIMOTO N, MATSUI I, ISHIZUKA S, et al. Lithocholic acid increases intestinal phosphate and calcium absorption in a vitamin D receptor dependent but transcellular pathway independent manner[J]. Kidney International, 2020, 97(6): 1164-1180. DOI:10.1016/j.kint.2020.01.032 |
| [57] |
APÁS A L, DUPRAZ J, ROSS R, et al. Probiotic administration effect on fecal mutagenicity and microflora in the goat's gut[J]. Journal of Bioscience and Bioengineering, 2010, 110(5): 537-540. DOI:10.1016/j.jbiosc.2010.06.005 |
| [58] |
MA Z Z, CHENG Y Y, WANG S Q, et al. Positive effects of dietary supplementation of three probiotics on milk yield, milk composition and intestinal flora in Sannan dairy goats varied in kind of probiotics[J]. Journal of Animal Physiology and Animal Nutrition, 2019, 104(1): 44-55. |
| [59] |
XU H Y, HUANG W Q, HOU Q C, et al. The effects of probiotics administration on the milk production, milk components and fecal bacteria microbiota of dairy cows[J]. Science Bulletin, 2017, 62(11): 767-774. DOI:10.1016/j.scib.2017.04.019 |
| [60] |
ZHANG F M, CUI B T, HE X X, et al. Microbiota transplantation: concept, methodology and strategy for its modernization[J]. Protein & Cell, 2018, 9(5): 462-473. |
| [61] |
吴兆海. 粪菌移植对被动免疫失败犊牛肠道屏障功能及肠道菌群构建的影响[D]. 博士学位论文. 北京: 中国农业大学, 2018: 36-61. WU Z H. Effects of fecal microbiota transplantation on establishment on intestial barrier function and microbiota establishment in calves with failure of passive immune transfer[D]. Ph. D. Thesis. Beijing: China Agricultural University, 2018: 36-61. (in Chinese) |
| [62] |
BRUNSE A, MARTIN L, RASMUSSEN T S, et al. Effect of fecal microbiota transplantation route of administration on gut colonization and host response in preterm pigs[J]. The ISME Journal, 2019, 13(3): 720-733. DOI:10.1038/s41396-018-0301-z |
| [63] |
MCCORMACK U M, CURIÃO T, WILKINSON T, et al. Fecal microbiota transplantation in gestating sows and neonatal offspring alters lifetime intestinal microbiota and growth in offspring[J]. mSystems, 2018, 3(3): e00134-17. |
| [64] |
YU S B, ZHANG G Y, LIU Z B, et al. Repeated inoculation with fresh rumen fluid before or during weaning modulates the microbiota composition and co-occurrence of the rumen and colon of lambs[J]. BMC Microbiology, 2020, 20: 29. DOI:10.1186/s12866-020-1716-z |
| [65] |
吴婷婷. 补喂4种方法处理的瘤胃液制备物对新生羔羊肠道黏膜免疫的影响[D]. 硕士学位论文. 乌鲁木齐: 新疆农业大学, 2013: 28-45. WU T T. The effect of supplement with four kinds of ruminal fluid preparations on the intestinal mucosal immune of newborn lambs[D]. Master's Thesis. Urumqi: Xinjiang Agricultural University, 2013: 28-45. (in Chinese) |
| [66] |
BELANCHE A, PALMA-HIDALGO J M, NEJJAM I, et al. Inoculation with rumen fluid in early life as a strategy to optimize the weaning process in intensive dairy goat systems[J]. Journal of Dairy Science, 2020, 103(6): 5047-5060. DOI:10.3168/jds.2019-18002 |




