2. 四川水利职业技术学院, 成都 611230
2. Sichuan Water Conservancy Vocational College, Chengdu 611230, China
酵母菌属于兼性厌氧菌,其家族庞大,种类超过1 000种,其中的酿酒酵母(Saccharomyces cerevisiae)作为饲料添加剂在反刍动物中使用已超过70年。众多研究表明,反刍动物饲粮中添加酿酒酵母或酵母培养物可提高饲粮纤维消化率[1-4],提高奶产量,改善乳品质[5-7],提高日增重[8]。但是,前人的报道多认为酵母起作用主要是因为其能够消耗瘤胃内氧气,提高乳酸利用菌如巨型球菌属(Megasphaera)和月形单胞菌属(Selenomonas)和纤维分解菌如纤维杆菌属(Fibrobacter)和瘤胃球菌属(Ruminococcus)等的相对丰度,降低蛋白质降解菌如普雷沃氏菌属(Prevotella)、互养球菌属(Syntrophococcus)和光岗菌属(Mitsuokella)等的相对丰度[9-11],从而加速瘤胃内乳酸分解代谢,稳定瘤胃pH[8],降低氧化还原电位,改善瘤胃内环境,改善动物健康,提高生产性能。但是,这些研究主要从细菌的角度解释了酿酒酵母起作用的原因。厌氧真菌于1975年被发现是瘤胃微生物重要的组成部分[12],因为具有纤维小体和氢化酶体而被认为是瘤胃内最为有效的降解木质纤维素的微生物类群[13],工业上用作帮助降解纤维从而更高效率地生产生物能源[14-15]。目前,饲粮添加活性干酵母对瘤胃真菌的影响未见报道。因此,本试验旨在研究饲粮添加活性干酵母对肉牛营养物质表观消化率和瘤胃真菌群落组成的影响,并从瘤胃真菌的角度解释活性干酵母提高饲粮营养物质消化率的原因,为酿酒酵母在反刍动物饲粮中的应用提供理论支撑。
1 材料与方法 1.1 试验材料试验使用的活性干酵母为反刍动物专用酿酒酵母,活细胞数≥2×1010 CFU/g。
1.2 试验设计与饲养管理试验选取体重(110±13) kg和日龄相近的18头肉牛(西杂牛♂×犏牛♀)为研究对象,采用完全随机设计,分为3个组,每组6头牛。各组分别饲喂基础饲粮(对照组)、基础饲粮+2 g/(d·头)活性干酵母(ADY2组)、基础饲粮+4 g/(d·头)活性干酵母(ADY4组)。预试期10 d,正试期110 d。
基础饲粮参照我国《肉牛饲养标准》(NY/T 815-2004)[16],按照体重150 kg、日增重1.1 kg设计配方,基础饲粮组成及营养水平见表 1。牛只单栏饲养,试验前对场地进行消毒,并对供试牛进行驱虫。每天08:00和20:00按时饲喂,将活性干酵母与饲粮拌均匀后供牛只采食,自由饮水。
|
|
表 1 基础饲粮组成及营养水平(干物质基础) Table 1 Composition and nutrient levels of the basal diet (DM basis) |
于正试期第80天开始,每2周取1次饲粮样品,每次4份,每份2 kg,混匀后按照四分法收集饲粮样品,装入密封袋并于-20 ℃冰箱保存待测。于正式试验结束的前4天开始对所有18头牛采用全收粪法连续收集新鲜粪便,用10%硫酸固氮保存。将饲粮和粪便样品于65 ℃烘干后粉碎过1 mm筛保存,用以测定营养物质含量。干物质(DM)含量参照GB/T 6435-2014测定,粗蛋白质(CP)含量参照GB/T 6432-2018测定,粗脂肪(EE)含量参照GB/T 6433-2006测定,中性洗涤纤维(NDF)和酸性洗涤纤维(ADF)含量分别参照GB/T 20806-2006和NY/T 1459-2007测定,钙含量参照GB/T 6436-2002测定,磷含量参照GB/T 6437-2002测定。各营养物质表观消化率的计算公式如下:

|
试验结束后全部牛只屠宰后分别在瘤胃背囊、腹囊、左纵沟和右纵沟等部位共收集瘤胃内容物约100 g,混匀后置于-80 ℃冰箱保存,用以测定瘤胃真菌组成。
1.4 瘤胃液微生物总DNA提取、扩增和测序采用十六烷基三甲基溴化铵(hexade-cyltrimethy ammonium bromide,CTAB)法提取瘤胃液样品的基因组总DNA,检测纯度和浓度后,稀释至1 ng/μL备用。以稀释后的基因组DNA为模板,使用ITS1-5F(200~400 bp)进行瘤胃液真菌PCR扩增,通用引物为:ITS5-1737F:5′-GGAAGTAAAAGTCGTAACAAGG-3′;ITS2-2043R:5′-GCTGCGTTCTTCATCGATGC-3′。PCR产物经2%凝胶电泳检测,使用Qiagen Gel Extraction Kit(Qiagen公司,德国)纯化PCR产物。使用TruSeq DNA PCR-Free Sample Preparation Kit建库试剂盒进行文库构建,构建好的文库通过Qubit和PCR进行定量,构建文库合格后,使用Illumina HiSeq 2500进行上机测序。
1.5 序列分析根据Barcode和PCR扩增引物序列从原始数据中拆分出各样品数据,截去Barcode和引物序列后使用FLASH对每个样品的reads进行拼接,得到的拼接序列为原始Tags(raw Tags)数据,经过严格的过滤处理得到高质量Tags(clean Tags)数据。高质量Tags数据序列通过UCHIME Algorithm与数据库进行比对检测嵌合体序列,并最终去除其中的嵌合体序列,得到有效Tags(effective Tags)数据。
1.6 操作分类单元(operational taxonomic units,OTUs)聚类和物种注释利用Uparse软件对所有样品的全部有效Tags进行聚类,默认以97%的一致性(identity)将序列聚类成为OTUs,同时筛选出OTUs中出现频数最高的序列作为OTUs的代表序列进行物种注释,用QIIME 1.9.1软件[17]中的Blast方法与Unit数据库进行物种注释分析,并分别在门(phylum)、纲(class)、目(order)、科(family)、属(genus)、种(species)各个分类水平统计各样本的群落组成。对各样品的数据进行均一化处理,以样品中数据量最少的为标准进行均一化处理,后续的多样性分析均基于均一化处理后的数据。
1.7 数据统计分析数据采用Excel 2016进行初步处理,然后采用SAS 9.3统计软件中的随机区组设计进行组间差异显著性比较,数据用平均值和均值标准误(SEM)表示,P < 0.05为差异显著,P < 0.01为差异极显著。
2 结果 2.1 饲粮添加活性干酵母对肉牛营养物质表观消化率的影响由表 2可知,ADY2组和ADY4组的NDF和ADF表观消化率显著高于对照组(P < 0.05),但ADY2组和ADY4组之间无显著差异(P>0.05)。各组之间DM、CP和EE表观消化率无显著差异(P>0.05)。
|
|
表 2 饲粮添加活性干酵母对肉牛营养物质表观消化率的影响 Table 2 Effects of dietary active dried yeast on nutrient apparent digestibilities of beef cattle |
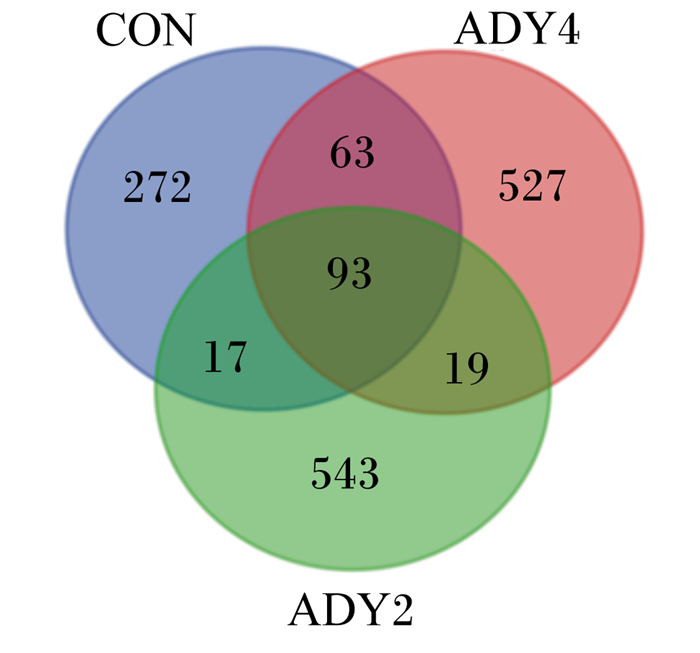
3个组的OTUs比较韦恩图见图 1。按97%一致性的原则对序列进行聚类,对照组仅鉴定出272个OTUs,ADY2组和ADY4组分别鉴定出了543和527个OTUs。对照组与ADY2组共有17个OTUs,对照组与ADY4组共有63个OTUs,ADY2组与ADY4组共有19个OTUs,3个组共有93个OTUs。
|
CON:对照组control group;ADY2:ADY2组ADY2 group;ADY4:ADY4组ADY4 group。下图同the same as below. 图 1 OTUs比较韦恩图 Fig. 1 Venn graph of OTUs comparison |
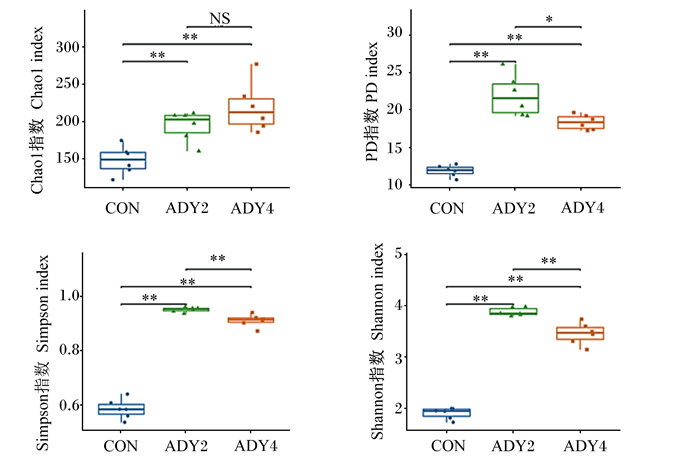
由图 2可知,ADY2组和ADY4组的Simpson和Shannon指数均极显著高于对照组(P < 0.01),且ADY2组极显著高于ADY4组(P < 0.01)。ADY2组和ADY4组的Chao1和PD指数均极显著高于对照组(P < 0.01),ADY2组的PD指数显著高于ADY4组(P < 0.05),ADY2组的Chao1指数与ADY4组差异不显著(P>0.05)。
|
*:差异显著(P < 0.05);* *:差异极显著(P < 0.01);NS:差异不显著(P>0.05)。 *: significant difference (P < 0.05); * *: extremely significant difference (P < 0.01); no significant difference (P>0.05). 图 2 α多样性指数比较 Fig. 2 α diversity indexes comparison |
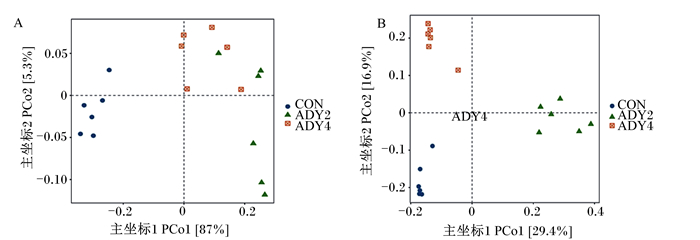
3个组基于Unweighted和Weighted Unifrac距离的PCoA见图 3。Weighted Unifrac距离的PCoA见图 3-A,对照组在纵坐标方向与ADY2组和ADY4组区分开来,主坐标1(PCo1)解释87%的变量。ADY2组和ADY4组的样品相互交叉,不能清晰地区分开。基于Unweighted Unifrac距离的PCoA见图 3-B,3个组的样本组内差异较小因而距离较小,对照组与ADY2组在纵坐标上能够明确区分开,PCo1解释29.4%的变量。对照组与ADY4组在横坐标上能够明确区分开,主坐标2(PCo2)解释16.9%的变量。
|
图 3 基于Unweighted和Weighted Unifrac距离的PCoA Fig. 3 PCoA based on Unweighted and Weighted Unifrac distance |
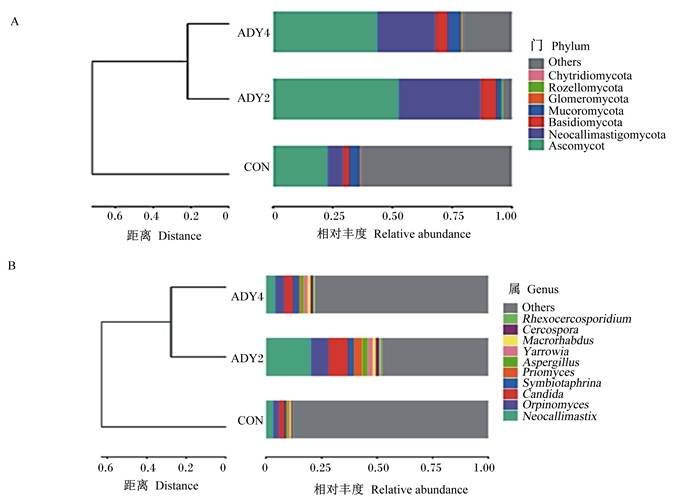
通过Mothur软件将18个样本进行UPGMA分类树分类,结果见图 4。由图 4-A可知,ADY2组和ADY4组在门水平上在0.2的距离上聚为一类,之后再与对照组在>0.6的距离上进行聚类。由图 4-B可知,在属水平也与门水平相似,ADY2组和ADY4组在属水平上在0.3的距离上聚为一类,之后再与对照组在>0.6的距离上进行聚类。
|
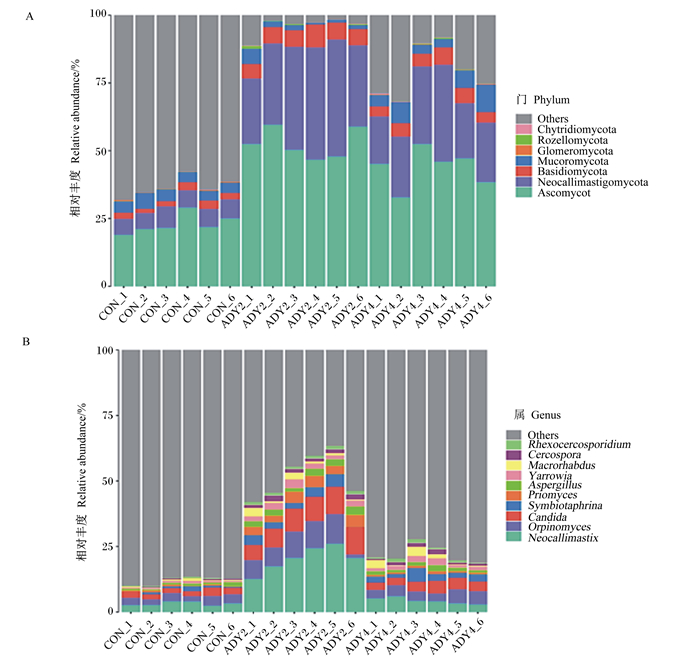
Ascomycot:子囊菌门;Neocallimastigomycota:新丽鞭毛菌门;Basidiomycota:担子菌门;Mucoromycota:毛霉门;Glomeromycota:球囊菌门;Rozellomycota:隐真菌门;Chytridiomycota:壶菌门;Others其他。Neocallimastix:新美鞭菌属;Orpinomyces:根囊鞭菌属;Candida:念珠菌属;Symbiotaphrina:烟草甲体内共生菌;Priomyces:梨囊鞭菌属;Aspergillus:曲霉菌属;Yarrowia:亚罗酵母属;Macrorhabdus:鹦鹉胃癌酵母菌;Cercospora:尾孢菌属;Rhexocercosporidium:山豆根内生真菌。图 5同the same as Table 5. 图 4 门和属水平上基于Weighted Unifrac距离的UPGMA聚类树 Fig. 4 UPGMA clustering tree based on Weighted Unifrac distance at phylum and genus levels |
从表 3和图 5-A可知,从18个瘤胃样品中共鉴定出7个门,分别是子囊菌门(Ascomycota)、新丽鞭毛菌门(Neocallimastigomycota)、担子菌门(Basidiomycota)、毛霉门(Mucoromycota)、球囊菌门(Glomeromycota)、隐真菌门(Rozellomycota)和其他真菌门(Fungi_NA)。ADY2和组ADY4组的子囊菌门、新丽鞭毛菌门、担子菌门的相对丰度显著高于对照组(P < 0.05);ADY2组和ADY4组其他真菌门的相对丰度显著低于对照组(P < 0.05),且ADY2组显著低于ADY4组(P < 0.05)。
|
|
表 3 饲粮添加活性干酵母对肉牛瘤胃菌群门水平相对丰度差异分析 Table 3 Analysis of difference in relative abundance of rumen microflora at phylum level of beef cattle supplemented with active dried yeast |
|
图 5 在门和属水平上真菌的相对丰度 Fig. 5 Relative abundance of fungus at phylum and genus levels |
本试验共鉴定出98个真菌属,表 4列出了相对丰度较高的26个属。由表 4和图 5-B可知,ADY2组和ADY4组未分类真菌属(unclassified_fungi)的相对丰度显著低于对照组(P < 0.05);ADY2组新丽鞭菌属(Neocallimastix)、根囊鞭菌属(Orpinomyces)和念珠菌属(Candida)的相对丰度显著高于对照组和ADY4组(P < 0.05),ADY4组与对照组之间差异不显著(P>0.05)。
|
|
表 4 饲粮添加活性干酵母对肉牛瘤胃菌群属水平相对丰度差异分析 Table 4 Analysis of difference in relative abundance of rumen microflora at genus level of beef cattle supplemented with active dried yeast |
直到1975年,Orpin[12]在绵羊体内发现了带鞭毛的游动孢子,这种孢子具有活动阶段(孢子)和非活动阶段(营养体),从而正式确认其为严格厌氧的真菌。之所以这么晚才发现真菌可能与研究者对瘤胃液的采集方法有关。一般采用纱布过滤收集瘤胃液作为研究对象,而这样主要获得了游动的孢子,而忽略了瘤胃内容物上附着的大量真菌菌体。此后也有研究表明,瘤胃上皮栖息的菌群对于保持瘤胃健康、提高挥发性脂肪酸的吸收也起着至关重要的作用[18-19]。本试验则通过采集瘤胃内容物,进而从内容物上通过离心分离获得真菌菌体进行研究。
酿酒酵母在反刍动物中已经成功应用70多年。诸多报道表明,外源添加酵母能够提高饲粮NDF和ADF表观消化率[2, 20],但也有报道表明,外源添加酵母对饲粮的NDF和ADF等表观消化率没有显著改善[23-24]。导致不一致结果的主要原因可能是因为不同试验的饲粮结构和动物品种不同。尽管都是反刍动物,奶牛和绵羊的生产目的不同,饲粮结构差异较大,因而瘤胃菌群结构组成大相庭径[10]。此前对于外源添加活性干酵母提高饲粮纤维消化率的解释则主要集中在瘤胃纤维降解菌方面,如提高了瘤胃产琥珀酸纤维杆菌(Fibrobacter succinogenes)、黄色瘤胃球菌(Ruminococcus flavefaciens)、白色瘤胃球菌(Ruminococcus albus)等的相对丰度[25],提高了瘤胃内纤维降解酶活性[10, 26],而外源添加活性干酵母对瘤胃真菌组成有何影响则少有涉及[25]。
OTUs数量是衡量一个生境中菌群多样性的指标。ADY2组和ADY4组的OUTs数量较对照组提高了约1倍,说明饲粮添加活性干酵母促进了瘤胃内真菌的生长。ADY2组和ADY4组的Chao1和PD指数均高于对照组,说明饲粮添加活性干酵母提高了瘤胃内真菌菌群的丰富度。ADY2组的PD指数高于ADY4组,说明饲粮添加2 g/(d·头)活性干酵母提高瘤胃真菌丰富度的效果优于添加4 g/(d·头)。ADY2组和ADY4组的Simpson和Shannon指数均高于对照组,表明饲粮添加活性干酵母改善了瘤胃内真菌组成的均匀度。ADY2组Simpson和Shannon指数高于ADY4组,说明饲粮添加2 g/(d·头)活性干酵母提高瘤胃真菌均匀度的效果也优于添加4 g/(d·头)。饲粮添加活性干酵母影响瘤胃内真菌菌群组成存在显著的剂量效应。
Weighted和Unweighted Unifrac PCoA的结果显示,对照组、ADY2组和ADY4组之间的样品能清晰地分开,进一步说明饲粮添加活性干酵母改变了瘤胃生境中真菌群落组成,使ADY2组和ADY4组能清晰地区别于对照组。对照组的几个样本间的距离较远,而ADY2组和ADY4组内的几个点距离较近,进一步说明饲粮添加外源活性干酵母提高了瘤胃真菌菌群均匀度。此外,Weighted Unifrac PCoA的结果显示,ADY2组和ADY4组也能清晰地区分开,再一次说明饲粮添加活性干酵母影响瘤胃内真菌菌群组成存在显著的剂量效应。对照组、ADY2组和ADY4组之间的样品能清晰区分开的结果在UPGMA聚类图中也体现出来,ADY2组和ADY4组先在较近的距离上聚为一个小分支,再与对照组在较远的距离上进行聚类,说明饲粮添加活性干酵母后的瘤胃真菌组成的相似性更高。
前人的研究表明,滩羊瘤胃中的相对丰度最高的是子囊菌门和新丽鞭毛菌门[13],这与本研究中ADY2组的结果相似。但是本研究结果显示,对照组中相对丰度最高(63.6%)的是其他真菌门,而ADY2组的其他真菌门的相对丰度则显著降低至0.041。与此同时,ADY2组子囊菌门、新丽鞭毛菌门和担子菌门的相对丰度显著提高。由此产生的结果是饲粮的NDF和ADF表观消化率提高,说明其他真菌门降解纤维的能力低于子囊菌门、新丽鞭毛菌门和担子菌门。饲粮添加2 g/(d·头)活性干酵母显著提高了将新丽鞭菌属、根囊鞭菌属和念珠菌属的相对丰度,说明新丽鞭菌属、根囊鞭菌属和念珠菌属真菌在降解饲粮纤维成分方面起重要作用[26]。但是,饲粮添加4 g/(d·头)活性干酵母后其新丽鞭菌属、根囊鞭菌属和念珠菌属的相对丰度则与对照组无显著差异,这再次表明外源饲粮添加活性干酵母对瘤胃内真菌菌群组成存在剂量效应,过高的添加剂量并不利于提高纤维消化率。在本试验条件下,饲粮添加2 g/(d·头)活性干酵母对于改善瘤胃真菌组成最为有利。
4 结论① 饲粮添加活性干酵母提高了肉牛瘤胃子囊菌门、新丽鞭毛菌门和担子菌门的相对丰度,提高了新丽鞭菌属、根囊鞭菌属和念珠菌属的相对丰度,降低了未分类真菌属的相对丰度。
② 饲粮添加2 g/(d·头)活性干酵母足以改变肉牛瘤胃真菌菌群组成,从而提高饲粮纤维物质表观消化率。
| [1] |
CAGLE C M, FONSECA M A, CALLAWAY T R, et al. Evaluation of the effects of live yeast on rumen parameters and in situ digestibility of dry matter and neutral detergent fiber in beef cattle fed growing and finishing diets[J]. Applied Animal Science, 2020, 36(1): 36-47. DOI:10.15232/aas.2019-01888 |
| [2] |
SOUSA D O, OLIVEIRA C A, VELASQUEZ A V, et al. Live yeast supplementation improves rumen fibre degradation in cattle grazing tropical pastures throughout the year[J]. Animal Feed Science and Technology, 2018, 236(2): 149-158. |
| [3] |
牛建康, 李妍, 高艳霞, 等. 饲粮中添加活性干酵母对泌乳牛瘤胃发酵特性和营养物质表观消化率的影响[J]. 动物营养学报, 2019, 31(7): 3338-3345. NIU J K, LI Y, GAO Y X, et al. Effects of dietary active dry yeast on rumen fermentation characteristics and nutrient apparent digestibility of lactating cows[J]. Chinese Journal of Animal Nutrition, 2019, 31(7): 3338-3345 (in Chinese). DOI:10.3969/j.issn.1006-267x.2019.07.047 |
| [4] |
郑玮才, 郝小燕, 张春香, 等. 不同非纤维性碳水化合物与中性洗涤纤维比例饲粮中添加酿酒酵母对绵羊体外瘤胃发酵的影响[J]. 动物营养学报, 2019, 31(11): 5354-5366. ZHENG W C, HAO X Y, ZHANG C X, et al. Effects of Saccharomyces cerevisiae supplementation in diets with different non-fiber carbohydrate to neutral detergent fiber ratios on in vitro ruminal fermentation of sheep[J]. Chinese Journal of Animal Nutrition, 2019, 31(11): 5354-5366 (in Chinese). |
| [5] |
ELAREF M Y, HAMDON H A M, NAYEL U A, et al. Influence of dietary supplementation of yeast on milk composition and lactation curve behavior of Sohagi ewes, and the growth performance of their newborn lambs[J]. Small Ruminant Research, 2020, 191: 106176. DOI:10.1016/j.smallrumres.2020.106176 |
| [6] |
ABD EL-GHANI A A. Influence of diet supplementation with yeast culture (Saccharomyces cerevisiae) on performance of Zaraibi goats[J]. Small Ruminant Research, 2004, 52(3): 223-229. DOI:10.1016/j.smallrumres.2003.06.002 |
| [7] |
AMIN A B, MAO S Y. Influence of yeast on rumen fermentation, growth performance and quality of products in ruminants: a review[J]. Animal Nutrition, 2020. DOI:10.1016/j.aninu.2020.10.005 |
| [8] |
SILVA L G T, COOKE R F, SCHUBACH K M, et al. Supplementing a yeast-derived product to enhance productive and health responses of beef steers[J]. Animal, 2018, 12(8): 1576-1583. DOI:10.1017/S1751731117003585 |
| [9] |
PENG Q H, CHENG L, KANG K, et al. Effects of yeast and yeast cell wall polysaccharides supplementation on beef cattle growth performance, rumen microbial populations and lipopolysaccharides production[J]. Journal of Integrative Agriculture, 2020, 19(3): 810-819. DOI:10.1016/S2095-3119(19)62708-5 |
| [10] |
ALZAHAL O, LI F Y, GUAN L L, et al. Factors influencing ruminal bacterial community diversity and composition and microbial fibrolytic enzyme abundance in lactating dairy cows with a focus on the role of active dry yeast[J]. Journal of Dairy Science, 2017, 100(6): 4377-4393. DOI:10.3168/jds.2016-11473 |
| [11] |
MALEKKHAHI M, TAHMASBI A M, NASERIAN A A, et al. Effects of supplementation of active dried yeast and malate during sub-acute ruminal acidosis on rumen fermentation, microbial population, selected blood metabolites, and milk production in dairy cows[J]. Animal Feed Science and Technology, 2016, 213: 29-43. DOI:10.1016/j.anifeedsci.2015.12.018 |
| [12] |
ORPIN C G. Studies on the rumen flagellate Neocallimastix frontalis[J]. Journal of General Microbiology, 1975, 91(2): 249-262. DOI:10.1099/00221287-91-2-249 |
| [13] |
李娜, 张洁, 郭婷婷, 等. 基于内转录间隔区测序分析不同饲养方式对滩羊羔羊瘤胃真菌组成及多样性的影响[J]. 动物营养学报, 2020, 32(2): 784-794. LI N, ZHANG J, GUO T T, et al. Effects of different feeding methods on rumen fungal composition and diversity of Tan sheep based on internal transcriptional spacer sequencing[J]. Chinese Journal of Animal Nutrition, 2020, 32(2): 784-794 (in Chinese). DOI:10.3969/j.issn.1006-267x.2020.02.034 |
| [14] |
AYDIN S, YILDIRIM E, INCE O, et al. Rumen anaerobic fungi create new opportunities for enhanced methane production from microalgae biomass[J]. Algal Research, 2017, 23: 150-160. DOI:10.1016/j.algal.2016.12.016 |
| [15] |
YILDIRIM E, INCE O, AYDIN S, et al. Improvement of biogas potential of anaerobic digesters using rumen fungi[J]. Renewable Energy, 2017, 109: 346-353. DOI:10.1016/j.renene.2017.03.021 |
| [16] |
中华人民共和国农业部. NY/T 815-2004肉牛饲养标准[S]. 北京: 中国农业出版社, 2004. Ministry of Agriculture of the People's Republic of China. NY/T 815-2004 Feeding standard of beef cattle[S]. Beijing: China Agricultural Press, 2004. (in Chinese) |
| [17] |
LOZUPONE C A, STOMBAUGH J I, GORDON J I, et al. Diversity, stability and resilience of the human gut microbiota[J]. Nature, 2012, 489(7415): 220-230. DOI:10.1038/nature11550 |
| [18] |
PETRI R M, WETZELS S U, QUMAR M, et al. Adaptive responses in short-chain fatty acid absorption, gene expression, and bacterial community of the bovine rumen epithelium recovered from a continuous or transient high-grain feeding[J]. Journal of Dairy Science, 2019, 102(6): 5361-5378. DOI:10.3168/jds.2018-15691 |
| [19] |
SADET-BOURGETEAU S, MARTIN C, MORGAVI D P. Bacterial diversity dynamics in rumen epithelium of wethers fed forage and mixed concentrate forage diets[J]. Veterinary Microbiology, 2010, 146(1/2): 98-104. |
| [20] |
MPOFU I D T, NDLOVU L R. The potential of yeast and natural fungi for enhancing fibre digestibility of forages and roughages[J]. Animal Feed Science and Technology, 1994, 48(1/2): 39-47. |
| [21] |
ARCOS-GARCÍA J L, CASTREJÓN F A, MENDOZA G D, et al. Effect of two commercial yeast cultures with Saccharomyces cerevisiae on ruminal fermentation and digestion in sheep fed sugar cane tops[J]. Livestock Production Science, 2000, 63(2): 153-157. DOI:10.1016/S0301-6226(99)00116-5 |
| [22] |
CORONA L, MENDOZA G D, CASTREJÓN F A, et al. Evaluation of two yeast cultures (Saccharomyces cerevisiae) on ruminal fermentation and digestion in sheep fed a corn stover diet[J]. Small Ruminant Research, 1999, 31(3): 209-214. DOI:10.1016/S0921-4488(98)00146-1 |
| [23] |
KHEJORNSART P, WANAPAT M, ROWLINSON P. Diversity of anaerobic fungi and rumen fermentation characteristic in swamp buffalo and beef cattle fed on different diets[J]. Livestock Science, 2011, 139(3): 230-236. DOI:10.1016/j.livsci.2011.01.011 |
| [24] |
TRIPATHI M K, KARIM S A. Effect of yeast cultures supplementation on live weight change, rumen fermentation, ciliate protozoa population, microbial hydrolytic enzymes status and slaughtering performance of growing lamb[J]. Livestock Science, 2011, 135(1): 17-25. DOI:10.1016/j.livsci.2010.06.007 |
| [25] |
BAYAT A R, KAIRENIUS P, STEFAŃSKI T, et al. Effect of camelina oil or live yeasts (Saccharomyces cerevisiae) on ruminal methane production, rumen fermentation, and milk fatty acid composition in lactating cows fed grass silage diets[J]. Journal of Dairy Science, 2015, 98(5): 3166-3181. DOI:10.3168/jds.2014-7976 |
| [26] |
GRUNINGER R J, RIBEIRO G O, CAMERON A, et al. Invited review: application of meta-omics to understand the dynamic nature of the rumen microbiome and how it responds to diet in ruminants[J]. Animal, 2019, 13(9): 1843-1854. DOI:10.1017/S1751731119000752 |




