2. 山西农业大学动物科学学院, 太原 030801
2. College of Animal Science, Shanxi Agricultural University, Taiyuan 030801, China
角蛋白结构稳定、疏水性极强[1-2],动物内源蛋白酶难以将其有效降解[3]。家禽屠宰副产物羽毛中粗蛋白质含量在90%以上,若能高效利用可有助于缓解我国蛋白质饲料资源供需矛盾。传统物理、化学方法处理角蛋白存在诸多不足,产品氨基酸营养品质差,且易产生环境污染[4-5]。目前,已经发掘出多种角蛋白降解菌,包括细菌[6-9]、真菌[10-12]、放线菌等[13-14],这些菌株均可分泌能特异性降解角蛋白的蛋白酶——角蛋白酶。角蛋白酶可水解不溶性角蛋白[15-16],是处理羽毛等角蛋白资源的较优的生物制品[5, 17],可用于处理羽毛,提高其营养价值[18-19],或直接用作饲料添加剂[20]。
角蛋白热稳定性强,可耐受饲料制粒温度,在高温条件下可提高其酶促反应速率。本实验室前期研究发现,地衣芽孢杆菌CP-16角蛋白酶的热稳定性不佳[15]。因此,改善角蛋白酶热稳定性,对提高其酶解羽毛角蛋白效率和在畜禽饲粮中应用具有重要意义。目前有关角蛋白酶结构与功能的研究报道较少,大多设计均参考同源性较高的其他类型蛋白酶[21]。易错PCR无需了解蛋白酶结构功能关系,可快速建立突变体文库,实现角蛋白酶分子改良[22-23]。因此,本研究拟通过易错PCR构建角蛋白酶突变体文库,以期获得活力高、耐温性好的角蛋白酶突变体,提高其在饲料工业中的适用性。
1 材料与方法 1.1 主要试剂、质粒和培养基易错PCR试剂盒(北京天恩泽基因科技有限公司);Prime STAR® Max DNA Polymerase(宝日医生物技术有限公司);地衣芽孢杆菌CP-16为本实验室前期筛选保存,并获得了其角蛋白酶基因全长序列[15];pWB980-K-His为本实验室保藏。
筛选培养基:脱脂奶粉20 g/L,琼脂粉15 g/L,高压灭菌15 min后,加入卡那霉素至终浓度50 μg/mL,制作平板,冷却备用。
发酵培养基:LB液体培养基(50 μg/mL卡那霉素)。
1.2 角蛋白酶突变文库的构建以含角蛋白酶基因的重组质粒pWB980-K- His为模板,使用特异基因的上游、下游引物对角蛋白酶基因进行易错PCR扩增,使用引物对载体pWB980-K-His进行线性化扩增。
|
|
表 1 引物序列 Table 1 Primer sequences |
参考Zhang等[24]的方法,将纯化后的线性化载体和角蛋白酶基因片段进行延长重叠延伸PCR(POE-PCR),以实现质粒多聚化。PCR产物转入枯草芽孢杆菌(Bacillus subtilis)SCK6感受态细胞,稀释涂布于牛奶筛选平板。
1.3 突变体初筛与复筛为同时获得活性高、热稳定性好的突变体,挑选水解圈与菌落直径比值大于野生型的阳性转化子,摇瓶发酵,检测粗酶液角蛋白酶活性和热稳定性,进行复筛。以100目脱脂羽毛粉(0.5%)为底物,测定经70 ℃热处理5 min后的粗酶液残留酶活性(以未经处理的粗酶液酶活性为100%)。角蛋白酶活性测定参考王德山[15]的方法。
1.4 野生型和突变型角蛋白酶的纯化以复筛后的正向突变菌株为出发菌,制备粗酶液,经超滤浓缩、镍柱纯化获得目的蛋白,十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(SDS-PAGE)验证纯化后酶蛋白纯度及分子质量。
1.5 野生型和突变型角蛋白酶的酶学性质研究真实底物羽毛粉为不溶底物,为保证数据的精准性,酶学性质测定均使用可溶性蛋白-酪蛋白为底物[15],所有处理均设置3个重复。
分别于不同pH(7.0、8.0、8.5、9.0、9.5、10.0、11.0、12.0、13.0)缓冲液体系,55 ℃,测定野生型和突变型角蛋白酶活性,确定适宜pH。以最高酶活性为100%,绘制酶活性随pH变化曲线。所用缓冲体系:Tris-HCL缓冲体系(200 mmol/L,pH 7.0~9.0),硼砂-氢氧化钠缓冲体系(100 mmol/L,pH 9.5~12.0),氯化钾-氢氧化钠缓冲体系(pH 13.0)。
在适宜pH和不同反应温度(40、45、50、55、60、65、70、75 ℃)条件下,测定野生型和突变型角蛋白酶活性,确定适宜反应温度,最高的酶活性定义为100%,绘制相对酶活性随反应温度变化曲线。
纯酶液分别在55、65、75、85 ℃下热处理5 min,适宜条件下测定酶活性,以未经处理的酶活性为100%,计算不同温度处理后的酶活性残留率。
配制不同浓度(0.5、0.8、1.0、1.3、1.5、2.0、2.5、5.0、8.0、10.0、12.0 mg/mL)酪蛋白底物溶液,适宜条件下反应10 min,测定酶活性。利用双倒数法作图,计算动力学参数。
1.6 野生型和突变型角蛋白酶晶体结构模型构建与分析运用SWISS-MODEL[24]和Discovery Studio Visualizer 4.0对野生型和突变型角蛋白酶晶体进行同源建模及三维结构分析。
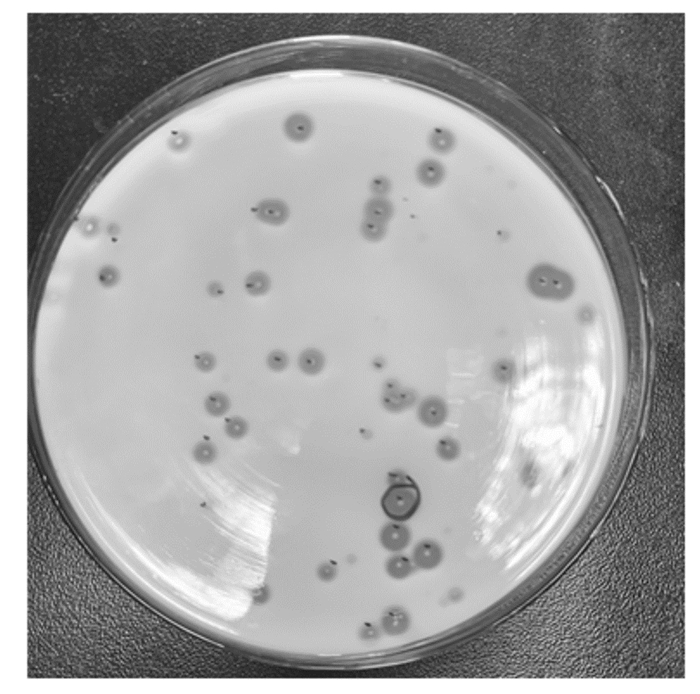
2 结果 2.1 突变型角蛋白酶初筛与复筛经易错PCR成功构建出角蛋白酶突变体文库(图 1),在牛奶平板挑选水解圈与菌落直径比值不小于野生型的转化子共564株进行摇瓶发酵,测定角蛋白酶的热处理后残留酶活性,获得3株正向突变体。由表 2可知,角蛋白酶突变体A307V/S346T、R70G、N245K热处理后残留酶活性分别为72.12%、86.12%、74.82%,均高于野生型角蛋白酶(50.32%)。
|
图 1 阳性转化子在牛奶平板上产生的水解圈 Fig. 1 Hydrolysis circle formed by positive transformants on milk plate |
|
|
表 2 野生型与突变型角蛋白酶发酵液酶活性及热处理后残留酶活性 Table 2 Enzyme activity of fermentation broth and residual enzyme activity after heat treatment of wild-type and mutant-type keratinase |
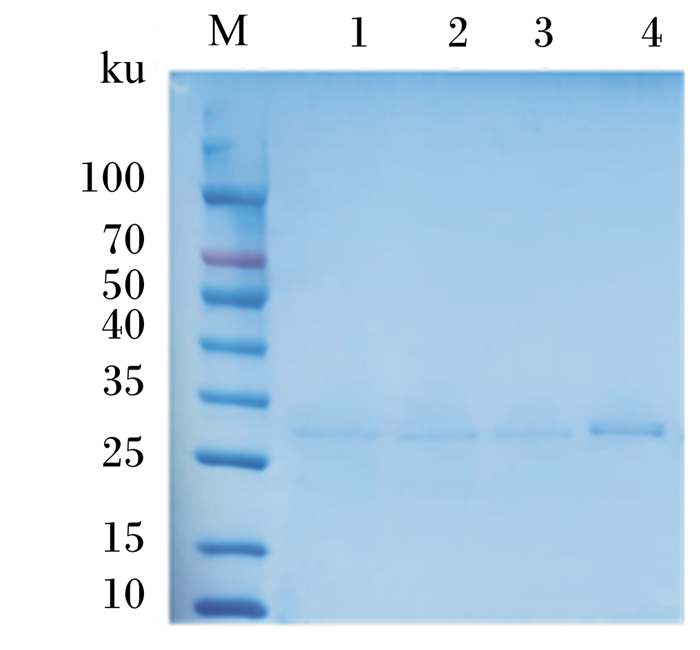
野生型和突变型角蛋白酶纯化后酶蛋白的SDS-PAGE分析结果见图 2,目的蛋白条带分子质量与预期一致,单一、无杂带,可用于下一步分析。
|
M:中分子质量蛋白marker;1:野生型wild-type;2:突变体A307V/S346T mutant A307V/S346T;3:突变体R70G mutant R70G;4:突变体N245K mutant N245K。 图 2 SDS-PAGE分析 Fig. 2 SDS-PAGE analysis |
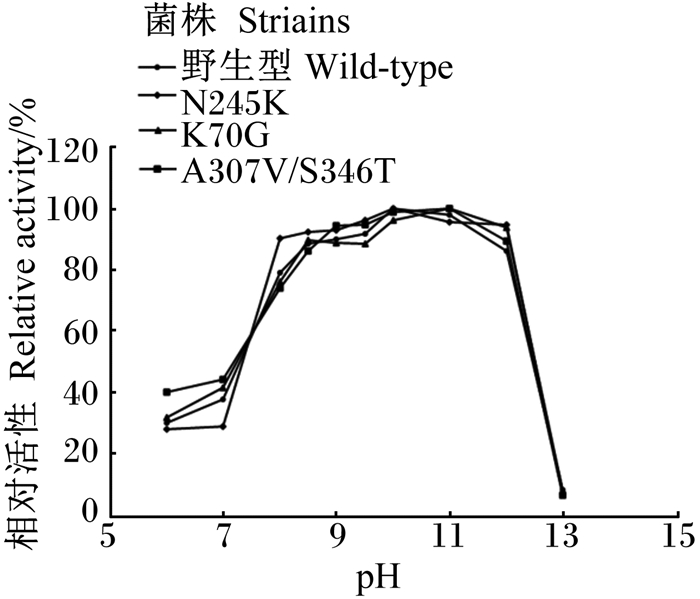
经不同pH条件下蛋白酶活性测定分析,野生型角蛋白酶和角蛋白酶突变体A307V/S346T适宜pH为10,角蛋白酶突变体R70G、N245K适宜pH为11(图 3)。这说明突变位点影响了其适宜pH。
|
图 3 野生型与突变型角蛋白酶适宜pH Fig. 3 Optimum pH of wild-type and mutant-type keratinase |
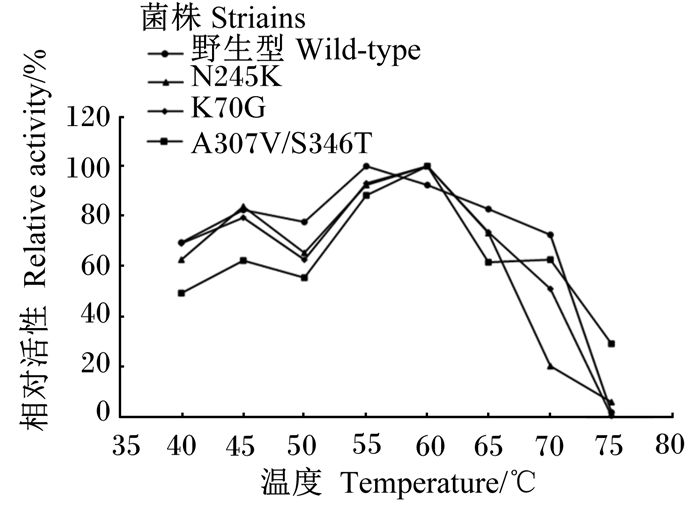
不同温度条件下,角蛋白酶突变体A307V/S346T、R70G、N245K适宜反应温度相较野生型发生偏移(图 4);同时,角蛋白酶突变体A307V/S346T、R70G、N245K的热稳定性也相应提升(表 3)。这说明突变位点均影响了其适宜反应温度和热稳定性。
|
图 4 野生型与突变型角蛋白酶适宜反应温度 Fig. 4 Optimum reaction temperature of wild-type and mutant-type keratinase |
|
|
表 3 野生型与突变型角蛋白酶5 min热处理后残留酶活性 Table 3 Residual enzyme activity after heat treatment 5 min of wild-type and mutant-type keratinase |
采用双倒数法作图计算角蛋白酶动力学参数,与野生型角蛋白酶相比,角蛋白酶突变体A307V/S346T、R70G、N245K对底物的亲和力与催化效率均有所提升(表 4)。
|
|
表 4 野生型与突变型角蛋白酶动力学参数 Table 4 Kinetic parameters of wild-type and mutant-type keratinase |
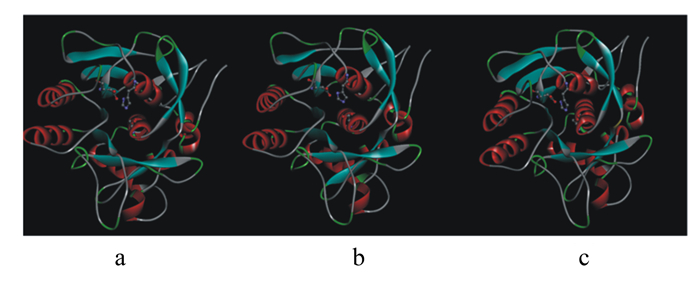
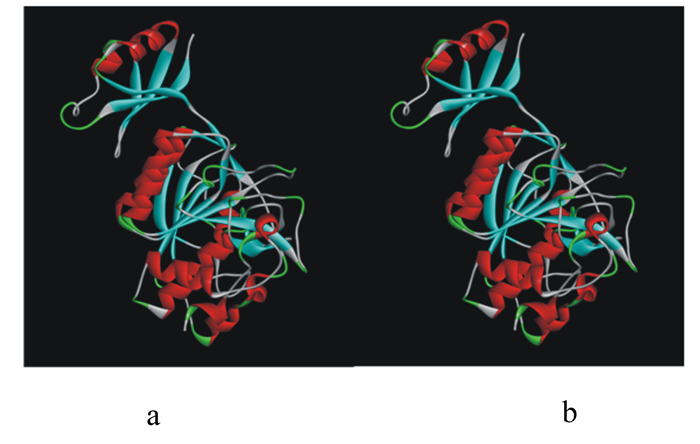
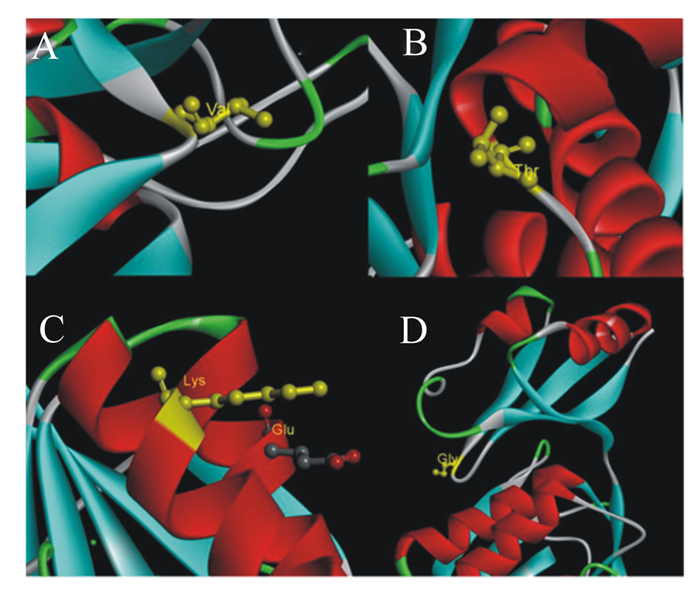
角蛋白酶突变体A307V/S346T、N245K的突变位点位于成熟肽区域,直接以成熟肽氨基酸序列建模,以地衣芽孢杆菌的枯草菌溶素(PDB蛋白质数据库ID: 3unxA)为模板得到野生型角蛋白酶和角蛋白酶突变体A307V/S346T、N245K晶体的三维结构模型图(图 5)。角蛋白酶突变体R70G的突变位点位于前导肽区域,用前导肽与成熟肽氨基酸序列建模,以枯草蛋白酶E(PDB蛋白质数据库ID: 3whi.1.A)为模板进行同源建模得到野生型角蛋白酶和角蛋白酶突变型R70G前导肽的三维结构模型图(图 6)。角蛋白酶突变体A307V/S346T、N245K、R70G突变位点分析如图 7所示。据结构模型显示,3种突变型角蛋白酶突变前后的蛋白质结构均无明显变化。
|
图 5 野生型角蛋白酶(a)和角蛋白酶突变体A307V/S346T(b)、N245K(c)晶体的三维结构模拟示意图 Fig. 5 3D structure simulation diagram of crystal of wild-type keratinase (a) and keratinase mutant A307V/S346T (b), N245K (c) |
|
图 6 野生型角蛋白酶(a)和角蛋白酶突变体R70G(b)前导肽的三级结构模拟示意图 Fig. 6 3D structure simulation diagram of propeptide of wild type keratinase (a) and keratinase mutant R70G (b) |
|
Val:缬氨酸valine;Thr:苏氨酸threonine;Lys:赖氨酸lysine;Glu:谷氨酸glutamate;Gly:甘氨酸glycine。 图 7 角蛋白酶突变体A307V/S346T(a, b)、N245K(c)、R70G(d)突变位点分析 Fig. 7 Analysis of mutant sites of keratinase mutants A307V/S346T (a, b), N245K (c) and R70G (d) |
本试验利用易错PCR方法,对角蛋白酶进行定向进化,获得了3株耐温性较好的角蛋白酶突变体A307V/S346T,R70G、N245K,反映出易错PCR在酶分子改造方面具有较好的便捷性。初筛所选转化子水解圈均不小于野生型,但角蛋白酶突变体A307V/S346T、R70G的粗酶液角蛋白酶活性为野生型角蛋白酶的89%左右,仅角蛋白酶突变体N245K的发酵液酶活性相对野生型角蛋白酶提升了19.43%,这说明水解圈与发酵酶活性水平间存在不一致性,复筛十分必要。
野生型角蛋白酶与3个突变型角蛋白酶在pH 8~12的缓冲体系中酶活性变化趋势平稳。角蛋白酶突变体R70G、N245K的适宜pH后移至11,在pH 12表现出比野生型更高的相对活性,在碱性pH宽泛性增加,利于在碱性条件下快速处理羽毛角蛋白,在洗涤与纺织业也有较好利用前景。但在畜禽小肠pH(6.0~7.0)条件下,野生型和突变型角蛋白酶活性均大幅降低,影响其作用发挥,后续研究需进一步通过定向进化、C端截短、结构域重组等分子改造手段提高其pH稳定性[21-22]。
根据纯化酶蛋白热处理后酶活性残留情况可见,部分突变型角蛋白酶出现75 ℃组活性高于65 ℃组的现象,这可能与在特定条件下出现蛋白酶自溶现象有关[25-26]。与此同时还发现,纯酶热稳定性明显低于粗酶液,可能是粗酶液中细菌代谢产生的蛋白与角蛋白酶结合暂时形成酶-底物复合物[27],构象相对稳定。以上现象产生的原因与机制尚需进一步深入研究。
本研究共获得4个角蛋白酶突变位点。角蛋白酶突变体A307V/S346T的307位的丙氨酸(Ala)位于2个β折叠之间的loop区域,Ala的构象不稳定,被替换为缬氨酸(Val)后,构象的稳定性和氨基酸的疏水性均有所增强。疏水作用是蛋白质折叠主要驱动力[28-29],有利于蛋白质三级结构稳定。Val疏水性增强有助于反应区疏水环境的形成,降低结合状态的自由能,有利于底物与酶的结合,促进催化反应进行,这可能是角蛋白酶突变体A307V/S346T在适宜反应温度与热稳定性提升的同时,还保留了较高活性的主要原因。而346位氨基酸丝氨酸(Ser)与苏氨酸(Thr)的结构功能均较为接近,可能不是影响酶学性质改变的主要突变因素。角蛋白酶突变体N245K的245位氨基酸天冬酰胺(Asn)位于α螺旋,Asn在高温下易发生脱酰胺作用,不利于蛋白质的热稳定性,嗜热蛋白中一般含有较少的Asn和谷氨酰胺[30-32]。同时,已有研究揭示增加蛋白质表面负电荷氨基酸可提高酶稳定性[33],Asn被替换为赖氨酸(Lys)后可能更有利于α螺旋的稳定。Lys在蛋白表面引入负电荷可能通过静电作用力,形成离子键,稳定了蛋白质结构。角蛋白酶突变体R70G的70位氨基酸位于酶前导肽2个β折叠间的loop上,由精氨酸(Arg)变为结构最为简单的甘氨酸(Gly),侧链仅有1个氢原子,空间位阻小。空间位阻的改变可能对前导肽结构起优化作用,并进一步影响成熟肽空间结构,从而提升了突变体的热稳定性。已有研究探明前导肽与脂肪酶的热稳定性相关[34],但在角蛋白酶的研究中多将前导肽与酶活性进行关联分析,前导肽与蛋白酶稳定性间的关系尚无相关报道,该研究结果或许可为今后耐热蛋白酶理性设计提供新的思路。
4 结论本试验通过易错PCR成功对地衣芽胞杆菌CP-16来源的角蛋白酶进行了定向进化,筛选到热稳定性提升的角蛋白酶突变体,拓展了角蛋白酶的适用性,并分析了其热稳性提高的分子基础,为耐热蛋白酶理性设计提供新思路。
| [1] |
SUZUKI Y, TSUJIMOTO Y, MATSUI H, et al. Decomposition of extremely hard-to-degrade animal proteins by thermophilic bacteria[J]. Journal of Bioscience and Bioengineering, 2006, 102(2): 73-81. DOI:10.1263/jbb.102.73 |
| [2] |
KREPLAK L, DOUCET J, DUMAS P, et al. New aspects of the alpha-helix to beta-sheet transition in stretched hard alpha-keratin fibers[J]. Biophysical Journal, 2004, 87(1): 640-647. DOI:10.1529/biophysj.103.036749 |
| [3] |
JAGADEESAN Y, MEENAKSHISUNDARAM S, SARAVANAN V, et al. Sustainable production, biochemical and molecular characterization of thermo-and-solvent stable alkaline serine keratinase from novel Bacillus pumilus AR57 for promising poultry solid waste management[J]. International Journal of Biological Macromolecules, 2020, 163: 135-146. DOI:10.1016/j.ijbiomac.2020.06.219 |
| [4] |
SHARMA S, GUPTA A. Sustainable management of keratin waste biomass: applications and future perspectives[J]. Brazilian Archives of Biology and Technology, 2016, 59: 1-14. |
| [5] |
BRANDELLI A, SALA L, KALIL S J. Microbial enzymes for bioconversion of poultry waste into added-value products[J]. Food Research International, 2015, 73: 3-12. DOI:10.1016/j.foodres.2015.01.015 |
| [6] |
CHENG S W, HU H M, SHEN S W, et al. Production and characterization of keratinase of a feather-degrading Bacillus licheniformis PWD-1[J]. Bioscience, Biotechnology, and Biochemistry, 1995, 59(12): 2239-2243. DOI:10.1271/bbb.59.2239 |
| [7] |
EL-REFAI H A, ABDELNABY M A, GABALLA A, et al. Improvement of the newly isolated Bacillus pumilus FH9 keratinolytic activity[J]. Process Biochemistry, 2005, 40(7): 2325-2332. DOI:10.1016/j.procbio.2004.09.006 |
| [8] |
MACEDO A J, BEYS DA SILVA W O, TERMIGNONI C. Properties of a non collagen-degrading Bacillus subtilis keratinase[J]. Canadian Journal of Microbiology, 2008, 54(3): 180-188. DOI:10.1139/W07-124 |
| [9] |
SUH H J, LEE H K. Characterization of a keratinolytic serine protease from Bacillus subtilis KS-1[J]. Journal of Protein Chemistry, 2001, 20(2): 165-169. DOI:10.1023/A:1011075707553 |
| [10] |
ASAHI M, LINDQUIST R, FUKUYAMA K, et al. Purification and characterization of major extracellular proteinases from Trichophyton rubrum[J]. The Biochemical Journal, 1985, 232(1): 139-144. DOI:10.1042/bj2320139 |
| [11] |
FARAG A M, HASSAN M A. Purification, characterization and immobilization of a keratinase from Aspergillus oryzae[J]. Enzyme and Microbial Technology, 2004, 34(2): 85-93. DOI:10.1016/j.enzmictec.2003.09.002 |
| [12] |
ANBU P, GOPINATH S C B, HILDA A, et al. Purification of keratinase from poultry farm isolate-Scopulariopsis brevicaulis and statistical optimization of enzyme activity[J]. Enzyme and Microbial Technology, 2005, 36(5/6): 639-647. |
| [13] |
GUSHTEROVA A, VASILEVA-TONKOVA E, DIMOVA E, et al. Keratinase production by newly isolated Antarctic actinomycete strains[J]. World Journal of Microbiology and Biotechnology, 2005, 21(6/7): 831-834. |
| [14] |
BRANDELLI A, DAROIT D J, RIFFEL A. Biochemical features of microbial keratinases and their production and applications[J]. Applied Microbiology and Biotechnology, 2010, 85(6): 1735-1750. DOI:10.1007/s00253-009-2398-5 |
| [15] |
王德山. 地衣芽孢杆菌CP-16降解羽毛角蛋白相关蛋白酶的分离、表达和相互关系[D]. 硕士学位论文. 北京: 中国农业科学院, 2014. WANG D S. Isolation, expression and correlation of feather keratin degrading protease from Bacillus licheniformis CP-16[D]. Master's Thesis. Beijing: Chinese Academy of Agricultural Sciences, 2014. (in Chinese) |
| [16] |
廖朝勇. 地衣芽孢杆菌CP-16降解羽毛关键酶高效表达研究[D]. 硕士学位论文. 北京: 中国农业科学院, 2019. LIAO C Y. Study on the high-efficient expression of key enzymes in degradation of feather by Bacillus licheniformis CP-16[D]. Master's Thesis. Beijing: Chinese Academy of Agricultural Sciences, 2019. (in Chinese) |
| [17] |
SRIVASTAVA B, KHATRI M, SINGH G, et al. Microbial keratinases: an overview of biochemical characterization and its eco-friendly approach for industrial applications[J]. Journal of Cleaner Production, 2020, 252: 119847. DOI:10.1016/j.jclepro.2019.119847 |
| [18] |
NNOLIM N, UDENIGWE C, OKOH A, et al. Microbial keratinase: next generation green catalyst and prospective applications[J]. Frontiers in Microbiology, 2020. DOI:10.3389/fmicb.2020.580164 |
| [19] |
EAKSUREE W, PRACHAYAKITTI A, UPATHANPREECHA T, et al. In vitro and in vivo evaluation of protein quality of enzymatic treated feather meals[J]. SpringerPlus, 2016, 5(1): 971. DOI:10.1186/s40064-016-2626-2 |
| [20] |
谭权, 赵剑, 陶青燕, 等. 外源性蛋白酶在饲料工业中的应用[J]. 中国畜牧杂志, 2020, 56(5): 32-35. TAN Q, ZHAO J, TAO Q Y, et al. Application of exogenous protease in feed industry[J]. Chinese Journal of Animal Science, 2020, 56(5): 32-35 (in Chinese). |
| [21] |
方真. Stenotrophomonas maltophilia角蛋白酶的分子改造[D]. 博士学位论文. 无锡: 江南大学, 2017. FANG Z. Molecular modification of Stenotrophomonas maltophilia keratinase[D]. Ph. D. Thesis. Wuxi: Jiangnan University, 2017. (in Chinese) |
| [22] |
GONG J S, WANG Y, ZHANG D D, et al. Biochemical characterization of an extreme alkaline and surfactant-stable keratinase derived from a newly isolated actinomycete Streptomyces aureofaciens K13[J]. RSC Advances, 2015, 5(31): 24691-24699. DOI:10.1039/C4RA16423G |
| [23] |
王海燕. 脱毛蛋白酶产生菌的选育及其碱性蛋白酶基因的克隆与表达[D]. 博士学位论文. 成都: 四川大学, 2006. WANG H Y. Breeding of unhairing protease producing strain and cloning and expression of alkaline protease gene[D]. Ph. D. Thesis. Chengdu: Sichuan University, 2006. (in Chinese) |
| [24] |
ZHANG X Z, ZHANG Y H P. Simple, fast and high-efficiency transformation system for directed evolution of cellulase in Bacillus subtilis[J]. Microbial Biotechnology, 2011, 4(1): 98-105. DOI:10.1111/j.1751-7915.2010.00230.x |
| [25] |
刘柏宏. Bacillus licheniformis角蛋白酶的高效表达、热稳定性及底物特异性改造[D]. 博士学位论文. 无锡: 江南大学, 2015. LIU B H. High expression, thermal stability and substrate specific modification of Bacillus licheniformis keratinase[D]. Ph. D. Thesis. Wuxi: Jiangnan University, 2015. (in Chinese) |
| [26] |
VRIEND G, EIJSINK V. Prediction and analysis of structure, stability and unfolding of thermolysin-like proteases[J]. Journal of Computer-Aided Molecular Design, 1993, 7(4): 367-396. DOI:10.1007/BF02337558 |
| [27] |
NOPPEN B, FONTEYN L, AERTS F, et al. Autolytic degradation of ocriplasmin: a complex mechanism unraveled by mutational analysis[J]. Protein Engineering, Design & Selection, 2014, 27(7): 215-223. |
| [28] |
王镜岩, 朱圣庚, 徐长法. 生物化学: 上册[M]. 3版. 北京: 高等教育出版社, 2002. WANG J Y, ZHU S G, XU C F. Biochemistry: volume Ⅰ[M]. 3rd ed. Beijing: Higher Education Press, 2002 (in Chinese). |
| [29] |
CHAN M K, MUKUND S, KLETZIN A, et al. Structure of a hyperthermophilic tungstopterin enzyme, aldehyde ferredoxin oxidoreductase[J]. Science, 1995, 267(5203): 1463-1469. DOI:10.1126/science.7878465 |
| [30] |
王珏, 胡丽丽, 张育敏, 等. 基于前导肽和成熟区共进化提高米黑霉脂肪酶热稳定性研究[J]. 生物学杂志, 2021, 38(1): 41-45. WANG J, HU L L, ZHANG Y M, et al. Enhancing the thermostability of Rhizomucor miehei lipase through coevolution of the propeptide and the mature region[J]. Journal of Biology, 2021, 38(1): 41-45 (in Chinese). DOI:10.3969/j.issn.2095-1736.2021.01.041 |
| [31] |
TINA K G, BHADRA R, SRINIVASAN N. PIC: protein interactions calculator[J]. Nucleic Acids Research, 2007, 35(Web Server Issue): W473-W476. |
| [32] |
GRAZIANO G, MERLINO A, et al. Molecular bases of protein halotolerance[J]. Biochimica et Biophysica Acta, 2014, 1844(4): 850-858. DOI:10.1016/j.bbapap.2014.02.018 |
| [33] |
CHAKRAVARTY S, VARADARAJAN R. Elucidation of determinants of protein stability through genome sequence analysis[J]. FEBS Letters, 2000, 470(1): 65-69. DOI:10.1016/S0014-5793(00)01267-9 |
| [34] |
LIU Y, XIE W P, YU H W. Enhanced activity of Rhizomucor miehei lipase by deglycosylation of its propeptide in Pichia pastoris[J]. Current Microbiology, 2014, 68(2): 186-191. DOI:10.1007/s00284-013-0460-0 |




