我国是肉鸭生产和消费大国,肉鸭年出栏量遥居世界首位[1]。在养殖生产中,通过改善肠道黏膜结构和微生物区系增强肉鸭消化功能和免疫机能已经成为保障肉鸭健康的有效途径[2]。肉鸭肠道中定植的菌群与肠道复杂的稳态环境有着密切联系。肠道菌群影响着宿主营养物质的消化和吸收、肠道黏膜的发育、氨基酸和维生素的合成、肠道黏膜免疫系统的反应和病原微生物的增殖[3-4]。同时短链脂肪酸(short chain fatty acids,SCFAs)作为肠道菌群的代谢产物能够为宿主肠道上皮细胞提供能量,并抑制病原菌致病因子的表达[5]。因而肠道菌群组成和结构对肉鸭的生长发育具有重要影响。目前,基于高通量测序技术的发展,不同样本环境中的微生物群落图谱得到进一步的补充完善,肠道菌群作为家禽肠道疾病预防和诊断的生物标记物逐渐得到关注[6-7]。肉鸭肠道微生物方面的研究主要集中在饲粮、日龄和添加剂等因素与肠道微生物组成和结构变化关系的研究[1, 8-9],这些研究表明肠道微生物在肉鸭营养物质消化及肠道健康中起到非常重要的作用。肉鸭盲肠作为微生物活动的主要场所,主要以微生物消化为主。回肠作为肉鸭营养物质化学消化的部位,其营养物质的消化吸收也与微生物群落有着密不可分的关联[10]。由此可见,肉鸭肠道功能的发挥与回肠和盲肠菌群的空间分布密切相关。目前,肉鸭肠道菌群的研究主要集中在盲肠,而回肠和盲肠菌群的群落组成和空间分布的研究鲜有报道。因此,本试验旨在研究35日龄北京鸭回肠和盲肠菌群空间分布的变化,揭示回肠和盲肠菌群空间分布差异,为维持肠道正常功能和保障肠道健康提供基础理论依据,为进一步开展肠道健康与微生物关系研究和新型饲用微生物制剂研发提供技术支撑。
1 材料与方法 1.1 试验设计试验选用36只健康且体重基本一致的15日龄雄性北京鸭,随机分为6个重复,每个重复6只鸭。试验采用玉米-豆粕型基础饲粮,饲粮营养水平满足我国《肉鸭饲养标准》(NY/T 2122—2012)要求,基础饲粮组成及营养水平见表 1。试验期为21 d。
|
|
表 1 基础饲粮组成及营养水平(风干基础) Table 1 Composition and nutrient levels of the basal diet (air-dry basis) |
动物养殖试验在中国农业科学院北京畜牧兽医研究所昌平试验基地进行。试验鸭网上平养,自由采食与饮水,24 h光照。其他按试验基地常规管理进行。
1.3 样品采集35日龄时,从每个重复中随机选取2只北京鸭,然后进行屠宰,解剖并分离回肠和盲肠。将回肠和盲肠的内容物快速地挤压到1.5 mL灭菌离心管中,液氮速冻,-80 ℃保存。
1.4 回肠与盲肠微生物测定使用十二烷基苯磺酸钠(SDS)法提取基因组DNA[11],采用NanoDrop-2000检测样本DNA浓度和纯度,并用1%琼脂糖凝胶电泳检测DNA的完整性。将DNA浓度稀释至10 ng/μL之后,用带有Barcode的特异性引物对样本DNA的V3~V4区进行扩增。用2%的琼脂糖凝胶电泳检测扩增产物。使用TruSe®DNA PCR-Free Sample Preparation Kit进行文库的构建,构建完成的文库经Qubit和荧光定量PCR检测合格后使用MiSeq进行上机测序。
1.5 16S rRNA数据处理根据Barcode将测序得到的reads拆分出各样本数据,通过Trimmomatic软件去除Barcode和引物序列后利用Flash软件将各个样本的reads进行拼接,从而得到原始序列(raw reads)。利用Qiime 2软件对raw reads进行质量控制,得到质量值小于20的干净序列(clean reads),然后使用UCHIME Algorithm去除clean reads中的嵌合体序列获得有效序列(effective reads)。用Usearch(v7.0)按97%的相似性对样本中的effective reads进行操作分类单元(operational taxonomic units,OTUs)聚类,选取97%相似水平的OTUs代表序列。通过Qiime 2将OTUs代表序列与Silva的SSU rRNA数据库进行物种注释分析,从而获得样本在各个分类学上的物种组成。使用Qiime 2对样本进行alpha多样性和beta多样性分析,其中alpha多样性指数为Ace、Chao1、Simpson和Shannon指数,beta多样性中的主坐标分析(principal co-ordinates analysis,PCoA)基于Unweighted UniFrac距离算法。使用线性判别分析效应大小(linear discriminant analysis effect size,LEfSe)软件分析回肠与盲肠中相对丰度显著差异的微生物,回肠与盲肠中的生物标记物筛选的线性判别分析(linear discriminant analysis,LDA)阈值大于4。
1.6 数据分析采用SAS 9.4软件对alpha多样性指数及相关数据进行独立样本t检验,结果用平均值和标准误(SE)表示,以P<0.05为差异显著性水平,P<0.01为差异极显著水平。
2 结果与分析 2.1 15~35日龄肉鸭生长性能35日龄肉鸭的平均体重为(2 396.8±10.9) g,15~35日龄肉鸭料重比为2.21±0.02,表明肉鸭在养殖过程中生长发育正常。
2.2 35日龄肉鸭回肠和盲肠微生物菌群OTUs聚类和alpha多样性比较分析本试验应用Illumina MiSeq测序平台进行双末端(paired-end)测序,通过对reads进行拼接质控后测得每样本平均clean reads数目为53 309个,每样本平均effective reads数目为43 944个,reads平均长度为418 bp。基于97%相似性对序列进行OTUs聚类,共获得1 268个OTUs。由图 1可知,样品稀释曲线随着测序深度的增加逐渐趋于平缓,表明测序数据量合理。
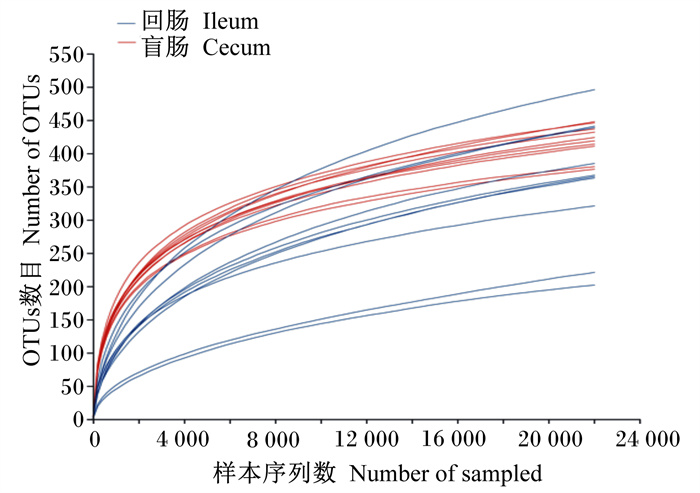
|
图 1 样品稀释曲线分析图 Fig. 1 Rarefaction curve analysis figure of samples |
由图 2可知,回肠和盲肠中共有的OTUs数目为509个,占到了总OTUs数目的40.14%,且盲肠中的OTUs数目多于回肠中的OTUs数目。与回肠相比,肉鸭盲肠中Simpson指数显著降低(P<0.05),Shannon指数极显著增加(P<0.01),表明肉鸭盲肠微生物的多样性高于回肠;而肉鸭盲肠中Ace和Chao1指数差异不显著(P>0.05),表明肉鸭回肠和盲肠微生物的丰富度大致相同。
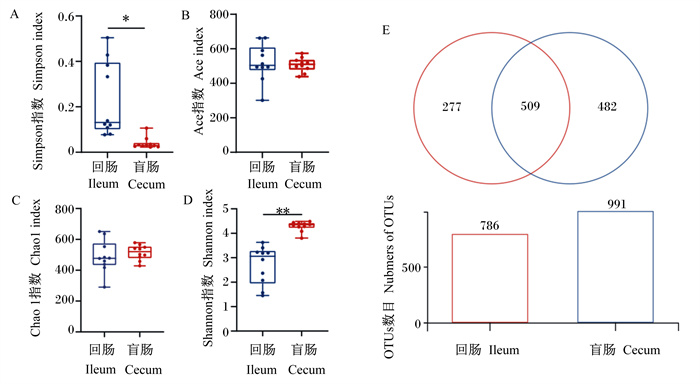
|
数据箱形标记*表示差异显著(P<0.05),标记* *表示差异极显著(P<0.01)。 Value boxes with * mean significant difference (P < 0.05), and with * * mean extremely significant difference (P < 0.01). 图 2 肉鸭回肠和盲肠微生物菌群alpha多样性分析 Fig. 2 Alpha diversity analysis of ileum and cecal microbiota of meat ducks |
本试验共鉴定出23个门(phylum),53个纲(class),128个目(order),211个科(family),455个属(genus)。由图 3可知,在回肠中厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidita)、放线菌门(Actinobacteria)、变形菌门(Proteobacteria)为优势菌门(相对丰度>1%),在盲肠中厚壁菌门、拟杆菌门、放线菌门、变形菌门和脱铁杆菌门(Desulfobacterota)为优势菌门(相对丰度>1%)。厚壁菌门是回肠和盲肠微生物菌群中相对丰度(>60%)最高的菌门;拟杆菌门是盲肠中主要的优势菌门(相对丰度>20%),而其在回肠中的相对丰度低于2%;变形菌门和放线菌门是回肠中主要的优势菌门(相对丰度>20%),而在盲肠中二者的相对丰度低于8%;脱铁杆菌门是盲肠中特有的优势菌门,相对丰度高于2%。
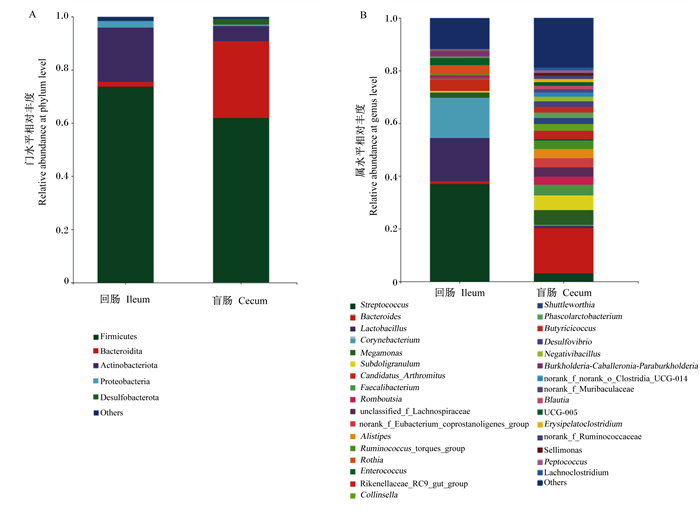
|
Firmicutes:厚壁菌门;Bacteroidita:拟杆菌门;Actinobacteriota:放线菌门;Proteobacteria:变形菌门;Desulfobacterota:脱铁杆菌门;Streptococcus:链球菌属;Bacteroides:拟杆菌属;Lactobacillus:乳酸菌属;Corynebacterium:棒杆菌属;Megamonas:巨单细胞菌属;Subdoligranulum:罕见小球菌属;Candidatus_Arthromitus:分节丝状菌属;Faecalibacterium:粪杆菌属;Romboutsia:罗姆布茨菌属;unclassified_f_Lachnospiraceae:毛螺菌科未定义菌属;Rothia:罗思氏菌属;Enterococcus:肠球菌属;Ruminococcus_torques_group:瘤胃球菌属扭链群;Rikenellaceae_RC9_gut_group:理研菌科RC9肠道群;Collinsella:柯林斯氏菌属;Shuttleworthia:沙特尔沃思菌属;Phascolarctobacterium:考拉杆菌属;Butyricicoccus:丁酸球菌属;Desulfovibrio:脱硫弧菌属;Negativibacillus:负芽孢杆菌属;Burkholderia-Caballeronia-Paraburkholderia:伯克霍尔德氏菌属;norank_f_norank_o_Clostridia_UCG-014:梭菌目UCG-014;Erysipelatoclostridium:毛霉菌属;norank_f_Ruminococcaceae:瘤胃菌科未定义属;Peptococcus:肽球菌属;Blautia:布劳特氏菌属;others:其他。 图 3 肉鸭回肠和盲肠微生物群落组成 Fig. 3 Ileum and cecal microbial community composition of meat ducks |
在回肠和盲肠鉴定出的455个属中,相对丰度高于1%的优势菌属共有32个。回肠和盲肠中主要的菌属为拟杆菌属(Bacteroides)、链球菌属(Streptococcus)、乳杆菌属(Lactobacillus)和棒杆菌属(Corynebacterium)。链球菌属、乳杆菌属和棒杆菌属是回肠中的主要菌属(相对丰度>50%),而其在盲肠中的相对丰度低于5%;拟杆菌属是盲肠中的优势菌属,相对丰度达17%,而在回肠中的相对丰度仅为1%。
2.4 35日龄肉鸭回肠和盲肠微生物群落结构图 4表明了回肠和盲肠微生物群落结构的相似性与差异性。PCoA分析展示了回肠和盲肠的微生物群落形成了2个独立的簇,表明不同肠段影响着微生物群落结构的形成。同时在科水平上展示了定植在回肠和盲肠微生物菌群相对丰度的差异性比较分析。棒杆菌科(Corynebacteriaceae)、乳杆菌科(Lactobacillaceae)和链球菌科(Streptococcaceae)是回肠微生物群落中主要的菌科,并形成了一个独立的簇;毛螺菌科(Lachnospiraceae)、拟杆菌科(Bacteroidaceae)和瘤胃菌科(Ruminococcaceae)是盲肠微生物群落中主要的菌科,并聚类形成了一个独立的簇,然而气球菌科(Aerococcaceae)和微球菌科(Micrococcaceae)在盲肠中的相对丰度低于回肠。
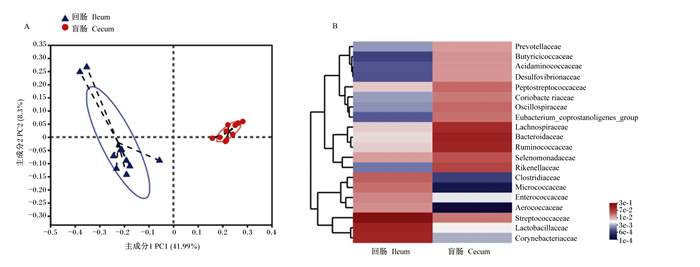
|
Prevotellaceae:普雷沃氏菌科;Butyricicoccaceae:丁酸球菌科;Acidaminococcaceae:氨基酸球菌科;Desulfovibrionaceae:脱硫弧菌科;Peptostreptococcaceae:消化链球菌科;Coriobacte riaceae:肠杆菌科;Oscillospiraceae:颤杆菌科;Eubacterium_coprostanoligenes_group:产粪甾醇真杆菌科;Lachnospiraceae:毛螺菌科;Bacteroidaceae:拟杆菌科;Ruminococcaceae:瘤胃菌科;Selenomonadaceae:月形单细胞菌科;Rikenellaceae:理研菌科;Clostridiaceae:梭菌科;Micrococcaceae:微球菌科;Enterococcaceae:肠球菌科;Aerococcaceae:气球菌科;Streptococcaceae:链球菌科;Lactobacillaceae:乳杆菌科;Corynebacteriaceae:棒杆菌科。 图 4 肉鸭回肠和盲肠微生物群落结构 Fig. 4 Ileum and cecal microbial community structure of meat ducks |
LEfSe分析筛选出了回肠和盲肠的差异微生物。由图 5可知,盲肠中共获得32个差异微生物,其中在科水平上具有显著差异的微生物菌群为拟杆菌科、瘤胃菌科、毛螺菌科、理研菌科(Rikenellaceae)、颤杆菌科(Oscillospiraceae)、消化链球菌科(Peptostreptococcaceae)、肠杆菌科(Coriobacteriaceae)、氨基酸球菌科(Acidaminococcaceae)、丁酸球菌科(Butyricicoccaceae)和产粪甾醇真杆菌科(Eubacterium coprostanoligenes group);回肠中共获得22个差异微生物,其中在科水平上具有显著差异的微生物菌群为链球菌科、乳杆菌科、棒杆菌科、微球菌科、气球菌科、梭菌科(Clostridiaceae)、伯克氏菌科(Burkholderiaceae)和肠球菌科(Enterococcaceae)。
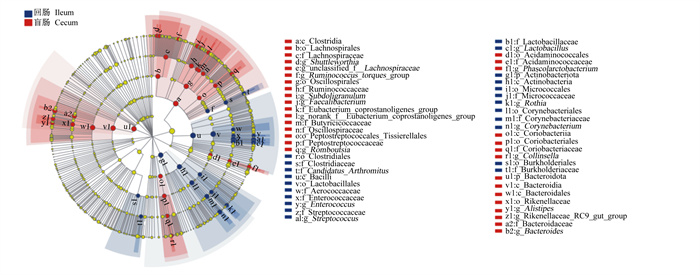
|
Clostridia:梭菌纲;Lachnospirales:毛螺菌目;Lachnospiraceae:毛螺菌科;Shuttleworthia:沙特尔沃思菌属;unclassified_f_Lachnospiraceae:毛螺菌科未定义属;Ruminococcus_torques_group:瘤胃球菌属扭链群;Oscillospirales:颤杆菌目;Ruminococcaceae:瘤胃菌科;Subdoligranulum:罕见小球菌属;Faecalibacterium:粪杆菌属;Eubacterium_coprostanoligenes_group:产粪甾醇真杆菌科;norank_f__Eubacterium_coprostanoligenes_group:产粪甾醇真杆菌科未定义属;Butyricicoccaceae:丁酸球菌科;Oscillospiraceae:颤杆菌科;Peptostreptococcaceae:消化链球菌科;Romboutsia:罗姆布茨菌属;Clostridiales:梭菌目;Clostridiaceae:梭菌科;Candidatus_Arthromitus:念珠菌属;Bacilli:杆菌纲;Lactobacillales:乳杆菌目;Aerococcaceae:气球菌科;Enterococcaceae:肠球菌科;Enterococcus:肠球菌属;Streptococcaceae:链球菌科;Streptococcus:链球菌属;Lactobacillaceae:乳杆菌科;Lactobacillus:乳酸菌属;Acidaminococcales:氨基酸球菌目;Acidaminococcaceae:氨基酸球菌科;Phascolarctobacterium:考拉杆菌属;Actinobacteriota:放线菌门;Actinobacteria:放线菌纲;Micrococcales:微球菌目;Micrococcaceae:微球菌科;Rothia:罗思氏菌属;Corynebacteriales:棒杆菌目;Corynebacteriaceae:棒杆菌科;Corynebacterium:棒杆菌属;Coriobacteriia:肠杆菌纲;Coriobacteriales:肠杆菌目;Coriobacteriaceae:肠杆菌科;Collinsella:柯斯林氏菌属;Burkholderiales:伯克氏菌目;Burkholderiaceae:伯克氏菌科;Bacteroidota:拟杆菌门;Bacteroidia:拟杆菌纲;Rikenellaceae:理研菌科;Alistipes:另枝菌属;Rikenellaceae_RC9_gut_group:理研菌科RC9肠道群;Bacteroidaceae:拟杆菌科;Bacteroides:拟杆菌属;Peptostreptococcales_Tissierellales:Peptostreptococcales_Tissierellales菌目。 图 5 差异微生物LEfSe分析 Fig. 5 LEfSe analysis on differential microbiota |
家禽肠道中定植的微生物群落参与宿主的营养吸收和免疫防御过程,对家禽的生长和健康起到了重要的作用[12]。回肠是肉鸭营养物质吸收的主要部位,而盲肠是主要的发酵场所,进而使回肠和盲肠微生物菌群的多样性与肠道功能有着密切的联系[9]。盲肠中Simpson指数的降低和Shannon指数的增加表明了盲肠中微生物更加具有多样性。Bjerrum等[13]报道,肉鸡盲肠中定植的微生物菌群较回肠中的微生物菌群呈现出多样化的特点。这一结果也得到了Mohdasrore等[14]研究结果的支持,在该研究中,通过对肉鸡回肠和盲肠微生物菌群16S rRNA基因测序,表明盲肠微生物菌群的多样性更加丰富。肉鸭盲肠中定植的微生物菌群多为专性厌氧菌,其在降解多糖、尿酸和纤维素中起关键作用[15]。肉鸭盲肠中的OTUs数目多于回肠中的OTUs数目,而二者之间的丰富度基本相同,表明回肠中菌群分布均匀性较低,部分OTUs中含有大量的effective reads。消化道中定植的微生物菌群之间在饲粮营养物质的利用过程中存在竞争抑制效应,导致肠道菌群均匀度降低。因此,肉鸭生长过程中不仅要控制肠道中潜在的病原菌,而且调节肠道菌群的平衡也至关重要。
3.2 35日龄肉鸭回肠和盲肠微生物群落组成在门水平上,厚壁菌门、拟杆菌门、变形菌门和放线菌门是肉鸭回肠和盲肠中共有的优势菌门,其中厚壁菌门是回肠和盲肠中主要的菌门。拟杆菌门主要存在盲肠中,而在回肠中的相对丰度较低。研究发现,厚壁菌门和拟杆菌门参与肠道中SCFAs的代谢,其中厚壁菌门参与丙酸和丁酸的合成,拟杆菌门主要参与丙酸的代谢途经[16]。拟杆菌门在肉鸭盲肠中相对丰度的增加表明盲肠中参与SCFAs代谢途径的菌群更为发达,促进饲粮营养物质的微生物发酵,尤其是饲粮纤维的利用,进而为宿主提供生长所需的能量和氨基酸等营养物质[17]。此外,家禽肠道中厚壁菌门与拟杆菌门的比值与其生长性能和营养物质的消化相关[18]。Singh等[18]研究发现,肉鸡盲肠中厚壁菌门和拟杆菌门比值的增加,提高了肉鸡对养分的利用效率,促进了肉鸡的生长发育。与盲肠相比,肉鸭回肠中厚壁菌门与拟杆菌门比值的增加有利于提高回肠对营养物质的消化能力,从而使回肠更好地执行其消化功能。Wei等[4]研究报道,变形菌门和放线菌门是肉鸡盲肠中主要的菌门,然而本试验中发现变形菌门和放线菌门在回肠中的相对丰度高于盲肠,可能是由于宿主和饲养环境的差异而造成菌群多样性的不同。
在属水平上,肉鸭回肠和盲肠中主要的菌属为链球菌属、乳杆菌属、棒杆菌属和拟杆菌属。链球菌属、乳杆菌属和棒杆菌属在肉鸭回肠中的相对丰度高于盲肠。这与肉鸡回肠中乳杆菌属、棒杆菌属相对丰度高于盲肠方面的研究结果[13, 21-22]一致。研究表明,链球菌属、乳杆菌属和棒杆菌属是淀粉分解菌,其能够促进饲粮中的淀粉分解为可被宿主吸收的葡萄糖分子形式[19]。因而,肉鸭回肠中淀粉分解菌相对丰度的增加有利于饲粮中淀粉在回肠中消化吸收。家禽盲肠作为微生物发酵的主要场所,其肠腔内氧分压和氧化还原电位低于回肠,从而有利于专性厌氧菌在盲肠生态位中的增殖[4]。本试验发现,在肉鸭盲肠生态位中专性厌氧菌拟杆菌属和粪杆菌属的相对丰度高于回肠。这一结果也得到了Mohdasrore等[14]研究结果的支持,在该研究发现肉鸡盲肠中定植的拟杆菌属和粪杆菌属的相对丰度高于回肠。拟杆菌属和粪杆菌属是发酵产生SCFAs的主要菌群,其中拟杆菌属在含氮物质的利用以及胆汁酸和其他类固醇的生物转化中也具有重要作用[20]。由此可见,肉鸭盲肠中产SCFAs菌群相对丰度的增加有利于执行盲肠的发酵功能。
3.3 35日龄肉鸭回肠和盲肠菌群结构和差异微生物分析PCoA分析展示了肉鸭回肠和盲肠微生物菌群分别形成了2个独立的簇,进而表明了回肠微生物菌群的组成与盲肠有着明显的不同。Xiao等[22]应用PCoA发现肉鸡小肠的微生物菌群存在相似性,而与盲肠的微生物菌群组成存在明显的差别。Lu等[23]研究表明,肉鸡肠道微生物菌群的组成与其定植的肠道部位相关。通过在科水平上的分层聚类热图发现,瘤胃菌科、毛螺菌科和拟杆菌科是肉鸭盲肠中主要的菌科;而棒杆菌科、乳杆菌科和链球菌科是肉鸭回肠中主要的菌科,进一步表明了肉鸭肠道微生物在不同肠段中群落的组成和相对丰度各不相同。
LEfSe分析筛选出肉鸭盲肠中的差异微生物在科水平上主要为拟杆菌科、瘤胃菌科、毛螺菌科、理研菌科、颤杆菌科。瘤胃菌科、毛螺菌科和颤杆菌科是丁酸的主要生产者,其通过发酵饲粮中难以被肉鸭消化利用的多聚糖从而产生可被机体利用的SCFAs[24]。Polansky等[24]研究报道,SCFAs不仅为肠道上皮细胞提供能量,而且能够抑制病原菌产生致病因子。Hu等[25]研究发现,毛螺菌科的相对丰度与肉鸡生长性能之间存在显著的正相关关系。此外,拟杆菌科能够刺激肠道上皮细胞产生抗炎细胞因子[25]。肉鸭回肠中的差异微生物在科水平上主要为链球菌科、乳杆菌科、棒杆菌科。乳杆菌科中的乳酸菌属是定植在家禽肠道中益生菌,其不仅能够产生细胞素,而且生成大量的乳酸和乙酸,从而降低了肠道中的pH,有利于抑制病原菌的增殖[22]。因而,肉鸭回肠和盲肠中的差异微生物为其肠道生长发育提供了有利的微生态环境,为肉鸭的健康生长提供了保障。
4 结论① 肉鸭回肠和盲肠中共有的OTUs数目为509个,回肠和盲肠微生物的丰富度没有显著差异,盲肠微生物的多样性高于回肠。
② 链球菌属、乳杆菌属和棒杆菌属是肉鸭回肠中的主要菌属,其为淀粉分解菌,可能影响回肠中淀粉的消化吸收。
③ 拟杆菌属和巨单细胞菌属是肉鸭盲肠中的主要菌属,其为SCFAs的生产者,可能影响盲肠菌群繁殖和盲肠发育。
| [1] |
QIN S M, ZHANG KY, APPLEGATE T J, et al. Dietary administration of resistant starch improved cecal barrier function by enhancing intestinal morphology and modulating microbiota composition in meat duck[J]. British Journal of Nutrition, 2019, 123(2): 1-27. |
| [2] |
CRHANOVA M, HRADECKA H, FALDYNOVA M, et al. Immune response of chicken gut to natural colonization by gut microflora and to Salmonella enterica serovar enteritidis infection[J]. Infection and Immunity, 2011, 79(7): 2755-2763. DOI:10.1128/IAI.01375-10 |
| [3] |
RINTTILÄ T, APAJALAHTI J. Intestinal microbiota and metabolites-implications for broiler chicken health and performance[J]. Journal of Applied Poultry Research, 2013, 22(3): 647-658. DOI:10.3382/japr.2013-00742 |
| [4] |
WEI S, MORRISON M, YU Z. Bacterial census of poultry intestinal microbiome[J]. Poultry Science, 2013, 92(3): 671-683. DOI:10.3382/ps.2012-02822 |
| [5] |
TOPPING D L, CLIFTON P M. Short-chain fatty acids and human colonic function: roles of resistant starch and nonstarch polysaccharides[J]. Physiological Reviews, 2001, 81(3): 1031-1064. DOI:10.1152/physrev.2001.81.3.1031 |
| [6] |
FORTE C, MANUALI E, ABBATE Y, et al. Dietary Lactobacillus acidophilus positively influences growth performance, gut morphology, and gut microbiology in rurally reared chickens[J]. Poultry Science, 2018, 97(3): 930-936. DOI:10.3382/ps/pex396 |
| [7] |
NEISH A S. Microbes in gastrointestinal health and disease[J]. Gastroenterology, 2009, 136(1): 65-80. DOI:10.1053/j.gastro.2008.10.080 |
| [8] |
ZHAO L L, YIN H C, LU T F, et al. Application of high-throughput sequencing for microbial diversity detection in feces of specific-pathogen-free ducks[J]. Poultry Science, 2018, 97(7): 2278-2286. DOI:10.3382/ps/pex348 |
| [9] |
VASAÏ F, BRUGIRARD RICAUD K, BERNADET M D, et al. Overfeeding and genetics affect the composition of intestinal microbiota in Anas platyrhynchos (Pekin) and Cairina moschata (Muscovy) ducks[J]. FEMS Microbiology Ecology, 2014, 87(1): 204-216. |
| [10] |
STANLEY D, HUGHES R J, MOORE R J. Microbiota of the chicken gastrointestinal tract: influence on health, productivity and disease[J]. Applied Microbiology and Biotechnology, 2014, 98(10): 4301-4310. DOI:10.1007/s00253-014-5646-2 |
| [11] |
JOHANSEN C H, BJERRUM L, PEDERSEN K. Impact of salinomycin on the intestinal microflora of broiler chickens[J]. Acta Veterinaria Scandinavica, 2007, 49(1): 30. DOI:10.1186/1751-0147-49-30 |
| [12] |
KOGUT M H. The effect of microbiome modulation on the intestinal health of poultry[J]. Animal Feed Science and Technology, 2019, 250: 32-40. DOI:10.1016/j.anifeedsci.2018.10.008 |
| [13] |
BJERRUM L, ENGBERG R M, LESER T D, et al. Microbial community composition of the ileum and cecum of broiler chickens as revealed by molecular and culture-based techniques[J]. Poultry Science, 2006, 85(7): 1151-1164. DOI:10.1093/ps/85.7.1151 |
| [14] |
MOHDASRORE M S, SIEO C C, CHONG C W, et al. Deciphering chicken gut microbial dynamics based on high-throughput 16S rRNA metagenomics analyses[J]. Gut Pathogens, 2015, 7: 4. DOI:10.1186/s13099-015-0051-7 |
| [15] |
REHMAN H U, VAHJEN W, AWAD W A, et al. Indigenous bacteria and bacterial metabolic products in the gastrointestinal tract of broiler chickens[J]. Archives of Animal Nutrition, 2007, 61(5): 319-335. DOI:10.1080/17450390701556817 |
| [16] |
PANDIT R J, HINSU A T, PATEL N V, et al. Microbial diversity and community composition of caecal microbiota in commercial and indigenous Indian chickens determined using 16s rDNA amplicon sequencing[J]. Microbiome, 2018, 6(1): 115. DOI:10.1186/s40168-018-0501-9 |
| [17] |
WAITE D W, TAYLOR M W. Exploring the avian gut microbiota: current trends and future directions[J]. Frontiers in Microbiology, 2015, 6: 673. |
| [18] |
SINGH P, KARIMI A, DEVENDRA K, et al. Influence of penicillin on microbial diversity of the cecal microbiota in broiler chickens[J]. Poultry Science, 2013, 92(1): 272-276. DOI:10.3382/ps.2012-02603 |
| [19] |
REGMI P R, METZLER-ZEBELI B U, GÄNZLE M G, et al. Starch with high amylose content and low in vitro digestibility increases intestinal nutrient flow and microbial fermentation and selectively promotes Bifidobacteria in pigs[J]. The Journal of Nutrition, 2011, 141(7): 1273-1280. DOI:10.3945/jn.111.140509 |
| [20] |
LAN P T N, SAKAMOTO M, SAKATA S, et al. Bacteroides barnesiae sp. nov., Bacteroides salanitronis sp. nov. and Bacteroides gallinarum sp. nov., isolated from chicken caecum[J]. International Journal of Systematic and Evolutionary Microbiology, 2006, 56(12): 2853-2859. DOI:10.1099/ijs.0.64517-0 |
| [21] |
DUMONCEAUX T J, HILL J E, HEMMINGSEN S M, et al. Characterization of intestinal microbiota and response to dietary virginiamycin supplementation in the broiler chicken[J]. Applied and Environmental Microbiology, 2006, 72(4): 2815-2823. DOI:10.1128/AEM.72.4.2815-2823.2006 |
| [22] |
XIAO Y P, XIANG Y, ZHOU W D, et al. Microbial community mapping in intestinal tract of broiler chicken[J]. Poultry Science, 2017, 96(5): 1387-1393. DOI:10.3382/ps/pew372 |
| [23] |
LU J R, IDRIS U, HARMON B, et al. Diversity and succession of the intestinal bacterial community of the maturing broiler chicken[J]. Applied and Environmental Microbiology, 2003, 69(11): 6816-6824. DOI:10.1128/AEM.69.11.6816-6824.2003 |
| [24] |
POLANSKY O, SEKELOVA Z, FALDYNOVA M, et al. Important metabolic pathways and biological processes expressed by chicken cecal microbiota[J]. Applied Environmental Microbiology, 2015, 82(5): 1569-1576. |
| [25] |
HU Y, WANG L D, SHAO D, et al. Selectived and reshaped early dominant microbial community in the cecum with similar proportions and better homogenization and species diversity due to organic acids as AGP alternatives mediate their effects on broilers growth[J]. Frontiers in Microbiology, 2019, 10: 2948. |




