2. 河南省农业科学院畜牧兽医研究所, 郑州 450002
2. Animal Husbandry and Veterinary Research Institute, Henan Academy of Agricultural Sciences, Zhengzhou 450002, China
酪蛋白占牛奶总蛋白的80%,是新生儿氨基酸和磷酸钙的主要来源。牛奶中有4种类型的酪蛋白:αS1、αS2、β和κ。众所周知,κ-酪蛋白(κ-casein,CSN3)变异影响牛奶的工业生产特性,如乳浊状态[1]。由于糖基化形式的存在,CSN3形成一个高度水合的聚电解质层,具有空间和静电稳定性[2]。因此,CSN3是维持牛奶不透明状态的重要物质。
进化过程中,编码酪蛋白的核苷酸序列保守性很差,在所有的酪蛋白中,CSN3的核苷酸序列相对最保守[3]。目前,奶牛CSN3研究主要集中在CSN3基因多态性与乳产量或乳成分之间的关系上[4-5]。但是,对于奶牛CSN3基因表达调控方面的研究比较少,而CSN3的基因调控与奶牛乳产量或乳成分也密切相关。作为一类小的非编码RNA,miRNA可以在转录后调节基因表达,影响多种生物学过程[6],并且在奶牛的不同泌乳期,miRNA表达水平不同[7-8]。然而,还没有关于miRNA和CSN3基因之间调节的报道。因此,本研究的目的是寻找调节奶牛CSN3表达的miRNA,以期为miRNA的功能研究提供理论依据。
1 材料与方法 1.1 奶样采集采集中国荷斯坦奶牛3个泌乳阶段的牛奶样品,分别以产后60 d左右、产后120 d左右、产后240 d左右代表不同的泌乳阶段。同一泌乳阶段要求奶牛泌乳时间前后相差不超过10 d。具体方法为:选择15头奶牛,每个泌乳阶段选择5头,每头牛用50 mL离心管采集6管牛奶,当天将样品四周冰袋覆盖下运输至实验室。一部分样品用于检测牛奶中CSN3浓度;另一部分样品放入离心机离心,用于从牛奶中分离体细胞(somatic cell,SC)。分离方法:4 ℃、600×g离心10 min,弃去上层物质,沉淀即为体细胞。将分离出的体细胞加1 mL Trizol试剂,冻存于-80 ℃冰箱中,用于后续基因表达的研究。
1.2 酶联免疫吸附测定(ELISA)检测采用牛CSN3的ELISA试剂盒(货号:ml059486,上海酶联生物科技有限公司)测定牛乳中CSN3浓度,方法参照试剂盒说明书。简述如下:无菌管收集50 mL牛奶作为单个样本,-80 ℃贮存备用。样本在4 ℃、1 000×g下离心20 min,去除最上面的脂肪层。上清液用0.01 mol/L、pH=7.4的磷酸盐缓冲液(PBS)稀释10倍,之后进行ELISA试验。CSN3的ELISA试剂盒内含1套标准品,在450 nm处用光谱仪测定标准品光密度(OD)值,可以得到OD值与CSN3浓度的标准曲线。样品中CSN3浓度可以通过测定OD值与标准曲线比较来确定。
1.3 乳腺组织样品分别于产后60 d、产后120 d和产后240 d对3头健康荷斯坦奶牛的乳腺组织进行了3次活体取样,具体方法参考Li等[9]。简述如下:对采样的乳腺部位进行局部麻醉,用灭菌的活体取样器直接取样,插入深度约为10 cm,1次能取3 g左右的样品,用高压灭菌的手术剪和镊子将样品切割为绿豆大小的小块,放入1 mL冻存管,投入液氮中冻存。对手术部位进行消毒灭菌,缝合伤口,注射抗生素,手术后1周内的牛奶弃去不用。奶牛饲喂全混合日粮,自由饮水。
1.4 小RNA测序和数据加工乳腺组织RNA提取、小RNA测序和数据处理委托中国联川生物公司开展。Trizol试剂提取RNA后,用生物分析仪2100(Agilent,美国)测定RNA的完整数(RIN)。在Illumina HiSeq 2500平台上构建了3个小RNA文库(每个样本1 μg),用于单端测序(36或50 bp)。原始序列采用ACGT101-miR程序和miRBase 21.0过滤。通过校正计算对数据进行标准化处理,后续分析采用的均为标准化数据。低表达定义为所有样本的拷贝数小于10的miRNA;中表达定义为1个样本的拷贝数大于10,所有样本的拷贝数小于平均值;高表达定义为至少1个样本的拷贝数大于平均值。采用SAS 9.0的卡方检验(2×2)分析基于归一化的差异表达miRNA。
1.5 靶向CSN3的miRNA预测使用TargetScan 7.2(http://www.targetscan.org/vert_72/)预测靶向CSN3基因的miRNA。
1.6 交集筛选将2种方法(小RNA测序分析和生物信息学软件预测)交集筛选出miRNA作为下一步的研究对象。在差异表达miRNA中,删除低表达或不在软件预测范围的miRNA。采用RNAhybrid(https://bibiserv.cebitec.uni-bielefeld.de/rnahybrid)预测miRNA与CSN3-3’非编码区(3’untranslated region,3’UTR)之间的结合自由能。
1.7 载体构建利用表 1所示的引物,从牛基因组DNA中扩增出CSN3-3’UTR的全长(207 bp)。将PCR产物克隆到psiCHECK-Ⅱ,利用NotI和XhoI限制性酶切位点构建了psiCHECK-Ⅱ-CSN3-3’UTR质粒。
|
|
表 1 CSN3-3’UTR的引物 Table 1 Primers for CSN3-3'UTR |
293T细胞接种于96孔板中,每孔接种密度为2×104个细胞。采用PmirGLO载体和psiCHECK-Ⅱ载体作为系统控制和测试控制。将4种miRNA模拟物(60 nmol/L)和阴性对照(中国锐博公司)分别与1 μg质粒共转染293T细胞,转染试剂采用2 μL X-tremegene HP (罗氏公司,瑞士)。转染48 h后,用双荧光素酶报告试剂盒(E1910,Promega公司,美国)检测荧光素酶活性。
1.9 CSN3、bta-miR-193b和bta-miR-193a-3p的PCR检测应用SYBR Prime Script RT-PCR试剂盒(TaKaRa,日本)测定牛奶体细胞中CSN3、bta-miR-193b和bta-miR-193a-3p的相对表达量。甘油醛-3-磷酸脱氢酶(GAPDH)和U6分别作为CSN3和miRNA的内参基因,引物设计见表 2。扩增程序:95 ℃ 30 s,然后95 ℃ 5 s,40个循环,60 ℃ 34 s。采用2-ΔΔCt方法计算目的基因相对表达量。
|
|
表 2 CSN3和miRNA的引物设计 Table 2 Primer design for CSN3 and miRNA |
采用SPSS 22.0软件对牛奶中CSN3浓度、产奶量以及CSN3和miRNA基因相对表达量进行单因素方差分析;对2组之间相对荧光素酶活性进行t检验。数据显示为平均值±标准误,P < 0.05表示差异显著,P < 0.01表示差异极显著。使用SigmaPlot 14.0软件绘制图形。
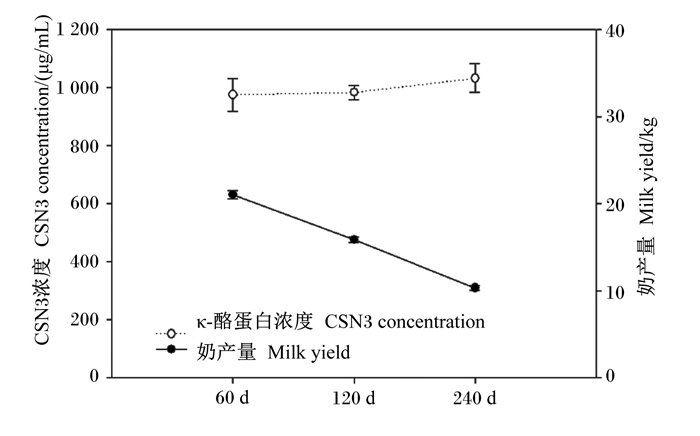
2 结果 2.1 CSN3浓度ELISA法测定不同泌乳阶段奶牛分泌的牛奶中CSN3浓度(图 1),在不同泌乳期,CSN3浓度差异不显著(P>0.05)。然而,从产后60 d到产后240 d,奶牛产奶量显著下降(P < 0.05)(图 1)。3个泌乳阶段平均产奶量分别为21.044、15.872、10.326 kg。因此,从产后60 d到产后240 d,牛奶中CSN3蛋白总量呈现降低趋势(CSN3蛋白总量=CSN3浓度×产奶量)。
|
产奶量的数据显示为5头奶牛的平均值。检测时间分别为产后60 d、产后120 d和产后240 d。 The data of milk yield showed an average milk yield of five cows. The determined time was 60th day, 120th day and 240th day after delivery. 图 1 牛奶中CSN3浓度和产奶量变化 Fig. 1 Changes in milk CSN3 concentration and milk yield |
本试验RNA的完整性指标RIN>7.0,样品可以用于后续测序。测序原始数据已存入美国国家生物技术信息中心(https://www.ncbi.nlm.nih.gov/bioproject/PRJNA589107)。本试验比较了不同泌乳期构建的3个miRNA文库,通过卡方检验(P≤0.05)和倍数改变(|log2(倍数改变)|>1)筛选差异表达的miRNA。3个文库中共检测到1 949个miRNA,其中有390个差异表达。在差异表达的miRNA中,有11个低表达的miRNA被删除。其中,中、高表达miRNA共379个,仅仅93个高表达的miRNA。不考虑miRNA前体和染色体位置的不同,只有77个miRNA呈现高表达,且在不同泌乳阶段差异显著(P<0.05),见表 3。
|
|
表 3 奶牛乳腺组织3个泌乳阶段均呈现高表达且差异显著的miRNA Table 3 Highly and differentially expressed miRNA in three lactation stages of cow mammary gland (n=77) |
根据TargetScan预测,CSN3-3’UTR上有2个保守位点和21个保守性差的miRNA结合位点(表 4)。
|
|
表 4 TargetScan预测的靶向CSN3的miRNA Table 4 Predicted miRNA targeting CSN3 with TargetScan |
删除低表达的miRNA后,只有4个中表达的miRNA保留在预测范围内(表 5)。它们是bta- miR-33a、bta-miR-2284ab、bta-miR-193a-3p、bta- miR-193b。这4个miRNA与CSN3-3’UTR结合的最小自由能分别为-16.1、-18.6、-21.8和-20.9 kcal/mol(1 kcal/mol≈4.184 kJ/mol)(表 6)。
|
|
表 5 交集中4个miRNA的测序数据分析 Table 5 Sequencing data analysis of four miRNA in intersection |
|
|
表 6 RNAhybrid预测miRNA与CSN3-3’UTR的结合情况 Table 6 Prediction for miRNA binding CSN3-3'UTR with RNAhybrid |
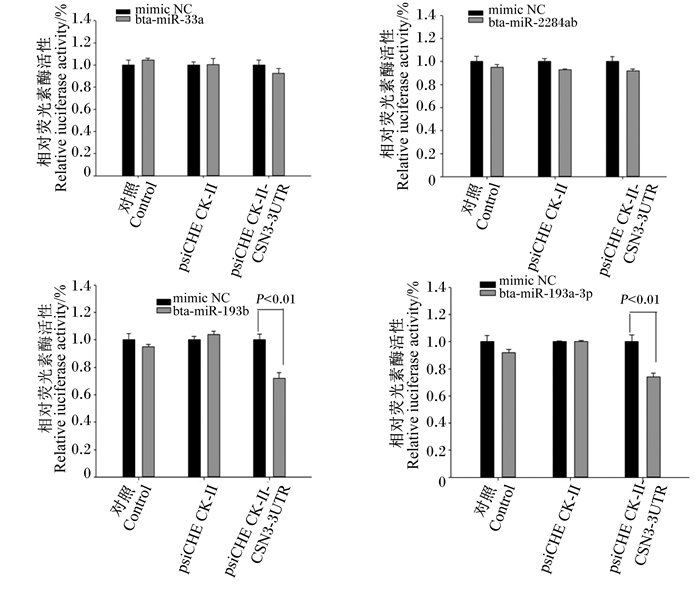
bta-miR-33a和bta-miR-2284ab模拟物(mimic)与阴性对照模拟物(mimic NC)相比,双荧光素酶活性无显著性差异(P>0.05)(图 2-a、图 2-b)。当bta-miR-193b和bta-miR-193a-3p模拟物被转染后,双荧光素酶活性极显著下降(P < 0.01)(图 2-c、图 2-d)。与阴性对照模拟物相比,bta-miR-193b和bta-miR-193a-3p模拟物导致萤火虫荧光素酶/肾素酶的比值分别下降28%和26%。综上所述,这些结果提示bta-miR-193b和bta-miR-193a-3p可直接结合CSN3-3’UTR,从而调节CSN3的表达。
|
mimic NC:阴性对照模拟物negative control mimic。P<0.01:差异极显著significant difference。 图 2 4种miRNA相对荧光素酶活性的变化 Fig. 2 Change of relative luciferase activity for four miRNA |
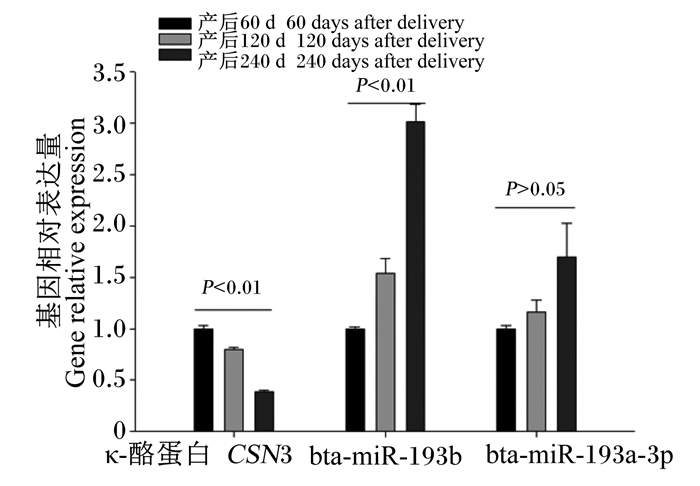
为了验证CSN3与bta-miR-193家族之间的关系,对牛奶体细胞样品中这些基因的相对表达量进行了检测。牛奶体细胞CSN3的相对表达量从产后60 d到产后120 d极显著下降(P < 0.01)(图 3)。相反,bta-miR-193b和bta-miR-193a-3p在产后60 d至产后120 d呈上升趋势,且bta-miR-193b变化产生统计学上的显著差异(P < 0.01)(图 3)。
|
P<0.01:差异极显著significant difference;P>0.05:差异不显著no significant difference。 图 3 不同时期牛奶体细胞中3个基因(CSN3、bta-miR-193b、bta-miR-193a-3p)的相对表达趋势 Fig. 3 Relative expression trends of three genes (CSN3, bta-miR-193b and bta-miR-193a-3p) in milk somatic cells at different stages |
作为小的非编码RNA分子,miRNA可以结合靶基因的3’UTR来抑制靶基因的表达[10]。每个miRNA可以调节上百个基因,每个基因可以受到上百个miRNA的调节[11]。牛奶中CSN3浓度在3个泌乳期间没有明显变化,但产奶量急剧下降,这意味着乳腺表达的CSN3的蛋白总量下降了。为了从miRNA角度探索CSN3表达的调控机制,采用TargetScan预测搜索靶向牛CSN3的miRNA。和其他预测工具一样,TargetScan预测也选择进化上保守的miRNA结合位点。假阳性是预测软件中常见的问题。因此,本试验将小RNA测序分析与软件预测相结合。为了获得尽可能多的候选miRNA,只放弃在3个阶段没有显著差异或者低表达水平的miRNA。软件预测范围内的大多数miRNA表达水平较低。通过这种方法,本试验筛选到4个可能靶向CSN3的miRNA。
在这项研究中,使用基因的3’UTR来搜索miRNA,这比使用miRNA搜索靶基因在方法上更易操作。研究人员只需构建一个CSN3-3’UTR的载体,逐个测试不同的miRNA。根据双荧光素酶活性测定结果,只有bta-miR-193b和bta-miR-193a-3p引起相对荧光素酶活性明显下降。而bta-miR-33a和bta-miR-2284ab没有这个功能,尽管依据软件预测bta-miR-33a具有保守的结合位点。
最小自由能和小RNA测序在避免预测的假阳性方面具有积极作用。与CSN3-3’UTR结合时,bta-miR-193b和bta-miR-193a-3p的最小自由能均低于bta-miR-33a和bta-miR-2284ab。自由能越低,结合的可能性就越高。与此同时,依据小RNA测序数据结果,bta-miR-193b的表达在3个泌乳阶段持续增加。这种现象与miRNA对靶基因的负调节作用相吻合。依据测序数据中bta-miR-33a和bta-miR-2284ab的表达趋势变化,可以将它们从预测的范围内剔除。同时,双荧光素酶试验结果证实了bta-miR-33a和bta-miR-2284ab不能靶向CSN3。
牛奶体细胞的基因表达数据为bta-miR-193b和bta-miR-193a-3p靶向CSN3提供了进一步的依据。关于miR-193家族的资料不多,目前的研究主要集中于miR-193b抑制肿瘤和促进脂肪生成方面[12-13],miR-193a也在很多肿瘤中表现为抑制作用[14]。这2个miRNA在牛上的报道很少,最近的一项研究发现,bta-miR-193b通过靶向乙酰辅酶A合成酶2(ACSS2)来调节牛脂肪细胞的发育[15];bta-miR-193a-3p在牛乳腺上皮细胞(MAC-T)中具有时间表达差异性[16]。但关于miR-193家族调控乳蛋白方面还未见任何报告。寻找调节乳蛋白的miRNA对于研究乳蛋白合成的调节机制至关重要。下一步试验,本课题组将进行bta-miR-193b和bta-miR-193a-3p靶向CSN3的细胞培养水平和活体动物水平验证。
4 结论通过小RNA高通量测序、生物信息学预测、结合位点自由能分析、双荧光结合分析、荧光定量分析,表明bta-miR-193b和bta-miR-193a-3p靶向CSN3,但还需进行奶牛乳腺细胞试验和活体动物试验的进一步验证。产后60 d至产后240 d,随着产奶量的下降,我们推测bta-miR-193b和bta-miR-193a-3p在维持牛奶中CSN3浓度稳定方面起关键作用。本试验为后期开展miRNA调控CSN3以及牛奶中CSN3稳定机制的研究指明了方向。
| [1] |
SCHAAR J, HANSSON B, PETTERSSON H E. Effects of genetic variants of κ-casein and β-lactoglobulin on cheesemaking[J]. Journal of Dairy Research, 1985, 52(3): 429-437. DOI:10.1017/S002202990002433X |
| [2] |
DE KRUIF C G. Skim milk acidification[J]. Journal of Colloid Interface Science, 1997, 185(1): 19-25. DOI:10.1006/jcis.1996.4548 |
| [3] |
BONSING J, MACKINLAY A G. Recent studies on nucleotide sequences encoding the caseins[J]. Journal of Dairy Research, 1987, 54(3): 447-461. DOI:10.1017/S0022029900025632 |
| [4] |
CIESLAK J, WODAS L, BOROWSKA A, et al. 5'-flanking variants of equine casein genes (CSN1S1, CSN1S2, CSN2, CSN3) and their relationship with gene expression and milk composition[J]. Journal of Applied Genetics, 2019, 60(1): 71-78. DOI:10.1007/s13353-018-0473-2 |
| [5] |
NEAMT R I, SAPLACAN G, ACATINCAI S, et al. The influence of CSN3 and LGB polymorphisms on milk production and chemical composition in Romanian Simmental cattle[J]. Acta Biochimica Polonica, 2017, 64(3): 493-497. DOI:10.18388/abp.2016_1454 |
| [6] |
AMBROS V, BARTEL B, BARTEL D P, et al. A uniform system for microRNA annotation[J]. RNA Society, 2003, 9(3): 277-279. DOI:10.1261/rna.2183803 |
| [7] |
GU Z L, ELESWARAPU S, JIANG H L. Identification and characterization of microRNAs from the bovine adipose tissue and mammary gland[J]. FEBS Letters, 2007, 581(5): 981-988. DOI:10.1016/j.febslet.2007.01.081 |
| [8] |
WANG M, MOISÁ S, KHAN M J, et al. MicroRNA expression patterns in the bovine mammary gland are affected by stage of lactation[J]. Journal of Dairy Science, 2012, 95(11): 6529-6535. DOI:10.3168/jds.2012-5748 |
| [9] |
LI C, CAI W T, ZHOU C H, et al. RNA-Seq reveals 10 novel promising candidate genes affecting milk protein concentration in the Chinese Holstein population[J]. Scientific Reports, 2016, 6: 26813. DOI:10.1038/srep26813 |
| [10] |
ZHANG L P, LIU J W, CHENG P, et al. Correlation between miRNA target site polymorphisms in the 3' UTR of AVPR1A and the risk of hypertension in the Chinese Han population[J]. Bioscience Reports, 2019, 39(5): BSR20182232. DOI:10.1042/BSR20182232 |
| [11] |
PINZÓN N, LI B, MARTINEZ L, et al. MicroRNA target prediction programs predict many false positives[J]. Genome Research, 2017, 27: 234-245. DOI:10.1101/gr.205146.116 |
| [12] |
WAHDAN-ALASWAD R S, COCHRANE D R, SPOELSTRA N S, et al. Metformin-induced killing of triple-negative breast cancer cells is mediated by reduction in fatty acid synthase via miRNA-193b[J]. Hormones & Cancer, 2014, 5(6): 374-389. |
| [13] |
MAZZU Y Z, HU Y L, SONI R K, et al. MiR-193b-regulated signaling networks serve as tumor suppressors in liposarcoma and promote adipogenesis in adipose-derived stem cells[J]. Cancer Research, 2017, 77(21): 5728-5740. DOI:10.1158/0008-5472.CAN-16-2253 |
| [14] |
ZHANG G X, ZOU X F, LIU Q L, et al. MiR-193a-3p functions as a tumour suppressor in human aldosterone-producing adrenocortical adenoma by down-regulating CYP11B2[J]. International Journal of Experimental Pathology, 2018, 99(2): 77-86. DOI:10.1111/iep.12267 |
| [15] |
KANG Z H, ZHANG S H, JIANG E H, et al. Mir-193b regulates the differentiation, proliferation, and apoptosis of bovine adipose cells by targeting the ACSS2/AKT axis[J]. Animals, 2020, 10(8): 1265. DOI:10.3390/ani10081265 |
| [16] |
JIN W, IBEAGHA-AWEMU E M, LIANG G X, et al. Transcriptome microRNA profiling of bovine mammary epithelial cells challenged with Escherichia coli or Staphylococcus aureus bacteria reveals pathogen directed microRNA expression profiles[J]. BMC Genomics, 2014, 15: 181. DOI:10.1186/1471-2164-15-181 |




